2. 北京工商大学 理学院 中国轻工业化妆品重点实验室,北京 100048
2. Key Laboratory of Cosmetic of China National Light Industry, School of Science, Beijing Technology and Business University, Beijing 100048, China
痤疮是世界上最常见的皮肤病之一,表现为一种毛囊皮脂腺的慢性炎症性,主要发生于面部、胸背部等皮脂溢出区,患者表现为粉刺、丘疹、脓疱、囊肿、结节及萎缩性瘢痕等皮损,大约会影响80%的青少年和青壮年[1]。痤疮普遍而且错误的被概括为只是患者经历的一个阶段,但对于一些人来说,痤疮可以持续多年,不仅影响患者的面容外观,还常伴有疼痛、瘙痒等躯体感觉,甚至会引起自卑、焦虑、抑郁等心理疾病,严重影响患者的身心健康[2-3]。因此在皮肤病学领域,痤疮的研究和治疗是一个广泛研究的问题。
痤疮的发病机制复杂,目前国内外公认的包括毛囊导管角化异常、微生物定植、皮脂分泌增加以及炎症反应等[4-5]。其中,微生物定植在痤疮发病中的作用是一个热点研究问题,大多工作是针对单一微生物的研究,如痤疮丙酸杆菌、金黄色葡萄球菌及表皮葡萄球菌等被认为和痤疮发病有一定的关联性[6-9]。
随着人类微生物组计划的开展,人们逐渐意识到,人的健康状况可以通过对人类微生物组的研究分析而评估,与宿主生活在一起的微生物在大部分情况下是作为一个整体发挥着重要作用[10]。微生物组是指存在于微生物群中的基因组和基因的集合。然而,对单个微生物基因组的研究存在着一定的限制,自然界中99%的微生物不能通过分离和培养进行研究,而且微生物更倾向于作为微生物群这样的整体发挥作用,因此,研究人员提出宏基因组学,即环境微生物中所有物种基因组信息的总和[11]。
在高通量测序技术迅速发展的推动下,宏基因组学吸引了大量研究人员,通过挖掘不同部位宏基因组的微生物群落结构,以及分析不同健康状态的宏基因组样本的差异,去探索人体健康与其寄宿的微生物之间玄妙的相互关系[12]。研究人员通过肠道微生物序列分析,发现肠道菌群紊乱与儿童孤独症发生有相关性[12]。此外,也发现许多了许多其他人类疾病,包括癌症、糖尿病,甚至神经发育障碍均与微生物组有关[13-14]。近年来,研究人员开始利用机器学习方法进行宏基因组学的研究工作。Huang等[15]使用主成分分析方法分析牙龈炎和健康牙龈的数据,获得主要影响牙龈炎的细菌。Wisittipanit等[16]利用支持向量机对炎症性肠炎的病人和健康人群的肠道微生物样本进行分类。Qin等[17]使用相关分析方法研究Ⅱ型糖尿病患者和健康人群的肠道宏基因组研究,发现可以区分样本的基因簇。
目前,关于痤疮宏基因组数据的研究比较缺乏,而基于机器学习方法在宏基因组数据上的有效应用已经有目共睹,本文尝试使用多重集典型相关分析(multi-set canonical correlation analysis, MCCA)方法分析痤疮的宏基因组测序数据,具体包括健康皮肤数据、痤疮患者的健康皮肤数据和患病皮肤数据。获得对不同样本集有不同影响的脂质,以及仅对其中一个样本集有显著影响的脂质,这些脂质可以有效地区分不同的皮肤状态,可用于指导痤疮的预防、诊断和治疗过程。
1 样本与方法 1.1 样本采集本次实验收集35名痤疮患者面部皮肤的感染细胞和健康细胞,同时收集没有患痤疮的35名志愿者的面部皮肤细胞作为正常对照组(NC)。使用色谱设备(Waters ACQUITY UPLC I-Class (Waters Corporation, Milford, Massachusetts, USA)),保持流速为0.3 mL/min,注射量为2.0 μL。使用流动相洗涤注射器针头在超高效液相色谱(ultra performance liquid chromatography, UPLC)运行期间,洗脱液出口连接到QTOF-MS来用于实体检测和表征。高分辨率质量测量使用设备(Waters Xevo G2-XS QTOF-MS (Waters Corporation, Milford, Massachusetts, USA)),该设备配有以正离子模式操作的电喷雾电离(electrospray ionization, ESI)界面。在操作色谱流动流速下,将UPLC系统洗脱物引入装置QTOF-MS,使用氮气作为雾化和脱溶剂化气体,通过系统(Masslynx 4.1 (Waters Corporation, Milford, Massachusetts, USA))收集UPLC-QTOF-MS数据作为质心原始数据。最终获得痤疮患者的患病皮肤(diseased skin, DS)样本集,健康皮肤(healthy skin, HS)样本集,以及正常对照组(normal control, NC)样本集,其中每个样本集包括有35名志愿者,每名志愿者收集2 520个序列。
1.2 多重集典型相关分析当研究两组样本集的数据分析时,典型相关分析(canonical correlation analysis, CCA)可以取得较好的效果,但是在分析多组样本集(不小于3组)时,CCA却很难得到令人满意的效果。为了同时分析3组样本集,本文选用MCCA方法,该方法是一种研究多组样本集之间关系的方法,给定多个样本集
| ${{ J}_{MCCA}}{{ = }}\frac{{\displaystyle\sum\limits_{{\rm{i = 1}}}^n {\displaystyle\sum\limits_{j = 1}^n {{ \omega} _i^{\rm T}{{ S}_{ij}}{{ \omega} _j}} } }}{{\sqrt {\displaystyle\sum\limits_{i = 1}^n {{ \omega} _i^{\rm T}{{ S}_{ii}}{{ \omega} _j}} } }}$ | (1) |
式中:
| ${\rm argmax}\beta = \sum\limits_{i = 1}^n {\sum\limits_{j = 1}^n {{ \omega} _i^{\rm T}{{ S}_{ij}}{{ \omega} _j}} } \;\; s.t.{\rm{ }}\sum\limits_{i = 1}^n {{ \omega} _i^{\rm T}{{ S}_{ii}}{{ \omega} _j} = 1} $ | (2) |
即当样本集之间的相关系数
| ${ L}({\omega _1},{\omega _2}, \cdots ,{\omega _n}) = \sum\limits_{i = 1}^n {\sum\limits_{j = 1}^n {{ \omega} _i^{\rm T}{{ S}_{ij}}{{ \omega} _j}} } - \beta \left(\sum\limits_{i = 1}^n {{ \omega} _i^{\rm T}{{ S}_{ii}}{{ \omega} _j}} - 1\right)$ | (3) |
令
| $({ C} - { D}){ \omega} = \beta { D \omega} $ | (4) |
其中,
| $ { C} = \left[ \!\!\!\!{\begin{array}{*{20}{c}} {{{ x}_1}{ x}_1^{\rm T}}& \cdots &{{{ x}_1}{ x}_N^{\rm T}} \\ \vdots & \ddots & \vdots \\ {{{ x}_N}{ x}_1^{\rm T}}& \cdots &{{{ x}_N}{ x}_N^{\rm T}} \end{array}}\!\!\!\! \right], \; { D} = \left[\!\!\!\! {\begin{array}{*{20}{c}} {{{ x}_1}{ x}_1^{\rm T}}& \cdots &0 \\ \vdots & \ddots & \vdots \\ 0& \cdots &{{{ x}_N}{ x}_N^{\rm T}} \end{array}}\!\!\!\! \right] $ |
得到每个样本集对应的典型变量
通过实验发现,使用MCCA方法能够有效分析痤疮宏基因组数据,算法复杂度为
| 表 1 MCCA获得同时对3组样本有影响的脂质 Tab.1 The lipids found by MCCA |

|
Download:
|
| 图 1 对DS、HS和NC样本集影响差异较大的脂质 Fig. 1 The effect of the lipids on DS, HS and NC samples | |
在图1(a)中,No.1205表示的脂质在NC样本集中有较低的贡献率,几乎可以忽略不计,但在DS和HS样本集中显示出较高且相似的影响,因此可以使用这种脂质来区分NC样本集和其他两个样本集。图1(b)显示出No.1304代表的脂质对DS、HS和NC样本的影响呈单调递减的趋势,可以认为这是有效区分DS、HS和NC样本集的一个脂质。
除此之外,使用MCCA方法还可以获得仅对其中一个样本集有显著影响,但是对其他两组样本集几乎没有影响的脂质,如图2所示。图2中编号所代表的脂质具体描述如表2所示。图2(a)和图2(b)显示No.95和No.1256代表的脂质对DS样本的影响普遍大于HS和NC样本。在图2(c)和图2(d)中,No.608和No.2334表示的脂质明显只会出现在HS样本集中,因此可以认为当这两种脂质出现时,痤疮患者的皮肤状态正在好转或者健康者的皮肤正在恶化。从图2(e)中明显看出,当No.2374表示的脂质在NC样本集有明显的增高,区别于HS和DS两个样本集,它可以反映受试者的皮肤状态是健康的,可以认为痤疮患者的治疗效果是显著的。
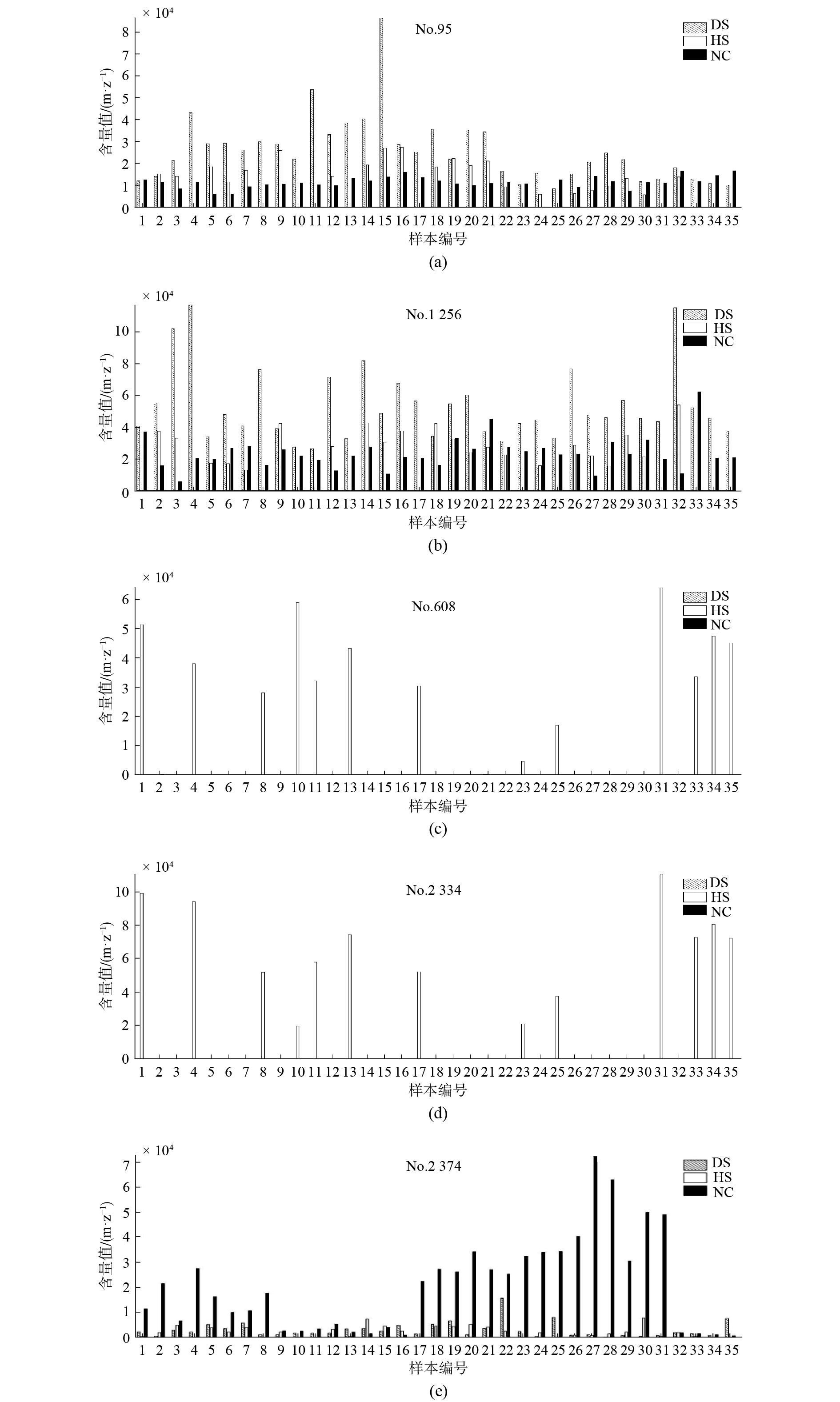
|
Download:
|
| 图 2 区分DS、HS和NC样本集的脂质 Fig. 2 The lipids which can distinguish the DS, HS and NC samples. | |
痤疮作为世界上最常见的皮肤疾病之一,患病人数多、病因复杂,并且复发率高,虽然部分患者在青春期之后,其症状会有所缓解,但是对于大部分患者来说,痤疮症状会持续很长时间,对患者的生活质量造成很大的影响,因此对于痤疮的研究和治疗是一个具有重要意义的课题。本文从宏基因组学的角度分析引起痤疮发病的脂质,并尝试使用MCCA方法分析DS、HS和NC3个样本集,可以得到仅对某一样本集有显著影响的脂质,以及同时有效区分3个样本集的脂质。实验结果显示,MCCA方法分析获得的脂质可以有效的区分3种不同的皮肤状态,并且对痤疮的预防、诊断和治疗有一定的辅助指导意义。在痤疮发病过程中,也许存在某种脂质的数值虽然很小,但却对痤疮有一定影响,而本文使用MCCA方法获取脂质对样本集的贡献时,会一定程度上弱化对这些脂质的分析,对于这些脂质还需要进一步的研究。
| [1] |
MARONI G, ERMIDORO M, PREVIDI F, et al. Automated detection, extraction and counting of acne lesions for automatic evaluation and tracking of acne severity[C]//Proceedings of 2017 IEEE Symposium Series on Computational Intelligence. Honolulu, USA, 2017: 1−6.
( 0) 0)
|
| [2] |
LUCUT S, SMITH M R. Dermatological tracking of chronic acne treatment effectiveness[C]//Proceedings of 2016 38th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. Orlando, USA, 2016: 5421−5426.
( 0) 0)
|
| [3] |
THIBOUTOT D M, DRéNO B, ABANMI A, et al. Practical management of acne for clinicians: an international consensus from the global alliance to improve outcomes in acne[J]. Journal of the American academy of dermatology, 2018, 78(2, Suppl 1): S1-S23.e1. ( 0) 0)
|
| [4] |
PAUGAM C, CORVEC S, SAINT-JEAN M, et al. Propionibacterium acnes phylotypes and acne severity: an observational prospective study[J]. Journal of the European academy of dermatology and venereology, 2017, 31(9): e398-e399. DOI:10.1111/jdv.2017.31.issue-9 ( 0) 0)
|
| [5] |
王鸿. 寻常型痤疮发病机制研究进展[J]. 西南医科大学学报, 2018, 41(4): 385-388. WANG Hong. Research progress on the pathogenesis of acne vulgaris[J]. Journal of Southwest Medical University, 2018, 41(4): 385-388. DOI:10.3969/j.issn.2096-3351.2018.04.021 (  0) 0)
|
| [6] |
FITZ-GIBBON S, TOMIDA S, CHIU B H, et al. Propionibacterium acnes strain populations in the human skin microbiome associated with acne[J]. Journal of investigative dermatology, 2013, 133(9): 2152-2160. DOI:10.1038/jid.2013.21 ( 0) 0)
|
| [7] |
DAGNELIE M, CORVEC S, SAINT-JEAN M, et al. 461 Diversity of Propionibacterium acnes phylotypes according to body localization in acne patients versus healthy controls[J]. Journal of investigative dermatology, 2017, 137(10, Suppl 2): S271. ( 0) 0)
|
| [8] |
ZOUBOULIS C C, JOURDAN E, PICARDO M. Acne is an inflammatory disease and alterations of sebum composition initiate acne lesions[J]. Journal of the European academy of dermatology and venereology, 2014, 28(5): 527-532. DOI:10.1111/jdv.2014.28.issue-5 ( 0) 0)
|
| [9] |
吴贇, 吉杰, 张玲琳, 等. 微生物在痤疮发病中的作用[J]. 中国皮肤性病学杂志, 2016, 30(3): 311-314. WU Yun, JI Jie, ZHANG Linglin, et al. Roles of microorganisms in the pathogenesis of acne[J]. The Chinese journal of dermatovenereology, 2016, 30(3): 311-314. (  0) 0)
|
| [10] |
ZHANG Xuegong, LIU Shansong, CUI Hongfei, et al. Reading the underlying information from massive metagenomic sequencing data[J]. Proceedings of the IEEE, 2017, 105(3): 459-473. ( 0) 0)
|
| [11] |
VAN OPSTAL E J, BORDENSTEIN S R. Rethinking heritability of the microbiome[J]. Science, 2015, 349(6253): 1172-1173. DOI:10.1126/science.aab3958 ( 0) 0)
|
| [12] |
KANG D W, PARK J G, ILHAN Z E, et al. Reduced incidence of Prevotella and other fermenters in intestinal microflora of autistic children[J]. PLoS one, 2013, 8(7): e68322. DOI:10.1371/journal.pone.0068322 ( 0) 0)
|
| [13] |
SEARS C L, GARRETT W S. Microbes, microbiota, and colon cancer[J]. Cell host & microbe, 2014, 15(3): 317-328. ( 0) 0)
|
| [14] |
HSIAO E Y, MCBRIDE S W, HSIEN S, et al. Microbiota modulate behavioral and physiological abnormalities associated with neurodevelopmental disorders[J]. Cell, 2013, 155(7): 1451-1463. DOI:10.1016/j.cell.2013.11.024 ( 0) 0)
|
| [15] |
HUANG Shi, LI Rui, ZENG Xiaowei, et al. Predictive modeling of gingivitis severity and susceptibility via oral microbiota[J]. The ISME journal, 2014, 8(9): 1768-1780. DOI:10.1038/ismej.2014.32 ( 0) 0)
|
| [16] |
WISITTIPANIT N, RANGWALA H, GILLEVET P, et al. SVM-based classification and feature selection methods for the analysis of Inflammatory Bowel disease microbiome data[C]//Proceedings of the 9th International Workshop on Data Mining in Bioinformatics. Washington, USA, 2010: 1−8.
( 0) 0)
|
| [17] |
QIN Junjie, LI Yingrui, CAI Zhiming, et al. A metagenome-wide association study of gut microbiota in type 2 diabetes[J]. Nature, 2012, 490(7418): 55-60. DOI:10.1038/nature11450 ( 0) 0)
|
 2020, Vol. 15
2020, Vol. 15


