2. 教育部 智能技术与教育应用教育部工程研究中心,北京 100816;
3. 中国科学院心理研究所 健康与遗传心理学研究室,北京 100101;
4. 华南师范大学 心理学院,广东 广州 510631
2. Intelligence Technology and Education Engineering Research Center, Ministry of Education, Beijing 100816, China;
3. Health and Genetic Psychology Research Laboratory, Institute of Psychology, Chinese Academy of Sciences, Beijing 100101, China;
4. School of Psychology, South China Normal University, Guangzhou 510631, China
脑是人类智能的核心,认知功能的脑机制研究是脑科学研究的重要方向[1-2]。脑功能可以从分子细胞、神经元集群、脑区等不同尺度层面展开研究。其中,利用功能磁共振成像(functional magnetic resonance image, fMRI)等无创脑影像技术在宏观层面监测和分析脑内不同区域的神经活动[3-4]是目前脑科学研究中的一个重要发展领域,特别是对于回答不同脑区的神经活动如何协同以实现各种认知过程和情绪情感体验[5],以及如何变化而产生各种神经精神疾病等一系列脑机制问题[6-7]。
基于脑影像数据的先进智能计算方法为人类高级认知功能脑机制的研究带来了新的机遇和挑战[8]。将智能计算方法引入到脑科学研究中建立脑计算模型有助于从数据中挖掘更多信息[9-10],从数据驱动角度为脑机制研究开辟新的方向,进而推进对人脑高级认知功能神经机制的理解。信息学科中的算法理论,尤其是基于数据驱动的一些智能计算方法,如独立成分分析(independent component analysis, ICA)[11]、贝叶斯网络[12]、判别字典学习[13]等在研究脑网络的组织结构和认知过程的加工机制,以及在脑疾病机制研究和影像学标记物识别等方面表现出很大潜力和应用前景。本文主要列举我们利用智能影像计算方法在识别脑认知网络成分、构建脑有向连接模型、预测认知行为变化、构建心理过程神经表征等几个方面的应用,来介绍智能影像计算在脑运作机理和脑疾病应用领域的重要前景,以及可能为认知心理学研究、脑疾病的识别和干预、以及类脑智能理论研究等提供的借鉴和参考。
1 认知网络成分识别功能分化和功能整合是人脑功能活动的重要组织原则[14]。脑内各区域既分工不同又相互作用、互相协调构成一个深度连接的复杂网络,以支持各种不同的认知功能[15-16]。准确识别和提取参与不同认知功能的网络成分是脑机制研究普遍关心的一个问题。传统的单变量方法尽管能观察到不同空间区域参与任务的脑区,但不能反映这些不同区域之间的连接关系;而普遍采用的基于时间序列相关的连接分析依赖于感兴趣区域种子点的准确定位作为先验信息。数据驱动的认知网络成分智能识别方法,如ICA在构建大尺度认知网络方面表现出显著优势[17-18]。ICA无需关于认知网络分布的先验信息,可以在分离多个认知网络的同时将噪声分解为独立成分,并且通过群体ICA组分析,进一步克服了脑影像数据信噪比低造成的难以精确识别不同脑网络成分的难题,实现数据驱动式的脑认知网络成分有效识别。
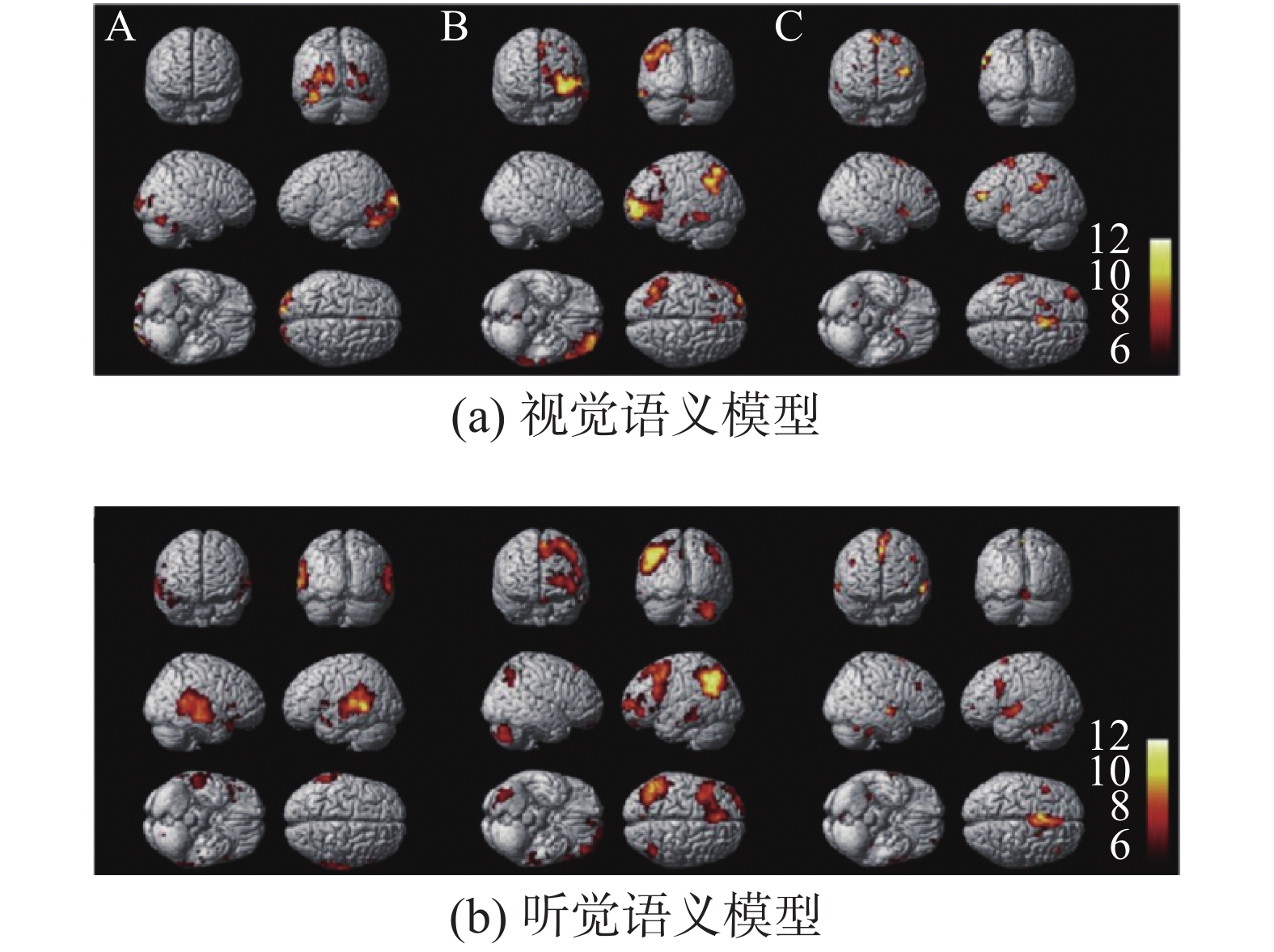
以语义通达模型为例,我们利用多变量ICA组分析方法对经典认知理论模型进行智能计算,实现对经典认知模型进行定量化表征[19]。通过给被试以视觉或听觉形式呈现词语并进行语义(是否具有危险性)判断任务,我们刻画了正常人在完成语义任务时脑内3个分离的功能网络模块:由枕叶(视觉语义任务)和颞叶(听觉语义任务)等感觉皮层区域构成的输入模块、由左半球额顶叶区域构成的语义通达模块、由前扣带回和感觉皮层以及部分额顶叶区域等构成的输出模块。通过对比视觉与听觉两种不同模态的任务网络,识别不同模态间共享语义系统的功能脑区定位(图1[9])。与视觉语义和听觉语义判断任务有关的3个认知网络模型分别对应输入 (A)、语义通达 (B) 和输出 (C) 3个模块。该研究结果不但实现了语义通达模型中3个模块的空间定位,而且直接验证了不同模态间共有的语义系统。
|
Download:
|
| 图 1 语义加工网络 Fig. 1 Semantic processing network | |
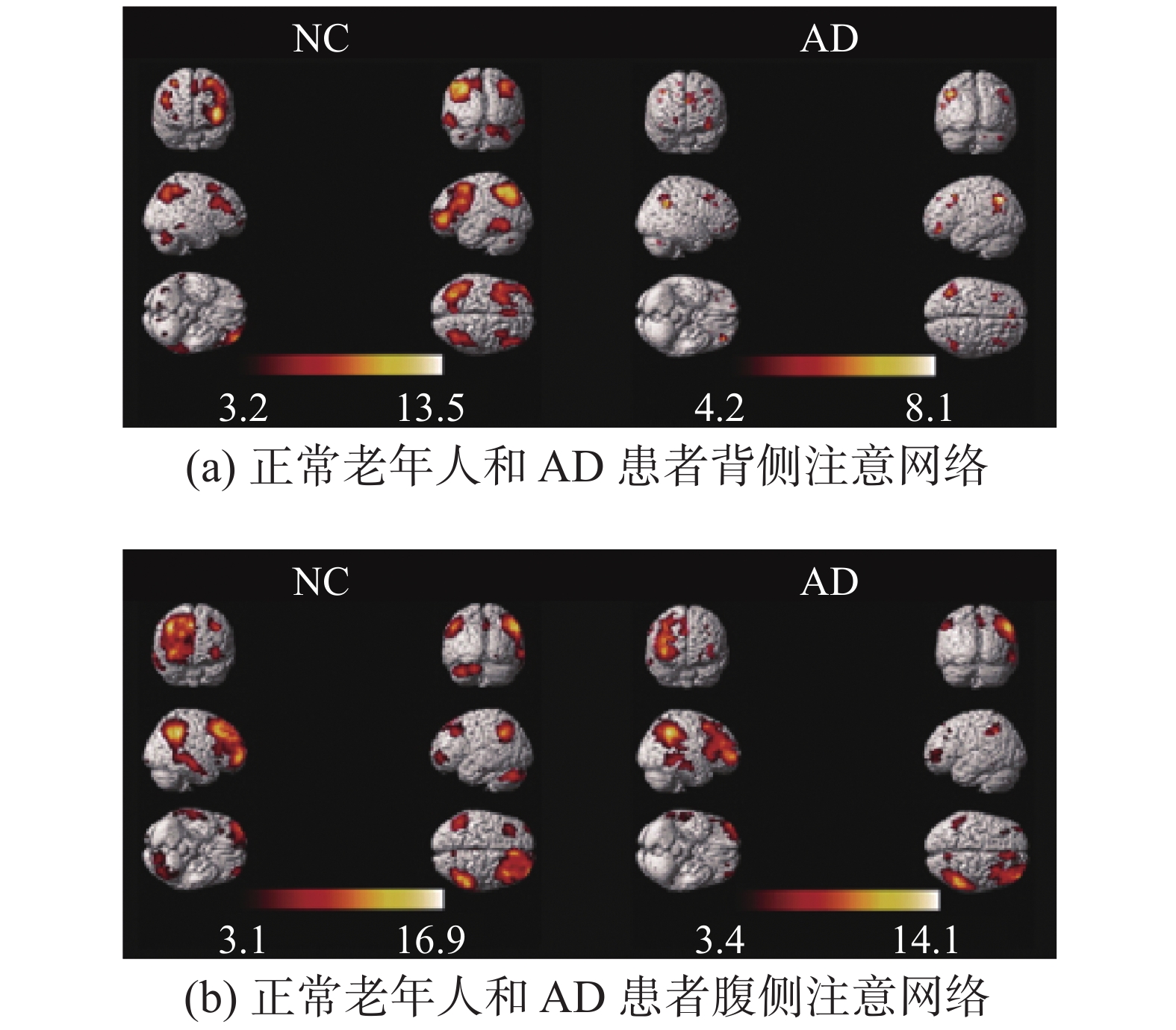
除了在任务下建立认知网络,ICA组分析在静息态下同样能够识别表征认知功能的固有脑网络,这对无法进行认知任务的脑疾病患者开展神经机制研究以及有关标记物研究具有重要应用前景。以阿尔茨海默病(alzheimer’s disease, AD)为例,本文利用ICA分离出参与自上而下注意指向的背侧注意网络(dorsal attention network, DAN)和参与自下而上处理新异刺激的腹侧注意网络(ventral attention network, VAN),发现AD选择性损伤DAN而相对保留VAN的功能连接(图2[10]),从而可以解释行为学上观察到AD在选择性注意任务时的功能缺陷和对新异刺激做出注意反应的现象[20- 21]。同时,基于受试者操作特征曲线发现在已有样本数据上,基于ICA的DAN功能活动强度和面积等特征比基于相关的传统方法构建的影像学特征在识别AD上表现出更高的敏感性和特异性[20]。这扩展了对于AD的探究仅限于以默认网络为研究对象的局限,为AD的识别和干预治疗效果评定提供更多参考,同时也为其他基于神经影像信号的脑疾病神经生物标记物临床诊断与检测提供借鉴。
|
Download:
|
| 图 2 注意网络 Fig. 2 Attention network | |
人脑静息态网络的存在体现了与任务刺激无关的脑自发活动内在的连接机制,即人脑功能网络并不是只有在执行任务时才临时组织起来,而是作为固有的连接网络存在于大脑中[22]。在这一理论观点的启发下,在分化出不同功能网络的同时,本文也关注这些功能网络在脑内如何连接起来形成一个深度连接的复杂功能网络,这对理解脑网络的组织机制和运作规律具有重要意义。基于时延关系的Granger因果模型[23]和基于条件概率关系的贝叶斯网络方法[24]作为数据驱动的有向连接智能计算方法在研究脑区间相互作用关系方面获得成功运用。其中贝叶斯网络方法因不依赖时间信息而避免了受fMRI数据时间分辨率低的影响,并能以更全局的方式通过挖掘节点变量间的条件独立和依赖关系构建连接模型,进而借助方向性连接推断节点间的信息传输和作用路径。本文将贝叶斯网络方法联合ICA引入到静息态fMRI数据分析,研究了静息态大尺度功能网络的分化和整合机制[25],并在默认网络[26]和网络间连接[27]层面研究了AD的失连接机制。
在利用ICA识别大尺度功能网络的基础上,本文基于贝叶斯网络方法构建了网络间的连接关系,发现静息态网络间按照功能层次属性以层级方式互连(图3[14]),如视觉、听觉、运动等感觉功能网络彼此之间存在显著连接并形成局部网络间连接优势性,从而利于人脑对各种外界刺激信息的感知和整合;同样认知网络之间存在紧密连接并接收来自感觉网络的方向性连接,使得认知网络可以有效合作,高效地加工处理来自感觉网络的信息,特别是发现默认网络在全脑信息的功能整合中可能发挥关键作用[14]。通过运用ICA和贝叶斯网络的方法学优势本文揭示了脑内功能网络的分化和整合特性,这种特性也是人类认知和行为灵活性的关键神经基础[17- 18]。

|
Download:
|
| 图 3 静息态下大尺度功能网络间的组织模式 Fig. 3 Organization mode of large-scale functional network in the resting state | |
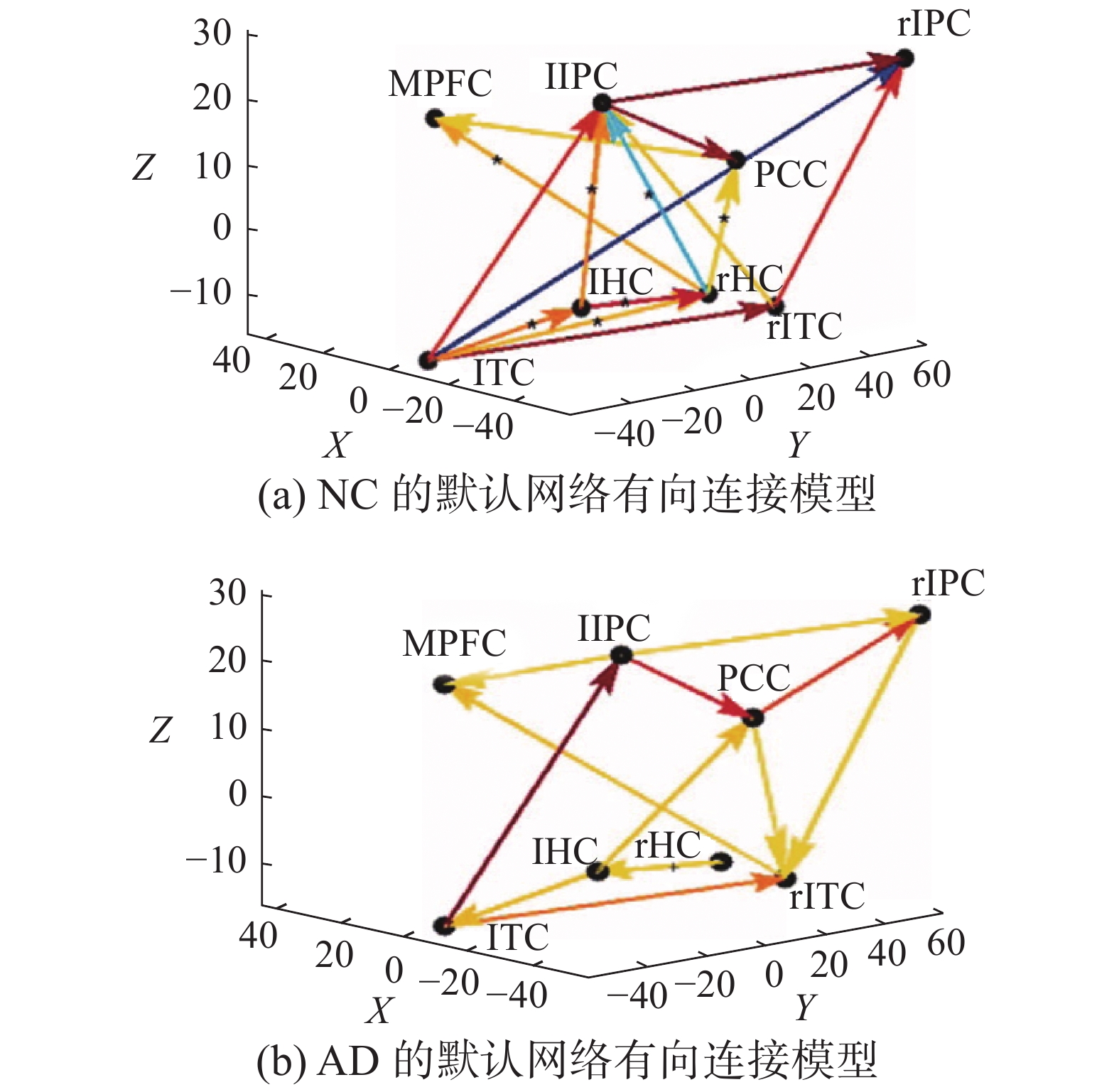
图3中:SMN为感觉运动网络;AN为听觉网络;VN1为外侧视觉网络;VN2为内侧视觉网络;VAN为腹侧注意网络;DAN为背侧注意网络;DMN为默认网络;SRN为自省网络[14]。基于贝叶斯网络的有向连接智能计算方法在定量验证脑疾病模型和机制阐释方面同样具有重要价值。如上所述,本文通过ICA揭示了AD认知网络的功能异常,采用贝叶斯网络方法从有向效应连接的变化考察网络通路上脑区间的作用关系,有助于进一步明确这些认知功能异常的失连接机制。本文首次构建了AD患者默认网络的有向连接,从连接效应的角度刻画了默认网络内脑区间依赖关系的改变,尤其是发现这些异常连接集中在海马与网络内其他脑区的效应连接(图4),结合海马是AD影响的早期靶点脑区,这个结果清晰地提示海马功能的异常是脑内广泛的失连接现象的重要原因[26]。在系统层面,本文基于ICA和贝叶斯网络模型发现AD选择性损伤高级认知网络的效应连接,而对感觉网络的效应连接影响较小,该模型可以很好地解释AD在记忆、注意等高级认知功能上的衰退[27]。对疾病模型的研究除了揭示其产生发展的机制,也为干预实践和干预效果提供指导,如老年期和AD相对保留的感知觉网络连接预示在相应功能区域具有较强的功能可塑性,这种神经可塑性资源是可以充分利用起来以寻找相应的干预方法或者开发有效干预技术的实践基础,而受损的通路和连接则是观察和评估干预效果的重要影像学指标[28-29]。此外,脑疾病模型也是研究脑运作机理的一个重要方法,如发现海马的功能异常导致连接的损伤实际上也支持海马驱动脑网络运作这一重要功能发现[30- 31]。
|
Download:
|
| 图 4 NC和AD的默认网络有向连接模型及组间比较 Fig. 4 Effective connectivity model of DMN and its comparison between NC and AD | |
模型中的节点表示默认网络脑区,其中lHC和rHC分别代表左侧和右侧海马;*表示AD减弱的连接,+表示AD增强的连接。3D 坐标系描绘了Talairach系统,其中x轴是大脑的左/右,y轴是大脑的后/前,z轴底部到大脑的顶部。
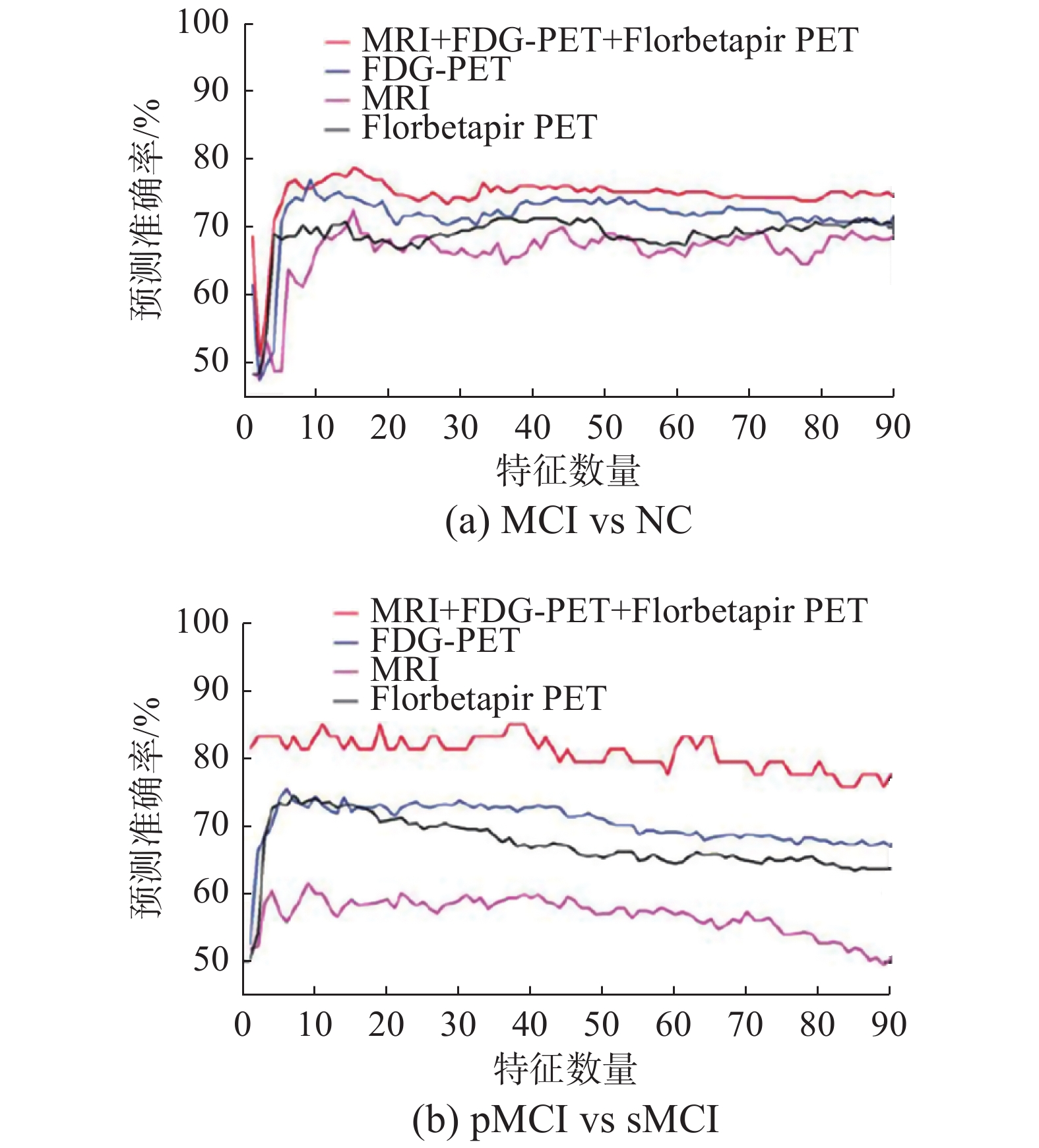
3 脑认知行为预测基于脑影像数据实现对认知功能的预测在脑科学研究领域有重要应用。在很多神经精神疾病中,认知行为的改变往往滞后于脑功能特性的变化[32- 33]。在这种情况下,如果能基于多模态神经影像数据,对认知功能改变和疾病发展轨迹进行预测,实现对脑功能性疾病的早期预防和精准干预,对患者和医院都具有重要意义[34-35]。多模态脑影像数据的异质性、特征维度高、数据复杂等问题使得传统方法无法应对这些复杂高维特征与行为之间的精确拟合[36-38]。以识别AD早期认知障碍和预测该疾病发展进程为例,本文利用国际AD神经影像计划(alzheimer’s disease neuroimaging iInitiative, ADNI)多模态影像学数据库[39],采用加权多模态稀疏表达智能算法,将加权组合的方式应用于脑影像多特征融合算法,识别出了能够区分不同疾病进程(认知正常–轻度认知障碍–AD)的模态特异性标记物,发现FDG-PET可能是相对更有效地可以鉴别轻度认知障碍及其转化的成像方法;尤其是发现融合多模态特征的模态特异性标记物对转化型轻度认知障碍(progressive mild cognitive impairment, pMCI)的预测准确率达到82.50%(图5[36]),验证了加权组合学习在预测AD认知功能行为变化轨迹方面的有效性[40]。这为退行性认知障碍疾病的早发现、早诊断提供了新思路,也提示基于智能计算实现面向大脑认知功能进行预测的应用前景。
|
Download:
|
| 图 5 加权多模态稀疏表达算法的认知障碍识别结果 Fig. 5 Cognitive impairment recognition results of the weighted multimodal sparse expression algorithm | |
心理过程的脑机制研究需要能够准确构建表征不同心理状态下的脑激活模式以及脑区间的交互关系[41-42]。当前对认知行为任务状态下大脑功能解码的研究并未充分考虑与任务条件间的因果关系,导致对心理过程的脑机制研究仍不完整。此外在实验设计或数据采集过程中,采集设备或任何实验步骤或参数的不同也都可能引起心理状态和脑功能状态的差异,进而影响神经表征的准确性。基于文献荟萃的元分析方法[43]通过对大量独立实验数据的系统定量分析可构建与特定任务相关的稳定可靠的神经表征,避免因采集设备、实验步骤或参数设置差异带来的不确定性问题。基于数据驱动的多变量Granger因果智能分析[44-45],能构建出特定行为任务状态下不同脑区间在时间上的因果作用关系,进而揭示心理过程背后的神经机制[46-47]。以社会决策任务为例,本文对过往利用最后通牒游戏范式研究公平相关神经机制的20项研究数据进行元分析,识别出公平规范执行依赖于两个神经环路来表征,由前脑岛和腹内侧前额叶构成的负责快速评估违反规范行为的直觉系统,以及由背侧前扣带回等负责整合社会规范和个人利益的反思系统[48]。在对公平规范执行进行神经编码基础上,本文采用奖惩回报刺激行为认知实验和Granger因果计算进一步揭示了这些脑区如何相互作用来参与公平规范行为,发现与惩罚规则有关的前脑岛区反应减弱,而与奖励回报提升的相关的腹侧前额区、楔前叶、背侧前额区反应增加[49]。有意思的是这与受行为主义心理学启发而发展起来的强化学习人工智能技术思想相吻合,其以回报收益最大化为目标,将一个单元作为惩罚网络的驱动者,驱动其他单元来调节惩罚代价行为[50-51]。通过对不同心理过程下的脑状态模式进行神经表征研究,不仅促进理解人类行为和认知加工的神经机制,也为类脑智能研究提供基于脑科学的理论见解。
5 结束语本文介绍的研究对人脑工作机制的理解只是冰山一隅,但看到智能计算方法在脑影像数据分析的应用对理解脑认知加工机制和脑信息处理机制提供新的途径,也为认知心理学研究及人工智能理论研究提供新的思路;同时智能影像计算在神经精神疾病领域也表现出重要应用前景,如揭示病理机制、预测疾病进程和构建生物标记等。但仍然需要注意的是,基于数据驱动的智能计算需要和认知理论驱动的实验发现有机结合起来。通过智能计算构建的脑连接等表征脑信息处理机制的模型,需要通过认知行为实验去理解这些连接方式对人类行为和认知的功能意义;而认知理论驱动下构建的脑认知模型也需要智能计算方法去验证并寻找新的线索进一步完善。从“行为”出发的传统认知实验研究和从“脑”出发的先进智能影像计算这两条研究思路主线的交叉融合和互相启发将共同推动对人脑这个复杂生命系统的理解。
| [1] |
WANG Q, MENG L, PANG J, et al. Characterization of EEG data revealing relationships with cognitive and motor symptoms in Parkinson’s disease: A systematic review[J]. Frontiers in aging neuroscience, 2020, 12: 373. ( 0) 0)
|
| [2] |
SADI M U, LANGELLA S, GIOVANELLO K S, et al. Accrual of functional redundancy along the lifespan and its effects on cognition[J]. NeuroImage, 2021, 229: 117737. DOI:10.1016/j.neuroimage.2021.117737 ( 0) 0)
|
| [3] |
YANG T, DONG X, LEI X. Hard to initiate sleep: a new paradigm for resting-state fMRI[J]. Cognitive neurodynamics, 2021: 15(1): 1-9. DOI:10.1007/s11571-020-09659-6 ( 0) 0)
|
| [4] |
HAMA T, KOEDA M, IKEDA Y, et al. Bupropion increases cerebral activation in auditory affective processing: A randomized controlled fMRI study[J]. Neuroscience letters, 2021, 749: 135716. DOI:10.1016/j.neulet.2021.135716 ( 0) 0)
|
| [5] |
GORDON I, VOOS A C, BENNETT R H, et al. Brain mechanisms for processing affective touch[J]. Human brain mapping, 2013, 34(4): 914-922. DOI:10.1002/hbm.21480 ( 0) 0)
|
| [6] |
蒲慕明. 脑科学研究的三大发展方向[J]. 中国科学院院刊, 2019, 34(7): 807-813. PU Muming. Three development directions of brain science research[J]. Bulletin of Chinese academy of science, 2019, 34(7): 807-813. (  0) 0)
|
| [7] |
APKARIAN A V, BUSHNELL M C, TREEDE R D, et al. Human brain mechanisms of pain perception and regulation in health and disease[J]. European journal of pain, 2005, 9(4): 463-484. DOI:10.1016/j.ejpain.2004.11.001 ( 0) 0)
|
| [8] |
LIU Feng, WANG Shouyi, ROSENBERGER J, et al. A sparse dictionary learning framework to discover discriminative source activations in EEG brain mapping[C]//Proceedings of the Thirty-First AAAI Conference on Artificial Intelligence. San Francisco, California, USA, 2017.
( 0) 0)
|
| [9] |
LI Kaiming, GUO Lei, NIE Jingxin, et al. Review of methods for functional brain connectivity detection using fMRI[J]. Computerized medical imaging and graphics, 2009, 33(2): 131-139. DOI:10.1016/j.compmedimag.2008.10.011 ( 0) 0)
|
| [10] |
CHUNG T, NORONHA A, CARROLL K M, et al. Brain mechanisms of change in addiction treatment: models, methods, and emerging findings[J]. Current addiction reports, 2016, 3(3): 332-342. DOI:10.1007/s40429-016-0113-z ( 0) 0)
|
| [11] |
COMON P. Independent component analysis, A new concept?[J]. Signal processing, 1994, 36(3): 287-314. DOI:10.1016/0165-1684(94)90029-9 ( 0) 0)
|
| [12] |
FRIEDMAN N, GEIGER D, GOLDSZMIDT M. Bayesian network classifiers[J]. Machine learning, 1997, 29(2/3): 131-163. DOI:10.1023/A:1007465528199 ( 0) 0)
|
| [13] |
TOŠIĆ I, FROSSARD P. Dictionary learning[J]. IEEE signal processing magazine, 2011, 28(2): 27-38. DOI:10.1109/MSP.2010.939537 ( 0) 0)
|
| [14] |
LORD L D, STEVNER A B, DECO G, et al. Understanding principles of integration and segregation using whole-brain computational connectomics: implications for neuropsychiatric disorders[J]. Philosophical transactions. series A, mathematical, physical, and engineering sciences, 2017, 375(2096): 1–21. ( 0) 0)
|
| [15] |
TURK-BROWNE N B. Functional interactions as big data in the human brain[J]. Science, 2013, 342(6158): 580-584. DOI:10.1126/science.1238409 ( 0) 0)
|
| [16] |
LYNN C W, BASSETT D S. The physics of brain network structure, function and control[J]. Nature reviews physics, 2019, 1(5): 318-332. DOI:10.1038/s42254-019-0040-8 ( 0) 0)
|
| [17] |
CALHOUN V D, ADALI T, PEARLSON G D, et al. A method for making group inferences from functional MRI data using independent component analysis[J]. Human brain mapping, 2001, 14(3): 140-151. DOI:10.1002/hbm.1048 ( 0) 0)
|
| [18] |
LONDEI A, D’AUSILIO A, BASSO D, et al. Brain network for passive word listening as evaluated with ICA and Granger causality[J]. Brain research bulletin, 2007, 72(4/5/6): 284-292. DOI:10.1016/j.brainresbull.2007.01.008 ( 0) 0)
|
| [19] |
WU Xia, LU Jie, CHEN Kewei, et al. Multiple neural networks supporting a semantic task: an fMRI study using independent component analysis[J]. NeuroImage, 2009, 45(4): 1347-1358. DOI:10.1016/j.neuroimage.2008.12.050 ( 0) 0)
|
| [20] |
LI Rui, WU Xia, FLEISHER A S, et al. Attention-related networks in Alzheimer's disease: a resting functional MRI study[J]. Human brain mapping, 2012, 33(5): 1076-1088. DOI:10.1002/hbm.21269 ( 0) 0)
|
| [21] |
FERNANDEZ-DUQUE D, BLACK S E. Selective attention in early Dementia of Alzheimer Type[J]. Brain and cognition, 2008, 66(3): 221-231. DOI:10.1016/j.bandc.2007.08.003 ( 0) 0)
|
| [22] |
BULLMORE E, SPORNS O. The economy of brain network organization[J]. Nature reviews neuroscience, 2012, 13(5): 336-349. DOI:10.1038/nrn3214 ( 0) 0)
|
| [23] |
ROEBROECK A, FORMISANO E, GOEBEL R. Mapping directed influence over the brain using Granger causality and FMRI[J]. NeuroImage, 2005, 25(1): 230-242. DOI:10.1016/j.neuroimage.2004.11.017 ( 0) 0)
|
| [24] |
MUMFORD J A, RAMSEY J D. Bayesian networks for fMRI: a primer[J]. NeuroImage, 2014, 86: 573−582.
( 0) 0)
|
| [25] |
LI Rui, CHEN Kewei, FLEISHER A S, et al. Large-scale directional connections among multi resting-state neural networks in human brain: a functional MRI and Bayesian network modeling study[J]. NeuroImage, 2011, 56(3): 1035-1042. DOI:10.1016/j.neuroimage.2011.03.010 ( 0) 0)
|
| [26] |
WU Xia, LI Rui, FLEISHER A S, et al. Altered default mode network connectivity in Alzheimer's disease-a resting functional MRI and Bayesian network study[J]. Human brain mapping, 2011, 32(11): 1868-1881. DOI:10.1002/hbm.21153 ( 0) 0)
|
| [27] |
LI R, WU X, CHEN K, et al. Alterations of directional connectivity among resting-state networks in Alzheimer disease[J]. American journal of neuroradiology, 2013, 34(2): 340-345. DOI:10.3174/ajnr.A3197 ( 0) 0)
|
| [28] |
VIDAURRE D, SMITH S M, WOOLRICH M W. Brain network dynamics are hierarchically organized in time[J]. Proceedings of the national academy of sciences of the United States of America, 2017, 114(48): 12827-12832. DOI:10.1073/pnas.1705120114 ( 0) 0)
|
| [29] |
LI Rui, ZHU Xinyi, YIN Shufei, et al. Multimodal intervention in older adults improves resting-state functional connectivity between the medial prefrontal cortex and medial temporal lobe[J]. Frontiers in aging neuroscience, 2014, 6: 39. ( 0) 0)
|
| [30] |
LI Rui, ZHANG Jing, WU Xia, et al. Brain-wide resting-state connectivity regulation by the hippocampus and medial prefrontal cortex is associated with fluid intelligence[J]. Brain structure and function, 2020, 225(5): 1587-1600. DOI:10.1007/s00429-020-02077-8 ( 0) 0)
|
| [31] |
CHAN R W, LEONG A T L, HO L C, et al. Low-frequency hippocampal-cortical activity drives brain-wide resting-state functional MRI connectivity[J]. Proceedings of the national academy of sciences of the United States of America, 2017, 114(33): E6972-E6981. DOI:10.1073/pnas.1703309114 ( 0) 0)
|
| [32] |
SOHEILI-NEZHAD S, JAHANSHAD N, GUELFI S, et al. Imaging genomics discovery of a new risk variant for Alzheimer's disease in the postsynaptic SHARPIN gene[J]. Human brain mapping, 2020, 41(13): 3737-3748. DOI:10.1002/hbm.25083 ( 0) 0)
|
| [33] |
LEVY J A, CHELUNE G J. Cognitive-behavioral profiles of neurodegenerative dementias: beyond Alzheimer’s disease[J]. Journal of geriatric psychiatry and neurology, 2007, 20(4): 227-238. DOI:10.1177/0891988707308806 ( 0) 0)
|
| [34] |
PAN Yongsheng, LIU Mingxia, LIAN Chunfeng, et al. Spatially-constrained fisher representation for brain disease identification with incomplete multi-modal neuroImages[J]. IEEE transactions on medical imaging, 2020, 39(9): 2965-2975. DOI:10.1109/TMI.2020.2983085 ( 0) 0)
|
| [35] |
HAO Xiaoke, BAO Yongjin, GUO Yingchun, et al. Multi-modal neuroimaging feature selection with consistent metric constraint for diagnosis of Alzheimer’s disease[J]. Medical image analysis, 2020, 60: 101625. DOI:10.1016/j.media.2019.101625 ( 0) 0)
|
| [36] |
LIU Cirong, YE F Q, YEN C C C, et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI[J]. NeuroImage, 2018, 169: 106-116. DOI:10.1016/j.neuroimage.2017.12.004 ( 0) 0)
|
| [37] |
CERCIGNANI M, BOUYAGOUB S. Brain microstructure by multi-modal MRI: is the whole greater than the sum of its parts?[J]. NeuroImage, 2018, 182: 117-127. DOI:10.1016/j.neuroimage.2017.10.052 ( 0) 0)
|
| [38] |
GROVES A R, SMITH S M, FJELL A M, et al. Benefits of multi-modal fusion analysis on a large-scale dataset: life-span patterns of inter-subject variability in cortical morphometry and white matter microstructure[J]. NeuroImage, 2012, 63(1): 365-380. DOI:10.1016/j.neuroimage.2012.06.038 ( 0) 0)
|
| [39] |
SHAW L M, VANDERSTICHELE H, KNAPIK‐CZAJKA M, et al. Cerebrospinal fluid biomarker signature in Alzheimer's disease neuroimaging initiative subjects[J]. Annals of neurology, 2009, 65(4): 403-413. DOI:10.1002/ana.21610 ( 0) 0)
|
| [40] |
XU Lele, WU Xia, LI Rui, et al. Prediction of progressive mild cognitive impairment by multi-modal neuroimaging biomarkers[J]. Journal of Alzheimer’s disease, 2016, 51(4): 1045-1056. DOI:10.3233/JAD-151010 ( 0) 0)
|
| [41] |
DAMASIO A R. How the brain creates the mind[J]. Scientific American, 1999, 281(6): 112-117. DOI:10.1038/scientificamerican1299-112 ( 0) 0)
|
| [42] |
BROWN J W. Time, will, and mental process[M]. New York: Plenum Press, 1996.
( 0) 0)
|
| [43] |
VAN OVERWALLE F. Social cognition and the brain: a meta‐analysis[J]. Human brain mapping, 2009, 30(3): 829-858. DOI:10.1002/hbm.20547 ( 0) 0)
|
| [44] |
SETH A K, BARRETT A B, BARNETT L. Granger causality analysis in neuroscience and neuroimaging[J]. Journal of neuroscience, 2015, 35(8): 3293-3297. DOI:10.1523/JNEUROSCI.4399-14.2015 ( 0) 0)
|
| [45] |
DESHPANDE G, LACONTE S, JAMES G A, et al. Multivariate granger causality analysis of fMRI data[J]. Human brain mapping, 2009, 30(4): 1361-1373. DOI:10.1002/hbm.20606 ( 0) 0)
|
| [46] |
KÓNYA L. Exports and growth: granger causality analysis on OECD countries with a panel data approach[J]. Economic modelling, 2006, 23(6): 978-992. DOI:10.1016/j.econmod.2006.04.008 ( 0) 0)
|
| [47] |
STOKES P A, PURDON P L. A study of problems encountered in Granger causality analysis from a neuroscience perspective[J]. Proceedings of the national academy of sciences of the United States of America, 2017, 114(34): E7063-E7072. DOI:10.1073/pnas.1704663114 ( 0) 0)
|
| [48] |
FENG Chunliang, LUO Yuejia, KRUEGER F. Neural signatures of fairness-related normative decision making in the ultimatum game: a coordinate-based meta-analysis[J]. Human brain mapping, 2015, 36(2): 591-602. DOI:10.1002/hbm.22649 ( 0) 0)
|
| [49] |
FENG Chunliang, DESHPANDE G, LIU Chao, et al. Diffusion of responsibility attenuates altruistic punishment: a functional magnetic resonance imaging effective connectivity study[J]. Human brain mapping, 2016, 37(2): 663-677. DOI:10.1002/hbm.23057 ( 0) 0)
|
| [50] |
KAELBLING L P, LITTMAN M L, MOORE A W. Reinforcement learning: a survey[J]. Journal of artificial intelligence research, 1996, 4: 237-285. DOI:10.1613/jair.301 ( 0) 0)
|
| [51] |
SUTTON R S, BARTO A G. Reinforcement learning: an introduction[M]. 2nd ed. Cambridge: MIT Press, 2018.
( 0) 0)
|
 2021, Vol. 16
2021, Vol. 16


