2. 哈尔滨工程大学 自动化学院,黑龙江 哈尔滨 150001
2. Automation College, Harbin Engineering University, Harbin 150001, China
乳腺癌是女性中最常见的癌症,也是全世界癌症死亡的主要原因之一,其死亡率仅低于肺癌[1],根据美国癌症协会(american cancer society)对2019年癌症患者的统计,在美国经确诊的浸润性乳腺癌有约268 600例新病例和非浸润性乳腺癌约62 930例新病例以及41 760例因乳腺癌死亡的病例,乳腺癌已经影响了约12%美国女性日常生活[2],早期发现可以增加患者治疗的机会进而提高患者的生存率[3-4],因此,乳腺癌的筛查及早期诊断尤为重要,现阶段,乳腺癌诊断的方法主要有:磁共振成像(magnetic resonance imaging, MRI)、正电子发射型计算机断层显像(positron emission computed tomography, PET)、超声成像和X线摄影等,其中MRI和PET价格较为昂贵且具有辐射性[5];X线摄影只能对特定的断面进行诊断,灵敏度低[6];相比于MRI、PET和X线,超声成像技术不仅价格低廉、不具有放射性,而且可以将各个断面的绝大部分的病变区域显示出来,尤其针对亚洲年轻女性较为紧密的乳腺组织,超声成像技术有着更好的检测效果,更适合于对乳腺肿瘤进行大规模的检查[7-8]。
虽然超声成像已成为早期诊断的最受欢迎的检测方式,但超声检测在实际应用中仍然存在较多的困难:
1) 超声成像过程较为复杂容易造成严重的噪声,同时鉴于乳腺肿瘤的浸润性,其对比度和分辨率比较低,边界模糊;
2) 由于超声波频率较高、缠头能力强,对于较多的小于10 mm的小肿瘤很难进行准确的鉴别;
3) 超声波束具有反射、折射等特点,而不同人体的组织器官的声阻抗具有较大差异,而极易导致超声图像生成伪像。
然而,影像科医生往往都是根据自己的工作经验来分析超声图像,借助肿瘤的形状、大小、能量、密度等相关的特征来分辨肿瘤良恶性,对于相同的一幅图像,不同医生的分析可能会有一定差异,不同时期图像的属性也有所不同,因此判读结果也会不同,除此之外,乳腺超声图像的诊断工作较为复杂,特别对于中型和大型医院病人较多、工作量较大的情况,单纯依赖医生诊断容易发生错误诊断,这将大大影响病人的有效治疗时间,给病人的生命财产造成严重的影响,基于以上原因,完全依赖医生的人工处理容易由于疲劳而造成误诊率上升的现象[8],随着人工智能相关理论的发展,基于深度学习的乳腺超声肿瘤识别技术有着重要的研究价值和意义。
1 相关工作目前乳腺肿瘤的识别方法主要可分为2类:乳腺肿瘤传统识别方法以及基于深度学习的乳腺肿瘤识别方法。
乳腺肿瘤传统识别方法包含了乳腺超声肿瘤图像预处理、区域分割、特征提取和分类4步。由于先前的过程会影响后续过程,因此这些方法都需要非常精确地处理每一个步骤,按照是否强调分类和强调目标感兴趣区域(region of interest, ROI)提取将传统方法分成2类,其中强调分类的方法通常需要人工手动或半自动获取ROI区域,更加侧重于开发区分良性恶性肿瘤的有效特征和分类器的选择;而强调ROI区域提取的方法都是侧重于开发更优的图像分割方法定位超声肿瘤的ROI区域。
Cai等 [9]首先提取出 138 个病例的乳腺超声图像的 ROI 区域,之后提出了一种结合相位一致性和局部二值模式(local binary pattern, LBP) 特征的局部纹理描述符特征,最后采用 支持向量机(support vector machine, SVM)分类器来区分乳腺超声图像中的良恶性肿瘤;Huang等[10]首先利用分水岭算法来获取乳腺超声图像初始轮廓,然后采用最小化能量函数进一步得到精确的肿瘤边界,最后采用支持向量机对乳腺肿瘤超声图像做良恶性识别;Shahriar 等[11]将得到的超声图像先寻找轮廓,之后提取纹理和统计特征,最后采用神经网络来分类;Menon等[12]首先采用基于局部区域的主动轮廓法对预处理后的乳腺超声图像精确地确定 ROI 区域,接着提取纹理及形态等特征,最后采用 SVM 对其进行分类。
这些传统的识别方法虽然在一定程度上实现了超声肿瘤识别,但都基本需要进行人工的特征提取,往往识别精度较低、鲁棒性较差、通用性不强,故很难应用到实际的诊断中。
近几年,随着深度学习的兴起,部分研究人员又提出基于深度学习的方法来进行肿瘤识别,例如:Xiao Ting等[13]提出采用深度迁移融合的方法来实现乳腺超声肿瘤识别,其采用 Resnet50、Xception 和 Inception V3 这3种卷积神经网络提取的不同的特征进行迁移融合,接着采用前向神经网络来分类。Han等[14] 首先对获取的 5 151 例患者的 7 408 张超声图像进行了 ROI 区域人工提取,之后将其输入到改进后的 GoogLeNet 网络中进行特征提取和分类,结果显示,AUC 大于 0.9,准确性为 90%,灵敏度为 86%,特异度为 96%。梁舒和王恒立等[15-16]分别采用优化后的 U-net 和全卷积网络进行超声乳腺肿瘤 ROI 区域的分割,最终在数据集上分别实现了 82.5% 、83.48% 的准确率。
目前基于深度学习乳腺肿瘤识别方法,基本都是仅限于采用图像分割的方法定位ROI区域或将给定的ROI区域分类为良性或恶性,而并不能同时实现对ROI区域的定位和对肿瘤的分类,给医护人员的诊断带了极大的不变,最近,Osman [17-18]提出采用基于改进的 FCN-AlexNet 和基于改进的 Mask R-CNN 语义分割模型实现端到端的超声肿瘤识别,同时执行定位和分类,目前这种采用图像分割的乳腺超声肿瘤识别的方法,虽然较为精确地定位超声肿瘤的边界且实现了良恶性识别,但这种图像语义分割的方法需要手动标注大量肿瘤分割的数据集,而制作分割数据集的过程非常繁琐,极大地增加了专业医生标注的负担,因此并不适合大规模的应用。
2019年5月Shin等[19]在 IEEE Transac-tions on Medical Imaging 首次提出采用基于深度学习中的目标检测算法来同时实现乳腺超声肿瘤的定位和分类,其采用Faster R-CNN算法并借助合作医院所提供的数据集对模型进行训练和测试,但由于Faster R-CNN算法检测速率和检测精度较低,无法直接应用到实际中,因此本文仍沿着这一思路,首先使用检测速率和精度均较高的YOLOv3算法进行肿瘤识别,然后针对乳腺超声肿瘤识别中的相关问题对算法进行改进。
2 传统YOLOv3算法YOLOv3算法将目标检测问题转化为回归问题,合并分类和定位任务到一个步骤,直接预测物体的位置及类别,其包含了新特征提取网络Darknet-53以及3种尺度的YOLO层,分别用于特征提取和与多尺度预测,网络结构如图1所示[20]。
其中Darknet-53层由1个卷积块DBL和5个残差块Resn(n=1,2,4,8)组成,DBL是卷积(conv)、批规范化(BN)和激活函数(leaky relu)的集合,这也是YOLOv3中的最小组件,Resn中的n表示残差网络Res的个数,而Res是2个DBL加上短连接(shortcut)组成,经过Darknet-53网络特征提取后,输出大小为13×13×1024特征图,经过上采样与浅层特征图拼接(见图1中的concat),输出3个尺度的特征图用于YOLO层的检测结果的预测,也就是每个box负责对3个Anchor box进行回归,其预测结果包括目标的中心位置x和y、宽高w和h、置信度以及类别,在YOLOv1版本中,x、y、w、h是直接预测物体实际值,预测值的微小变化都会被放大到整个图像的范围,导致坐标波动较大,预测不准确,YOLOv3采用改进后的算法求解预测值,之后通过对置信度设定阈值过滤掉低分的预测框,然后对剩下的预测框执行非极大值抑制算法(non-maximum suppression, NMS)处理,得到网络最终的预测结果。

|
Download:
|
| 图 1 YOLOv3网络结构 Fig. 1 YOLOv3 network structure | |
尽管YOLOV3在目标识别算法中表现优异,但乳腺超声肿瘤图像相对其他图像而言,边界更加模糊、噪声更大、对比度更低,导致特征更难提取,更容易出现错误识别和遗漏目标的问题,本节针对乳腺超声图像特征难提取的问题,从3个角度对YOLOv3的特征提取网络DarkNet-53进行改进并进行实验验证,以提高网络图像特征的提取能力。
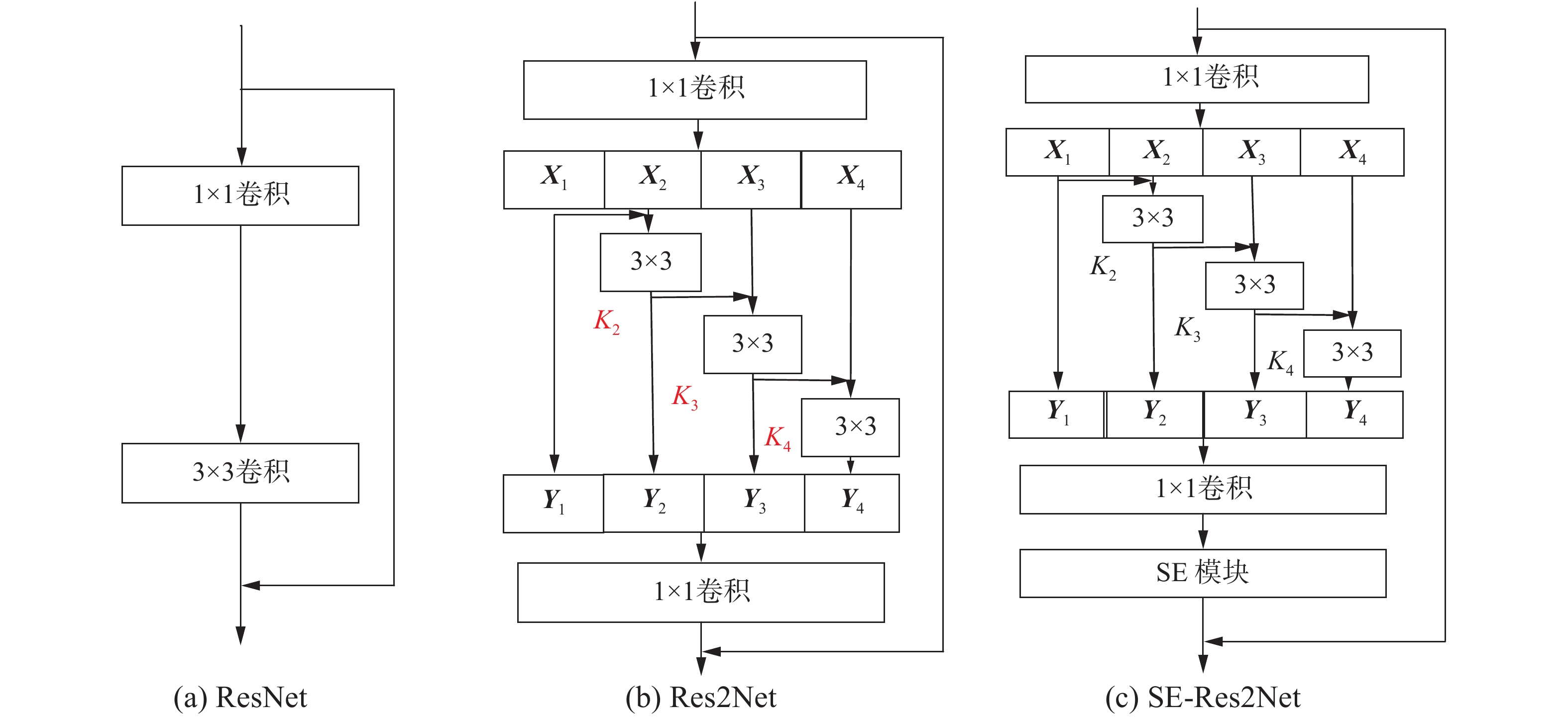
3.1 残差模块设计YOLOv3中的Residual残差模块结构在特征提取过程中参考了ResNet结构,ResNet克服了梯度消失问题,增强了特征表达力,本节中,为了扩大每个网络层的感受野,提取更加有效的特征,充分利用单层内的特征,考虑通过改进ResNet网络多样性角度来改进网络结构,提出将SE (sequeze and excitation)模块和Res2Net 2种网络进行融合组成SE-Res2Net以改进原始的ResNet结构。
SE模块是2017年由胡杰[21]提出的,SE模块则是从卷积的通道特征角度通过局部的感受野将深度信息和空间信息进行融合,其结构如图2所示。SE模块可分为Squeeze和Excitation 2个部分,其中

|
Download:
|
| 图 2 SE模块网络结构 Fig. 2 Network structure of SE block | |
SE模块具有迁移性强的特点,可以嵌入到任何的网络结构中,为最大化利用提取到的超声影像中的乳腺肿瘤特征,本节将其与Res2Net[22]结合组成SE-Res2Net,Res2Net是2019年的IEEE国际计算机视觉与模式识别会议提出的新网络,网络结构如图3(b)所示,Res2Net结构采用新的卷积方式来代替ResNet中3×3卷积,首先采用一个1×1卷积核将输入的特征图分成4个特征子图,每个特征子图均对应一个3×3卷积 (第1个特征子图除外),将每个特征子图与上一个特征子图对应的卷积的输出的和作为该子图对应的3×3卷积的输入,然后将4个通道的特征图进行融合,显然Res2Net中每个特征子图下的3×3卷积均可利用之前的特征,并且它的输出可以获得更大的感受野,其在单层内增加了尺度,扩大了感受野范围,更好地利用了上下文信息,充分结合上下文信息可以令分类器更容易检测出具体目标,同时使用多尺度的方法来提取特征使得网络的整体语义表征能力更加出色,特征表现力更强,最后在1×1卷积之后融合SE模块,能进一步结合SE模块的优点:对通道特征进行重新加权,抑制无用的特征,充分利用了单层中的特征。
|
Download:
|
| 图 3 残差网络和SE-Res2Net网络 Fig. 3 Residual network and SE-Res2Net network | |
DarkNet-53在每个残差模块前均会有一个步长为2,大小为3×3的卷积核做下采样,DarkNet-53中一共有5个这样的卷积操作,该模块容易造成特征丢失,会在一定程度上影响识别的性能,因此本节综合考虑各种因素,引入类似Inception的结构Downsample下采样模块来改进DarkNet-53的下采样结构,其首先采用多个1×1的卷积核将特征图扩展,之后采用池化层和几个不同大小的卷积核来做下采样,接着将下采样后的特征图进行融合,这种融合与ResNet残差网络的融合不一样,其只是通道的融合,特征图的尺寸不会发生改变,最后使用1×1滤波器来降低尺寸,将其尺寸变成整个Downsample下采样模块输入模块的一半,如图4所示。

|
Download:
|
| 图 4 Downsample下采样模块 Fig. 4 Downsample network | |
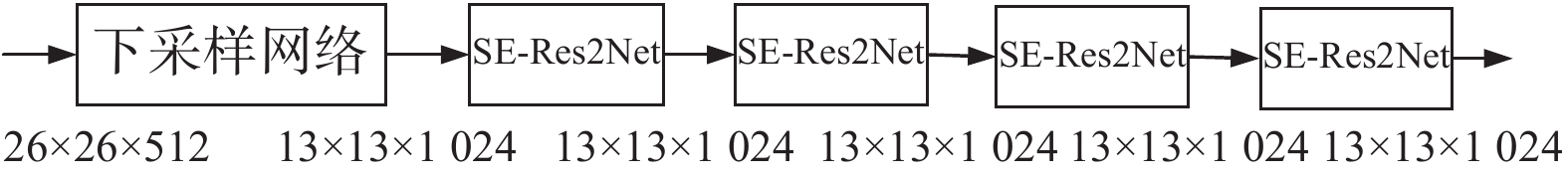
DarkNet-53每次下采样输出特征图后,均会进入一个Residual的残差模块,3.1节将网络中的ResNet替换为SE-Res2Net,3.2节将3×3的下采样模块替换为Downsample模块,为进一步提高网络的特征提取能力,在前文基础上,本节引入残差网络ResNet和密集连接网络DenseNet并将其合并为残差密集连接网络Res-DenseNet,为了方便描述改进后的网络,本节以第5个Residual的残差模块为例解释这一过程,如图5所示,其由一个Downsample下采样模块和4个残差网络Res2Net组成,输入特征图尺寸是26×26×512,先经过一个Downsample进行下采样特征图变成13×13×1024,连续经过4个SE-Res2Net后输出。
|
Download:
|
| 图 5 原始残差模块网络结构 Fig. 5 Network structure of original residual module | |
在引入密集连接网络DensetNet和ResNet后,改进后的Res-DenseNet网络如图6所示,残差密集网络Res-DenseNet在DenseNet网络基础上引入ResNet,以充分利用图像的特征,前面所有模块的输出均作为下一个模块的输入,这样越靠后的模块,特征数量就会越多,容易造成特征的冗余,为了消除冗余,提取更加高效的特征,在最后一个模块后加入1×1卷积进行降维,随着网络深度的增加,特征的增多,会造成网络难以训练,因此借鉴残差连接的思想,将残差密集网络中的所有模块均当作ResNet中的卷积层,将Downsample下采样的输出与当前模块的输出进行像素相加,这样不仅实现了内部特征上的融合,而且还会加快网络的收敛速度,除此之外,就参数上讲,添加1×1卷积层,对网络总参数量几乎没有影响。
显然,整个网络输入特征图尺寸是26×26×512,在经过下采样模块后,特征图是13×13×1024,先将所有SE-Res2Net模块的输出都两两进行了连接,使得网络中每一层输出都接受它前面所有层的特征作为输入,则每个SE-Res2Net输出后,经过特征拼接,输出特征图大小分别为13×13×1280、13×13×1536、13×13×1792、13×13×2048,考虑到最后一个残差网络SE-Res2Net输出后,经过特征拼接,特征图维度是经过下采样后特征图维度的2倍,采用一个conv1进行特征降维输出13×13×1024的特征图,之后借鉴残差网络ResNet对应通道像素相加的思想,将第一个conv3下采样输出的特征图与经过conv1特征降维后的特征图进行对应通道像素的相加,最后输出大小为13×13×1024的特征图。

|
Download:
|
| 图 6 Res-DenseNet网络 Fig. 6 Res-DenseNet Network | |
本文在Ubuntu 16.04操作系统下完成软件环境平台的搭建和实验,GPU选用NVIDIA GeForce 1080Ti,软件:Python (3.6);keras ≥ 2.1.5;tensorflow-gpu (≥ 1.4.1)。
4.1 数据集本文实验采用的乳腺超声数据是由专业医生从相关医院的数据库选取从2015-2019年不同年龄层次、地区和人种的2 011例原始病例,共计有13 586张原始图像,图像大小为768×576,如图7所示,经过专业医生筛选剔除后,得到含有肿瘤的有效图片共计3 259例,其中良性患者共计1 199例,恶性患者共计2 060例,考虑到虽然原始图像边框中含有较多的标注信息,如影像保存时间、超声探头发射频率、探测深度和部位,同时标注位置不固定,但为了提高算法的鲁棒性,方便实际应用,本实验不再进行人工的区域裁剪,而是直接使用经过筛选的原始图片作为目标图片。

|
Download:
|
| 图 7 超声乳腺肿瘤原始图像 Fig. 7 Original ultrasound images of breast tumor | |
该数据库中除了包含有图像信息外,还提供了所有病例的病理诊断结果,其良恶性均经过活检证实,经过专业医生借助ImageLab软件结合病例信息对进行肿瘤位置和类别标注,生成的PASCAL VOC格式的文件构建了本实验所需的乳腺肿瘤数据集,考虑到数据集较小,直接将数据集按照3∶1∶1分成训练集、验证集和测试集,其中训练集1 955张,良性719张、恶性1 236张;测试集652张,良性240张、恶性412;验证集652张,良性240张、恶性412,如表1所示。
| 表 1 乳腺超声数据集划分 Tab.1 Dividing breast ultrasound data sets |
本文采用了与YOLOv3[20]相同的损失函数,整体的损失函数由3部分组成:类别误差、置信度误差以及目标框误差。为了加快网络的训练速度和防止过拟合,本文采用Adma算法来做梯度优化,其中设置初始学习率为0.001,每100次迭代后学习率衰减为原来的1/10,冲量为0.9,衰减系数为0.0 002,epoch设置为25,batch size设置为8,之后对原始的YOLOv3和不同配置的YOLOv3算法在预训练模型上进行训练,不同配置的YOLOv3算法见表2所示,其中YOLOv3(1)表示在原YOLOv3算法上引入SE-Res2Net网络,YOLOv3(2)表示在原YOLOv3上引入SE-ResNet和Downsample下采样模块,YOLOv3(3)表示在YOLOv3上引入SE-Res2Net、Downsample下采样模块和残差密集网络,最后通过查准率−查全率(precision-recall, PR)曲线、精度均值(average precision, AP)和平均精度均值(mean average precision, mAP)评价模型的性能。
| 表 2 不同配置的YOLOv3算法 Tab.2 YOLOV3 algorithm with different configurations |
1) AP和mAP
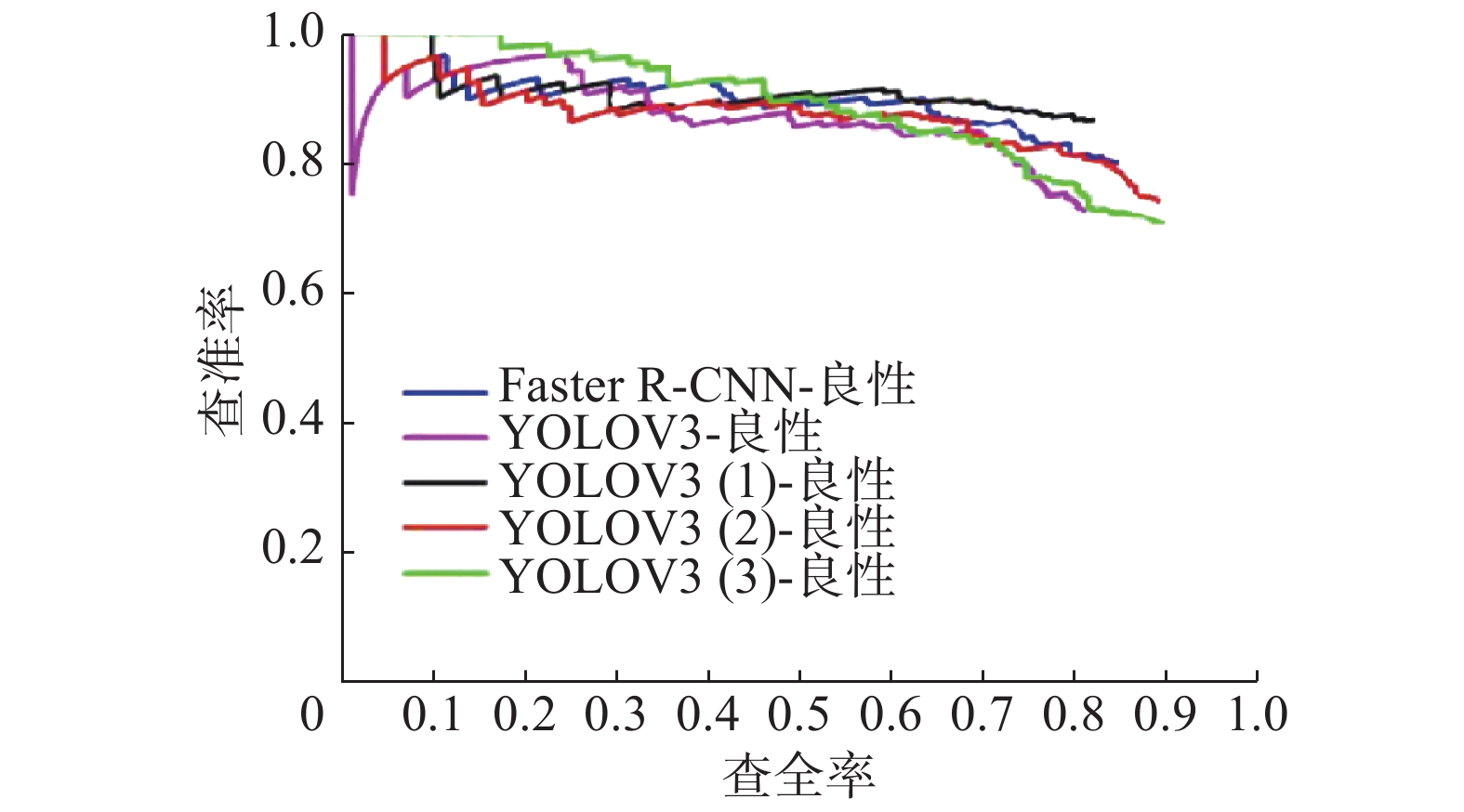
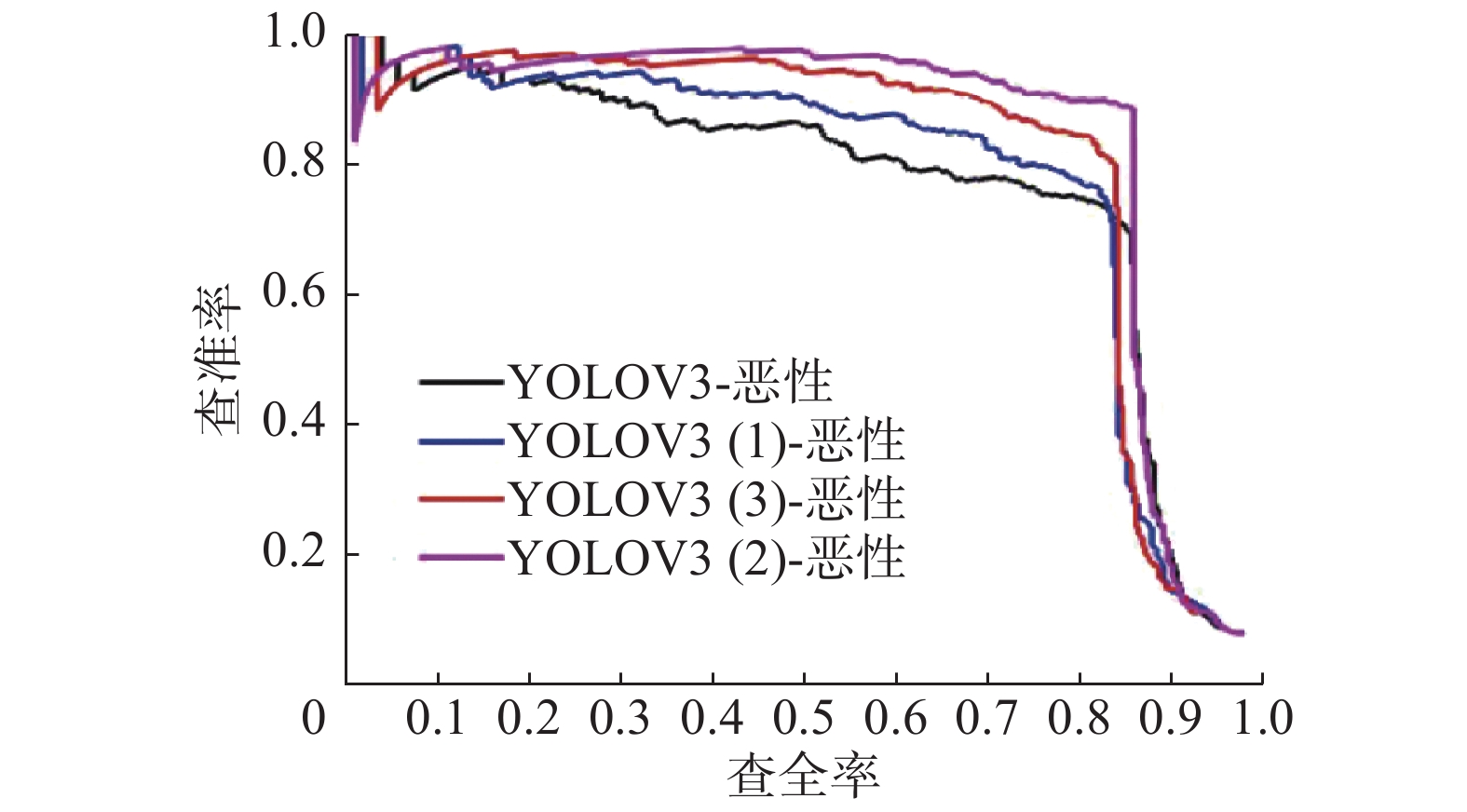
待训练完成后将模型分别在测试集测试,得到良性样本的PR曲线和恶性样本的PR曲线如图8、9所示,之后通过计算PR曲线下的面积可得到良恶性样本的AP,YOLOv3(1)良恶性AP相对YOLOV3算法提高1.59%和1.73%,其mAP提高1.66%,这是因为Res2Net在单层内增加了尺度,充分利用了单层内的特征;YOLOv3(2)良恶性AP相对于YOLOv3(1)提高了1.18%和3.08%,mAP提高2.13%,这是由于DownSample模块在一定程度上克服了3×3下采样容易丢失特征的缺陷;YOLOV3(3)良恶性AP相对于YOLOv3(2)提高了0.99%和0.56%,mAP提高0.775%,这是由于残差密集网络充分利用了前面所有层特征;YOLOv3(3)相对YOLOv3良恶性AP提高了3.76%和5.37%,mAP提高了4.56%,证实了本文在引入Res2Net、DownSample模块和残差密集网络后,YOLOv3模型的整体性能得到较大的提升。
|
Download:
|
| 图 8 测试集上良性样本的PR曲线 Fig. 8 PR curves of benign samples on test set | |
|
Download:
|
| 图 9 测试集上恶性样本的PR曲线 Fig. 9 PR curves of malignant samples on test set | |
| 表 3 乳腺超声数据集划分 Tab.3 Dividing breast ultrasound data sets |
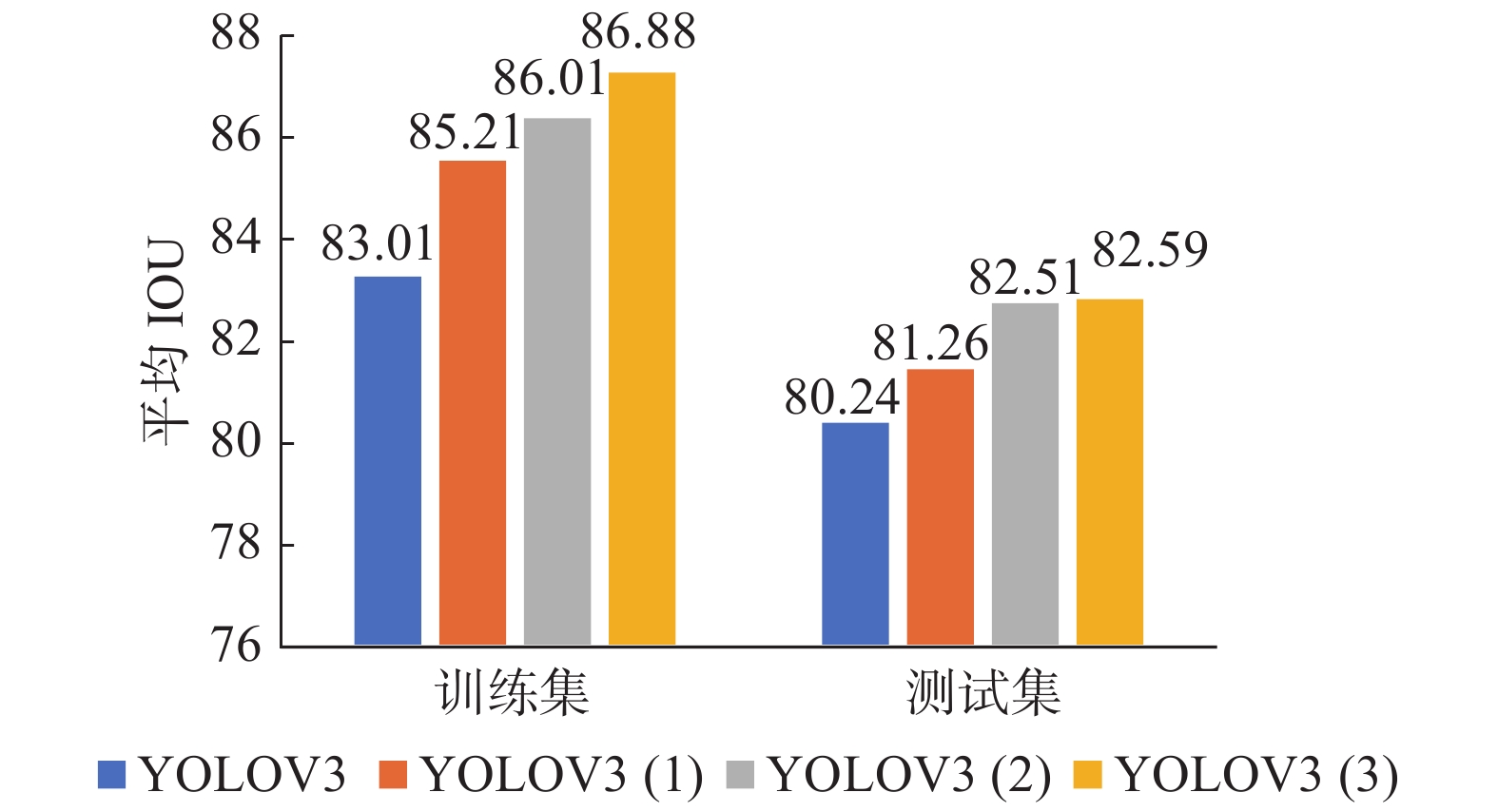
2)平均IOU
除了以上评价指标外,本文还引入平均重叠度(intersection over union, IOU)来判定肿瘤的定位误差,4种算法的在测试集上结果如图10所示,显然,在训练集上的4种算法平均IOU明显高于测试集上对应算法的平均IOU,YOLOV3、YOLOV3(1)、YOLOV3(2)和YOLOV3(3)算法在测试集和训练集上平均IOU依次增加,这也证实了本文在引入Res2Net、DownSample模块和残差密集网络后,YOLOv3特征提取能力更强。
4.3 实际测试效果从数据库中随机抽取一个恶性样本,采用改进后的YOLVO3算法进行识别,如图11所示,YOLOV3、YOLOV3(1)、YOLOV3(2)和YOLOV3(3)算法均将其识别为恶性,识别的置信度分别为95.39%、97.652%、99.31%和99.97%;从数据库中随机抽取一个良性样本,如图12所示,YOLOV3、YOLOV3(1)、YOLOV3(2)和YOLOV3(3)算法均将其识别为恶性,识别的置信度分别为97.28%、98.48%、98.89%和99.97%,显然本文在引入Res2Net、DownSample模块和残差密集网络后,YOLOV3特征提取能力更强。
|
Download:
|
| 图 10 训练集和测试集上的平均IOU Fig. 10 Average IOU on training set and test set | |
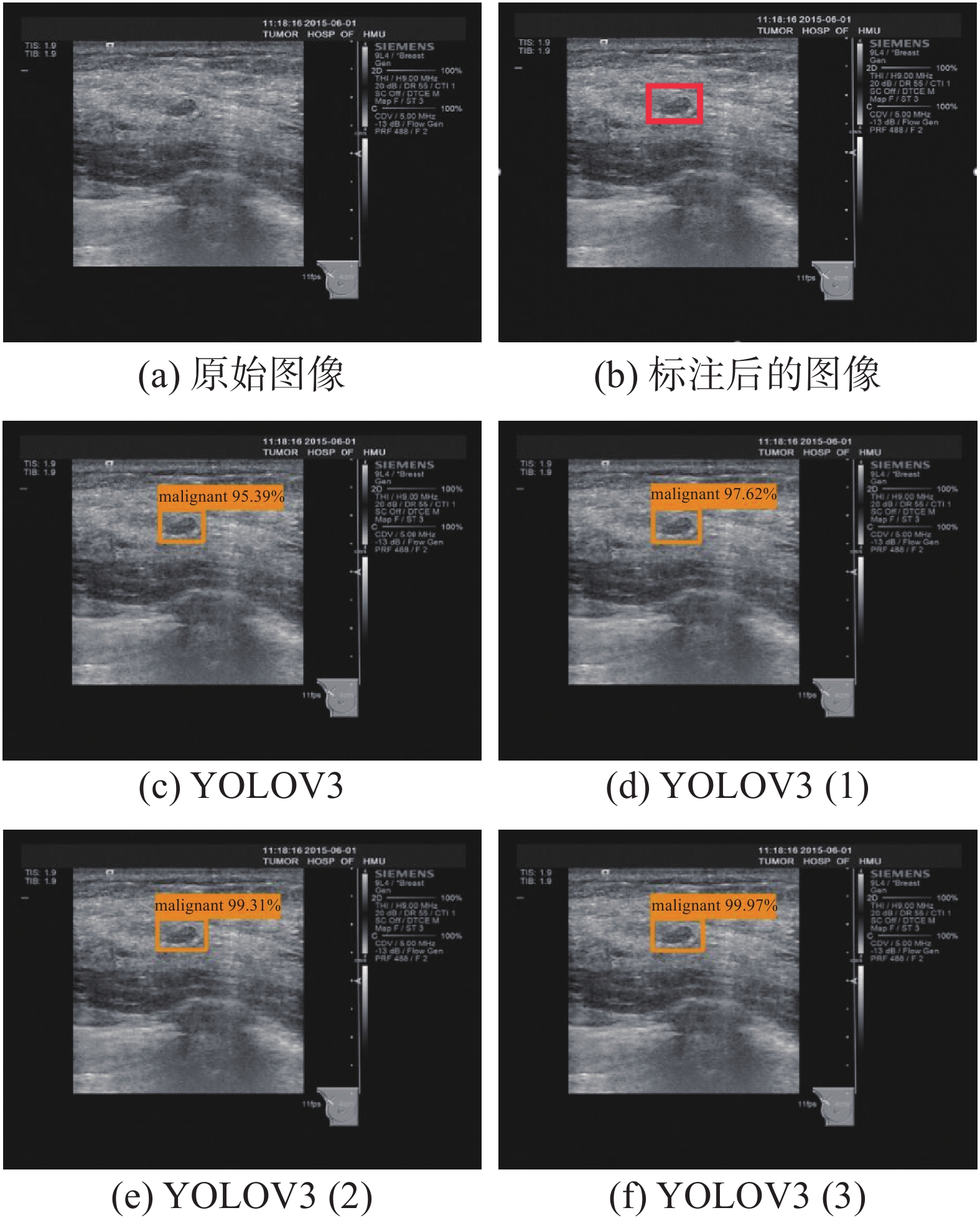
|
Download:
|
| 图 11 恶性测试样本效果展示 Fig. 11 Malignant test sample effect display | |

|
Download:
|
| 图 12 良性测试样本效果展示 Fig. 12 Benign test sample effect display | |
针对传统乳腺超声肿瘤识别方法均采用人工提取的特征逐步实现ROI区域定位和肿瘤分类往往识别精度低、鲁棒性较差且通用性不强,目前基于深度学习的方法又仅限于肿瘤ROI区域的定位或对给定的肿瘤ROI区域进行分类,本文提出采用深度学习中的YOLOv3算法同时实现了对良恶性的分类和肿瘤ROI区域的定位,同时针对乳腺肿瘤识别中的问题对算法进行了改进,经过实验证明,在引入Res2Net、DownSample模块和残差密集网络后YOLOv3算法有着更高的检测精度,其在测试集上mAP达到0.7959,平均IOU达到0.8259,相比于传统的YOLOv3算法分别提高了4.56%和2.35%,今后可进一步优化算法提高检测精度。
经专业医生鉴定,采用改进后的YOLOv3算法不仅同时实现对肿瘤ROI区域的定位和良恶性的分类,而且取得了较好的检测效果,使得人工智能应用更接近实际操作环境,有效提升基层医生诊断能力,降低专科医生工作强度,有着极大的应用价值。
| [1] |
CHEN Wanqing, ZHENG Rongshou, BAADE P D, et al. Cancer statistics in China, 2015[J]. CA: a cancer journal for clinicians, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 ( 0) 0)
|
| [2] |
SIEGEL R L, MILLER K D, JEMAL A. Cancer statistics, 2016[J]. CA: a cancer journal for clinicians, 2016, 66(1): 7-30. DOI:10.3322/caac.21332 ( 0) 0)
|
| [3] |
LO C S, WANG C M. Support vector machine for breast MR image classification[J]. Computers and mathematics with applications, 2012, 64(5): 1153-1162. DOI:10.1016/j.camwa.2012.03.033 ( 0) 0)
|
| [4] |
GINSBURG O, BRAY F, COLEMAN M P, et al. The global burden of women’s cancers: a grand challenge in global health[J]. The lancet, 2017, 389(10071): 847-860. DOI:10.1016/S0140-6736(16)31392-7 ( 0) 0)
|
| [5] |
MENEZES G L, KNUTTEL F M, STEHOUWER B L, et al. Magnetic resonance imaging in breast cancer: a literature review and future perspectives[J]. World journal of clinical oncology, 2014, 5(2): 61-70. DOI:10.5306/wjco.v5.i2.61 ( 0) 0)
|
| [6] |
袁红梅, 余建群, 褚志刚, 等. 动态增强MRI、超声及X射线对乳腺良恶性病灶诊断的对比研究[J]. 中国普外基础与临床杂志, 2015, 22(2): 246-250. YUAN Hongmei, YU Jianqun, CHU Zhigang, et al. Comparative study of dynamic contrast-enhanced breast MRI, ultrasound, and X-ray mammography in differential diagnosis of benign and malignant breast lesions[J]. Chinese journal of bases and clinics in general surgery, 2015, 22(2): 246-250. (  0) 0)
|
| [7] |
中国抗癌协会乳腺癌专业委员会. 中国抗癌协会乳腺癌诊治指南与规范(2017年版)[J]. 中国癌症杂志, 2017,27(9): 695-759. Breast cancer professional committee of Chinese anti-cancer association. Guidelines and specifications for breast cancer diagnosis and treatment of China anti cancer association (2017 Edition)[J]. China oncology, 2017,27(9): 695-759. (  0) 0)
|
| [8] |
周星彤, 沈松杰, 孙强. 中国乳腺癌筛查现状及进展[J]. 中国医学前沿杂志, 2020, 12(3): 6-11. ZHOU Xingtong, SHEN Songjie, SUN Qiang. Current situation and progress of breast cancer screening in China[J]. Chinese journal of the frontiers of medical science (electronic version), 2020, 12(3): 6-11. (  0) 0)
|
| [9] |
Cai L, Wang X, Wang Y, et al. Robust phase-based texture descriptor for classification of breast ultrasound images[J]. BioMedical Engineering OnLine, 2015, 14(1): 1–21.
( 0) 0)
|
| [10] |
HUANG Y L, JIANG Y R, CHEN D R, et al. Computer-aided diagnosis with morphological features for breast lesion on sonograms[J]. Ultrasound in obstetrics and gynecology, 2008, 32(4): 565-572. DOI:10.1002/uog.5205 ( 0) 0)
|
| [11] |
KABIR S M, BHUIYAN M I H. Classification of breast tumour in contourlet transform domain[C]//2018 10th International Conference on Electrical and Computer Engineering (ICECE). Dhaka, Bangladesh, 2018: 289–292.
( 0) 0)
|
| [12] |
MENON R V, RAHA P, KOTHARI S, et al. Automated detection and classification of mass from breast ultrasound images[C]//2015 5th National Conference on Computer Vision, Pattern Recognition, Image Processing and Graphics. Patna, India, 2015: 1–4.
( 0) 0)
|
| [13] |
肖婷. 基于深度特征迁移与融合的乳腺超声图像分类[D]. 重庆: 重庆大学, 2018. XIAO Ting. Breast ultrasound image classification on deep feature based transfer learning and feature fusion[D]. Chongqing: Chongqing University, 2018. (  0) 0)
|
| [14] |
HAN S, KANG H K, JEONG J Y, et al. A deep learning framework for supporting the classification of breast lesions in ultrasound images[J]. Physics in medicine and biology, 2017, 62(19): 7714-7728. DOI:10.1088/1361-6560/aa82ec ( 0) 0)
|
| [15] |
梁舒. 基于残差学习U型卷积神经网络的乳腺超声图像肿瘤分割研究[D]. 广州: 华南理工大学, 2018. LIANG Shu. Research on breast ultrasound image segmentaion based on residual U-shaped convolution neural network[D]. Guangzhou: South China University of Technology, 2018. (  0) 0)
|
| [16] |
王恒立. 基于全卷积网络的乳腺超声图像语义分割方法[D]. 哈尔滨: 哈尔滨工业大学, 2018. WANG Hengli. Semantic segmentation method for breast ultrasound images based on fully convolutional network[D]. Harbin: Harbin Institute of Technology, 2018. (  0) 0)
|
| [17] |
YAP M H, GOYAL M, OSMAN F M, et al. End-to-end breast ultrasound lesions recognition with a deep learning approach[C]//Proceedings Volume 10578, Medical Imaging 2018: Biomedical Applications in Molecular, Structural, and Functional Imaging. Houston, Texas, United States, 2018: 1057819.
( 0) 0)
|
| [18] |
CHIAO J Y, CHEN K Y, LIAO K Y, et al. Detection and classification the breast tumors using mask R-CNN on sonograms[J]. Medicine, 2019, 98(19): e15200. DOI:10.1097/MD.0000000000015200 ( 0) 0)
|
| [19] |
SHIN S Y, LEE S, YUN I D, et al. Joint weakly and semi-supervised deep learning for localization and classification of masses in breast ultrasound images[J]. IEEE transactions on medical imaging, 2019, 38(3): 762–774.
( 0) 0)
|
| [20] |
REDMON J, FARHADI A. Yolov3: an incremental improvement[J]. arXiv preprint: arXiv: 1804.02767, 2018.
( 0) 0)
|
| [21] |
HU Jie, SHEN Li, SUN Gang. Squeeze-and-excitation networks[C]//Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City, USA, 2018: 7132–7141.
( 0) 0)
|
| [22] |
GAO Shanghua, CHENG Mingming, ZHAO Kai, et al. Res2Net: a new multi-scale backbone architecture[J]. IEEE transactions on pattern analysis and machine intelligence, 2019:1–10.
( 0) 0)
|
 2021, Vol. 16
2021, Vol. 16


