2. 云南省高校智能传感网络及信息系统创新团队,云南 昆明 650500
2. Program for Innovative Research Team (in Science and Technology) in University of Yunnan Province, Kunming 650500, China
阿尔兹海默症(Alzheimer's disease,AD)是最常见的一种老年痴呆症,占所有痴呆症的60%~70%[1]。AD的形成原因尚未完全研究清楚[1],目前还没有任何药物可以阻止或逆转其发展,只有在AD早期时通过一些治疗来改善症状和干预其进一步的恶化[2],从而减轻患者的病情和延长存活时间。因此,AD的早期诊断对于该疾病的治疗具有非常积极的作用。在AD发病之前,患者会经历早期轻度认知障碍(early mild cognitive impairment,EMCI),该阶段是老年正常认知衰退和痴呆症严重认知衰退的中间阶段,此时的大脑已具有轻微的认知和记忆障碍,但不会影响患者的日常生活,因此在临床检测中很难被发现。已有研究表明,患有EMCI的个体患AD的风险高于正常人,约为正常人的10倍[3-5]。因此,准确诊断EMCI对AD的治疗有着十分积极的作用。
目前,较准确的EMCI诊断方法为生物标志物诊断,如通过脑脊髓液完成[6],然而脑脊髓液抽取属有创诊断,且诊断过程也较为复杂。另外一种对EMCI的诊断方法是让患者完成神经心理学问卷[7],再经神经心理学医师进行评估,但这种诊断往往带有一定主观性,只能作为一种辅助诊断方式。近年来,随着神经影像技术的发展,具有无创的核磁共振成像(magnetic resonance imaging,MRI)和功能性磁共振成像(functional magnetic resonance imaging,fMRI)技术被越来越多的用于AD的诊断中[8]。fMRI反映了受试者执行特定任务时血氧水平依赖(blood oxygen level dependent, BOLD)信号的波动情况,相比MRI数据,其不仅包含结构性信息还包含功能性信息,只是获取fMRI数据需要设计合理的实验任务[9]。为避免实验任务因素对fMRI数据的影响,越来越多的研究[10]表明静息态fMRI(resting state fMRI, rs-fMRI)可替代fMRI来完成EMCI的诊断。由于在rs-fMRI数据获取时受试者不用完成任何特定任务,因此避免了实验任务对数据所带来的影响。
本文采用深度学习的分类方法,通过rs-fMRI数据来进行EMCI分类,以辅助AD的早期发现。首先将大脑划分成若干个兴趣点(region of interest, ROI)[11],依照ROI从rs-fMRI数据中抽取出时间序列来形成一个二维矩阵,再用一个二维卷积神经网络(convolutional neural networks,CNN)从该二维矩阵中提取特征信息,以此进行后面的分类。同时,为了提高运算速度和减少训练时间,CNN采用迁移学习网络,用一种轻量化CNN网络——拥有从ImageNet数据库[12]中预训练权重的MobileNet来提取ROI的特征信息。
1 相关工作AD症被视为一种神经退化性疾病,疾病早期的症状常被误认为是正常的老化,错过了最佳诊断时机。AD症的前期诊断一般通过病史收集和行为观察获得,耗时又耗力。若要较为准确的诊断,则需要进行详细的神经心理学检查才有可能判断是否存在认知障碍[13]。随着机器学习技术的发展,人们发现作为一种辅助诊断方法,机器学习可以提高诊断的效率和准确率。
在传统的机器学习分类技术中,支持向量机(support vector machine, SVM)是一类具有较高分类准确率的诊断方法[14],但SVM的准确率依赖于提取的特征值,当特征值本身未能准确反映疾病的特征,则其准确率往往会达到一个瓶颈。近年来随着深度学习的兴起,出现了很多可以自动从分类数据中提取有效特征值的方法,如堆叠自动编码器(stacked auto-encoders, SAE)[15]和CNN[16]等,这些方法均使得分类准确率得到了很大的提高。通常,深度学习的网络较为复杂,权重数量巨大,且需要海量的训练数据作支持,这都使得深度学习的训练耗时耗力。然而,公开的AD病症数据非常有限,AD早期病症的数据更为稀少,因此深度学习的分类准确率将严重依赖于疾病数据的完备性。为了解决该问题,近年来迁移学习被提出来对AD进行诊断,例如AlexNet[16]和VGG16[17]等。由于迁移学习网络已在其他数据库进行了预训练,只需在AD数据库中只训练其顶层即可,因此可以缩短训练时间,并可减少分类对训练数据的依赖。然而,迁移学习的预训练网络是否对EMCI的诊断有效,目前仍无文献进行报道,这也是本文关注的一个重点。
机器学习可以同神经影像学相结合以完成疾病的诊断,常用的影像技术包括电子计算机断层扫描(computed tomography, CT)、正电子发射型计算机断层显像(positron emission computed tomography,PET)、MRI和fMRI[18]等,其中又以MRI和fMRI的低辐射和无创性而得到了广泛的关注。对于MRI,其成像较为清晰,且包含信息丰富,因此被广泛应用于对AD的诊断[19]。通过对皮质体积、皮质厚度及皮质下体积等脑部结构数据,MRI可以较准确预测MCI到AD的转变。然而,对EMCI患者来说,其脑部结构变化并不明显,因此仅使用MRI数据对EMCI的检测还是具有一定限制。最近有研究表明,AD还表现在某些脑区功能连接的变化上[20],而fMRI又恰恰能反映功能连接特性,因此可以使用fMRI数据来有效诊断AD和EMCI。然而,采集fMRI通常需要被试者完成特定的实验任务,因此fMRI数据又往往与完成的任务有关。其实,大脑即使在不完成任何特定任务时,各脑区仍然在相互作用,存在一个默认的功能网络,而这种默认模式在一些认知障碍的大脑疾病中通常遭到了破坏,因此rs-fMRI也逐渐地用在EMCI诊断中[21]。与fMRI数据相比,rs-fMRI的获取不需要被试者完成预先设定好的实验动作,仅需要被试者保持休息状态,因此避免了人为因素对数据的干扰。
通常,rs-fMRI为一四维数据,用于深度分类网络需先降维。常用的一种降维方式是,将脑区划分成一些ROI,从这些ROI中提取BOLD信号,再从这些BOLD信号提取特征值,如利用SAE[22]或受限玻尔兹曼机(restricted Boltzmann machine,RBM)[23]提取特征值,最后利用这些特征值进行分类。由于输入数据的降维不可避免带来信息丢失,而不降维又容易产生较复杂的分类网络,因此,在rs-fMRI中如何解决分类网络的数据输入是本文需要关注的另一个重点。
2 问题提出由于深度学习能自动提取特征值,且具有较高的分类准确率,因此本文将采用深度学习的方法来进行EMCI的诊断。同时,由于rs-fMRI无需完成任何实验任务,本文也采用rs-fMRI数据来进行诊断。
将rs-fMRI应用于深度学习的EMCI诊断首先需要解决数据输入的问题,如图1所示,既要保证输入到分类网络的数据降维后应包含丰富的分类信息,又能降低分类网络的复杂性,以确保较少的训练时间。
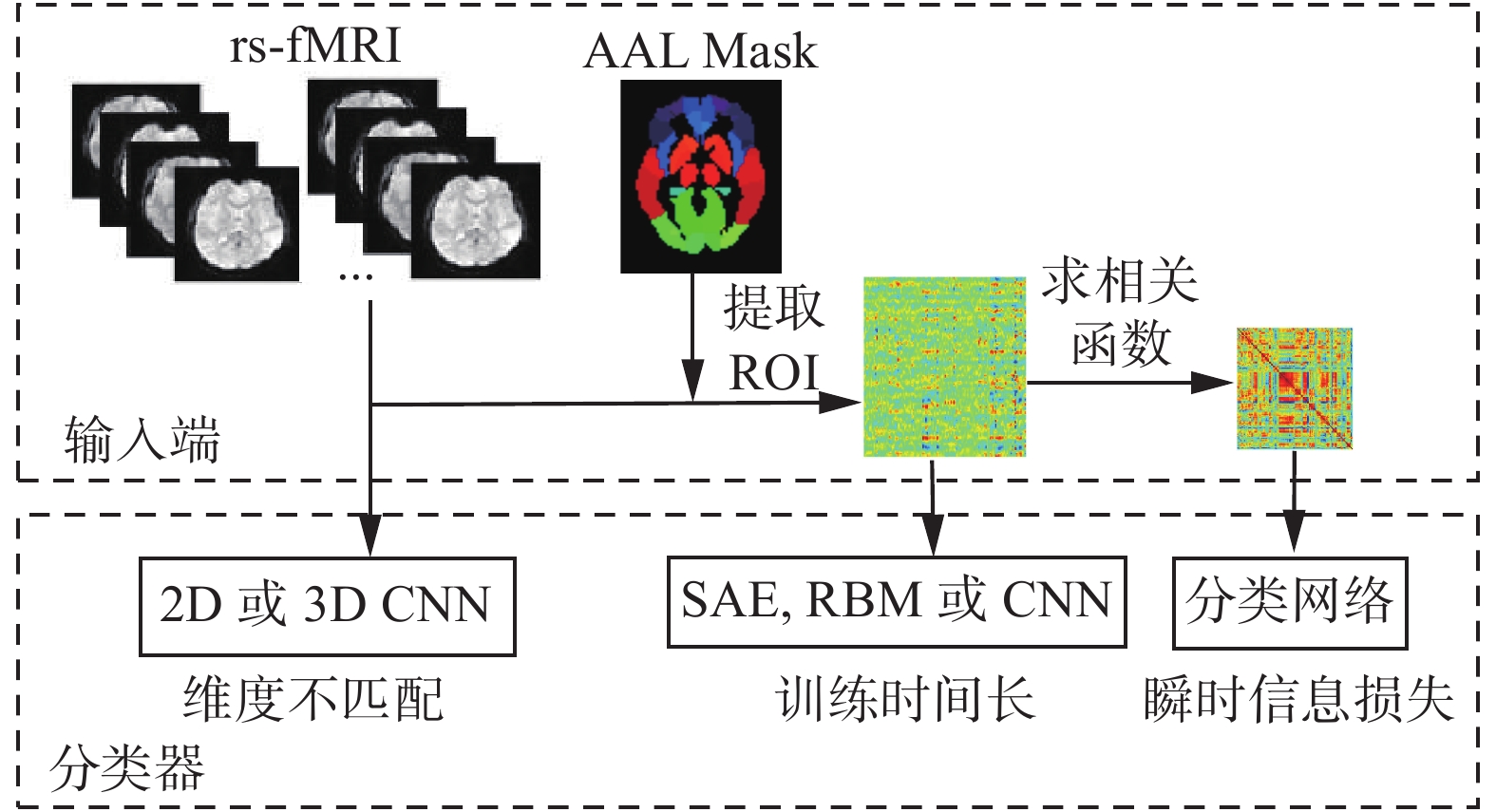
|
Download:
|
| 图 1 特征提取的相关问题 Fig. 1 Issues related to feature extraction | |
若用
一种常用的降维方式是使用自动解剖标记(automated anatomical labeling,AAL)[25]图谱来得到一个
| ${W_{{\rm{SAE}}}}{\rm{ = }}2{N_T}{N_A}{S_1}{S_2}\cdots{S_{{N_s}}}$ | (1) |
从式(1)看到,由于SAE采用的是全连接网络,因此其权重的数量与神经元数量和隐藏层数相关,当这两者数量增大时将不可避免使得整个网络权重数变得庞大。当然,CNN也可以自动提取特征值,目前的CNN利用卷积运算对权重进行共享,相比全连接层可以减少权重数量。假设一个CNN网络共有
| ${W_{{\rm{CNN}}}} = \sum\limits_{i = 1}^{{N_c}} {{F_i}{F_i}{L_i}({M_i}{M_i} + b)} $ | (2) |
其中
利用脑区功能性连接网络理论来提取特征值的方法可以进一步对数据降维[20],其本质是求统计相关系数,若
| ${R_{xy}} = \frac{1}{T}\sum\limits_{t = 0}^{T - 1} {{x_t}{y_t}} $ | (3) |
因此,对于一个
本文使用rs-fMRI数据以及迁移学习的CNN网络来对EMCI进行诊断,方法总体框架如图2所示,把患有EMCI和正常控制组(normal control,NC)的rs-fMRI数据进行配准、头动校正、颅骨剥离、归一化、平滑及滤波等预处理操作,然后使用AAL对完成预处理数据提取ROI得到相应的二维时间序列,随后输入到已预训练的迁移网络,以此得到瓶颈(bottneck)特征,最后将该特征输入到设计好的TOP层中得到分类结果,完成疾病诊断。
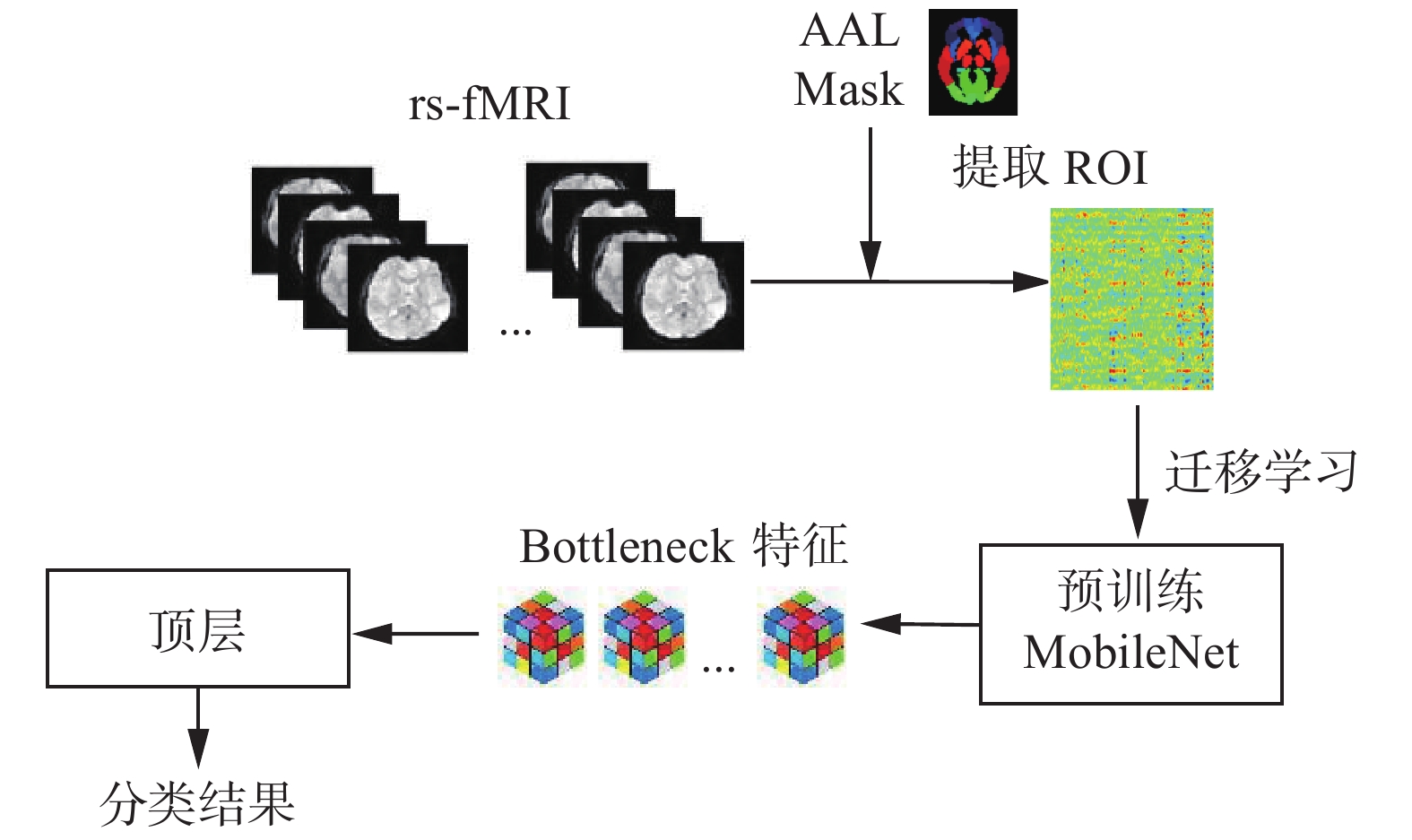
|
Download:
|
| 图 2 算法总体框架 Fig. 2 Algorithm overall framework | |
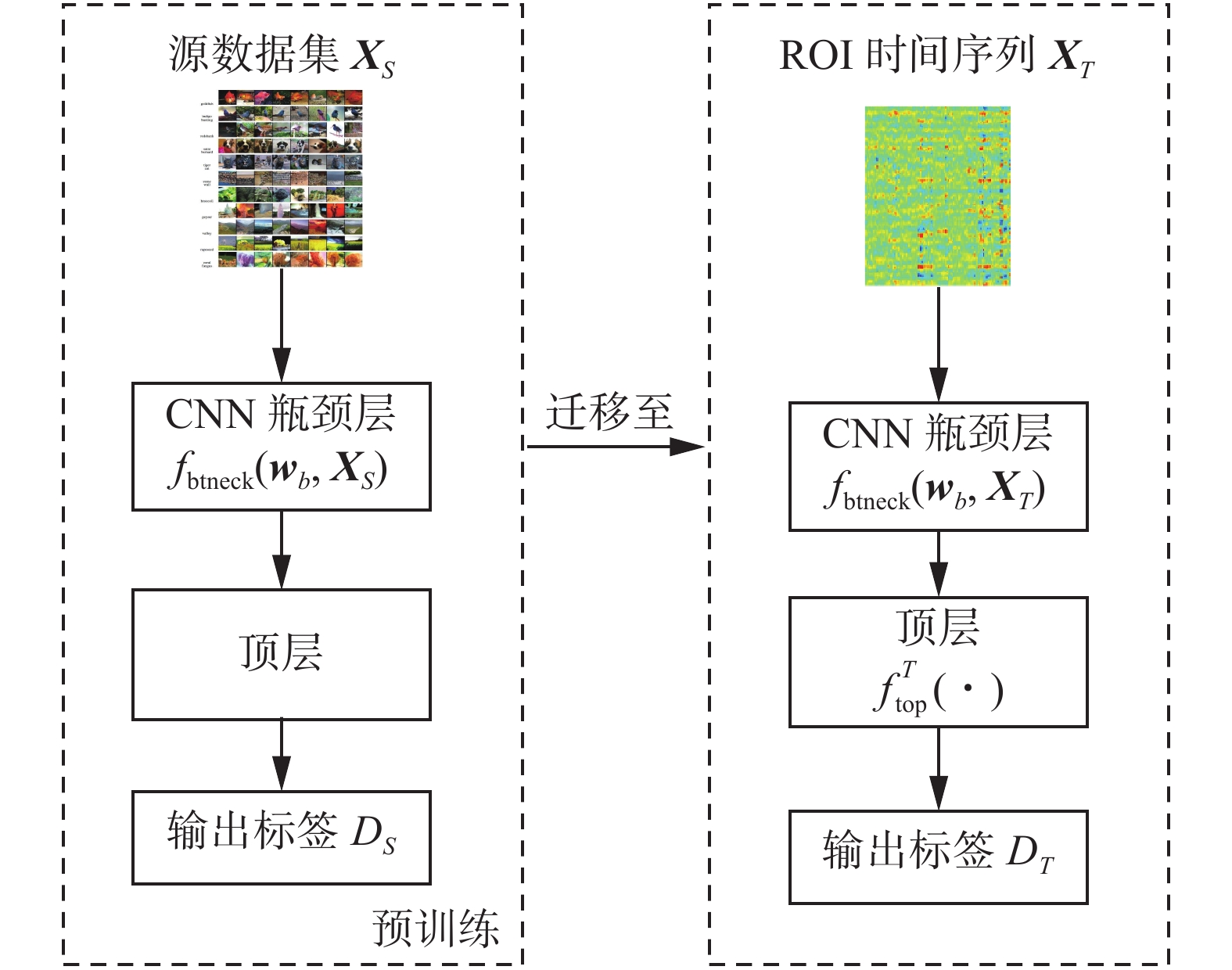
将一个具有脑区空间和时间维度的四维rs-fMRI信号直接作为分类器的输入,维度较高,可从其中提取ROI时间序列来降维,但从ROI时间序列提取特征值,仍需训练。如前所述,SAE和CNN网络提取特征将面临权重数庞大和训练时间增多的问题,而采用相关法提取特征又容易丢失瞬时信息,因此本文考虑采用迁移学习方法从ROI提取特征值。
设
| ${D_S}=f_{{\rm{top}}}^S\{ {\boldsymbol{w}}_t^S,{f_{{\rm{btneck}}}}({{\boldsymbol{w}}_b},{{\boldsymbol{X}}_S})\} $ | (4) |
式中:
预训练完成后,将由四维rs-fMRI数据张量
| ${\boldsymbol{F}} = {f_{{\rm{btneck}}}}({{\boldsymbol{w}}_b},{{\boldsymbol{X}}_T})$ | (5) |
其中
| ${D_T}=f_{{\rm{top}}}^O\{ {{\boldsymbol{w}}_t^O},{\boldsymbol{F}}\} $ | (6) |
则本迁移学习任务完成,其中
图3给出了式(6)的迁移学习过程,图中预训练部分通常无需在本地端完成,即使瓶颈层的权重
|
Download:
|
| 图 3 迁移学习提取ROI特征 Fig. 3 Transfer learning to extract ROI features | |
其实,图3的EMCI目标网络训练仅仅对一顶层网络进行训练,而顶层只是一个浅层神经网络,其权重
作为一种轻型的CNN网络,Mobilenet[26]使用深度可分离卷积替代原有的传统卷积以减少计算量,本文将采用该网络来作为迁移学习函数

|
Download:
|
| 图 4 深度可分离卷积 Fig. 4 Depth separable convolution | |
对于一个
| ${D_K} \times {D_K} \times M \times N \times {N_T} \times {N_A}$ | (7) |
在深度可分离卷积中,先使用深度卷积为每个输入通道应用单个滤波器,那么深度卷积的计算成本为
| ${D_K} \times {D_K} \times M \times {N_T} \times {N_A}$ | (8) |
然后使用点卷积(1×1的卷积核)来创建深度层的输出的线性组合,其计算成本为
| $M \times N \times {N_T} \times {N_A}$ | (9) |
所以深度可分离卷积的计算成本为
| ${D_K} \times {D_K} \times M \times {N_T} \times {N_A}{\rm{ + }}M \times N \times {N_T} \times {N_A}$ | (10) |
因此,深度可分离卷积的乘法次数与标准卷积之比为
| $\frac{{{D_K} \times {D_K} \times M \times {N_T} \times {N_A}{\rm{ + }}M \times N \times {N_T} \times {N_A}}}{{{D_K} \times {D_K} \times M \times N \times {N_T} \times {N_A}}}{\rm{ = }}\frac{1}{N}{\rm{ + }}\frac{1}{{D_K^2}}$ | (11) |
由式(11)可知,由MobileNet得到瓶颈层所需计算量与相同规模网络比,可大大减少。因此从计算量角度,选择MboileNet作为迁移网络是一个较好的选择。
3.4 顶层分类网络将ROI时间序列经预训练的瓶颈层MobileNet就得到瓶颈特征,随后再将瓶颈特征输入到设计的顶层
| $\left( {{N_{{\rm{GAP}}}} \times {N_{{\rm{GAP}}}} \times {C_1} + \sum\limits_i^{m - 1} {{C_i} \times {C_{i + 1}} + 2{C_m}} } \right) \times \left( {1 - r} \right)$ | (12) |
式中:

|
Download:
|
| 图 5 顶层设计 Fig. 5 Top-level design | |
另外,顶层中的一些参数设定会影响最后的分类结果,如全连接层层数
最后给出本文EMCI分类算法训练的步骤,如下所示:
1)预处理:对rs-fMRI信号的观测值
2)ROI提取:由AAL图谱从预处理数据中提取ROI时间序列
3)瓶颈特征提取:由
4)训练:在训练集中,通过式(6)训练得到顶层权重
5)验证:由验证集,通过式(6)得到分类结果,并计算分类准确率;
6)重复执行步骤4)~5)至分类准确率收敛。
4 实验设置及结果 4.1 数据来源及预处理在本实验中,所采用的rs-fMRI数据均来源于阿尔茨海默病神经影像学数据库(Alzheimer’s disease neuroimaging initiative,ADNI),其网址为
| 表 1 rs-fMRI数据相关参数 Tab.1 rs-fMRI data related parameters |
数据预处理采用数据处理和脑成像分析(data processing & analysis of brain imaging,DPABI)工具箱 [27],具体过程如下:
1)去除每个被试者前10帧图像,仅保留130帧图像;
2)以第48帧图像为基准对所有的图像进行校准,使每个切片上的数据具有相同时间点;
3)对所有被试进行头动校正,将头动校正到同一位置,并为接下来的图像质量控制提供数据;
4)将与rs-fMRI数据匹配的MRI数据进行颅骨剥离,然后同功能图像进行配准,将MRI数据变换到rs-fMRI数据的空间,使得rs-fMRI数据可以被分割成脑脊髓液、灰质和白质信号;
5)去除一些混淆因素,将全局平均信号、脑脊髓液和白质信号被作为无关变量去除,且以6个头动参数(包含3个转动参数和3个平动参数)去除头动带来的影响;
6)评估被试者头部的旋转和平移,发现所有参与者都没有表现出过度(任一方向平移超过2 mm或任一方向转动超过2°)的头部运动;
7)进行归一化和平滑,并将所有数据都过滤到一个频率范围(0.01~0.08 Hz);
8)使用AAL图谱将大脑分为116个ROI,如图6所示,每个半脑球包含58个区域,最后得到一个116

|
Download:
|
| 图 6 大脑节点 Fig. 6 Top-level design | |
在实验中,采用两种方法对rs-fMRI进行降维,一种仅通过AAL提取ROI,一种为提取ROI后再求pearson相关系数,因为pearson相关系数通常用于衡量功能连接的强度[28],因此也可以使用它来衡量不同大脑区域之间的功能连接。其具体参数详见表2。在对数据降维后,采用两种方法提取特征值和分类,分别采用SAE和迁移网络MobileNet,具体参数也由表2列出,由此我们分别得到以下4种方法的分类结果:
1)SAE_ROI:由AAL得到ROI,再由SAE提取特征值后分类;
2)SAE_Corr:由AAL得到ROI,再求pearson相关系数,再由SAE提取pearson相关系数的特征值后分类。
3)Transf_Corr:由AAL得到ROI,再求pearson相关系数,再由MoblieNet提取pearson相关系数特征值后进入TOP层分类;
4)Transf_ROI:由AAL得到ROI,再由MoblieNet提取ROI特征值后进入TOP层进行分类,计算步骤也可参见表1。
以上方法的分类方法都将采用
| 表 2 相关实验方法参数 Tab.2 Related experimental method parameters |
除了统计分类准确率,实验还使用敏感性和特异性来评估分类方法的性能。灵敏度为检测出真阳性数与实际真阳性数比值,越大的值检测EMCI的性能越好。特异度为检测出真阴性数与实际真阴性数比值,越大的值检测非EMCI的性能越好。
另外,文中所用的SAE基于DeepLearnToolbox-master,下载地址为
首先,给出各方法的分类准确率,如表3所示。从表3可以看到,SAE_ROI的分类准确率为59%,其余3种方法均超过了59%,这表明利用SAE提取ROI特征的方法分类准确率并不高。在用相关系数降维的方法中,利用迁移学习提取相关系数特征值的方法要优于用SAE的方法约0.17%。分类准确率最高的是利用迁移学习提取ROI特征值的方法,它能使分类准确率达到约74%,与其余的SAE_ROI、SAE_Corr和Transf_Corr3种方法的分类准确率相比,分别提升了近14.67%、10.17%和10%。该结果也表明了,利用迁移学习提取ROI特征值在分类信息的损失上要小于提取相关系数特征值,因此分类准确率也得到了提高。
| 表 3 分类方法的分类准确率 Tab.3 Classification accuracy of classification method |
表3还给出了灵敏度和特异度的实验结果。从该结果中可以发现,Transf_ROI的灵敏度为0.760,高于Transf_Corr(0.703)、SAE_Corr(0.650)和 SAE_ROI(0.583)。而Transf_ROI的特异度为0.713,这在所讨论的方法中也是最高的,高于Transf_Corr(0.570)、SAE_Corr(0.620)和 SAE_ROI(0.597)。
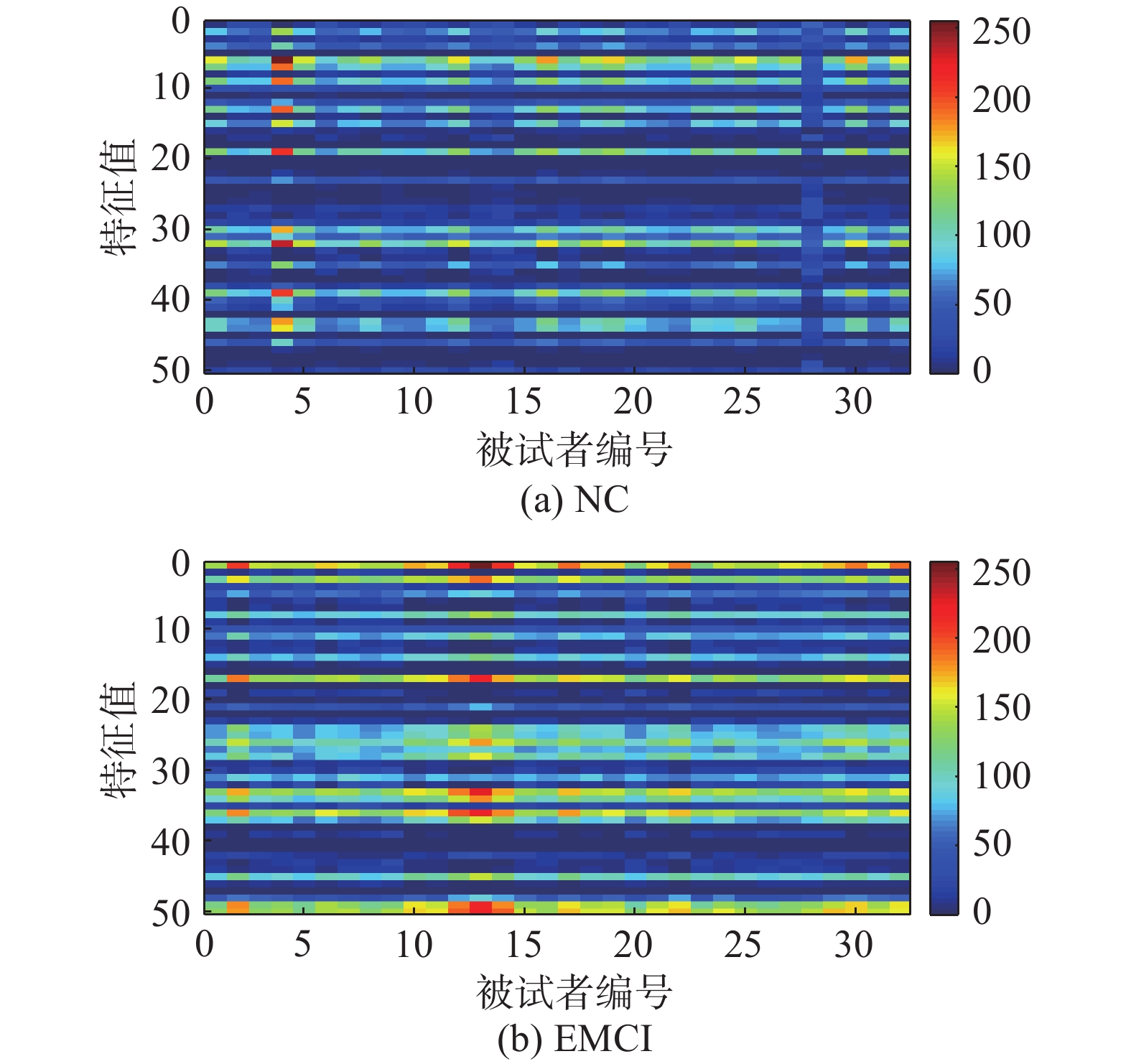
图7给出了采用Transf_ROI方法所提取的特征值结果图,其中图7(a)是在TOP层分类网络的第3全连接层所提取的所有NC特征值均值,图7(b)是相应所有EMCI特征值均值。从图中可以看到,虽然两幅特征值图的绝大部分像素值均在50左右,但是还是存在一定的差异性。例如,对于EMCI组,其在第13列的3、17、35和50行的像素值一道道200左右,而NC组相应位置仍在50左右。该结果也与表3的结果一致,表明利用迁移学习提取特征值在两个组别确实存在差异性,因而可以将两个组别的被试进行分类。
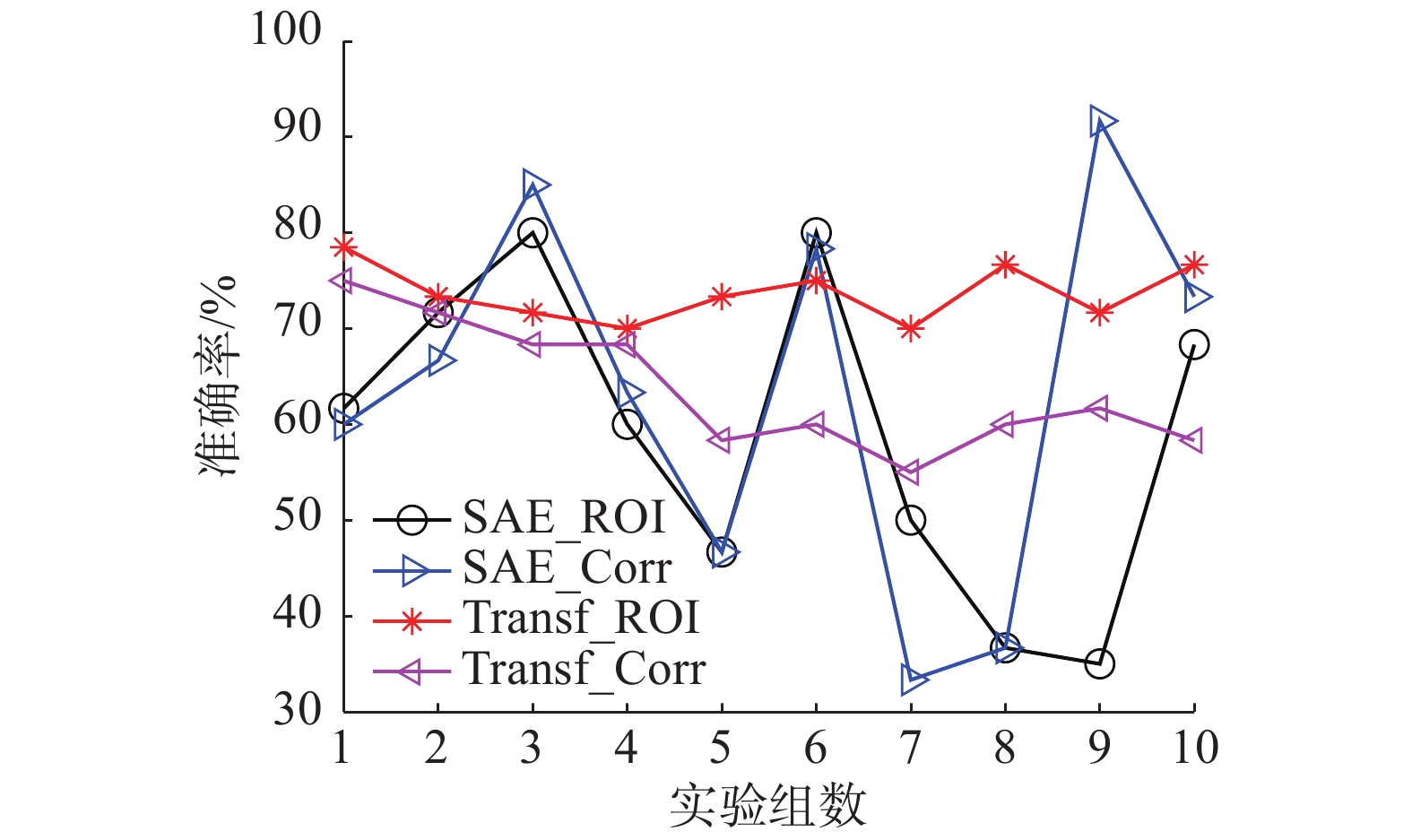
图8给出了4种分类方法在10次实验中的分类准确率波动情况,整体看,其曲线由高到低排列大致是Transf_ROI、Transf_Corr、SAE_Corr以及SAE_ROI,与表3中的结果一致。并且在10次实验中,Transf_ROI方法在7次实验中都取得了最高的分类准确率,这也表明,不仅在平均值上Transf_ROI方法分类准确率较高,而且对于单次实验,Transf_ROI的方法的分类准确率也大概率高于其他方法。同时,在10次实验中,Transf_ROI和Transf_Corr的分类准确率分别稳定在70%~80%和60%~75%,而SAE_Corr和SAE_ROI的分类准确率在10次实验中的波动较大,可能是由于数据量不足。
|
Download:
|
| 图 7 Transf_ROI中第3层全连接层中提取的特征值 Fig. 7 Feature values extracted from the 3rd fully connected layer in Transf_ROI | |
|
Download:
|
| 图 8 10次实验中的分类准确率 Fig. 8 Classification accuracy in ten experiments | |
给出在不同数据输入及不同模型的条件下提取特征时间和分类时间,其中提取特征时间为分别从时间序列数据及相关系数数据中提取特征以供分类网络所占用的时间;分类时间是对全部的被试者,将其从时间序列数据及相关系数数据中提取到的特征,送入分类网络获得分类结果所用的时间。所有实验方法均在Inter(R) Core(TM) i5-6300HQ(4核)的中央处理单元(central processing unit, CPU)环境下运行,未采用任何图形处理器(graphics processing unit, GPU)。表4给出了在不同模型和不同数据输入时的提特征取和分类时间,从表中可以看出,与SAE方法相比,Transf方法在特征提取时所用的时间减少了近80%,且所用的分类时间也都有减少,网络总运行时间减少了约60%。这都表明在相同的环境下,使用迁移学习对EMCI进行分类所耗费的时间少于SAE的方法,其原因也在于迁移学习其实具有更少的权重数量。
| 表 4 模型性能指标 Tab.4 Model performance indicators |
本文主要分析Transf_ROI的顶层网络的全连接层数对分类性能的影响,其最终参数在表2列出。在这里,给出不同的顶层网络参数设置对最终分类结果带来的不同影响。图9分别给出了采用Transf_ROI中池化层所提取的NC组和EMCI组特征值的结果图,从图中可以看到,两个组别的大部分像素值均在50左右,当然,存在一部分像素值超过了150,但其所在两个组别的位置也没有存在明显差异性。该结果表明,仅靠池化层提取的特征还不能将两组进行区分,需要加入全连接层。

|
Download:
|
| 图 9 Transf_ROI中池化层中提取的特征值 Fig. 9 Feature values extracted from the pooling layer in Transf_ROI | |
全连接层的层数也会对实验结果产生影响,图10和图11分别给出了在1~4层全连接层时的分类准确率和运行时所用的迭代次数。从图10中可以看出,在使用3层全连接层时,分类准确率最高,且在10组实验中,有6次是最高值。如图7所示的第3层全连接层中提取的特征值的组间差异性明显大于如图9所示的池化层中提取的特征值的组间差异性,因此加入全连接层是十分有必要的。

|
Download:
|
| 图 10 TOP层具有1、2、3和4个全连接层时的测试精度 Fig. 10 Test accuracy when TOP layer has 1, 2, 3 and 4 fully connected layers | |
|
Download:
|
| 图 11 TOP层具有1,2,3和4个全连接层时的Epoch Fig. 11 Epoch when TOP layer has 1, 2, 3 and 4 fully connected layers | |
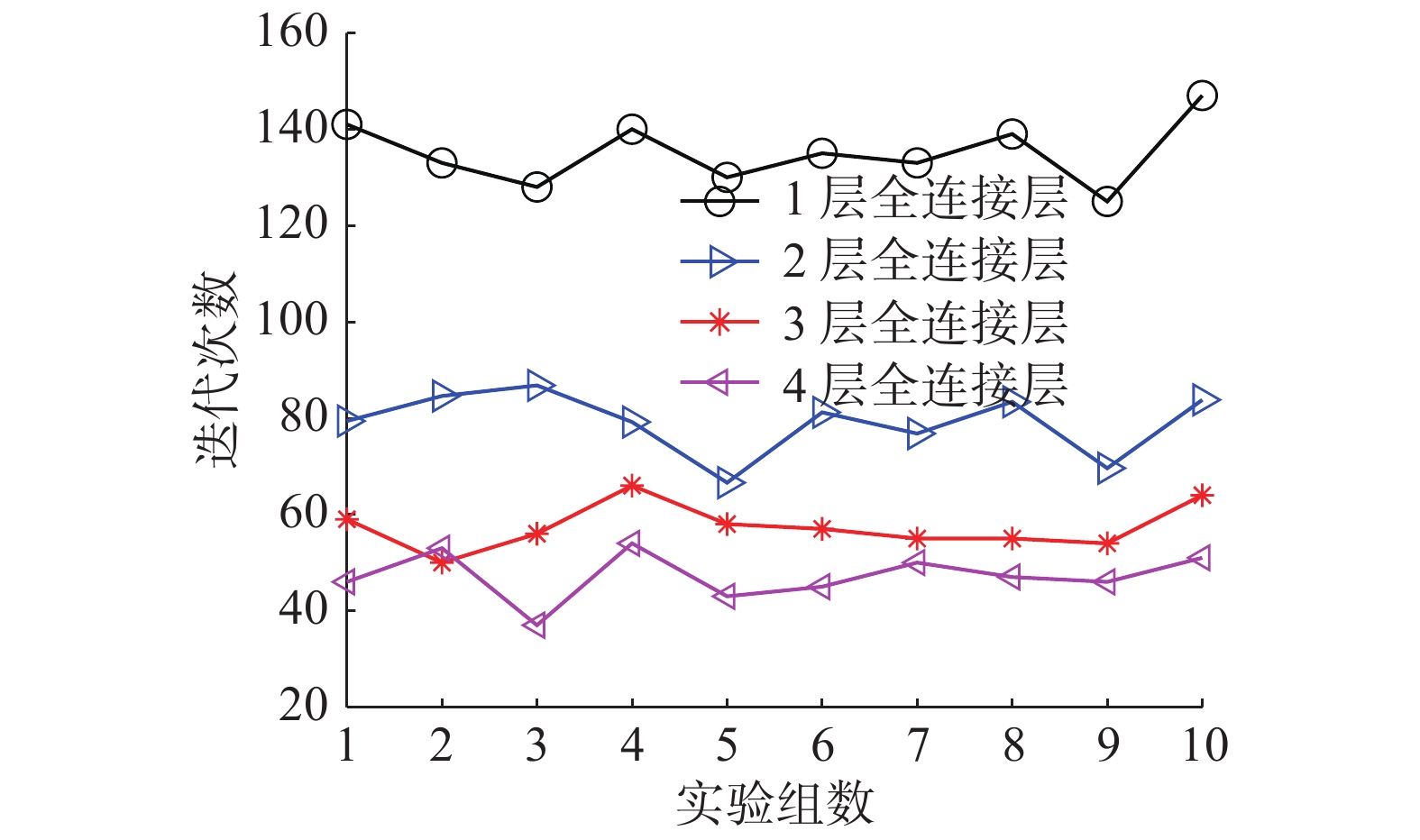
而在图11中可以看出,随着全连接层层数的增多,迭代次数在相应地减少,但减少的程度在降低。根据图10和图11综合考虑分类准确率和迭代次数后,我们的实验选用了3层全连接层的设置。
5 讨论针对rs-fMRI数据的EMCI分类问题,本文采用迁移学习MoblieNet网络对ROI时间序列进行特征提取,再进入到分类网络进行分类。实验结果表明,本文的方法较传统方法的分类准确率有所提升,且运行时间也有较大程度的减少,但还有以下几点需要进一步进行讨论。
实验中,本文方法的分类准确率为73.67%,虽然比其他几种传统方法高,但低于文献[22]给出的86%的分类结果,该文献采用SAE来提取Pearson相关系数的特征,其主要原因在于,所使用的数据集较小,被试者数为64,而文献[22]所用被试数为170。虽然本文所采用的ADNI-2数据库中,有关EMCI的rs-fMRI数据确实较多,但与其对应的MRI数据却相对较少。没有对应MRI数据将无法进行良好配准,这会导致一些混淆变量不能有效去除,如脑脊液和白质等,而该混淆变量又会影响分类结果。因此,最后能够满足实验条件的rs-fMRI数据量就较小。当然,实验中的所采用方法无论是迁移学习还是传统方法,面向的数据集是相同的,即本文方法的性能提升是在同样数据条件下所获得的。在未来的工作中,我们将会尝试更多的可用数据,以进一步来验证本文的研究。
另外,在顶层参数设置的实验中,仅给出了全连接层数的实验结果,而未对其他参数作进一步讨论,这主要是因为相对于其他参数,全连接层数对分类结果的影响比较大。对于激活函数,我们也尝试了一些常用的函数,如ReLu、Softplus等[30],但发现这些函数的分类结果并没有较大差别,因此选择了较为常见的Softplus。当然,还有一些参数对顶层性能也非常重要,例如全连接层节点数。节点数量可以根据经验确定,若节点数太小,网络将无法适应复杂分类,若节点数太大,会增加训练时间且可能产生过拟合。在本文的顶层网络中,将3个全连接层的节点数分别设置为200、100和50。最后还有一个重要的顶层参数是Dropout层的神经元丢弃率,将其值设为大于0时,会降低分类网络对训练数据的拟合程度,从而得到较低的分类准确率,因此最后将其设置为0。
6 结束语针对通过rs-fMRI信号来诊断EMCI问题,本文提出了一种采用MobileNet的迁移网络来提取ROI时间序列特征的方法。对于4D的rs-fMRI信号,采用迁移学习来提取特征值可以使网络的训练时间大幅度下降,并能减少瞬时信息的丢失。本文在实验中,采用了ADNI-2的rs-fMRI数据,并用DPABI工具箱对这些数据进行预处理,将本文算法与从相关系数提取特征值的方法进行了对比,结果显示本文算法的分类准确率比传统的相关系数方法提高了约10%,而网络的分类时间大约只有用SAE提取相关系数特征方法的25%。该结果说明,ImageNet数据库中预训练的迁移网络从ROI提取特征的分类准确率要高于从脑区网络的功能性连接提取特征的方法,并且这种方法还可减少特征提取的时间和分类网络运行时间。
| [1] |
BURNS A, ILIFFE S. Alzheimer’s disease[J]. British medical journal, 2009, 338(7692): 467-471. ( 0) 0)
|
| [2] |
Alzheimer’s Association. 2018 Alzheimer’s disease facts and figures[J]. Alzheimer’s & dementia, 2018, 14(3): 367-429. ( 0) 0)
|
| [3] |
DAVATZIKOS C. Baseline and longitudinal patterns of brain atrophy in MCI patients and their use in prediction of short-term conversion to Alzheimer’s disease: results from ADNI[J]. Alzheimer’s & dementia, 2009, 5(4S): P21-P22. ( 0) 0)
|
| [4] |
GRUNDMAN M, PETERSEN R C, FERRIS S H. Mild cognitive impairment can be distinguished from Alzheimer disease and normal aging for clinical trials[J]. Archives of neurology, 2004, 61(1): 59-66. DOI:10.1001/archneur.61.1.59 ( 0) 0)
|
| [5] |
BISCHKOPF J, BUSSE A, ANGERMEYER M C. Mild cognitive impairment-a review of prevalence, incidence and outcome according to current approaches[J]. Acta psychiatrica scandinavica, 2002, 106(6): 403-414. DOI:10.1034/j.1600-0447.2002.01417.x ( 0) 0)
|
| [6] |
SHARMA N, SINGH A N. Exploring biomarkers for Alzheimer's disease[J]. Journal of clinical and diagnostic research, 2016, 10(7): KE01-KE06. ( 0) 0)
|
| [7] |
MCKAHNN G. Clinical diagnosis of Alzheimer's disease: report of the NINCDS-ADRDA Work Group* under the auspices of department of health and human services task force on Alzheimer's disease[J]. Neurology, 1984, 34(7): 939. DOI:10.1212/WNL.34.7.939 ( 0) 0)
|
| [8] |
SCHROETER M L, STEIN T, MASLOWSKI N, et al. Neural correlates of Alzheimer’s disease and mild cognitive impairment: a systematic and quantitative meta-analysis involving 1351 patients[J]. NeuroImage, 2009, 47(4): 1196-1206. DOI:10.1016/j.neuroimage.2009.05.037 ( 0) 0)
|
| [9] |
HALLER S, BARTSCH A J. Pitfalls in fMRI[J]. European radiology, 2009, 19(11): 2689-2706. DOI:10.1007/s00330-009-1456-9 ( 0) 0)
|
| [10] |
BRIER M R, THOMAS J B, SNYDER A Z, et al. Loss of intranetwork and internetwork resting state functional connections with Alzheimer’s disease progression[J]. Journal of neuroscience, 2012, 32(26): 8890-8899. DOI:10.1523/JNEUROSCI.5698-11.2012 ( 0) 0)
|
| [11] |
STANLEY M L, MOUSSA M N, PAOLINI B M, et al. Defining nodes in complex brain networks[J]. Frontiers in computational neuroscience, 2013, 7: 169. ( 0) 0)
|
| [12] |
RUSSAKOVSKY O, DENG J, SU H, et al. ImageNet large scale visual recognition challenge[J]. International Journal of Computer Vision, 2015, 115(3): 211-252. DOI:10.1007/s11263-015-0816-y ( 0) 0)
|
| [13] |
BÄCKMAN L, JONES S, BERGER A K, et al. Multiple cognitive deficits during the transition to Alzheimer's disease[J]. Journal of internal medicine, 2004, 256(3): 195-204. DOI:10.1111/j.1365-2796.2004.01386.x ( 0) 0)
|
| [14] |
WEE C Y, YAP P T, DENNY K, et al. Resting-state multi-spectrum functional connectivity networks for identification of MCI patients[J]. PLoS One, 2012, 7(5): e37828. DOI:10.1371/journal.pone.0037828 ( 0) 0)
|
| [15] |
GEHRING J, MIAO Y J, METZE F, et al. Extracting deep bottleneck features using stacked auto-encoders[C]//Proceedings of 2013 IEEE International Conference on Acoustics, Speech and Signal Processing. Vancouver, BC, Canada, 2013: 3377−3381.
( 0) 0)
|
| [16] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks[J]. Communications of the ACM, 2017, 60(6): 84-90. DOI:10.1145/3065386 ( 0) 0)
|
| [17] |
HON M, KHAN N M. Towards Alzheimer's disease classification through transfer learning[C]//Proceedings of 2017 IEEE International Conference on Bioinformatics and Biomedicine. Kansas City, MO, USA, 2017: 1166−1169.
( 0) 0)
|
| [18] |
JOHNSON K A, FOX N C, SPERLING R A, et al. Brain Imaging in Alzheimer Disease[J]. Cold spring harbor perspectives in medicine, 2012, 2(4): a006213. ( 0) 0)
|
| [19] |
CUINGNET R, GERARDIN E, TESSIERAS J, et al. Automatic classification of patients with Alzheimer’s disease from structural MRI: a comparison of ten methods using the ADNI database[J]. Neuroimage, 2011, 56(2): 766-781. DOI:10.1016/j.neuroimage.2010.06.013 ( 0) 0)
|
| [20] |
BARKHOF F, HALLER S, ROMBOUTS S A R B. Resting-state functional MR imaging: a new window to the brain[J]. Radiology, 2014, 272(1): 29-49. DOI:10.1148/radiol.14132388 ( 0) 0)
|
| [21] |
MACHULDA M M, WARD H A, BOROWSKI B, et al. Comparison of memory fMRI response among Normal, MCI, and Alzheimer's patients[J]. Neurology, 2003, 61(4): 500-506. DOI:10.1212/01.WNL.0000079052.01016.78 ( 0) 0)
|
| [22] |
JU Ronghui, HU Chenhui, ZHOU Pan, et al. Early diagnosis of Alzheimer’s disease based on resting-state brain networks and deep learning[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2019, 16(1): 244-257. DOI:10.1109/TCBB.2017.2776910 ( 0) 0)
|
| [23] |
HJELM R D, CALHOUN V D, SALAKHUTDINOV R, et al. Restricted boltzmann machines for neuroimaging: an application in identifying intrinsic networks[J]. NeuroImage, 2014, 96: 245-260. DOI:10.1016/j.neuroimage.2014.03.048 ( 0) 0)
|
| [24] |
NIE Dong, LU Junfeng, ZHANG Han, et al. Multi-channel 3D deep feature learning for survival time prediction of brain tumor patients using multi-modal neuroimages[J]. Scientific reports, 2019, 9(1): 1103. DOI:10.1038/s41598-018-37387-9 ( 0) 0)
|
| [25] |
TZOURIO-MAZOYER N, LANDEAU B, PAPATHANASSIOU D, et al. Automated anatomical labeling of activations in SPM using a macroscopic anatomical parcellation of the MNI MRI single-subject brain[J]. Neuroimage, 2002, 15(1): 273-289. DOI:10.1006/nimg.2001.0978 ( 0) 0)
|
| [26] |
HOWARD A G, ZHU Menglong, CHEN Bo, et al. Mobilenets: efficient convolutional neural networks for mobile vision applications[J/OL]. Computer Vision and Pattern Recognition. (2017-04-17) [2020-07-24] https://arxiv.org/abs/1704.04861v1.
( 0) 0)
|
| [27] |
YAN Chaogan, ZANG Yufeng. DPARSF: a MATLAB toolbox for “pipeline” data analysis of resting-state fMRI[J]. Frontiers in Systems Neuroscience, 2010, 4: 13. ( 0) 0)
|
| [28] |
XIONG Hui, SHEKHAR S, TAN Pangning, et al. Exploiting a support-based upper bound of Pearson’s correlation coefficient for efficiently identifying strongly correlated pairs[C]//Proceedings of the 10th ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. Seattle, WA, USA, 2004: 334−343.
( 0) 0)
|
| [29] |
MOORE A W. Cross-validation for detecting and preventing overfitting[M]. Pittsburgh, USA: Carnegie Mellon University, 2001.
( 0) 0)
|
| [30] |
ZHENG Hao, YANG Zhanlei, LIU Wenju, et al. Improving deep neural networks using softplus units[C]//Proceedings of 2015 International Joint Conference on Neural Networks. Killarney, Ireland, 2015: 1−4.
( 0) 0)
|
 2021, Vol. 16
2021, Vol. 16


