前列腺癌在欧美等国家是最常见的泌尿系恶性肿瘤,在所有直接导致死亡原因的肿瘤中排在第2位,随着我国人口老龄化,前列腺癌的发生率也越来越高[1]。对前列腺癌进行影像引导放射治疗是诊治该疾病的关键步骤之一。
影像引导放射治疗前列腺,通常是对患者进行CT等影像检查后,由医生手动分割影像图像前列腺癌病变部分。但是手动分割病变需要耗费一定的时间与精力,且不同的医生依据自己的工作经验对病变分割的结果存在一定的差异性,在某种程度上会影响分割的精度,进而影响对病症的诊断。因此,准确地自动分割对前列腺癌的诊断是十分重要的。
CT检查具有快速、薄层、大范围扫描等优势,且检查时间较短,但是由于图像的低对比度,使得前列腺与周围组织结构显像并不清晰,给图像的自动分割造成了一定的困难。而MRI检查软组织分辨率高,图像对比度好,在显示肿瘤本身以及前列腺包膜、周围组织等方面较 CT 更有优势[2]。为了提高CT图像的分割精度,对CT图像进行更有效地自动分割,我们提出利用MRI的高分辨率特点去帮助CT图像分割前列腺癌的病变部分。
针对这一想法,本文提出一种新的基于深度学习的多模态U形网络图像分割模型MM-unet:1)运用深度学习迁移的思想,借用其他MRI数据集(即PROMISE12)辅助训练MRI图像的初始分割模型,进而将训练好的MRI图像初始分割模型进行迁移到CT图像的初始分割模型中;2)通过设计一种新型的多模态损失函数MM-Loss,衡量MRI与CT不同模态分割模型间的一致性,并建立它们之间的联系;联合训练基于MRI与CT 图像的MM-unet,使用监督学习的方式让两个模态互相学习,使CT图像能够借助MRI提供的影像信息来提高自身的分割精度。
1 相关工作医学图像不同于一般的图像,具有一定的复杂性、多样性、不规则性等特点,因此很多用于处理一般图像的分割方法不能直接用于医学图像处理。而CT图像由于软组织结构对比度低的特点,使得其分割难度加大。为解决这一问题,近年来有不少研究者提出若干CT前列腺图像分割方法,如文献[3]提出基于特征学习的框架实现精准定位的分割方法,文献[4]提出从之前分割好的训练图像中建立可变形的器官模型实现自动分割的方法,文献[5]提出基于稀疏表示的分类器分割的方法等。上述方法都是利用单模态图像信息对CT图像实现分割,而本文提出的多模态U形网络分割方法是充分利用多模态图像信息。
单模态,是使用一种成像设备得到的图像;而多模态图像是使用两种以上成像设备得到。不同模态的图像有着各自的成像特点,能为同一组织结构提供不尽相同的影像信息;而使用多模态图像能够结合不同模态的影像信息以帮助更全面的疾病诊断。如文献[6]提出的多模态堆叠深度多项式网络通过融合与学习多个模态的特征表示来诊断阿尔茨海默病;文献[7]提出的变换深度卷积网络通过融合不同模态的图像特征对椎体识别更加有效。这两种方法与本文所提方法均是基于深度学习的方法。
深度学习具有强大的特征学习与表达能力,其概念起源于2006年Hinton等在Neural Computation发表的文献[8],但真正引起学术界广泛关注的是2012年Krizhevsky等提出的深度卷积神经网络Alexnet模型[9]。在那之后,深度学习迅猛发展,且多用于语音识别、图像分类检测、目标追踪等领域。此外,深度学习方法在医学图像分割中的应用也是十分广泛,如文献[10]提出的基于三维卷积神经网络分割椎体MRI图像,文献[11]提出的基于递归神经网络分割肌束膜病理图像,文献[12]提出的基于多尺度特征融合的深度三维卷积编码网络分割多发性硬化症病变等。
由于深度学习的模型训练往往需要大量的数据储备,为提升其模型精度且防止过拟合问题,研究者们运用迁移学习,将预训练好的其他网络模型迁移到目标模型中,如文献[13]提出使用基础数据集训练深度卷积神经网络学习模型,使用目标数据集微调该模型中学习到的特征,分别通过选择性迁移与直接迁移来训练目标模型,以提高其模型的泛化能力;文献[14]提出的级联深度自适应(CDDA)模型通过使用医生手工标注的掩模在具有不同掩膜比的原始CT图像上叠加来构建多个连续源域,并将这些源域上学习到的特征迁移到目标域中。
2 卷积神经网络学习模型 2.1 网络结构常用层卷积层[15] (convolution layer,Conv),是卷积神经网络的核心层,通常使用卷积核与输入的特征图进行卷积操作以实现特征的提取;池化层(pooling layer),通常是对卷积层输出的特征图进行下采样操作以降低维度并保留有用的信息,有效地防止过拟合,常见的方法有最大池化、均值池化等;修正线性单元[16] (rectified linear unit, ReLU),是一种激活函数,用于加快网络训练的收敛速度;反卷积层[17] (deconvolution layer,Deconv),可以看作是卷积层的逆操作,用于图像分割中对输入特征图的重构。
2.2 单模态U形网络医学图像具有复杂多样化、线条不规律等特点,分割时对于图像中的组织结构等细节信息需要进行精准定位,才能有效地提高分割精度。而文献[18]提出的全卷积神经网络U-net模型有效地解决了这一问题,在医学图像分割中取得了好的表现。
U-net模型是由两个路径呈对称状组成,一个收缩路径用于获得图像信息,一个扩展路径用于实现精确定位。本文中,我们使用了U-net模型并作了一些调整,除最后一层卷积层外,每层卷积层后都增加了一层批处理归一化[19](batch normalization,BN),用来优化反向传播中的梯度问题,并将卷积层填充参数设为1,去掉原始的裁剪层,以确保输入、输出的图像大小保持一致,便于图像的处理。调整后的模型称为单模态U形网络。
本文训练的CT图像单模态U形网络结构如图1所示。我们将两层3×3大小的卷积层,每层后加上一层修正线性单元与批处理归一化(ReLU+BN)称为一个块结构(Block)。在左边的收缩路径中,输入的CT图像经过3次块结构与步长为2的2×2大小最大池化层(Pooling)的下采样操作,且每次下采样时都加倍通道数;之后经过一个块结构后进入扩展路径,再经过3次块结构与步长为2的2×2大小反卷积层(Deconv)的上采样操作,每次上采样时减半通道数:最后通过一层1×1大小的卷积层[20]映射到目标的类别,使用交叉熵损失函数计算输出的特征图与标记图像的像素值距离,得到输出图像。

|
Download:
|
| 图 1 训练CT图像的单模态U形网络 (CT-unet) Fig. 1 Singlemodal U-net for training CT images (CT-unet) | |
本节详细介绍所提出的CT图像分割方法,主要考虑3个方面:1)充分利用MRI模态的信息指导CT图像分割;2) 构造MRI与CT之间的中间模态图像,以缩小与CT模态图像的差异;3)设计多模态损失函数MM-Loss,衡量MRI与CT模态间的相关性,通过联合学习训练MM-unet模型。
3.1 单模态MRI模型指导CT图像分割MRI前列腺图像具有分辨率高、对比度好的特点,与CT图像相比,显像更为清晰。一般来说,使用同一标记图像的数据集训练时,MRI的分割结果会优于CT图像,但由于训练样本有限,MRI模型的分割精度会受影响。所以在利用MRI图像特点帮助CT图像分割时,应当尽可能地提高MRI模型的分割精度,有效地防止过拟合,才能指导CT图像更好地分割。因此,本文运用迁移学习的思想,首先充分利用其他前列腺图像的MRI数据集训练单模态U形网络,得到的参数模型去初始化训练与CT图像配准的MRI数据集,以期望获得分割精度较高的MRI模型,然后再将MRI模型参数作为初始模型参数,精化训练CT单模态U形网络(CT-unet),帮助提高CT图像的分割精度。
不同于CT图像分割的单模态U形网络,由于MRI图像展示的细节信息丰富且清晰,我们使用具有4次下采样、上采样操作的单模态U形网络模型(MRI-unet)来训练,以便在模型最底层能够更精确地捕获MRI的图像信息。
3.2 单模态CT-MRI模型指导CT图像分割MRI与CT两个模态图像因为成像原理不同,图像外观存在明显的差异性,可能会使模态之间的学习受到一些影响。为减小这种影响,需要减小MRI与CT图像外观的差异性。对此,本文将对应同一标记已经配准的MRI与CT图像都进行归一化处理,再对它们使用取均值操作构造出新的CT-MRI图像。从外观的表现来看,CT-MRI图像既有CT图像的特点,又有MRI图像的特点。
我们使用具有4次下采样、上采样操作的单模态U形网络模型训练CT-MRI图像 (CT-MRI-unet)。首先利用MRI模型参数作为初始模型,精化训练CT-MRI模型,进而将训练好的CT-MRI模型进行迁移训练CT-unet模型,帮助提高CT图像的分割精度。
3.3 基于多模态U形网络的CT图像分割由于不同的模态图像有各自的特点,提供的影像信息不完全相同,如果将多模态图像提供的信息结合起来,充分利用其互补性,则有利于提高单模态图像的分割精度。因此,本文提出基于多模态U形网络(MM-unet)的CT图像分割方法,如图2所示,由MRI-unet与CT-unet构成的,使用了一个MM-Loss损失函数用以建立MRI与CT两个单模态U形模型之间的联系。

|
Download:
|
| 图 2 MM-unet模型 Fig. 2 Multimodal U-unet model | |
两个单模态U形网络同时训练各自的模态图像,通过收缩路径获取底层图像信息,扩展路径实现高层信息的提取,在最后一层卷积层使用MM-Loss损失函数对MRI与CT图像进行相似性度量。MM-Loss损失函数的定义如下:
| $L = \frac{1}{\rm{2NCHW}}\sum\limits_{i = 1}^N y ||{{{a}}_i} - {{{b}}_i}||_2^2 + \sum\limits_{i = 1}^N {(1 - y)} ||{{{a}}_i} + {{{b}}_i}||_2^2$ | (1) |
式中:ai、bi为输入的MRI与CT图像对应的分割结果概率图,N、C、H、W为输入图像的数量、通道、高度、宽度,y为标记图像。
输入MRI与CT图像,对于它们共同对应的标记图像的分割部分,约束它们相似的部分尽可能接近;对标记图像的背景部分,促使它们成为分割目标的概率值尽可能地小。以此使两个模态图像的分割模型能够对于分割目标进行互相学习。
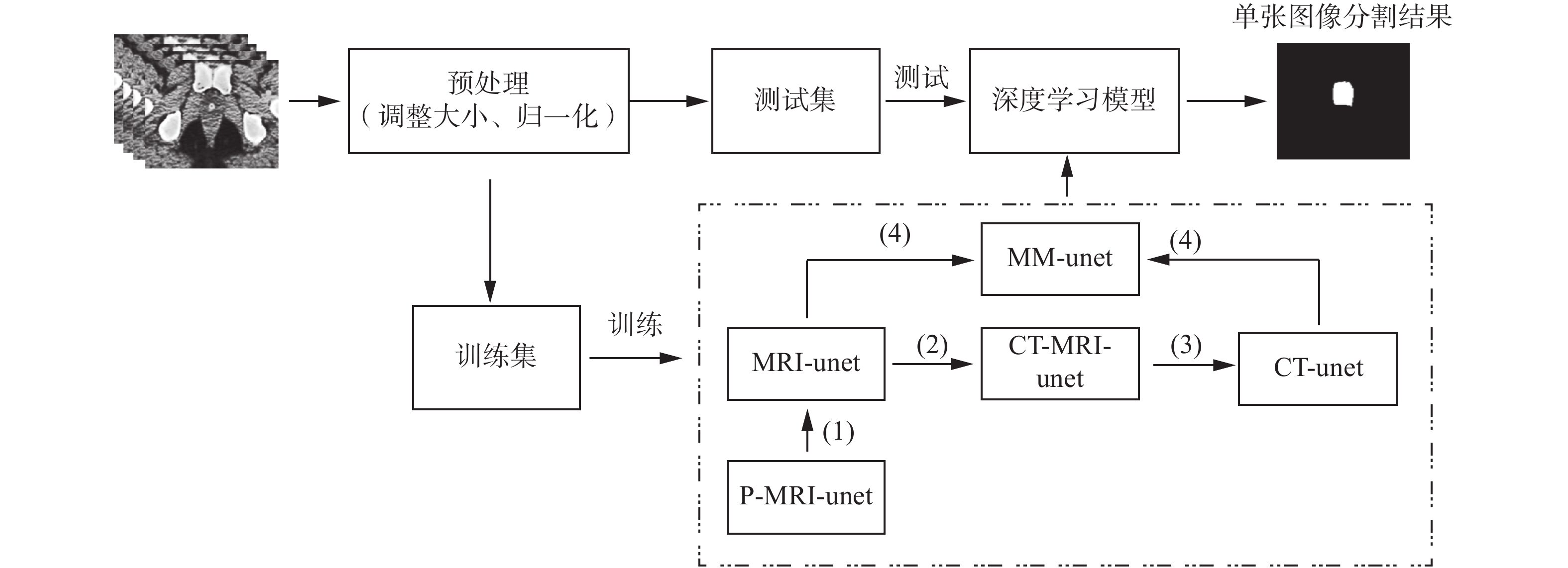
3.4 算法流程本文方法的整个流程如图3所示。数据集经过预处理工作后分为训练集与测试集,在训练阶段,使用训练集训练分割模型,算法如图3虚线框部分,其操作步骤如下:
|
Download:
|
| 图 3 基于深度学习模型的前列腺CT图像分割流程图 Fig. 3 The flowchart of deep learning model for prostate segmentation in CT images | |
1)使用PROMISE12数据集[21]预训练得到的P-MRI-unet模型(详见4.5节实验1),进行迁移训练 MRI-unet模型;
2)构造CT-MRI图像,将1)中得到的MRI-unet模型进行迁移训练CT-MRI-unet模型;
3)将2)中得到的CT-MRI-unet模型进行迁移训练CT- unet模型;
4)将1)中得到的MRI-unet模型与3)中得到的CT-unet模型进行迁移训练MM-unet模型。
在测试阶段,使用训练阶段最终得到的MM-unet模型对测试集里的CT图像进行分割,得到CT图像的分割结果图。
4 实验 4.1 数据集我们使用的前列腺数据集有两个,一个是网上公开的PROMISE12数据集[21],一个是某合作医院提供的Prostate数据集。PROMISE12数据集含有50个患者的MRI图像与相对应的手工标记图像,图像大小不一。删去无病变的MRI图像与标记图像,并将其余的癌病变图像大小统一调整到256×256,共得到778张MRI图像与778张标记图;使用前40个患者的样本作为训练集,后10个患者的样本作为验证集训练模型。Prostate数据集含有22个患者的CT、MRI与相对应的病变标记图像,已经过配准处理,大小为153×193。为便于训练,我们将图像大小统一调整到256×256,共得到658张CT、658张MRI与658张标记图;采用二折交叉验证的方式,第一折使用前11个患者的样本作为训练集,后11个患者的样本作为测试集进行测试;第二折使用后11个患者的样本作为训练集,前11个患者的样本作为测试集进行测试。
4.2 预处理工作为加快模型的训练速度,优化训练过程,我们对数据集进行了归一化处理。归一化操作定义为
| $I = \frac{{X - \mu }}{\sigma }$ | (2) |
式中:X为图像像素,μ为图像像素值的均值,σ为图像像素值的标准差。
4.3 实验设置本文是在深度学习框架Caffe[22]上进行的实验。在模型的训练阶段批处理参数设置为2,测试阶段的批处理设置为1,使用随机梯度下降[23]求解器与0.9的动量参数,固定策略的学习率,大小为10-6,权重衰减为0.005。
4.4 评价标准我们使用常用的重合率(Dice)、查准率(Precision)、查全率(Recall)指标[24]来度量自动分割图像与手工标记图像之间的差异,以用于评价实验结果。
定义公式如下:
| ${\rm{Dice}} = \frac{{2 \times |{\rm{AS}} \cap {\rm{MS}}|}}{{|{\rm{AS}}| + |{\rm{MS}}|}} \times 100\text{%} $ | (3) |
| ${\rm{Precision}} = \frac{{|{\rm{AS}} \cap {\rm{MS}}|}}{{|{\rm{AS}}|}} \times 100\text{%} $ | (4) |
| ${\rm{Recall}} = \frac{{|{\rm{AS}} \cap {\rm{MS}}|}}{{|{\rm{AS}}|}} \times 100\text{%} $ | (5) |
式中:AS为自动分割图像,MS为医生手工标记图像。
4.5 实验与结果本节通过3个实验来验证我们的方法的有效性。
实验1 为评估MRI模型指导CT图像分割的精度,我们使用PROMISE12数据集训练MRI-unet,获得的模型作为初始模型用于训练Prostate数据集里的MRI图像,训练后得到MRI参数模型。将MRI参数模型进行迁移学习得到CT-unet模型,用于对CT测试图像进行分割,该方法记作MRI→CT-unet;
实验2 为评估CT-MRI模型指导CT图像分割的精度,我们使用Prostate数据集里的MRI与CT图像构造CT-MRI图像,使用实验1里的MRI参数模型作为初始模型训练CT-MRI图像,得到的CT-MRI模型再迁移到用于CT图像分割的CT-unet中,用于对CT测试图像进行分割,该方法记作CT-MRI→CT-unet;
实验3 为评估并验证文中提出的MM-unet模型的有效性,我们使用实验1的MRI参数模型、实验2中通过CT-MRI参数模型迁移学习后的CT参数模型,分别作为MM-unet模型的MRI部分与CT部分的初始模型进行训练,得到的多模态参数模型用于对CT测试图像进行分割,该方法记作MM-unet。
3个实验的CT图像分割结果如表1所示。从表格中可看出,第2个方法因为构造的CT-MRI图像减少了MRI与CT图像的外观差异,使其指导CT图像分割的重合率、查准率、查全率均高于第一个方法;相比前两个方法,第3个方法的重合率、查全率最高。
| 表 1 CT图像分割实验结果 Tab.1 Experimental results of CT segmentation |
使用统计性t检验对MM-unet模型方法与其他方法的重合率Dice进行比较,P值小于0.05,说明MM-unet显著地优于其他方法。
4.6 比较实验本节将我们的方法(即上述实验中表现最好的MM-unet方法)与U-net模型方法[18]、CDDA模型[14]方法、无初参的MM-unet模型方法作比较,评价分割结果如表2所示。
| 表 2 不同模型的实验结果对比 Tab.2 Comparisons of experimental results on different models % |
为进一步展示所提方法的分割优势,部分图像可视化结果如图4所示,红色方框为医生手工标注的前列腺癌病变部分。第1排图像绿色方框为U-net模型分割的病变部分,第2排图像蓝色方框为迁移学习的MM-unet模型分割病变的部分。

|
Download:
|
| 图 4 两个模型分割CT图像的可视化结果 Fig. 4 Visual results of two models for CT segmentation | |
通过表2与图4显示,与U-net模型相比,我们的MM-unet模型方法分割效果有明显优势,重合率、查准率、查全率分别提升了3.31%、0.48%、5.61%,分割边界更接近于由医生标注的真实标记。
5 结束语本文中,我们提出了基于多模态U形网络实现CT图像前列腺癌分割的方法。该方法利用了MRI图像高分辨率与多模态图像信息互补的特点,构建基于MRI与CT图像的多模态深度学习模型指导CT图像学习前列腺癌分割。实验结果表明,我们的方法可以有效地提高CT图像分割精度。本文提出的多模态U形模型分割CT图像的方法适用于CT/MRI已进行配准匹配的前列腺癌数据集,对于未进行配准的数据集和其他非前列腺癌的数据集有待进一步验证。
| [1] |
黄高贤. 前列腺癌中CT、MR诊断的研究进展[J]. 吉林医学, 2010, 31(4): 529-531. HUANG Gaoxian. Progress in diagnosis of CT and MR in prostate cancer[J]. Jilin medical journal, 2010, 31(4): 529-531. DOI:10.3969/j.issn.1004-0412.2010.04.067 (  0) 0)
|
| [2] |
陈识, 宋曼. MRI与CT诊断不同病理分期前列腺癌患者的准确率比较观察[J]. 中国老年保健医学, 2016, 14(6): 85-87. CHEN Shi, SONG Man. MRI and CT diagnosis of different pathological stages of prostate cancer patients' accuracy rate comparison[J]. Chinese journal of geriatric care, 2016, 14(6): 85-87. DOI:10.3969/j.issn.1672-4860.2016.06.039 (  0) 0)
|
| [3] |
LIAO Shu, SHEN Dinggang. A feature-based learning framework for accurate prostate localization in CT images[J]. IEEE transactions on image processing, 2012, 21(8): 3546-3559. DOI:10.1109/TIP.2012.2194296 ( 0) 0)
|
| [4] |
CHEN Siqi, LOVELOCK D M, RADKE R J. Segmenting the prostate and rectum in CT imagery using anatomical constraints[J]. Medical image analysis, 2011, 15(1): 1-11. ( 0) 0)
|
| [5] |
GAO Yaozong, LIAO Shu, SHEN Dinggang. Prostate segmentation by sparse representation based classification[J]. Medical physics, 2012, 39(10): 6372-6387. DOI:10.1118/1.4754304 ( 0) 0)
|
| [6] |
SHI Jun, ZHENG Xiao, LI Yan, et al. Multimodal neuroimaging feature learning with multimodal stacked deep polynomial networks for diagnosis of Alzheimer's disease[J]. IEEE journal of biomedical and health informatics, 2018, 22(1): 173-183. DOI:10.1109/JBHI.2017.2655720 ( 0) 0)
|
| [7] |
CAI Yunliang, LANDIS M, LAIDLEY D T, et al. Multi-modal vertebrae recognition using transformed deep convolution network[J]. Computerized medical imaging and graphics, 2016, 51: 11-19. DOI:10.1016/j.compmedimag.2016.02.002 ( 0) 0)
|
| [8] |
HINTON G E, OSINDERO S, THE Y W. A fast learning algorithm for deep belief nets[J]. Neural computation, 2006, 18(7): 1527-1554. DOI:10.1162/neco.2006.18.7.1527 ( 0) 0)
|
| [9] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks[C]//Proceedings of the 25th International Conference on Neural Information Processing Systems. Lake Tahoe, Nevada, 2012: 1097–1105.
( 0) 0)
|
| [10] |
KOREZ R, LIKAR B, PERNUŠ F, et al. Model-based segmentation of vertebral bodies from MR images with 3D CNNs[C]//Proceedings of the 19th International Conference on Medical Image Computing and Computer-Assisted Intervention. Athens, Greece, 2016: 433–441.
( 0) 0)
|
| [11] |
XIE Yuanpu, ZHANG Zizhao, SAPKOTA M, et al. Spatial clockwork recurrent neural network for muscle perimysium segmentation[C]//Proceedings of the 19th International Conference on Medical Image Computing and Computer-assisted Intervention. Athens, Greece, 2016: 185–193.
( 0) 0)
|
| [12] |
BROSCH T, TANG L Y W, YOO Y, et al. Deep 3D convolutional encoder networks with shortcuts for multiscale feature integration applied to multiple sclerosis lesion segmentation[J]. IEEE transactions on medical imaging, 2016, 35(5): 1229-1239. DOI:10.1109/TMI.2016.2528821 ( 0) 0)
|
| [13] |
YOSINSKI J, CLUNE J, BENGIO Y, et al. How transferable are features in deep neural networks?[J]. arXiv:1411.1792, 2014. ( 0) 0)
|
| [14] |
SHI Yinghuan, YANG Wanqi, GAO Yang, et al. Does manual delineation only provide the side information in CT prostate segmentation?[C]//Proceedings of the 20th International Conference on Medical Image Computing and Computer-Assisted Intervention. Quebec City, Canada, 2017: 692–700.
( 0) 0)
|
| [15] |
KOUSHIK J. Understanding convolutional neural networks[J]. arXiv: 1605.09081, 2016. ( 0) 0)
|
| [16] |
XU Bing, WANG Naiyan, CHEN Tianqi, et al. Empirical evaluation of rectified activations in convolutional network[J]. arXiv: 1505:00853, 2015. ( 0) 0)
|
| [17] |
ZEILER M D, TAYLOR G W, FERGUS R. Adaptive deconvolutional networks for mid and high level feature learning[C]//Proceedings of the 2011 International Conference on Computer Vision. Barcelona, Spain, 2011: 2018–2025.
( 0) 0)
|
| [18] |
RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation[C]//Proceedings of the 18th International Conference on Medical Image Computing and Computer-Assisted Intervention. Munich, Germany, 2015: 234–241.
( 0) 0)
|
| [19] |
IOFFE S, SZEGEDY C. Batch normalization: accelerating deep network training by reducing internal covariate shift[J]. arXiv: 1502.03167, 2015. ( 0) 0)
|
| [20] |
LIN Min, CHEN Qiang, YAN Shuicheng. Network in network[J]. arXiv: 1312.4400, 2013. ( 0) 0)
|
| [21] |
FRANCE N. MICCAI grand challenge: prostate MR image segmentation 2012[EB/OL]. [2012-10-01]. https://promise12.grand-challenge.org/home/.
( 0) 0)
|
| [22] |
JIA Yangqing, SHELHAMER E, DONAHUE J, et al. Caffe: convolutional architecture for fast feature embedding[C]//Proceedings of the 22nd ACM International Conference on Multimedia. Florida, USA, 2014: 675–678.
( 0) 0)
|
| [23] |
KETKAR N. Stochastic gradient descent[M]//KETKAR N. Deep Learning with Python. Berkeley: Apress, 2017.
( 0) 0)
|
| [24] |
TAHA A A, HANBURY A. Metrics for evaluating 3D medical image segmentation: analysis, selection, and tool[J]. BMC medical imaging, 2015, 15: 29. DOI:10.1186/s12880-015-0068-x ( 0) 0)
|
 2018, Vol. 13
2018, Vol. 13


