扩展功能
文章信息
- 刘世梁, 林玉成
- LIU Shiliang, LIN Yucheng
- 云南洞密蛛的种群遗传结构初探
- A Preliminary Study on Population Genetic Structure of Trogloneta yunnanense
- 四川动物, 2022, 41(3): 256-263
- Sichuan Journal of Zoology, 2022, 41(3): 256-263
- 10.11984/j.issn.1000-7083.20220024
-
文章历史
- 收稿日期: 2022-01-23
- 接受日期: 2022-03-31
2. 四川大学生命科学学院,四川省濒危野生动物保护生物学重点实验室,成都 610065
2. Sichuan Key Laboratory of Conservation Biology on Endangered Wildlife, College of Life Sciences, Sichuan University, Chengdu 610065, China
洞穴是一个相对封闭的特殊生态系统,几乎没有阳光、没有植物生长,有较为恒定且通常低于地表的温度和有限的食物供应(Culver et al., 2009)。由于受洞穴极端环境的影响,生活在其中的专性洞穴物种结构相对简单,并在时间和空间上相互隔离(Poulson & White,1969)。因此,在研究岛屿模型的进化过程、物种形成以及生物地理学等方面,洞穴被认为是天然的实验室(Cooper et al., 2007;Page et al., 2008)。中国西南山地有世界上面积最大的喀斯特地区,是全球34个生物多样性热点地区之一(Luo et al., 2016)。云贵高原是中国南方喀斯特地区的核心地带,地下有数千个洞穴,但洞穴中的主要生物群——陆生节肢动物却很少被研究(Zhang & Li,2014)。其中洞穴蜘蛛在洞穴生态系统中担任顶级捕食者的角色(Mammola et al., 2016),并产生了多种对洞穴环境的功能适应性(Cardoso et al., 2011),被认为是一个用来了解动物在极端环境下适应进化及过程的理想模型。对云贵高原的洞穴类球蛛的生物地理学研究发现,类球蛛科Nesticidae Simon, 1894小类球蛛属Nesticella Lehtinen & Saaristo, 1980起源于始新世,中新世时期的气候变化迫使地表蜘蛛种群迁移到洞穴中,并随着时间的推移定居下来(Ballarin & Li,2018)。许多地区正面临着气候变化、旅游开发和地下水污染等威胁,而分布在热带洞穴和大多数岩溶地区内的洞穴动物群几乎未被研究过,迫切需要进行密集和广泛的生物调查(Howarth,1983;Shu et al., 2013)。
在动物种群遗传研究方面,由于母系遗传、在不同物种间的同源性、受到强烈的选择压力而快速进化等特点,线粒体基因是适合用来探究遗传多样性的工具(Avise,2009),其中,线粒体细胞色素c氧化酶亚基Ⅰ(CO Ⅰ)基因已被成熟应用于蜘蛛的分子系统学研究(刘名信等,2012)。
云南洞密蛛Trogloneta yunnanense隶属蛛形纲Arachnida蜘蛛目Araneae密蛛科Mysmenidae,是一种个体微小的洞穴蜘蛛,最早发现于云南省个旧市郊的飞霞洞,宋大祥和朱明生(1994)将其命名为云南困蛛P. yunnanense,放入球蛛科Theridiidae。Lin和Li(2008)根据云贵高原多个洞穴采获的样本,将其描述为齿勺洞密蛛T. denticocleari并置于密蛛科。此后,二者被认为是同物异名(Li & Lin,2019),根据国际动物命名法规的优先律原则,该种有效学名应为T. yunnanense。
在地下相对稳定的极端环境条件下,洞穴隔离怎样维持洞穴蜘蛛的种群稳定,对洞穴物种的遗传多样性有何影响等问题的研究较少,尚未得到合理的解释。云南洞密蛛仅在云贵高原的8个洞穴中分布(宋大祥,朱明生,1994;Lin & Li,2008;Li & Lin,2019),作为相互独立的地理种群,其种群结构、遗传多样性及演化历史等方面的研究未见报道。本研究选取6个相互隔离的云南洞密蛛地理种群[关牛洞(GN)、野佬大洞(YLD)、清虚洞(QX)、十里大洞(SLD)、仙人洞(XR)、白岩洞(BY)],通过分析、比较不同种群的遗传多样性和种群分化,以期探讨洞穴隔离对云南洞密蛛种群遗传结构的影响。
1 材料与方法 1.1 研究材料2020年8月在云贵高原的6个洞穴中采获503号样本(表 1)。所有样本均用95%的分析纯乙醇浸泡,-20 ℃冻存。标本保存于四川大学自然博物馆(NHMSU)动物标本室。
| 洞名 Cave |
行政位置 Locations |
洞口坐标 Coordinates | 海拔 Elevation/m |
样本数 Sample size |
|
| 东经 Eastern longitude/° | 北纬 Northern latitude/° | ||||
| 关牛洞(GN) | 贵州省遵义市深溪镇龙江村 | 106.969 101 | 27.613 715 | 848 | 101 |
| 野佬大洞(YLD) | 贵州省大方县文阁乡三合村 | 105.465 684 | 27.184 263 | 1 429 | 85 |
| 清虚洞(QX) | 贵州省大方县羊场镇陇东村 | 105.669 876 | 27.103 043 | 1 476 | 22 |
| 十里大洞(SLD) | 贵州省盘县珠东乡十里坪村 | 104.756 632 | 25.623 685 | 1 658 | 103 |
| 仙人洞(XR) | 云南省武定县猫街镇大麦地 | 102.172 899 | 25.464 836 | 2 097 | 30 |
| 白岩洞(BY) | 云南省宜良县九乡大哲宗村 | 103.401 000 | 25.151 000 | 1 875 | 162 |
每个洞穴种群选取20~30只个体作为分子材料,用于开展种群遗传研究。每个采集位点随机选取包含雌雄和幼体在内的个体样本进行实验,为避免腹部肠道内容物带来的外源基因影响,仅取每只个体的头胸甲用于分子实验。使用QIAGEN血液和组织提取试剂盒(Qiagen;P/N:69506)提取基因组DNA。雄性的触肢器和雌、雄腹部均保留作为分子凭证。CO Ⅰ基因的扩增和测序引物为LCOI490(5'-GGTCAACAAATCATAAAGATATTGG-3')和HCO2198(5'-TAAACTTCAGGGTGACCAAAAAA TCA-3')。PCR反应体系25 μL:模板4 μL,引物各0.5 μL(10 mmol·L-1),ddH2O 7.5 μL,2×M5 HiPer plus Taq HiFi PCR mix(with blue dye)12.5 μL。扩增条件:95 ℃ 30 min;94 ℃ 30 s, 48.95 ℃ 30 s, 72 ℃ 30 s, 35个循环;72 ℃ 6 min。PCR产物通过1%琼脂糖凝胶电泳检测,送北京擎科生物科技有限公司成都分公司纯化和测序。
1.3 序列分析测序数据使用Bioedit 7.2.5(Hall,1999)进行检查和编辑。MEGA X(Kumar et al., 2018)用于翻译和对齐CO Ⅰ序列,并基于Kimura双参数距离模型计算碱基组成、转换与颠换值。基于P-distance计算种群间的遗传距离,并构建邻接(NJ)树。利用DNAsp 6.0(Rozas et al., 2017)计算平均核苷酸差异数(k)、单倍型数量(h)、单倍型多样性(Hd)和核苷酸多样性(π),并进行中性检验和核苷酸错配分布检验。使用PopArt 1.7(Leigh & Bryant,2015)构建Templeton-Crandall-Sing(TCS)网络(Clement et al., 2000)。利用Arlequin 3.5(Excoffier et al., 2005)进行分子方差分析,并计算种群遗传分化指数(FST)和种群间基因流(Nm)。
利用贝叶斯推断(BI)和最大似然法(ML)重建系统发育树。基于AIC准则使用PartitionFinder2(Lanfear et al., 2017)选择最适合的核苷酸替换模型为GTR+G,并使用MrBayes(Ronquist et al., 2012)构建BI系统发育树,4个马尔可夫链蒙特卡罗(MCMCs)采用默认参数运行200万代,抽样频率为200代一次,独立运行2次。基于AIC准则使用ModelFinder(Kalyaanamoorthy et al., 2017)选择的最适合核苷酸替换模型为TIM3+F+I+G4,使用IQTREE 1.6.12(Nguyen et al., 2015)构建ML系统发育树,节点支持值使用1 000次自展分析重复检测。
2 结果与分析 2.1 CO Ⅰ基因序列变异分析共获得159个样本部分CO Ⅰ基因,序列长度为630 bp,变异位点32个,包括2个单变异位点和30个简约信息位点。转换/颠换比为20.09,碱基组成为:T(44.00%)、A(26.16%)、C(11.89%)、G(17.95%)。A+T含量高于C+G含量。159个样本共包含16个单倍型(GenBank登录号分别为MZ577427、MZ577392、MZ577393、MZ577418、MZ577440、MZ577446、MZ577404、MZ577405、MZ577410、MZ577379、MZ577417、MZ577425、OM920931、OM920932、OM920935、OM920975)。
2.2 种群遗传多样性和遗传结构6个地理种群间未发现共享单倍型,种群内单倍型之间的差异不超过4个核苷酸。QX遗传多样性最高(k=1.305,Hd=0.553,π=0.002 07),其次为BY(k=1.073,Hd=0.607,π=0.001 70)。而遗传多样性最低的是GN,仅有1个单倍型(表 2)。整体而言,云南洞密蛛总体的k为10.021,Hd为0.888,π为0.015 91。
| 种群 Population |
样本数 Samples |
单倍型数 Haplotypes |
平均核苷酸差异数 Mean of nucleotide differences (k) |
单倍型多样性 Haplotype diversity (Hd) |
核苷酸多样性 Nucleotide polymorphism (π) |
| XR | 26 | 2 | 0.271 | 0.271 | 0.000 43 |
| QX | 20 | 4 | 1.305 | 0.553 | 0.002 07 |
| SLD | 29 | 3 | 0.315 | 0.305 | 0.000 50 |
| YLD | 28 | 3 | 0.376 | 0.362 | 0.000 60 |
| GN | 30 | 1 | 0 | 0 | 0 |
| BY | 26 | 3 | 1.073 | 0.607 | 0.001 70 |
以单倍型构建的NJ树与TCS单倍型网络结构相符,6个地理种群可分为2个大的谱系,其中YLD、GN与XR聚为一支;SLD、BY与QX聚为一支(图 1),2大谱系下的次级分支显示各个地理种群均为独立的谱系,它们之间不存在共享单倍型。
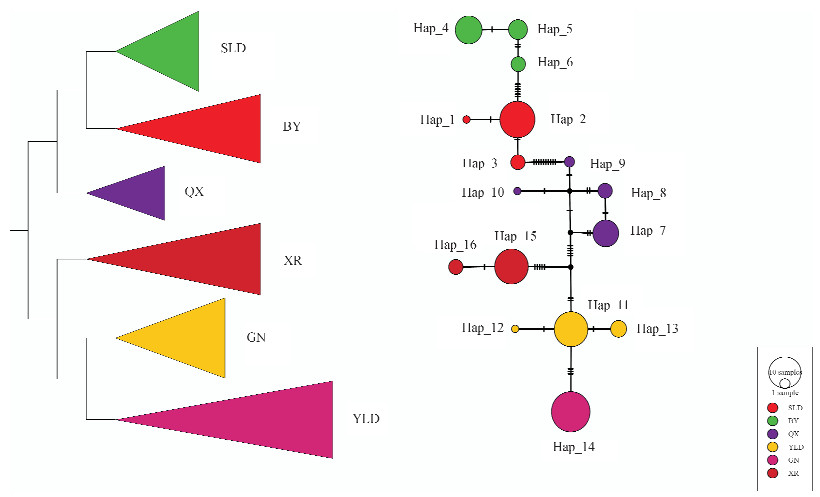
|
| 图 1 基于种群遗传距离构建的NJ树(左)和单倍型构建的TCS网络(右) Fig. 1 NJ tree (left) based on the population genetic distance and TCS network (right) based on haplotypes |
| |
6个地理种群中,YLD和GN之间的遗传距离最近(P=0.005 10),最远的是BY和GN(P=0.026 29),其他种群间的遗传距离在0.011 70~0.025 48之间(表 3)。6个种群间的FST值均在0.9以上, 存在极显著差异(P < 0.01)(表 4)。AMOVA分析显示,种群间变异占总变异的95.75%,种群内变异占总变异的4.25%,并且统计检验极显著(P < 0.001;表 5)。根据Hudson等(1992)的基因多样性算法,6个种群间的遗传分化指数为0.953 4,说明总变异的95.34%来自不同种群间的差异。
| 种群 Populations |
SLD | BY | QX | YLD | GN |
| BY | 0.013 35 | — | — | — | — |
| QX | 0.020 47 | 0.021 84 | — | — | — |
| YLD | 0.020 81 | 0.021 86 | 0.014 94 | — | — |
| GN | 0.025 23 | 0.026 29 | 0.018 25 | 0.005 10 | — |
| XR | 0.025 48 | 0.023 36 | 0.019 61 | 0.011 70 | 0.016 12 |
| 种群 Populations |
SLD | BY | QX | YLD | GN |
| BY | 0.920 7** | ||||
| QX | 0.944 2** | 0.914 5** | |||
| YLD | 0.973 7** | 0.948 8** | 0.918 7** | ||
| GN | 0.990 1** | 0.970 0** | 0.953 7** | 0.942 6** | |
| XR | 0.981 7** | 0.954 9** | 0.941 6** | 0.955 9** | 0.987 4** |
| 注 Note: ** P < 0.01 | |||||
| 变异来源 Source of variation |
df | 方差总数 Total variance |
方差分量 Variance components |
变异百分比 Percent of variation/% |
P |
| 种群间 | 5 | 743.455 | 5.689 10 | 95.75 | < 0.001 |
| 种群内 | 153 | 38.150 | 0.252 65 | 4.25 | < 0.001 |
| 总计 | 158 | 781.605 | 5.941 75 | 100 |
使用BI法和ML法构建的系统发育拓扑结构基本相同。除YLD外(PP=0.45,BP=43),其他种群的支持率都较高(PP>0.6,BP>50)。云南洞密蛛的6个种群分化为6个相互独立的地理谱系,第1支为BY,第2支为SLD,第3支为QX,第4支为XR,第5支为YLD,第6支为GN(图 2)。

|
| 图 2 基于6个云南洞密蛛种群CO Ⅰ基因构建的贝叶斯树 Fig. 2 Bayes inference tree based on CO Ⅰ genes from 6 Trogloneta yunnanense populations 节点旁的数字代表后验概率(PP)和自展支持度(BP) Numbers beside nodes correspond to Bayesian posterior probability (PP) and Maximum Likelihood bootstrap support values (BP) |
| |
使用中性检验和核苷酸错配分布检验并推测云南洞密蛛的种群历史动态。中性检验结果显示Tajima'D值为2.23(P=0.941),Fu's FS值为7.512(P=0.995)。核苷酸错配分布图显示(图 3),观测值呈多峰分布,与期望值不符。
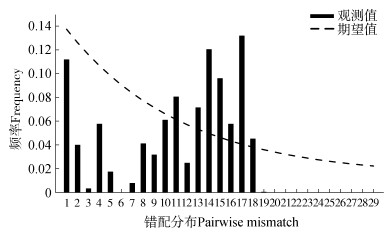
|
| 图 3 云南洞密蛛的错配分布图 Fig. 3 Mismatch distribution test of Trogloneta yunnanense |
| |
迄今为止,云南洞密蛛的分布记录仅来自云贵高原的8个地下洞穴(宋大祥,朱明生,1994;Lin & Li,2008;Li & Lin,2019),目前未发现在地表有分布。
3.1 云南洞密蛛的遗传多样性与种群分化云南洞密蛛的遗传多样性总体上较高,但各种群内的遗传多样性较低。6个种群间高遗传分化指数、不存在共享单倍型和低基因流表明,云南洞密蛛在洞穴之间没有发生迁移,6个种群间的Nm值总体为0.01,几乎不存在基因交流,形成各自独立演化的“岛屿”种群。这与Snowman等(2010)对洞穴类球蛛遗传结构的研究结果相似。种群间高度分化、种群内序列相似性较高的模式,与破碎生境下的种群模型预期结果一致(Hedin,1997)。受困于洞穴中的种群个体无法与外界交流,导致各洞穴种群进一步的遗传分化(Snowman et al., 2010)。由于封闭的洞穴环境的特殊性和相对较小的种群规模,洞穴动物群可能形成极端的遗传结构(Hedin,1997)。
系统发育分析表明,云南洞密蛛不同地理种群的单系性支持良好,6个种群遗传分化明显。在地理上相邻的地区观察到高度分化的谱系,这表明“岛屿”化的溶洞限制了种群间个体的迁移。从云贵高原整个地理区域来看,该物种的6个谱系并未表现出明显的地理分布格局。云南洞密蛛种群的地理分布可能与洞穴隔离有关,而与地表的地理距离无关。另外,本研究仅使用CO Ⅰ基因作为遗传标记,如果使用更多的基因序列,可能会得到更理想的结果。
3.2 云南洞密蛛的种群历史动态本研究中使用Tajima' D、Fu's FS 2种中性检验方法和核苷酸错配分布检验,推测了云南洞密蛛种群历史动态。2种中性检验结果均为正值,且P值不显著,这与核苷酸错配分布检验结果相符,表明云南洞密蛛自进入洞穴拓殖适应之后,未经历过种群扩张,群体大小保持稳定。推测造成这一历史动态的原因可能是,互不连通的洞穴系统将多洞穴分布的物种切割成为各自独立的“岛屿”种群,这种地理阻隔促进了物种种群间的遗传分化;与此同时,由于相对封闭的黑暗洞穴中的环境条件极端稳定,云南洞密蛛无法实现种群扩张。
3.3 云贵高原地下洞穴的物种形成云贵高原位于中国地势的第二阶梯,受青藏高原造山运动的影响,气候复杂多变。Ballarin和Li(2018)的研究发现,在云贵高原范围内分布的小类球蛛属大多为洞穴类群,根据分化时间推断,其起源于始新世末期的地表,中新世的气候变冷促使小类球蛛迁入地下洞穴避难并定殖。体型更小的云南洞密蛛目前已知仅在洞穴中有分布,其地下栖息生境与小类球蛛相似。猜测它可能与小类球蛛有着相似的演化适应模式,不同的是它的地表种群可能已经绝灭,只有地下离散分布的洞穴种群得以保留,因洞穴“岛屿”隔离效应,阻断了该物种种群之间的基因交流,各种群的基因突变累积增加了整体的遗传多样性。
云南洞密蛛种群间高水平的分化程度支持Barr(1968)的假说:在洞穴殖民过程中,在初创事件之后,多样性会减少,随着一个物种在新的空白的地下生境拓殖定居,拓展了其种群在各自洞内的分布范围,通过长时间的演化,可累积增加遗传多样性,同时洞穴隔离和极端的生境条件会加大不同地理种群的遗传分化,有利于种群加速分化,最终形成新物种。
致谢: 感谢西华师范大学的李云春博士在野外帮助采集样本,四川大学生命科学学院的李亚和何怿婷在野外工作和样本鉴定方面提供的帮助。
刘名信, 彭作刚, 张志升. 2012. 蜘蛛分子系统学研究进展[J]. 蛛形学报, 21(2): 103-114. DOI:10.3969/j.issn.1005-9628.2012.02.010 |
宋大祥, 朱明生. 1994. 我国数种洞穴蛛形类记述. 中国动物学会成立60周年: 纪念陈桢教授诞辰100周年论文集[M]. 北京: 中国科学技术出版社, 35-46.
|
Avise JC. 2009. Phylogeography: retrospect and prospect[J]. Journal of Biogeography, 36(1): 3-15. DOI:10.1111/j.1365-2699.2008.02032.x |
Ballarin F, Li SQ. 2018. Diversification in tropics and subtropics following the mid-Miocene climate change: a case study of the spider genus Nesticella[J]. Global Change Biology, 24(2): 577-591. DOI:10.1111/gcb.13958 |
Barr TC. 1968. Cave ecology and the evolution of troglobites[M]//Dobzhansky T, Hecht MK, Steere WC. Evolutionary biology. Boston: Springer: 35-102.
|
Cardoso P, Pekar S, Jocque R, et al. 2011. Global patterns of guild composition and functional diversity of spiders[J/OL]. PLoS ONE, 6(6): e21710[2021-10-09]. https://doi.org/10.1371/journal.pone.0021710.
|
Clement M, Posada D, Crandall KA. 2000. TCS: a computer program to estimate gene genealogies[J]. Molecular Ecology, 9(10): 1657-1659. DOI:10.1046/j.1365-294x.2000.01020.x |
Cooper S, Bradbury J, Saint K, et al. 2007. Subterranean archipelago in the Australian arid zone: mitochondrial DNA phylogeography of amphipods from central Western Australia[J]. Molecular Ecology, 16(7): 1533-1544. DOI:10.1111/j.1365-294X.2007.03261.x |
Culver D, Pipan T, Schneider K. 2009. Vicariance, dispersal and scale in the aquatic subterranean fauna of karst regions[J]. Freshwater Biology, 54(4): 918-929. DOI:10.1111/j.1365-2427.2007.01856.x |
Excoffier L, Laval G, Schneider S. 2005. Arlequin (version 3.0): an integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics, 1: 47-50. |
Hall TA. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 41: 95-98. |
Hedin MC. 1997. Molecular phylogenetics at the population/species interface in cave spiders of the southern Appalachians (Araneae: Nesticidae: Nesticus)[J]. Molecular Biology and Evolotion, 14(3): 309-324. DOI:10.1093/oxfordjournals.molbev.a025766 |
Howarth FG. 1983. Ecology of cave arthropods[J]. Annual Review of Entomology, 28: 365-389. DOI:10.1146/annurev.en.28.010183.002053 |
Hudson RR, Slatkin M, Maddison WP. 1992. Estimation of levels of gene flow from DNA sequence data[J]. Genetics, 132(2): 583-589. DOI:10.1093/genetics/132.2.583 |
Kalyaanamoorthy S, Minh BQ, Wong TKF, et al. 2017. ModelFinder: fast model selection for accurate phylogenetic estimates[J]. Nature Methods, 14(6): 587-589. DOI:10.1038/nmeth.4285 |
Kumar S, Stecher G, Li M, et al. 2018. MEGA X: molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolotion, 35(6): 1547-1549. DOI:10.1093/molbev/msy096 |
Lanfear R, Frandsen PB, Wright AM, et al. 2017. PartitionFinder 2: new methods for selecting partitioned models of evolution for molecular and morphological phylogenetic analyses[J]. Molecular Biology and Evolotion, 34(3): 772-773. |
Leigh JW, Bryant D. 2015. POPART: full-feature software for haplotype network construction[J]. Methods in Ecology and Evolotion, 6(9): 1110-1116. DOI:10.1111/2041-210X.12410 |
Li Y, Lin YC. 2019. Taxonomic review of the Asian Trogloneta species (Araneae, Mysmenidae)[J]. ZooKeys, 817: 41-60. DOI:10.3897/zookeys.817.30468 |
Lin YC, Li SQ. 2008. Mysmenid spiders of China (Araneae: Mysmenidae)[J]. Annales Zoologici, 58(3): 487-520. DOI:10.3161/000345408X364337 |
Luo ZH, Tang SH, Jiang ZG, et al. 2016. Conservation of terrestrial vertebrates in a global hotspot of karst area in southwestern China[J/OL]. Scientific Reports, 6: 25717[2021-10-09]. https://doi.org/10.1038/srep25717
|
Mammola S, Piano E, Isaia M. 2016. Step back! Niche dynamics in cave-dwelling predators[J]. Acta Oecologica, 75: 35-42. DOI:10.1016/j.actao.2016.06.011 |
Nguyen LT, Schmidt HA, von Haeseler A, et al. 2015. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum likelihood phylogenies[J]. Molecular Biology and Evolotion, 32(1): 268-274. DOI:10.1093/molbev/msu300 |
Page T, Humphreys W, Hughes J. 2008. Shrimps down under: evolutionary relationships of subterranean crustaceans from Western Australia (Decapoda: Atyidae: Stygiocaris)[J/OL]. PLoS ONE, 3(2): e1618[2021-10-09]. https://doi.org/10.1371/journal.pone.0001618.
|
Poulson TL, White WB. 1969. The cave environment[J]. Science, 165(3897): 971-981. DOI:10.1126/science.165.3897.971 |
Ronquist F, Teslenko M, van der Mark P, et al. 2012. MrBayes 3.2: efficient Bayesian phylogenetic inference and model selection across a large model space[J]. Systematic Biology, 61(3): 539-542. DOI:10.1093/sysbio/sys029 |
Rozas J, Ferrer-Mata A, Sanchez-DelBarrio JC, et al. 2017. DnaSP 6: DNA sequence polymorphism analysis of large datasets[J]. Molecular Biology and Evolotion, 34(12): 3299-3302. DOI:10.1093/molbev/msx248 |
Shu SS, Jiang WS, Whitten T, et al. 2013. Drought and China's cave species[J]. Science, 340(6130): 272. |
Snowman CV, Zigler KS, Hedin M. 2010. Caves as islands: mitochondrial phylogeography of the cave-obligate spider species Nesticus barri (Araneae: Nesticidae)[J]. Journal of Arachnology, 38: 49-56. DOI:10.1636/A09-057.1 |
Zhang YY, Li SQ. 2014. A spider species complex revealed high cryptic diversity in South China caves[J]. Molecular Phylogenetics and Evolution, 79: 353-358. DOI:10.1016/j.ympev.2014.05.017 |
 2022, Vol. 41
2022, Vol. 41




