扩展功能
文章信息
- 李宗宪, 李寄仟, 徐伟江, 范丽仙
- LI Zongxian, LI Jiqian, XU Weijiang, FAN Lixian
- 黑斑蛙重盘吸虫的线粒体基因组特征及其系统发育研究
- Mitochondrial Genome Characterization and Phylogeny of Diplodiscus nigromaculati
- 四川动物, 2022, 41(3): 248-255
- Sichuan Journal of Zoology, 2022, 41(3): 248-255
- 10.11984/j.issn.1000-7083.20210372
-
文章历史
- 收稿日期: 2021-11-01
- 接受日期: 2022-01-21
2. 生物能源持续开发利用教育部工程研究中心,昆明 650500
2. Engineering Research Center of Sustainable Development and Utilization of Biomass Energy, Ministry of Education, Kunming 650500, China
黑斑蛙重盘吸虫Diplodiscus nigromaculati隶属复殖亚纲Digenea同盘总科Paramphistomoidea Fischoeder, 1901重盘科Diplodiscidae Cohn, 1904重盘属Diplodiscus Diesing, 1836。汪溥钦(1977)首次于黑斑蛙Pelophylax nigromaculatus消化道中检获并命名为黑斑蛙重盘吸虫,高利宾(2007)在泽蛙Rana limnocharis肠道内也记录了该种吸虫自然寄生。黑斑蛙重盘吸虫主要于江西、福建等地被报道,迄今仅见形态分类学研究(高利宾,2007;周庆安,2013)。
复殖吸虫系统发育研究大多基于核基因小分子片段(18S、28S和ITS 1-5.8S-ITS 2 rDNA联合序列)和线粒体cox1 (Littlewood & Olson,2001;Razo-Mendivil et al., 2004, 2006;Laidemitt et al., 2017;丁健等,2018;李寄仟等,2020)。这些分子标记较短、所含信息量难以全面呈现物种各分类阶元的关系。扁形动物的线粒体DNA进化速率是核基因单拷贝速率的5~10倍,相比单基因承载了更多的信息量,比任何1个相对较短的DNA序列具有更多、更广泛的特征(Boore,1999;Arnason et al., 2007;张娟,2011;Locke et al., 2018)。因此,线粒体基因组已被用作物种的准确鉴定依据,基于线粒体基因组对吸虫的系统学研究表明,线粒体基因组对研究吸虫的种间和种内变异具有更高可信度,在分子诊断学、系统分类学、基因进化和物种进化等方面也体现较大潜力(Le et al., 2002;Littlewood,2008;Hegedusova et al., 2014;Ma et al., 2015, 2017;聂宗恒等,2018;许姝歆等,2019)。
本文首次报道了重盘科吸虫线粒体基因组,补充了该类吸虫的遗传学数据,并揭示了其线粒体基因组特征,为后续开展基于线粒体基因组学的流行病学和系统发育研究提供新的基础资料。
1 材料与方法 1.1 吸虫的采集宿主滇蛙Dianrana pleuraden捕获于云南省曲靖市陆良县寨子山(103°30′2.81″E,25°3′54.66″N,海拔1 958 m)。将宿主双毁髓处死,收集消化道内的吸虫,部分做成装片。
1.2 DNA的提取与线粒体基因组的获取根据D3399-01石蜡DNA试剂盒说明书步骤提取总DNA。线粒体基因组由北京擎科生物科技有限公司通过二代测序获得,包括构建文库、测序、clean reads、组装和比对等流程。
1.3 线粒体基因组注释和数据分析tRNA通过ARWEN(Laslett & Canbck,2008)和tRNAscan-SE 2.0(Lowe & Chan,2016)注释,蛋白编码基因和rRNA通过Mitos(Brent et al., 2013)注释。采用PhyloSuite(Zhang et al., 2020)计算密码子使用、同义密码子相对使用度,使用R语言ggplot2(Wilkinson,2011)可视化。
1.4 系统发育分析为研究黑斑蛙重盘吸虫在同盘总科的系统发育地位,从NCBI下载同盘总科吸虫以及与其亲缘关系较近的棘口总科Echinostomatoidea吸虫:Paramphistomum cervi(KF475773)、P. cervi(NC023095)、P. leydeni(KP34 1657)、Fischoederius elongatus(KM397348)、Gastrothylax crumenifer(KM400624)、Echinostoma miyagawai(MH393928)、E. revolutum(MN116706)、E. hortense(KR062182)、Fasciola hepatica(AF216697)和F. gigantica(MH621335),并选取Schistosoma japonicum(AF215860)为外群,构建系统发育树。采用PhyloSuite(Zhang et al., 2020)完成对12个蛋白编码基因的提取、比对、修剪、串联、数据分区、模型选择和构建贝叶斯(BI)系统发育树,之后导入iTOL(Letunic & Bork,2021)编辑和美化。
2 结果 2.1 形态学虫体前端狭小,中部膨大,后端圆钝,呈纺锤形。口吸盘较小,其后有1对口支囊。食道存在。腹吸盘位于体末端,中央盘状突起存在。食管球明显。两肠支长而宽大,具有5~10个弯曲,延伸至腹吸盘的前缘。睾丸光滑,位于虫体中段。阴茎囊呈带状。生殖孔位于肠分叉之后。卵巢位于睾丸后,偏右侧,类圆形。子宫环分布于两肠之间,含少数虫卵。卵黄腺呈滤泡团块状,在肠支内外侧或与其重叠。排泄囊位于腹吸盘背侧,开孔于腹吸盘水平的前缘。根据上述形态特征确定所采集样本为黑斑蛙重盘吸虫。
2.2 线粒体基因组特征和核苷酸的使用黑斑蛙重盘吸虫线粒体基因组序列全长14 697 bp(GenBank登录号:MW698822),由22个tRNA基因、12个蛋白编码基因(nad1~6、nad4L、cox1~3、cytb、atp6)、2个rRNA基因和1个非编码区组成。各基因间有1~33 bp的基因间隔序列或无基因间隔序列的情况下彼此相邻或重叠1~41 bp(图 1;表 1)。基因组序列A、T、G、C含量分别为19.3%、40.7%、28.5%、11.5%,其中,蛋白编码基因的AT含量为59.7%,与线粒体全基因组的AT含量(60.0%)接近;nad4L基因的AT含量最高(65.2%);cox2基因的AT含量最低(57.1%)。

|
| 图 1 黑斑蛙重盘吸虫Diplodiscus nigromaculati线粒体基因组结构 Fig. 1 Mitochondrial genome structure of Diplodiscus nigromaculati |
| |
| 基因 Gene |
编码区域 Coding position (5'-3') |
长度 Length/bp |
起始/终止密码子 Start/stop codons |
氨基酸数 No. of amino acids |
基因间隔核苷酸数 Intergenic nucleotides |
| cox3 | 1~645 | 645 | ATG/TAG | 214 | 8 |
| trnH(gtg) | 654~719 | 66 | 3 | ||
| cytb | 723~1 838 | 1 116 | ATG/TAG | 371 | 8 |
| nad4L | 1 847~2 110 | 264 | GTG/TAG | 87 | -40 |
| nad4 | 2 071~3 357 | 1 287 | ATG/TAA | 428 | 7 |
| trnQ(ttg) | 3 365~3 429 | 65 | 2 | ||
| trnF(gaa) | 3 432~3 499 | 68 | 18 | ||
| trnM(cat) | 3 518~3 582 | 65 | 2 | ||
| atp6 | 3 585~4 100 | 516 | ATG/TAA | 171 | 20 |
| nad2 | 4 121~4 990 | 870 | ATG/TAA | 288 | 29 |
| trnV(tac) | 5 020~5 090 | 71 | 24 | ||
| trnA(tgc) | 5 115~5 181 | 67 | 6 | ||
| trnD(gtc) | 5 188~5 252 | 65 | 2 | ||
| nad1 | 5 255~6 157 | 903 | GTG/TAG | 300 | 10 |
| trnN(gtt) | 6 168~6 237 | 70 | 33 | ||
| trnP(tgg) | 6 271~6 338 | 68 | -1 | ||
| trnI(gat) | 6 338~6 398 | 61 | 21 | ||
| trnK(ctt) | 6 420~6 489 | 70 | 0 | ||
| nad3 | 6 490~6 843 | 354 | GTG/TAG | 117 | 2 |
| trnS1(gct) | 6 846~6 905 | 60 | 18 | ||
| trnW(tca) | 6 924~6 993 | 70 | 2 | ||
| cox1 | 6 996~8 543 | 1 548 | GTG/TAA | 515 | 13 |
| trnT(tgt) | 8 557~8 613 | 57 | -5 | ||
| rrnL | 8 609~9 665 | 1 057 | -41 | ||
| trnC(gca) | 9 625~9 690 | 66 | 3 | ||
| rrnS | 9 694~10 423 | 730 | 24 | ||
| cox2 | 10 448~11 032 | 585 | ATG/TAA | 194 | 17 |
| nad6 | 11 050~11 505 | 456 | ATG/TAG | 151 | 14 |
| trnY(gta) | 11 520~11 586 | 67 | 17 | ||
| trnL1(tag) | 11 604~11 675 | 72 | -3 | ||
| trnS2(tga) | 11 673~11 741 | 69 | 26 | ||
| trnL2(taa) | 11 768~11 834 | 67 | 12 | ||
| trnR(tcg) | 11 847~11 914 | 68 | 1 | ||
| nad5 | 11 916~13 478 | 1 563 | ATG/TAG | 520 | 20 |
| trnG(tcc) | 13 499~13 567 | 69 | 20 | ||
| trnE(ttc) | 13 588~13 655 | 68 | 0 | ||
| 非编码区 | 13 656~14 697 | 1 042 |
黑斑蛙重盘吸虫线粒体基因组序列的蛋白编码基因总长10 107 bp,包含了3 357个氨基酸。每个蛋白编码基因和密码子(123位)都呈现了较高的AT偏倚性。蛋白编码基因有2种起始密码子:ATG和GTG,2种终止密码子:TAA和TAG,且大多数蛋白编码基因的起始密码子为ATG。
在蛋白编码基因中最多使用的氨基酸是亮氨酸(Leu1:3.04%和Leu2:13.52%)、缬氨酸(Val:13.82%)和丝氨酸(Ser1:5.42%和Ser2:5.27%)(图 2)。在蛋白编码基因的密码子家族中使用最多的密码子是UUG(319)和UUU(310),最少的是ACC(4)和CUC(5)。
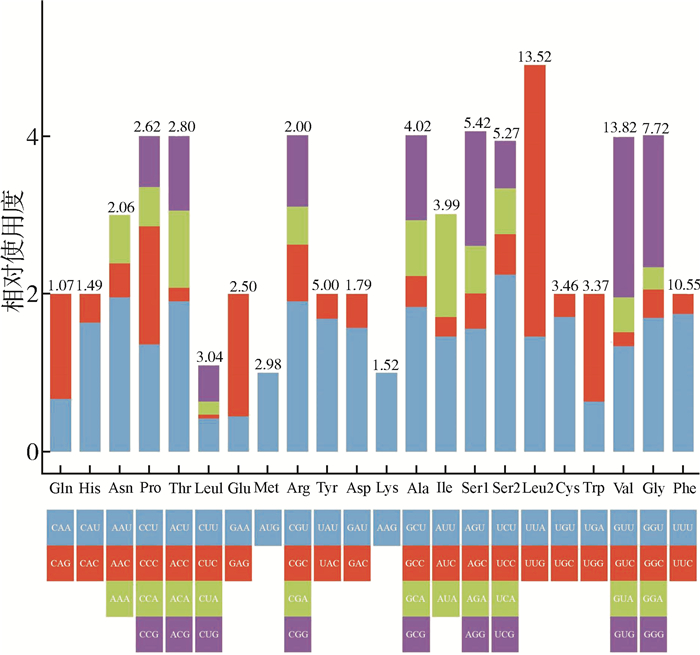
|
| 图 2 黑斑蛙重盘吸虫Diplodiscus nigromaculati线粒体基因组蛋白编码基因同义密码子相对使用度 Fig. 2 Relative synonymous codon usage of Diplodiscus nigromaculati mitogenome |
| |
黑斑蛙重盘吸虫线粒体基因组全序列共发现22个tRNA基因,长度为57~72 bp;除trnS1基因和trnT基因外,其他基因都呈传统的三叶草结构;trnS1基因缺失DHU臂;trnT基因缺失TΨC臂,这一结构特征在已发表的复殖吸虫线粒体基因组tRNA结构中未见。除trnM基因和trnK基因外,其他基因的反密码子起始都是T或G。
rrnL基因(16S)和rrnS基因(12S)分别长1 057 bp和730 bp,位于trnT基因和cox2基因之间,仅通过1个trnC基因分隔开。这一排列特征与已报道复殖吸虫线粒体基因组一致。
2.5 非编码区黑斑蛙重盘吸虫线粒体基因组的非编码区位于trnE基因和cox3基因之间,长1 042 bp,其余为1~33 bp较短的基因间隔序列。非编码区的AT含量为64.2%,与编码区的AT含量相近。在非编码区中通过人工比对,发现了2条重复序列,每条为112 bp,分别位于13 945~14 056和14 305~14 416;因其未相邻,所以未被Tandem Repeat Finder(Gary,1999)和mreps(Kolpakov et al., 2003)检测到。
2.6 系统发育分析系统发育树所有分支节点具有较高置信度(贝叶斯后验概率大部分为1)。系统发育树包括2个大分支:一支由同盘总科的5个物种组成(包括重盘科、腹袋科Gastrothylacidae和同盘科Paramphistomidae),另一支由片形科Fasciolidae的2个物种和棘口科Echinostomatidae的3个物种组成(图 3)。在同盘总科中,黑斑蛙重盘吸虫与其他物种关系较远。
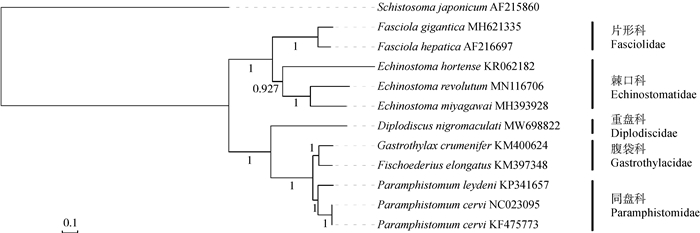
|
| 图 3 基于贝叶斯法的11种复殖吸虫线粒体12个蛋白编码基因序列构建的系统发育树 Fig. 3 Phylogenetic tree of concatenated 12 protein coding genes of mitochondrial genomes from 11 species of Digenea based on Bayesian inference |
| |
复殖吸虫的宿主特异性较弱,生活史中可选择不同属,甚至不同科的物种作为终末宿主(吴宝华,1991;唐崇惕,唐仲璋,2015)。研究者曾在黑斑蛙和泽蛙的消化道中发现自然感染的黑斑蛙重盘吸虫(高利宾,2007;周庆安,2013),本研究中的黑斑蛙重盘吸虫于滇蛙消化道内发现,滇蛙为其宿主新记录。
基因重排和互换是线粒体基因组的一个重要特征(张东,2020)。黑斑蛙重盘吸虫线粒体基因组除trnG基因和trnE基因发生互换外,其他基因排列与大多数复殖吸虫一致。从已报道的Paramphistomum leydeni、P. cervi(Yan et al., 2013)、Gastrothylax crumenifer(Yang et al., 2016)和Fischoederius elongatus(Han et al., 2020)4种同盘总科吸虫的线粒体基因组结果看,trnG和trnE基因互换似乎是同盘总科吸虫的一个重要遗传学特征。Littlewood等(2006)推测线粒体基因组中tRNA基因互换可能隐含了系统发育的相关信息(Littlewood et al., 2006)。本研究中黑斑蛙重盘吸虫线粒体基因组中trnG和trnE基因的互换现象为后续开展该类吸虫系统发育研究提供了明确的靶标。
碱基偏倚最能体现序列进化的特征,其可能来自自然突变或选择压力等其他因素(钟东等,2002)。本研究中黑斑蛙重盘吸虫的蛋白编码基因AT碱基占比为59.7%,比其他同盘总科吸虫低。因此,我们认为重盘属吸虫可能是一类GC→AT突变压力相对较小的类群。富含GC碱基密码子的基因通常指向了高度表达的基因(Lamolle et al., 2019),扁形动物绦虫的相关研究也证实了这一推论(Chen et al., 2013;Yang et al., 2014, 2015;Huang et al., 2017;Maldonado et al., 2018)。自然选择是基因高度表达的结果,密码子偏倚是碱基突变偏倚和自然选择之间平衡的结果(Sharp & Zeng,2010;Plotkin & Kudla,2011)。综上所述,本研究推测黑斑蛙重盘吸虫的这种密码子偏倚可能与重盘科吸虫的生态多样性和生理多样性有关。
基于线粒体基因组的系统发育分析结果更可靠(Waeschenbach et al., 2012)。复殖吸虫的物种多样性较高,加之形态结构特征的差异往往比较细微,仅依靠形态学手段往往存在“主观误差”:重盘属吸虫的形态特征与同盘科吸虫十分相似,唐崇惕和唐仲璋(2015)将其纳入同盘科。线粒体基因组的系统发育关系显示,黑斑蛙重盘吸虫单独为一支,腹袋科和同盘科的关系与黑斑蛙重盘吸虫更近。该结果支持Jones等(2005)的分类系统,认同重盘属隶属于重盘科。
目前仅有85种复殖吸虫(将其中未定种假定为不同的种)完成线粒体全基因组测序。该类吸虫记录物种超过18 000种(Bray et al., 2008),仍有极大部分复殖吸虫线粒体基因组信息未被研究和报道。而同盘总科的复殖吸虫是一类不容忽视的可导致动物吸虫病的寄生虫(Horak,1971;Yan et al., 2013),影响畜牧业的发展(Anuracpreeda et al., 2012;王根红,张志忠,2018)。从遗传学和基因组学的角度进一步揭示这类复殖吸虫对宿主的选择及致病机制,将有力推动吸虫病的防控。本研究首次揭示了黑斑蛙重盘吸虫线粒体基因组序列及其特征,为后续开展基于线粒体基因组信息的重盘科吸虫的分子系统发育和流行病学研究提供重要参考。
致谢: 感谢云南师范大学生命科学学院的敬凯老师在蛙类标本采集与鉴定中给予的帮助。
丁健, 李寄仟, 郭苗, 等. 2018. 澜沧江线鳢寄生叶形属复殖吸虫一新种[J]. 云南师范大学学报(自然科学版), 38(3): 63-69. |
高利宾. 2007. 南方四省无尾两栖类寄生复殖吸虫的分类研究[D]. 广州: 华南师范大学.
|
李寄仟, 李云臻, 范丽仙. 2020. 黄颡鱼寄生东肌吸虫属(复殖吸虫亚纲: 东肌吸虫科)3种复殖吸虫的分类学研究[J]. 四川动物, 39(6): 616-629. |
聂宗恒, 白俊, 许姝歆, 等. 2018. 复殖亚纲线粒体基因组特征分析及其应用进展[J]. 基因组学与应用生物学, 37(4): 48-57. |
唐崇惕, 唐仲璋. 2015. 中国吸虫学[M]. 北京: 科学出版社.
|
汪溥钦. 1977. 棘口、同盘两类吸虫新种记述和中华重盘吸虫的生活史研究[J]. 福建师范大学学报(自然科学版), 11(2): 62-77. |
王根红, 张志忠. 2018. 安徽省金寨县牛羊肠道寄生虫感染调查[J]. 畜牧与饲料科学, 39(8): 104-107. |
吴宝华. 1991. 浙江动物志[M]. 浙江: 浙江科学技术出版社.
|
许姝歆, 聂宗恒, 白俊, 等. 2019. 吸虫类线粒体基因组的特征及系统发生分析[J]. 基因组学与应用生物学, 38(2): 38-51. |
张东. 2020. 单殖吸虫的分子进化与系统发育研究及PhyloSuite平台开发[D]. 北京: 中国科学院大学.
|
张娟. 2011. 单殖吸虫线粒体基因组进化生物学研究[D]. 广州: 中山大学.
|
钟东, 赵贵军, 张振书, 等. 2002. 基因组内碱基分布整体均衡与局部不均衡的研究进展[J]. 遗传, 24(3): 351-355. DOI:10.3321/j.issn:0253-9772.2002.03.033 |
周庆安. 2013. 广西南宁市市售蛙, 蛇体内寄生虫调查[D]. 南宁: 广西大学.
|
Anuracpreeda P, Panyarachun B, Ngamniyom A, et al. 2012. Fischoederius cobboldi: a scanning electron microscopy investigation of surface morphology of adult rumen fluke[J]. Experimental Parasitology, 130(4): 400-407. DOI:10.1016/j.exppara.2012.02.001 |
Arnason U, Gullberg A, Janke A, et al. 2007. Mitogenomic analyses of caniform relationships[J]. Molecular Phylogenetics & Evolution, 45(3): 863-874. |
Boore JL. 1999. Animal mitochondrial genomes[J]. Nucleic Acids Research, 27(8): 1767-1780. DOI:10.1093/nar/27.8.1767 |
Bray RA, Gibson DI, Jones A. 2008. Keys to the Trematoda vol 3[M]. Wallingford: CABI Publishing and The Natural History Museum.
|
Brent M, Donath A, Jühling F, et al. 2013. MITOS: Improved de novo metazoan mitochondrial genome annotation[J]. Molecular Phylogenetics and Evolution, 69(2): 313-319. DOI:10.1016/j.ympev.2012.08.023 |
Chen L, Liu T, Yang D, et al. 2013. Analysis of codon usage patterns in Taenia pisiformis through annotated transcriptome data[J]. Biochemical & Biophysical Research Communications, 430(4): 1344-1348. |
Gary B. 1999. Tandem repeats finder: a program to analyze DNA sequences[J]. Nucleic Acids Research, 27(2): 573-580. DOI:10.1093/nar/27.2.573 |
Han ZQ, Li K, Luo H, et al. 2020. Characterization of the complete mitochondrial genome of Fischoederius elongatus derived from cows in Shanghai, China[J/OL]. BioMed Research International, 2020: 7975948[2021-09-10]. https://doi.org/10.1155/2020/7975948.
|
Hegedusova E, Brejova B, Tomaska L, et al. 2014. Mitochondrial genome of the basidiomycetous yeast Jaminaea angkorensis[J]. Current Genetics, 60(1): 49-59. DOI:10.1007/s00294-013-0410-1 |
Horak IG. 1971. Paramphistomiasis of domestic ruminants[J]. Advances in Parasitology, 9(8): 33-72. |
Huang X, Xu J, Chen L, et al. 2017. Analysis of transcriptome data reveals multifactor constraint on codon usage in Taenia multiceps[J/OL]. BMC Genomics, 18(1): 308[2021-08-10]. https://doi.org/10.1186/s12864-017-3704-8.
|
Jones A, Bray RA, Gibson DI. 2005. Keys to the Trematoda: volume 2[M]. Wallingford: CABI Publishing and The Natural History Museum.
|
Kolpakov R, Bana G, Kucherov G. 2003. mreps: efficient and flexible detection of tandem repeats in DNA[J]. Nucleic Acids Research, 31(13): 3672-3678. DOI:10.1093/nar/gkg617 |
Laidemitt MR, Zawadzki ET, Brant SV, et al. 2017. Loads of trematodes: discovering hidden diversity of paramphistomoids in Kenyan ruminants[J]. Parasitology, 144(2): 131-147. DOI:10.1017/S0031182016001827 |
Lamolle G, Fontenla S, Rijo G, et al. 2019. Compositional analysis of flatworm genomes shows strong codon usage biases across all classes[J/OL]. Frontiers in Genetics, 10(5): 771[2021-08-20]. https://doi.org/10.3389/fgene.2019.00771.
|
Laslett D, Canbck AB. 2008. ARWEN: a program to detect tRNA genes in metazoan mitochondrial nucleotide sequences[J]. Bioinformatics, 24(2): 172-175. DOI:10.1093/bioinformatics/btm573 |
Le TH, Blair D, Mcmanus DP. 2002. Mitochondrial genomes of parasitic flatworms[J]. Trends in Parasitology, 18(5): 206-213. DOI:10.1016/S1471-4922(02)02252-3 |
Letunic I, Bork P. 2021. Interactive tree of life (iTOL) v5: an online tool for phylogenetic tree display and annotation[J]. Nucleic Acids Research, 49(1): 293-296. |
Littlewood DT, Lockyer AE, Webster BL, et al. 2006. The complete mitochondrial genomes of Schistosoma haematobium and Schistosoma spindale and the evolutionary history of mitochondrial genome changes among parasitic flatworms[J]. Molecular Phylogenetics & Evolution, 39(2): 452-467. |
Littlewood DT, Olson P. 2001. Interrelationships of the Platyhelminthes[M]. London: Chemical Rubber Company Press.
|
Littlewood DT. 2008. Platyhelminth systematics and the emergence of new characters[J]. Parasite, 15(3): 333-341. DOI:10.1051/parasite/2008153333 |
Locke SA, Dam AV, Caffara M, et al. 2018. Validity of the Diplostomoidea and Diplostomida (Digenea, Platyhelminthes) upheld in phylogenomic analysis[J]. International Journal for Parasitology, 48(13): 1043-1059. DOI:10.1016/j.ijpara.2018.07.001 |
Lowe TM, Chan PP. 2016. tRNAscan-SE On-line: integrating search and context for analysis of transfer RNA genes[J]. Nucleic Acids Research, 24(2): 54-57. |
Ma J, He JJ, Liu GH, et al. 2015. Mitochondrial and nuclear ribosomal DNA dataset supports that Paramphistomum leydeni (Trematoda: Digenea) is a distinct rumen fluke species[J/OL]. Parasites & Vectors, 8(1): 201[2021-08-10]. https://doi.org/10.1186/s13071-015-0823-4.
|
Ma J, Sun MM, He JJ, et al. 2017. Fasciolopsis buski (Digenea: Fasciolidae) from China and India may represent distinct taxa based on mitochondrial and nuclear ribosomal DNA sequences[J]. Parasites & Vectors, 10(1): 101. |
Maldonado LL, Georgina S, Milone DH, et al. 2018. Whole genome analysis of codon usage in Echinococcus[J]. Molecular and Biochemical Parasitology, 225(18): 54-66. |
Plotkin JB, Kudla G. 2011. Synonymous but not the same: the causes and consequences of codon bias[J]. Nature Reviews Genetics, 12(1): 32-42. DOI:10.1038/nrg2899 |
Razo-Mendivil U, León-Règagnon V, Gerardo P. 2004. Description of two new species of Glypthelmins Stafford, 1905 (Digenea: Macroderoididae) in Rana spp. from Mexico, based on morphology and mtDNA and rDNA sequences[J]. Systematic Parasitology, 59(3): 199-209. DOI:10.1023/B:SYPA.0000048099.73779.f4 |
Razo-Mendivil U, Leónrégagnon V, Gerardo P. 2006. Monophyly and systematic position of Glypthelmins (Digenea), based on partial lsrDNA sequences and morphological evidence[J]. Organisms Diversity & Evolution, 6(4): 308-320. |
Sharp PM, Zeng EK. 2010. Forces that influence the evolution of codon bias[J]. Philosophical Transactions Biological Sciences, 365(1544): 1203-1212. DOI:10.1098/rstb.2009.0305 |
Waeschenbach A, Webster BL, Littlewood D. 2012. Adding resolution to ordinal level relationships of tapeworms (Platyhelminthes: Cestoda) with large fragments of mtDNA[J]. Molecular Phylogenetics & Evolution, 63(3): 834-847. |
Wilkinson L. 2011. ggplot2: elegant graphics for data analysis by Wickham, H[J]. Biometrics, 67(2): 671-679. DOI:10.1111/j.1541-0420.2011.01609.x |
Yan HB, Wang XY, Lou ZZ, et al. 2013. The mitochondrial genome of Paramphistomum cervi (Digenea), the first representative for the family Paramphistomidae[J/OL]. PLoS ONE, 8(8): e71300[2021-09-01]. https://doi.org/10.1371/journal.pone.0071300.
|
Yang X, Luo X, Cai X. 2014. Analysis of codon usage pattern in Taenia saginata based on a transcriptome dataset[J/OL]. Parasites & Vectors, 7(1): 527[2021-09-20]. https://doi.org/10.1186/s13071-014-0527-1.
|
Yang X, Ma X, Luo X, et al. 2015. Codon usage bias and determining forces in Taenia solium genome[J]. Korean Journal of Parasitology, 53(6): 689-697. DOI:10.3347/kjp.2015.53.6.689 |
Yang X, Wang LX, Chen HM, et al. 2016. The complete mitochondrial genome of Gastrothylax crumenifer (Gastrothylacidae, Trematoda) and comparative analyses with selected trematodes[J]. Parasitology Research, 115(6): 2489-2497. DOI:10.1007/s00436-016-5019-0 |
Zhang D, Gao F, Jakovli I, et al. 2020. PhyloSuite: an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Molecular Ecology Resources, 20(1): 348-355. DOI:10.1111/1755-0998.13096 |
 2022, Vol. 41
2022, Vol. 41




