扩展功能
文章信息
- 吕双娟, 舒林娟, 林啟研, 李思颖, 李丹宁, 肖越, 莫显明
- LYU Shuangjuan, SHU Linjuan, LIN Qiyan, LI Siying, LI Danning, XIAO Yue, MO Xianming
- 优化sgRNA序列提高斑马鱼CRISPR/Cas9系统的突变效率
- Enhancing the Mutative Efficiency of CRISPR/Cas9 System by Optimizing the sgRNA Sequences in Zebrafish
- 四川动物, 2021, 40(6): 622-631
- Sichuan Journal of Zoology, 2021, 40(6): 622-631
- 10.11984/j.issn.1000-7083.20210187
-
文章历史
- 收稿日期: 2021-06-01
- 接受日期: 2021-08-03
基因编辑技术是研究动物基因功能和物种进化基因规律的有效手段,其中最方便的基因编辑技术是CRISPR/Cas9技术。CRISPR/Cas9是一种在细菌和古细菌体内由sgRNA和Cas9蛋白组成复合体(Deltcheva et al., 2011)以抵御外源DNA的适应性免疫防御系统(Horvath & Barrangou,2010;Wiedenheft et al., 2012)。CRISPR/Cas9技术在生物医学等领域有着广泛的应用,已经成功地在多个物种中实现了基因组编辑,如斑马鱼Danio rerio(Chang et al., 2013;Hwang et al., 2013a, 2013b;Xiao et al., 2013;Yin et al., 2015;Brocal et al., 2016;Gasanov et al., 2021;Shaw & Mokalled,2021)。CRISPR/Cas9复合体通过碱基互补配对识别并与外源靶DNA的互补序列结合,在靶DNA的前间区序列邻近基序(protospacer adjacent motif,PAM)区上游3 bp引入双链断裂并通过非同源末端连接或同源定向修复以实现基因的特定编辑(Jinek et al., 2012;Cong et al., 2013;Jiang et al., 2013;Mali et al., 2013)。CRISPR/Cas9基因编辑系统依赖于sgRNA的识别序列与靶点序列的特异性结合。可以通过改变sgRNA序列来增加CRISPR/Cas9系统的靶效应,例如截断sgRNA的5’端碱基(Cho et al., 2014;Fu et al., 2014)或在sgRNA的5’端增加2个G(Cho et al., 2014)均可以降低脱靶效应;在斑马鱼体内针对同一基因使用2个sgRNAs造成目的基因的基因组大片段缺失(Xiao et al., 2013;Hoshijima et al., 2019)或者针对同一目标位点使用2个互补的sgRNA(Gasanov et al., 2021)可显著提高CRISPR/Cas9的突变效率。然而,由于基因组的复杂性和sgRNAs对特定碱基错配的忍耐性(Bolukbasi et al., 2016),sgRNA的识别序列同样可能与其他非靶DNA序列部分匹配,激活Cas9核酸内切酶活性,导致脱靶效应,从而降低靶DNA的切割活性(Yanni et al., 2014;Ewa et al., 2018)。因此,对sgRNA进行特异性和高效性预测具有重要意义。
本研究分析了针对斑马鱼35个基因的157个sgRNAs的序列组成,发现高效的sgRNA具有序列组成偏好性,能够最大化提高CRISPR/Cas9系统的切割效率。此外,对已发表的sgRNA序列特征进行重新分析以验证本研究所获得的序列特征是否具有普遍适用性,便于优化用于CRISPR/Cas9基因编辑和遗传筛选的sgRNA设计。
1 材料与方法 1.1 实验动物与实验试剂 1.1.1 实验动物斑马鱼品系为野生型斑马鱼AB和转基因型斑马鱼(Ifabp: Red/elastase: GFP)。所有动物实验均经四川大学华西医院实验动物伦理委员会批准。
1.1.2 实验试剂质粒pMD-18T由本实验室保存;Cas9蛋白(GenCrispr NLS-Cas9-NLS Nuclease)购自GenScript;体外转录试剂盒MEGAscript® T7 Kit购自Thermo Fisher Scientific;T7 EndonucleaseⅠ购自NEB。
1.2 CRISPR靶序列的设计和gRNA的合成使用CHOPCHOP(Montague et al., 2014)和Benchling预测和选择基因中最优的CRISPR/Cas9靶序列。为满足体外转录T7启动子的要求,本研究中使用的大部分靶点以5’-GG开头。然而,在某些基因上选择5’-GG开头的sgRNA较难,因此将序列要求放宽为5’-GN-3’。此外,每个基因至少设计3个不同的sgRNAs。
用特异性引物(表 1)以sgRNA骨架质粒pMD-18T(含T7启动子、RNA支架和氨苄青霉素抗性)为模板,使用Q5高保真酶进行PCR扩增,得到sgRNA体外转录模板。PCR产物经琼脂糖凝胶电泳进行单一条带(125 bp)确认后直接纯化回收。1 μg sgRNA模板通过试剂盒进行体外转录并通过LiCl沉淀法纯化回收。
| 引物 | 序列(5’-3’) |
| 上游引物F | TAATACGACTCACTATAG+GG/GN+18NT+GTTTTAGAGCTAGAAAATAGC |
| 下游引物R | AAAAAAAGCACCGACTCGGTGCCAC |
通过显微注射将1 nL的sgRNA和含有核定位信号(NLS)的Cas9蛋白的混合液(终浓度为250 ng sgRNA/μL和150 ng NLS-Cas9 NLS/μL)注射到野生型斑马鱼AB胚胎单细胞期的细胞质中。每种sgRNA注射300多枚胚胎,在检测切割活性后,将剩余的胚胎培养至成年,形成F0代。
T7EⅠ核酸内切酶特异性识别和切割异源双链DNA错配。显微注射24 h后,随机收集8枚未注射的胚胎作为对照,同时随机收集至少18枚(6枚/管)注射后的胚胎来鉴定靶点sgRNA的有效性。用碱裂解法提取胚胎基因组并以此为模板进行PCR扩增。利用T7EⅠ核酸内切酶选择性地消化PCR产物中的异源双链DNA,通过比较对照组与实验组的琼脂糖凝胶图谱上条带的数量来确定突变频率。将非单一条带的PCR产物进行Sanger测序,确保基因的靶向有效性和突变效率。
1.4 种系突变的筛选确认sgRNA活性后,将剩余的胚胎或2~3个有效sgRNAs共注射的胚胎培养至成体,并通过剪尾鳍确定其是否发生有效突变。经T7EⅠ核酸内切酶检测和Sanger测序初步筛选后,将F0代与野生型斑马鱼杂交获得F1代。待其长至1.5个月左右,对每条F1进行剪尾鳍鉴定和Sanger测序,获得携带移码突变的F1突变体。将具有相同突变类型的F1突变个体进行交配获得F2代。理论上F2代应包括纯合子突变个体、杂合子突变个体和野生型突变个体。将雌雄纯合子突变体F2进行交配,获得稳定遗传突变种系。
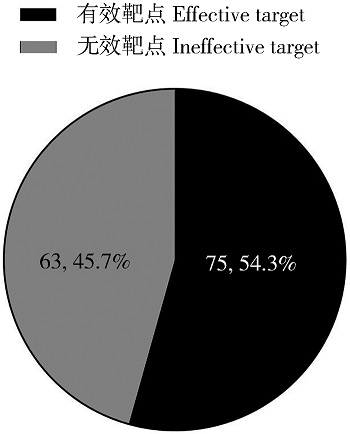
2 结果 2.1 CRISPR/Cas9是斑马鱼产生突变的有效方法运用ChopChop和Benchling设计并检测了35个斑马鱼基因中的157个sgRNAs(附件)。首先利用T7EⅠ核酸内切酶检测和Sanger测序,检测每个sgRNA的切割活性。结果表明,75个sgRNAs(47.8%)有效。为了获得更准确的序列特征,剔除了Tcf4基因,因为其19个sgRNAs的切割活性检测结果均显示无效。靶点有效率提高至54.3%(图 1),并且F1代鉴定测序显示35个基因中有34个(97%)均发生了可遗传的种系突变。为了验证157个sgRNAs活性序列特征的有效性,从现有的sgRNA文库中重新分析了959个sgRNAs。959个sgRNAs的效率显示出类似的结果(图 2:a)。
|
| 图 1 sgRNAs的靶点有效性 Fig. 1 The target effectiveness of sgRNAs |
| |

|
| 图 2 959个sgRNAs的靶点有效性及序列特征 Fig. 2 target effectiveness and sequence characteristics of 959 sgRNAs PAM. 前间区序列邻近基序protospacer adjacent motif; 下同,the same below |
| |
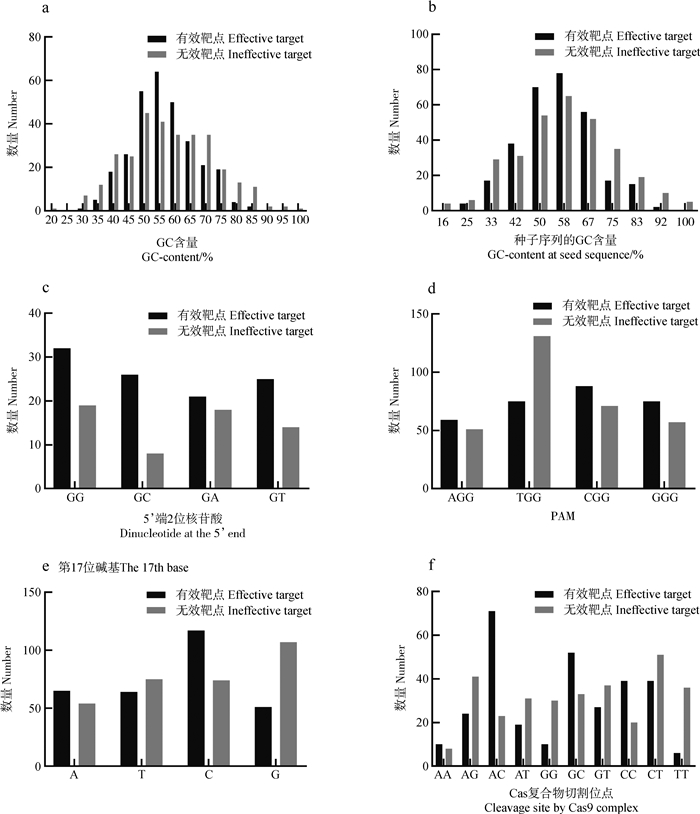
为了更好地探索CRISPR/Cas9系统的sgRNA序列组成在斑马鱼中的影响,分析了本研究的138个sgRNAs和已发表的959个sgRNAs的序列特征。首先比较所有靶点sgRNAs的GC含量分布(忽略PAM中NGG上的GG)。在整个靶点序列(20 bp)中,GC含量为50%~60%的有效靶点明显高于无效靶点(图 3:a),同时在已发表的959个sgRNAs中也存在这一趋势,尽管GC含量为45%和65%的sgRNAs在切割方面略有优势(图 2:b)。对sgRNAs的12个种子序列(近PAM端1~12位碱基)进行GC含量分析,50%~58%的GC含量在sgRNA种子区域有明显的富集现象(图 3:b),而已发表的959个sgRNAs中, 种子序列的GC含量为42%~67%(图 2:c)。

|
| 图 3 有效靶点与无效靶点的序列特征 Fig. 3 The sequence characteristics of effective and ineffective target sites |
| |
sgRNA序列中核苷酸的组成可能会影响CRISPR/Cas9系统的靶向切割效率,故分析了所有sgRNAs序列上重要位置的核苷酸偏好性。首先,比较了有效靶点和无效靶点的前2位碱基的组成。结果显示,与5’-GG sgRNAs相比,5’-GA sgRNAs的切割效率显著降低(P=0.037)(图 3:c)。对已发表的959个sgRNAs的前2位碱基偏好性统计表明,5’-GA sgRNAs序列的切割效率也明显较低(P=0.021)(图 2:d)。
2.4 PAM的可变核苷酸偏好C而排斥A为了评估sgRNA活性与PAM可变核苷酸之间的关系,以无活性sgRNAs作为阴性对照,比较了活性sgRNAs的PAM可变核苷酸的碱基偏好性。结果表明,AGG和CGG之间存在显著差异(P=0.026)(图 3:d)。PAM的第一个核苷酸为C能显著提高靶点的切割效率,而PAM可变核苷酸为A的sgRNAs则会明显降低靶点活性。在已发表的959个sgRNAs中(图 2:e)同样观察到PAM可变核苷酸对C的偏好。
2.5 第17位碱基为C的sgRNA具有较高的切割效率sgRNA的有效性同样取决于Cas9蛋白与sgRNA的结合效率。对sgRNA真正的“种子”区域(邻近PAM 5’端的5位碱基序列,即16~20 nt)的碱基分布进行评估。结果显示,除了在第17位碱基上存在明显的偏好C而排斥T外(P=0.034),第16、18、19、20位碱基上无显著的碱基偏好性,尽管与无效靶点相比,有效靶点在这4个位置出现G和C的频率都略高(图 4:a~e)。已发表的959个sgRNAs的第17位碱基上也存在对C的偏好性(P<0.000 1)(图 2:f)。同时还观察到,当CRISPR/Cas9系统的切割位点碱基对(第17/18位碱基)为AC时,sgRNAs具有较高的靶切割效率(图 4:f),但无显著差异(P=0.07)。对已发表的959个sgRNAs的切割位点碱基偏好性的统计分析表明,第17/18位碱基为AC的sgRNAs比其他sgRNAs具有显著的优势(P < 0.05)(图 2:g)。

|
| 图 4 有效sgRNAs与无效sgRNAs的碱基偏好性 Fig. 4 Base preference of effective and ineffective sgRNAs |
| |
为了验证sgRNA的设计原则在其他物种中的普遍适用性,再次分析959个sgRNAs中非斑马鱼基因相关基因的607个sgRNAs的序列特征。结果显示,607个sgRNAs的靶点序列和种子序列中的GC含量与之前的结果完全吻合(图 5:a,b);5’-GA sgRNAs和5’-GG sgRNAs之间仍然存在显著性差异(P=0.049)(图 5:c);PAM可变碱基偏好C,而排斥T(P=0.001),虽然与之前的结果略有不同,但都表明在设计sgRNA时,最好选择C作为PAM的可变碱基(图 5:d);最后对第17位碱基及CRISPR/Cas9切割位点的碱基分布统计发现,C在第17位碱基有明显优势(P<0.001)(图 5:e),而且切割位点的碱基对为AC的sgRNAs也有明显的优势(P<0.001)(图 5:f)。
|
| 图 5 607个sgRNAs的序列特征 Fig. 5 The sequence characteristics of 607 sgRNAs |
| |
本研究检测了影响sgRNA切割效率的重要参数,包括GC含量、5’端双核苷酸组成、PAM的可变碱基组成和Cas9核酸内切酶切割位点的碱基偏好性,发现通过优化相关参数可以提高斑马鱼CRISPR/Cas9系统的基因突变效率。同时为了验证结果的普遍适用性,本研究分析了已发表的大量sgRNA序列,包括352个斑马鱼相关基因的sgRNAs(219个有效和133个无效)、279个人类Homo sapiens相关基因的sgRNAs(133个有效和146个无效)和328个人类与小鼠Mus musculus相关基因的sgRNAs(164个有效和164个无效) (Doench et al., 2014;Gagnon et al., 2014;Chari et al., 2015;Shah et al., 2015;Varshney et al., 2015;Shaw & Mokalled,2021)。其中,631个斑马鱼和人类相关基因的sgRNAs的靶点序列及靶点有效性直接从文献中获得,但并未提及具体的突变效率。328个人类与小鼠相关基因的sgRNAs来源于Doench等(2014)中提及的1 841个sgRNAs。为了较准确得到有效靶点与无效靶点的序列特征,选择突变效率>0.5的164个sgRNAs为有效靶点,同时选择相应效率最低(突变效率<0.02)的164个sgRNAs作为无效靶点。
虽然现在有很多软件可以帮助设计出高效的sgRNA(Chari et al., 2015;Wong et al., 2015;Xu et al., 2015),但所获得的高效率靶点也存在一定的缺陷, 并且软件的效率评分高低不能代表真实的切割活性。因此,在设计sgRNA时,每个基因可设计3~4个sgRNAs,且必须在体内验证其切割活性后,选择高效率的sgRNA进行下一步实验。虽然已有研究表明,GC含量为40%~80%的sgRNAs具有切割效率(Gagnon et al., 2014;Wang et al., 2014),但本研究发现,GC含量为50%~60%的sgRNAs序列有更好的切割效率。碱基对错配的忍耐性(Hsu et al., 2013)以及5’端GG(Hwang et al., 2013a, 2013b)的存在会影响sgRNA的GC含量。本研究对sgRNA种子序列(Jinek et al., 2012;Shang et al., 2015)中的GC含量分布进行分析发现,GC含量为50%~60%的sgRNAs同样具有高效的切割效率。Gagnon等(2014)的研究表明,与5’-GG sgRNAs相比,5’-GA sgRNAs的切割效率只存在轻微下降(P>0.05);而本研究发现,与5’-GG sgRNAs相比,5’-GA sgRNAs的切割效率显著降低(P=0.037)。
CRISPR/Cas9系统利用PAM识别靶DNA序列中潜在的靶点, 同时PAM有助于Cas9蛋白与靶DNA的结合(Sternberg et al., 2014)。CRISPR/Cas9系统的PAM通常为NGG。对所有sgRNAs的PAM的可变碱基“N”进行统计分析,发现C的频率在有效靶点中占有绝对优势,这与以前的研究结果一致(Gagnon et al., 2014);但本研究结果显示,A大量存在于无效靶点中,而不是T(Doench et al., 2014)。因此在设计sgRNA时,最好选择C作为PAM的可变碱基,同时避免T和A。CRISPR/Cas9系统识别靶点后,Cas9蛋白的2个核酸内切酶(HNH核酸内切酶和RuvC核酸内切酶)分别在PAM 5’端上游3 bp和4 bp之间(第17/18位碱基)切割DNA链的互补链与非互补链,导致DNA双链断裂(Jinek et al., 2012;Shen et al., 2013)。对sgRNA真正“种子”区域(Wu et al., 2014)的碱基组成以及切割位点的碱基组成分析发现,C在第17位碱基有明显优势,而且切割位点为AC的sgRNAs也有明显的优势,但同时也应该避免切割位点为AA、TT和GG的sgRNAs。
对已发表的sgRNA相关参数的分析表明,本研究优化的sgRNA设计原则具有可使用性,并且在其他物种中具有普遍适用性。这些发现为动物(尤其是斑马鱼)体内设计最有效的sgRNA提供了一个可行的框架,也为发育生物学研究中的sgRNA设计提供了便利。
附录 Supplementary material附件 35个斑马鱼基因中的157个sgRNAs
Appendix 157 sgRNAs in 35 genes of zebrafish
Bolukbasi MF, Gupta A, Wolfe SA, et al. 2016. Creating and evaluating accurate CRISPR-Cas9 scalpels for genomic surgery[J]. Nature Methods, 13: 41-50. DOI:10.1038/nmeth.3684 |
Brocal I, White RJ, Dooley CM, et al. 2016. Efficient identification of CRISPR/Cas9-induced insertions/deletions by direct germline screening in zebrafish[J/OL]. BMC Genomics, 17: 259[2021-02-10]. https://doi.org/10.1186/s12864-016-2563-z.
|
Chang N, Sun C, Gao L, et al. 2013. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos[J]. Cell Research, 23(4): 465-472. DOI:10.1038/cr.2013.45 |
Chari R, Mali P, Moosburner M, et al. 2015. Unraveling CRISPR-Cas9 genome engineering parameters via a library-on-library approach[J]. Nature Methods, 12(9): 823-826. DOI:10.1038/nmeth.3473 |
Cho SW, Kim S, Kim Y, et al. 2014. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases[J]. Genome Research, 24(1): 132-141. DOI:10.1101/gr.162339.113 |
Cong L, Ran FA, Cox D, et al. 2013. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 339(6121): 819-823. DOI:10.1126/science.1231143 |
Deltcheva E, Chylinski K, Sharma CM, et al. 2011. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J]. Nature, 471(7340): 602-607. DOI:10.1038/nature09886 |
Doench JG, Hartenian E, Graham DB, et al. 2014. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation[J]. Nature Biotechnology, 32(12): 1262-1267. DOI:10.1038/nbt.3026 |
Ewa KK, Judyta J, Wojciech M, et al. 2018. CRISPR/Cas9 technology as an emerging tool for targeting amyotrophic lateral sclerosis (ALS)[J/OL]. International Journal of Molecular Sciences, 19(3): 906[2021-02-10]. https://doi.org/10.3390/ijms19030906.
|
Fu Y, Sander JD, Reyon D, et al. 2014. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs[J]. Nature Biotechnology, 32(3): 279-284. DOI:10.1038/nbt.2808 |
Gagnon JA, Valen E, Thyme SB, et al. 2014. Efficient mutagenesis by Cas9 protein-mediated oligonucleotide insertion and large-scale assessment of single-guide RNAs[J/OL]. PLoS ONE, 9(5): e98186[2021-02-10]. https://doi.org/10.1371/journal.pone.0098186.
|
Gasanov EV, Jędrychowska J, Pastor M, et al. 2021. An improved method for precise genome editing in zebrafish using CRISPR-Cas9 technique[J]. Molecular Biology Reports, 48: 1951-1957. DOI:10.1007/s11033-020-06125-8 |
Horvath P, Barrangou R. 2010. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 327(5962): 167-170. DOI:10.1126/science.1179555 |
Hoshijima K, Jurynec MJ, Klatt SD, et al. 2019. Highly efficient CRISPR-Cas9-based methods for generating deletion mutations and F0 embryos that lack gene function in zebrafish[J]. Developmental Cell, 51(5): 645-657. DOI:10.1016/j.devcel.2019.10.004 |
Hsu PD, Scott DA, Weinstein JA, et al. 2013. DNA targeting specificity of RNA-guided Cas9 nucleases[J]. Nature Biotechnology, 31(9): 827-832. DOI:10.1038/nbt.2647 |
Hwang WY, Fu Y, Reyon D, et al. 2013a. Efficient genome editing in zebrafish using a CRISPR-Cas system[J]. Nature Biotechnology, 31(3): 227-229. DOI:10.1038/nbt.2501 |
Hwang WY, Fu Y, Reyon D, et al. 2013b. Heritable and precise zebrafish genome editing using a CRISPR-Cas system[J/OL]. PLoS ONE, 8(7): e68708[2021-02-10]. https://doi.org/10.1371/journal.pone.0068708.
|
Jiang W, Bikard D, Cox D, et al. 2013. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology, 31(3): 233-239. DOI:10.1038/nbt.2508 |
Jinek M, Chylinski K, Fonfara I, et al. 2012. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 337(6096): 816-821. DOI:10.1126/science.1225829 |
Liang G, Zhang H, Lou D, et al. 2016. Selection of highly efficient sgRNAs for CRISPR/Cas9-based plant genome editing[J]. Scientific Reports, 6(21451): jkab089[2021-02-10]. https://doi.org/10.1093/g3journal/jkab089.
|
Lin YN, Cradick TJ, Brown MT, et al. 2014. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences[J]. Nucleic Acids Research, 42(11): 7473-7485. DOI:10.1093/nar/gku402 |
Mali P, Yang L, Esvelt KM, et al. 2013. RNA-guided human genome engineering via Cas9[J]. Science, 339(6121): 823-826. DOI:10.1126/science.1232033 |
Montague TG, Cruz JM, Gagnon JA, et al. 2014. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing[J]. Nucleic Acids Research, 42(W1): W401-W407. DOI:10.1093/nar/gku410 |
Shah AN, Davey CF, Whitebirch AC, et al. 2015. Rapid reverse genetic screening using CRISPR in zebrafish[J]. Nature Methods, 12(6): 535-540. DOI:10.1038/nmeth.3360 |
Shang C, Liu Z, Chen Z, et al. 2015. A parvalbumin-positive excitatory visual pathway to trigger fear responses in mice[J]. Science, 348(6242): 1472-1477. DOI:10.1126/science.aaa8694 |
Shaw DK, Mokalled MH. 2021. Efficient CRISPR/Cas9 mutagenesis for neurobehavioral screening in adult zebrafish[J/OL]. BioRxiv[2021-02-10]. https://doi.org/10.1101/2021.02.01.429280.
|
Shen B, Zhang J, Wu H, et al. 2013. Generation of gene-modified mice via Cas9/RNA-mediated gene targeting[J]. Cell Research, 23: 720-723. DOI:10.1038/cr.2013.46 |
Sternberg SH, Redding S, Jinek M, et al. 2014. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9[J]. Nature, 507(7490): 62-67. DOI:10.1038/nature13011 |
Varshney GK, Pei W, LaFave MC, et al. 2015. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9[J]. Genome Research, 25(7): 1030-1042. DOI:10.1101/gr.186379.114 |
Wang T, Wei JJ, Sabatini DM, et al. 2014. Genetic screens in human cells using the CRISPR-Cas9 system[J]. Science, 343(6166): 80-84. DOI:10.1126/science.1246981 |
Wiedenheft B, Sternberg SH, Doudna JA. 2012. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature, 482(7385): 331-338. DOI:10.1038/nature10886 |
Wong N, Liu W, Wang X. 2015. WU-CRISPR: characteristics of functional guide RNAs for the CRISPR/Cas9 system[J/OL]. Genome Biology, 16: 218[2021-01-30]. https://doi.org/10.1186/s13059-015-0784-0.
|
Wu X, Scott DA, Kriz AJ, et al. 2014. Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells[J]. Nature Biotechnology, 32(7): 670-676. DOI:10.1038/nbt.2889 |
Xiao A, Wang Z, Hu Y, et al. 2013. Chromosomal deletions and inversions mediated by TALENs and CRISPR/Cas in zebrafish[J/OL]. Nucleic Acids Research, 41(14): e141[2021-02-10]. https://doi.org/10.1093/nar/gkt464.
|
Xu H, Xiao T, Chen C, et al. 2015. Sequence determinants of improved CRISPR sgRNA design[J]. Genome Research, 25(8): 1147-1157. DOI:10.1101/gr.191452.115 |
Yin L, Jao LE, Chen W. 2015. Generation of targeted mutations in zebrafish using the CRISPR/Cas system[J]. Methods in Molecular Biology, 1332: 205-217. |
 2021, Vol. 40
2021, Vol. 40




