扩展功能
文章信息
- 蒋兰, 张雪艳, 王俊茵, 李静
- JIANG Lan, ZHANG Xueyan, WANG Junyin, LI Jing
- 豹猫转录组从头组装及组织特异性表达分析
- De novo Transcriptome and Tissues-Specific Expression Analysis of Prionailurus bengalensis
- 四川动物, 2021, 40(5): 497-508
- Sichuan Journal of Zoology, 2021, 40(5): 497-508
- 10.11984/j.issn.1000-7083.20210126
-
文章历史
- 收稿日期: 2021-04-06
- 接受日期: 2021-06-21
2. 峨眉山景区管委会, 峨眉山生物多样性保护研究所, 四川峨眉山 614200
2. Mount Emei Scenic Area Management Committee, Mount Emei Biodiversity Conservation Institute, Emeishan, Sichuan Province 614200, China
豹猫Prionailurus bengalensis属食肉目Carnivora猫科Felidae豹猫属,地理分布极为广泛,从亚洲南部和西南部的热带雨林到中国北方和俄罗斯远东地区的森林(Hughes,1998;Woolf et al., 2002),在中国,根据地理分布豹猫被分为5个亚种(Ross et al., 2015)。尽管豹猫资源丰富,但人类活动使其栖息地不断缩小,同时对其皮毛的需求也导致豹猫的生存受到严重威胁(Hughes,1998)。目前中国的野生豹猫种群数量正在下降,分布范围也逐渐缩小,一些原有的分布区域,如宁夏、青海、山东等地区的豹猫数量已非常稀少。《中国濒危动物红皮书》(汪松,1998)将豹猫列为易危种。
目前国内外有关豹猫的文献报道集中在遗传多样性和系统发育等研究。研究人员分别利用12s RNA、cyt b序列和cDNA序列探讨西表岛猫Prionailurus iriomotensis和豹猫系统发育关系,结果表明二者分歧时间较近,西表岛猫应为豹猫的一个亚种(Masuda et al., 1994;Suzuki et al., 1994)。Masuda和Yoshida(1995)基于cyt b序列认为马岛猫Prionailurus bengalensis euptilurus也是豹猫的一个亚种。Saka等(2018)发现西表岛和马岛的豹猫种群的MHC多样性远低于家猫Felis catus种群,暗示岛屿上的豹猫由于近亲繁殖或地理隔离,其对病原体的抵抗力降低。Ko等(2018)基于微卫星分析了韩国豹猫的遗传多样性,发现其平均等位基因数和杂合度分别为3.8和0.41,多样性低于其他12种猫科动物57个种群的平均值。白素英等(2004)利用随机扩增多态性DNA(RAPD)研究中国6个豹猫种群结构发现,各种群的关系与种群间的地理距离及亚种划分一致。目前国内关于豹猫的研究集中在分类、分布、食性以及解剖等方面(张淑云等,1988;白素英等,2004;Xiong et al., 2016),而关于其基因表达的研究还未见报道。
转录组测序技术(RNA-seq)是一种广泛应用于研究基因表达调控的分析方法,通过有效识别和量化RNA转录本,从而获得其特定生理状况的转录组信息(Feng et al., 2012;Spradling et al., 2013)。RNA-seq使得从整体水平分析动物基因结构及基因功能成为可能,更有利于发现生物学过程、免疫反应基因、疾病产生过程中的基因表达谱变化以及免疫受体多样化机制(Wang & Kirkness,2005)。由于RNA-seq技术对样本新鲜程度要求较高,一定程度上阻碍了其在野生动物相关研究中的应用。本研究基于1只意外死亡的野生豹猫个体,采用RNA-seq对其大脑、心脏、肾脏、肝脏、肺和骨骼肌6个组织进行转录组测序,通过从头组装、注释,报道了一个高质量的豹猫转录组。这些数据为理解豹猫的遗传背景及基因表达谱提供了宝贵的资源,也为这一小型猫科动物的基因组注释和深入开展保护遗传学研究奠定了基础。
1 材料与方法 1.1 样品采集与测序豹猫各器官的组织样品均来自于1只在峨眉山被汽车意外撞死的雌性成年个体,被管理人员发现后提供给本实验室。共采集了6个不同器官的组织样品,分别为大脑、心脏、肾脏、肝脏、肺和骨骼肌,新鲜的组织样品迅速置于液氮中保存,其余样品-80 ℃储存。样品的总RNA提取和转录组建库测序工作由北京诺禾致源生物信息科技有限公司完成,采用Illumina HiSeq 4000测序平台对6个样品的cDNA文库分别进行150 bp的双端测序。
1.2 质控与过滤转录组组装前需对原始序列进行质量控制,过滤标准为去除含N比例大于10%、低质量(质量值<5)碱基数比例大于50%、含有接头污染的序列,最终得到高质量的clean reads用于后续分析。
1.3 转录组从头组装使用Trinity(Haas et al., 2013)对过滤后的clean reads进行de novo组装,仅输出片段长度大于300 bp的contig序列。由于Trinity组装获得的转录组中通常含有大量相似的冗余序列,故最后使用Cd-hit-est (Li & Godzik,2006)去除冗余序列,得到非冗余转录本用于后续分析。
1.4 功能注释为了获得较为全面的基因功能信息,利用blastx(Camacho et al., 2009)将Unigene与NCBI非冗余蛋白质(NR)数据库、euKaryotic Ortholog Groups(KOG)数据库、Swiss-Prot数据库进行比对,E值设为1E-5。再根据NR数据库的比对结果进行Gene Ontology(GO)和Kyoto Encyclopedia of Genes and Genomes(KEGG)注释的分类统计,分别通过在线工具Web Gene Ontology Annotation Plot(WEGO)(Ye et al., 2006)和KEGG Automatic Annotation Server(KAAS)完成(Moriya et al., 2007)。在使用WEGO进行分类统计之前,需先将转录本与NR数据库比对的结果导入Blast2GO(Conesa et al., 2005)搜索相关的GO功能条目。
1.5 转录本的表达量和组织特异性分析为了解豹猫不同组织中基因的差异表达,统计了所有组装的转录本在豹猫6个组织中的表达量(FPKM值),并据此构建热图(Trapnell et al., 2010)。使用RSEM(Li & Dewey,2011)对组装的转录本进行表达定量。该过程需要调用Bowtie(Langmead,2010)将有效的clean reads回比到所有组装的转录本上。然后再由RSEM统计比对结果,统计出回比到每个转录本上的reads数目,并将该值转换为FPKM值。然后通过计算每个转录本的组织特异性指数(tissue-specific index,TSI)评估其表达的组织特异性: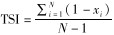
共获得171 370 884条150 bp的raw reads,共计51.4 Gb。质控过滤后得到154 489 126条(90.15%) clean reads,共计45.42 Gb,每个组织的数据量为5.54~13.46 Gb(表 1),表明测序数据质量符合要求。
| 样本 Sample |
原始序列数 Total raw reads |
原始序列碱基数 Total raw bases/Gb |
高质量序列数 Total clean reads |
高质量序列碱基数 Total clean bases/Gb(比例Ratio/%) |
| 肾脏 | 48 905 210 | 14.67 | 45 788 190 | 13.46 (91.75) |
| 肺 | 27 943 121 | 8.38 | 24 903 964 | 7.32 (87.34) |
| 大脑 | 25 113 222 | 7.53 | 22 631 186 | 6.65 (88.31) |
| 骨骼肌 | 21 477 196 | 6.44 | 18 849 518 | 5.54 (86.01) |
| 心脏 | 23 169 272 | 6.95 | 20 809 385 | 6.12 (88.02) |
| 肝脏 | 24 762 863 | 7.43 | 21 506 883 | 6.32 (85.11) |
使用Trinity将clean reads进行de novo组装,最终得到369 246条转录本,转录本长度301~31 696 bp,平均长度1 465 bp,Contig N50为2 660 bp(表 2)。拼接的序列中,155 985条(42.24%)序列长度在1 000 bp以上,组装质量较高。大多数转录本长度为300~400 bp和1 000~2 000 bp(图 1)。使用Cd-hit-est对Trinity组装的转录本去冗余,最终得到269 412条非冗余转录本用于注释分析。
| 项目Item | 数量Number |
| 原始序列总数 | 171 370 884 |
| 序列长度/bp | 150 |
| 高质量序列总数 | 154 489 126 |
| 组装所得contigs的总数 | 369 246 |
| Contigs的总长度/bp | 540 942 684 |
| Contigs的平均长度/bp | 1 465 |
| Contigs(≥1 000 bp)的数量 | 155 985 |
| Contigs(≥1 000 bp)占比/% | 42.24 |
| Contigs的最大长度/bp | 31 696 |
| 所有Contigs长度的中位数/bp | 301 |
| GC含量/% | 49.89 |
| Contig N50/bp | 2 660 |
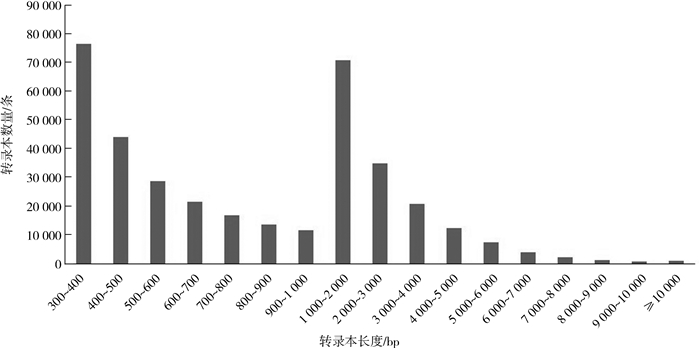
|
| 图 1 转录本的长度分布 Fig. 1 Length distribution of transcripts |
| |
使用blastx将去冗余后的转录本与NR、Swiss-Prot、KOG和KEGG 4个公共数据进行同源性比对注释。NR蛋白数据库注释到转录本110 997条(41.13%);Swiss-Prot蛋白数据库注释到转录本98 192条(36.39%);KOG数据库注释到转录本97 616条(36.17%);KEGG数据库获得注释信息转录本65 895条(24.42%)。4个数据库注释结果的韦恩图显示,共注释到转录本114 517条(42.44%),63 860条转录本在所有数据库中都有注释(图 2:A)。
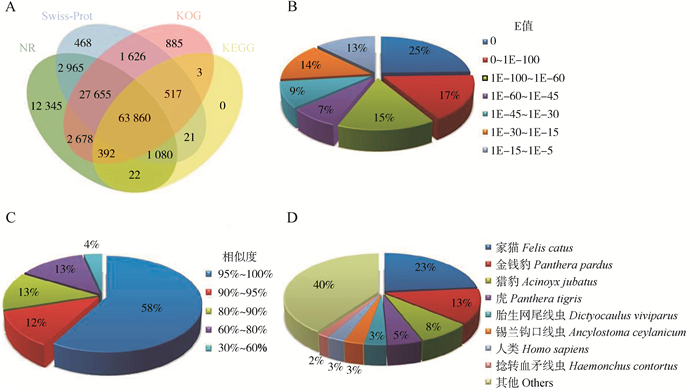
|
| 图 2 转录本与NR、Swiss-Prot、KOG和KEGG数据库同源性比对注释结果及与NR数据库比对的E值、相似度、物种分布 Fig. 2 Characteristics of homology search of transcripts against the NR, Swiss-Pro, KOG and KEGG database and the E value, similarity and species distribution by blast searches against the NR database A. 4个数据库比对结果的韦恩图, B. 与NR数据库比对结果的E值分布, C. 与NR数据库比对结果的同源相似度分布, D. 与NR数据库比对结果的物种分布 A. Venn diagram illustrating distribution of high-score matches among four public databases, B.distribution of E-value, C. distribution of similarity, D. distribution of top hit species |
| |
NR数据库比对表明,73%的序列有较强的同源性(E值≤1.0E-30),其中,25%的序列比对E值为0,39%的为0~1.0E-45,9%的为1.0E-45~1.0E-30(图 2:B)。相似度分布显示,83%的序列比对相似度≥80%,仅有17%的为30%~80%(图 2:C)。物种间比对结果显示,豹猫的转录本与猫科动物的相似性高,约49%的序列与猫科动物基因组(家猫:23%,金钱豹Panthera pardus:13%,猎豹Acinoyx jubatus:8%,虎Panthera tigris:5%)己经注释的基因同源(图 2:D)。
转录本的GO富集显示,33 262条转录本注释到247 376个GO功能条目(图 3:A),包括生物过程(113 399条,45.84%)、分子功能(37 773条,15.27%)和细胞组分(96 204条,38.89%)。生物过程中,细胞过程(20 431条)、代谢过程(16 659条)和生物调节(12 227条)最多;细胞组分中,细胞(18 428条)、细胞部分(18 261条)和细胞器(13 859条)最丰富;分子功能中,最丰富的为连接(18 061条)和催化活性(11 917条)。
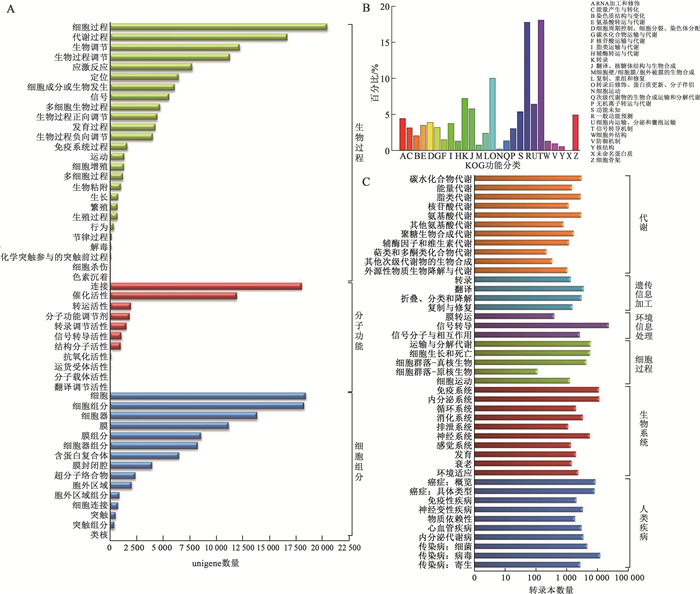
|
| 图 3 豹猫转录本功能注释结果 Fig. 3 The functional annotation results of transcripts of Prionailurus bengalensis A. GO功能条目分类分布, B. 转录本KOG功能分类, C. 转录本分配到KEGG通路6大一级分类中子分类的情况 A. GO functional classification, B. KOG function classification of transcripts, C. number of transcripts assigned to each subcategory of the 6 top KO categories |
| |
根据同源性比对KOG数据库,97 616条(36.17%)转录本分别在25种类别中被注释到(E值≤1E-5)(图 3:B),信号转导机制和一般功能预测的转录本最多,其次是翻译后修饰、蛋白转换、分子伴侣和转录。
KEGG注释结果显示有65 895条(24.42%)转录本被分配到386个KEGG通路上,其中与人类疾病相关的转录本(26 761条)最多,其次是生物系统(22 997条)(图 3:C)。在二级分类中,参与信号转导的转录本(10 039条)和传染病:病毒(10 003条)的转录本数量最多。
2.4 转录本的组织特异性表达转录本TSI值的频数分布图显示(图 4:A),仅17条转录本TSI值<0.15,它们在所有6个组织中表达差异很小,为低组织特异性基因,其中可能包括了一些重要的管家基因;而39.65%(66 763条) 的转录本TSI值在0.15~0.85之间,为中等组织特异性基因,它们的表达量在各组织中存在差异,TSI值越大差异越大;60.34%的转录本TSI值>0.85,为高组织特异性基因,其在部分组织中的表达与其他组织存在明显差异,其中24.5%(41 354条)的转录本TSI值达到1.0,它们严格地在特定的某一种组织中表达。由表达量热图(图 4:B)可见,这3类转录本在各组织中的表达模式不同。

|
| 图 4 转录本的组织特异性 Fig. 4 Tissue-specificities of transcripts A. 表达转录本的TSI值分布情况, B. 6个组织的转录本表达谱: LS. 低组织特异性基因, IS. 中等组织特异性基因, HS. 高组织特异性基因 A. the distribution of TSI values of expressed transcripts, B. transcript expression profiles of 6 tissues: LS. low tissue-specific gene, IS. intermediate tissue-specific gene, HS. high tissue-specific gene |
| |
为了解这些组织特异表达基因是否为各组织高表达基因,统计了6个组织中表达量最高的10条转录本(表 3),共包括39条转录本,其TSI值为0.48~1.0(平均0.83)。其中骨骼肌中10条转录本FPKM值均超过10 000,而脑组织中9条转录本FPKM值为1 213.09~3 074.01,仅1条>10 000,显示不同组织中基因表达量差异较大。这些转录本中,6条转录本(T_90962_c0_g2_i7、T_92170_c4_g8_i1、T_92170_c4_g7_i1、T_90443_c2_g7_i2、T_92619_c3_g1_i2和T_87893_c2_g3_i5)在骨骼肌和心脏中高表达,4条转录本(T_90962_c0_g2_i7、T_84299_c4_g5_i2、T_92588_c6_g2_i6和T_92288_c9_g4_i1)在肾脏和肝脏中高表达。39条转录本中26条(61.5%)为高组织特异性表达,包括8条在骨骼肌中高表达,7条在肺中高表达,6条在肝脏中高表达,肾脏、心脏、大脑中高表达的分别有5条、4条、3条;其他13个高表达转录本的组织特异性较低,如COX1在除肺以外的5个组织中都高表达。
| 样本 Sample |
转录本ID Transcript ID |
丰度 Abundance (FPKM) |
基因 Gene |
长度 Length/bp |
TSI值 TSI value |
| 肺 | T_89689_c2_g2_i10 | 13 775.78 | TMSB4 | 732 | 0.874 3 |
| T_82863_c0_g1_i3 | 6 480.79 | LOC109258830 | 781 | 0.999 9 | |
| T_92576_c5_g2_i1 | 5 489.70 | IgG1H | 750 | 0.948 1 | |
| T_89153_c3_g1_i1 | 3 929.31 | RPL34L | 379 | 0.645 2 | |
| T_86499_c1_g5_i3 | 3 690.71 | LOC109511281 | 851 | 0.821 2 | |
| T_90304_c1_g6_i2 | 3 428.04 | LOC102951493 | 448 | 0.923 0 | |
| T_83050_c3_g3_i1 | 3 303.70 | SFTPCX | 903 | 0.999 9 | |
| T_91428_c4_g2_i1 | 3 194.29 | LOC109497182 | 576 | 0.965 6 | |
| T_90023_c3_g1_i9 | 3 074.72 | HP | 2 061 | 0.881 6 | |
| T_92325_c4_g4_i3 | 3 012.00 | NA | 470 | 0.637 8 | |
| 肝脏 | T_90962_c0_g2_i7 | 16 218.81 | COX1 | 2 069 | 0.639 5 |
| T_85172_c3_g1_i5 | 14 931.79 | TTR | 494 | 0.983 7 | |
| T_84299_c4_g5_i2 | 6 814.43 | NR1I3 | 504 | 0.935 8 | |
| T_84293_c4_g1_i11 | 6 120.06 | RBP4 | 1 197 | 0.998 7 | |
| T_90023_c3_g1_i9 | 5 200.91 | HP | 2 061 | 0.881 6 | |
| T_92588_c6_g2_i6 | 5 005.22 | FTL | 1 337 | 0.799 8 | |
| T_84293_c4_g1_i8 | 4 421.93 | RBP4 | 1 436 | 0.999 3 | |
| T_89918_c3_g5_i3 | 3 485.62 | LOC101085148 | 550 | 0.995 8 | |
| T_90962_c1_g1_i4 | 3 260.37 | ND2 | 1 458 | 0.631 8 | |
| T_92288_c9_g4_i1 | 3 052.85 | COX2 | 811 | 0.642 6 | |
| 骨骼肌 | T_92170_c4_g8_i1 | 82 786.78 | NA | 379 | 0.939 4 |
| T_92170_c4_g7_i1 | 38 237.95 | ACTA1X | 777 | 0.978 6 | |
| T_90443_c2_g7_i2 | 38 227.76 | Ckm | 591 | 0.938 7 | |
| T_92619_c3_g1_i2 | 21 387.36 | CKMX | 1 308 | 0.949 2 | |
| T_91396_c6_g4_i5 | 18 453.34 | LOC102728128 | 787 | 1.000 0 | |
| T_90962_c0_g2_i7 | 17 374.92 | COX1 | 2 069 | 0.639 5 | |
| T_87394_c3_g1_i14 | 17 256.56 | ALDOAX | 680 | 0.996 8 | |
| T_84600_c3_g1_i1 | 12 153.30 | TNNC | 895 | 0.986 2 | |
| T_89879_c2_g3_i2 | 10 962.39 | TNNI | 558 | 0.997 6 | |
| T_87893_c2_g3_i5 | 10 528.89 | LOC109522138 | 916 | 0.825 7 | |
| 肾脏 | T_90962_c0_g2_i7 | 32 318.95 | COX1 | 2 069 | 0.639 5 |
| T_92675_c6_g1_i1 | 25 523.12 | NA | 1 491 | 0.956 2 | |
| T_92423_c0_g1_i12 | 15 951.09 | NA | 2 375 | 0.953 5 | |
| T_85227_c5_g1_i3 | 4 399.20 | OR7A17 | 1 174 | 0.521 3 | |
| T_92288_c9_g4_i1 | 4 075.73 | COX2 | 811 | 0.642 6 | |
| T_90030_c6_g1_i7 | 3 764.74 | LOC111557726 | 449 | 0.942 6 | |
| T_86564_c3_g2_i1 | 3 254.33 | LOC103017900 | 359 | 0.830 7 | |
| T_92588_c6_g2_i6 | 2 908.25 | FTL | 1 337 | 0.799 8 | |
| T_84299_c4_g5_i2 | 2 182.56 | NR1I3 | 504 | 0.935 8 | |
| T_89689_c2_g2_i10 | 2 130.08 | TMSB4 | 732 | 0.874 3 | |
| 心脏 | T_90962_c0_g2_i7 | 46 058.15 | COX1 | 2 069 | 0.639 5 |
| T_92170_c4_g8_i1 | 25 000.69 | NA | 379 | 0.939 4 | |
| T_87893_c2_g3_i5 | 12 082.28 | LOC109522138 | 916 | 0.825 7 | |
| T_90443_c2_g7_i2 | 11 701.42 | Ckm | 591 | 0.938 7 | |
| T_90962_c1_g1_i4 | 5 730.01 | ND2 | 1 458 | 0.631 8 | |
| T_92619_c3_g1_i2 | 5 421.07 | CKMX | 1 308 | 0.949 2 | |
| T_92170_c4_g7_i1 | 4 062.10 | ACTA1X | 777 | 0.978 6 | |
| T_85227_c5_g1_i3 | 3 877.12 | OR7A17 | 1 174 | 0.521 3 | |
| T_89993_c4_g4_i3 | 3 417.21 | MB | 497 | 0.848 0 | |
| T_92213_c4_g1_i3 | 2 300.82 | MYL3 | 464 | 0.845 7 | |
| 大脑 | T_90962_c0_g2_i7 | 14 575.18 | COX1 | 2 069 | 0.639 5 |
| T_85227_c5_g1_i3 | 3 074.01 | OR7A17 | 1 174 | 0.521 3 | |
| T_89689_c2_g2_i10 | 2 651.83 | TMSB4 | 732 | 0.874 3 | |
| T_90962_c1_g1_i4 | 2 611.93 | ND2 | 1 458 | 0.631 8 | |
| T_86236_c0_g1_i1 | 1 985.44 | PCP4 | 529 | 0.999 4 | |
| T_92325_c4_g4_i3 | 1 844.16 | NA | 470 | 0.637 8 | |
| T_89153_c3_g1_i1 | 1 547.20 | RPL34L | 379 | 0.645 2 | |
| T_83171_c3_g1_i3 | 1 380.63 | CST3 | 965 | 0.890 9 | |
| T_87640_c1_g2_i2 | 1 359.53 | RPS12 | 490 | 0.479 1 | |
| T_87056_c11_g1_i6 | 1 213.09 | CALM | 1 133 | 0.800 6 |
作为一种分布广泛的小型猫科动物,过去关于豹猫的研究大多关注其种群分布、遗传多样性水平和系统发育关系等(Mukherjee et al., 2010;Patel et al., 2017)。Ito等(2020)基于基因组SNP分析了豹猫遗传多样性,Bredemeyer等(2021)报道了豹猫的全基因组,但迄今为止关于豹猫基因表达调控相关研究仍非常缺乏。本研究采用RNA-seq对豹猫6个组织的转录组进行了测序、从头组装、功能注释及不同组织的基因表达谱分析,从而为丰富豹猫的遗传资源、辅助基因组注释、开发分子标记等提供了重要数据,也对于理解豹猫的基因表达调控,开展保护遗传学研究奠定了基础。
本研究基于豹猫6个组织的大量转录本从头组装了豹猫转录组,能更全面地体现豹猫基因表达情况,组装质量良好:N50长度为2 660 bp,42.24%的转录本长度超过1 000 bp。研究显示,越长的转录本序列越完整,组装质量越高,更容易获得较多关于基因的信息(Meng et al., 2015)。与发表的东北虎Panthera tigris altaica肺转录组(319 bp)和6个组织合并转录本(332 bp)(Lu et al., 2016)、小尾寒羊和杜泊羊骨骼肌转录组(735 bp和706 bp)(张春兰,2016)、梭子蟹Portunus trituberculatus雌性(954 bp)和雄性(823 bp)(Wang et al., 2018)转录组序列平均长度相比,豹猫转录组质量更高。GO、KOG和KEGG注释结果显示出豹猫各组织表达不同生理功能相关的多种基因。仅有42.44%(114 517条)的豹猫转录本具有注释信息,超过50%的转录本无法比对到现有数据库,这可能是由于豹猫的基因组和蛋白质相关信息仍不完善,大量转录本可能是豹猫特有的转录本,深入研究这些新转录本可揭示豹猫独特的生理、遗传特征。
豹猫6个组织的TSI分布显示,TSI<0.15的管家基因最少,仅17条(0.01%),60.34%的转录本TSI>0.85,其表达具有高度组织特异性。这显示尽管共有同一个基因组,但豹猫在不同组织中的基因转录和表达却存在很大差异,以保障不同组织执行不同的生理功能。Su等(2002)统计老鼠45个正常组织、Yanni等(2005)统计人类Homo sapiens 12种组织的TSI也显示,TSI值为0.9~1.0的高组织特异性表达基因最多,这与本研究结果一致。但不同的是,人类中57%、老鼠中56%的基因都属于中等组织特异性表达,即0.15≤TSI≤0.85。这可能是由于转录本注释质量的差异。人和老鼠具有高质量基因组,转录本注释完善,而豹猫缺乏相关的基因组和蛋白质信息,本研究从头组装的转录本高达26万余条,许多转录本并非全长基因,存在大量难以注释的短序列。
为进一步了解这些组织特异性表达的基因是否也是高表达的基因,统计了豹猫每个组织中表达量最高的前10条转录本。共计39个高表达的基因中,26个都属于在特定组织高表达的基因,这些基因都是与该组织特定功能密切相关的重要基因。如肝脏中特异高表达的RBP4与肝脏的脂肪降解功能相关,人的多组织基因表达图谱也显示该基因在肝脏和脂肪中特异表达(Fagerberg et al., 2014)。肺中特异高表达的SFTPC编码肺表面活性物质蛋白C,它通过降低覆盖肺部的液体的表面张力来维持肺组织的稳定性,SFTPC突变能引发婴幼儿间质性肺病,其过表达可抑制肺癌细胞的增殖(Hayasaka et al., 2018;Li et al., 2019)。大脑中特异高表达的PCP4与神经元的功能相关,小鼠胚胎发生过程中PCP4的过表达诱导神经元提前分化成熟,而在成年期过表达会导致小鼠学习障碍(Mouton-Liger et al., 2014;Renelt et al., 2014)。此外我们还鉴定了不同组织共同高表达的基因。如COX1编码细胞色素c氧化酶Ⅰ,是线粒体呼吸链的末端组分,该酶可将电子从还原的细胞色素c转移到分子氧,并有助于维持线粒体内膜的电化学梯度(Baklouti-Gargouri et al., 2013),为ATP合成酶合成ATP提供电化学梯度(Dennerlein & Rehling,2015),是细胞呼吸过程中重要的功能基因。COX1在心脏、肝脏、肾脏、大脑和骨骼肌中都是表达量前10的基因,在肺中的表达量也相对较高,证明该基因在各个组织中均发挥重要生理功能。TMSB4是一种广泛分布于真核细胞中的多功能肽,与细胞的移动性、分化能力及细胞分裂密切相关(Kuzan,2016)。TMSB4是肺、肾脏和大脑组织中共同高表达的基因,Du等(2015)发现其在大熊猫血液中也是高表达的基因之一,它可能也是维持细胞基本生理功能的重要基因。心脏和骨骼肌都属于肌肉组织,具有收缩性,本研究还鉴定到6条仅在心脏和骨骼肌中高表达转录本,这些转录本可能和肌肉组织的收缩功能密切相关。之前人类不同组织的基因表达谱研究也显示心脏和骨骼肌的表达谱相似性高(Shmueli et al., 2003)。此外肾脏和肝脏组织共同高表达的转录本有4条,这可能源于肝脏和肾脏之间有密切相关的生理功能。
白素英, 徐艳春, 周冬良, 等. 2004. 中国豹猫6个群体的RAPD分析[J]. 东北林业大学学报, 32: 52-54. |
汪松. 1998. 中国濒危动物红皮书兽类[M]. 北京: 科学出版社.
|
张春兰. 2016. 绵羊骨骼肌转录组高通量测序从头组装和特征分析[J]. 潍坊学院学报, 16(2): 6-10. DOI:10.3969/j.issn.1671-4288.2016.02.002 |
张淑云, 王玉玺, 吴玉琴. 1988. 豹猫消化系统解剖[J]. 野生动物学报, 25: 44. |
Baklouti-Gargourui S, Ghorbel M, Ben Mahmoud A, et al. 2013. A novel m.6307 A > G mutation in the mitochondrial COXI gene in asthenozoospermic infertile men[J]. Molecular Reproduction and Development, 80: 581-587. DOI:10.1002/mrd.22197 |
Bredemeyer KR, Harris AJ, Li G, et al. 2021. Ultracontinuous single haplotype genome assemblies for the domestic cat (Felis catus) and Asian leopard cat (Prionailurus bengalensis)[J]. Journal of Heredity, 112(2): 165-173. DOI:10.1093/jhered/esaa057 |
Camacho C, Coulouris G, Avagyan V, et al. 2009. BLAST+: architecture and applications[J/OL]. BMC Bioinformatics, 10: 421[2021-04-02]. https://doi.org/10.1186/1471-2105-10-421.
|
Conesa A, Götz S, García-Gómez JM, et al. 2005. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research[J/OL]. Bioinformatics, 21: 3674-3676[2021-04-02]. https://doi.org/10.1242/jcs.161729.
|
Dennerlein S, Rehling P. 2015. Human mitochondrial COX1 assembly into cytochrome c oxidase at a glance[J]. Journal of Cell Science, 128: 833. |
Du L, Li W, Fan Z, et al. 2015. First insights into the giant panda (Ailuropoda melanoleuca) blood transcriptome: a resource for novel gene loci and immunogenetics[J]. Molecular Ecology Resources, 15: 1001-1013. DOI:10.1111/1755-0998.12367 |
Fagerberg L, Hallström BM, Oksvold P, et al. 2014. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics[J]. Molecular & Cellular Proteomics, 13: 397-406. |
Feng C, Chen M, Xu CJ, et al. 2012. Transcriptomic analysis of Chinese bayberry (Myrica rubra) fruit development and ripening using RNA-Seq[J/OL]. BMC Genomics, 13: 19[2021-03-10]. https://www.biomedcentral.com/1471-2164/13/19.
|
Haas BJ, Papanicolaou A, Yassour M, et al. 2013. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis[J]. Nature Protocols, 8: 1494-1512. DOI:10.1038/nprot.2013.084 |
Hayasaka I, Cho K, Akimoto T, et al. 2018. Genetic basis for childhood interstitial lung disease among Japanese infants and children[J]. Pediatric Research, 83: 477-483. DOI:10.1038/pr.2017.217 |
Hughes GR. 1998. Wild cats-status survey and conservation action plan[J]. Biodiversity & Conservation, 7: 842-844. |
Ito H, Nakajima N, Onuma M, et al. 2020. Genetic diversity and genetic structure of the wild tsushima leopard cat from genome-wide analysis[J]. Animals, 10: 1375-1387. DOI:10.3390/ani10081375 |
Ko BJ, An J, Seomun H, et al. 2018. Microsatellite DNA analysis reveals lower than expected genetic diversity in the threatened leopard cat (Prionailurus bengalensis) in South Korea[J]. Genes & Genomics, 40: 521-530. |
Kuzan A. 2016. Thymosin β as an actin-binding protein with a variety of functions[J]. Advances in Clinical and Experimental Medicine, 25: 1331-1336. DOI:10.17219/acem/32026 |
Langmead B. 2010. Aligning short sequencing reads with bowtie[J]. Current Protocols in Bioinformatics, 32: 11.17.11-11.17.14. |
Li B, Dewey CN. 2011. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome[J/OL]. BMC Bioinformatics, 12: 323(2011-08-04) [2021-04-02]. https://doi.org/10.1186/1471-2105-12-323.
|
Li B, Meng YQ, Li Z, et al. 2019. MiR-629-3p-induced downregulation of SFTPC promotes cell proliferation and predicts poor survival in lung adenocarcinoma[J]. Artificial Cells, Nanomedicine, and Biotechnology, 47: 3286-3296. DOI:10.1080/21691401.2019.1648283 |
Li W, Godzik A. 2006. Cd-hit: a fast program for clustering and comparing large sets of protein or nucleotide sequences[J]. Bioinformatics, 22: 1658-1659. DOI:10.1093/bioinformatics/btl158 |
Lu T, Sun Y, Ma Q, et al. 2016. De novo transcriptomic analysis and development of EST-SSR markers in the Siberian tiger (Panthera tigris altaica)[J]. Molecular Genetics and Genomics, 291: 2145-2157. DOI:10.1007/s00438-016-1246-4 |
Masuda R, Yoshida MC, Shinyashiki F, et al. 1994. Molecular phylogenetic status of the iriomote cat Felis iriomotensis, inferred from mitochondrial DNA sequence analysis[J]. Zoology, 11: 597-604. |
Masuda R, Yoshida MC. 1995. Two Japanese wildcats, the tsushima cat and the iriomote cat, show the same mitochondrial DNA lineage as the leopard cat Felis bengalensis[J]. Zoological Science, 12: 655-659. DOI:10.2108/zsj.12.655 |
Meng XL, Liu P, Jia FL, et al. 2015. De novo transcriptome analysis of Portunus trituberculatus ovary and testis by rna-seq: identification of genes involved in gonadal development[J/OL]. PLoS ONE, 10(6): e0128659(2015-06-04)[2021-04-02]. https://doi.org/10.1371/journal.pone.0128659.
|
Moriya Y, Itoh M, Okuda S, et al. 2007. KAAS: an automatic genome annotation and pathway reconstruction server[J]. Nucleic Acids Research, 35: 182-185. DOI:10.1093/nar/gkm321 |
Mouton-Liger F, Sahún I, Collin T, et al. 2014. Developmental molecular and functional cerebellar alterations induced by PCP4/PEP19 overexpression: implications for down syndrome[J]. Neurobiology of Disease, 63: 92-106. DOI:10.1016/j.nbd.2013.11.016 |
Mukherjee S, Krishnan A, Tamma K, et al. 2010. Ecology driving genetic variation: a comparative phylogeography of jungle cat (Felis chaus) and leopard cat (Prionailurus bengalensis) in India[J/OL]. PLoS ONE, 5(10): e13724(2010-02-29)[2021-04-02]. https://doi.org/10.1371/journal.pone.0013724.
|
Patel RP, Wutke S, Lenz D, et al. 2017. Genetic structure and phylogeography of the leopard cat (Prionailurus bengalensis) inferred from mitochondrial genomes[J]. Journal of Heredity, 108(4): 349-360. DOI:10.1093/jhered/esx017 |
Renelt M, Von Bohlen Und Halbach V, Von Bohlen Und Halbach O. 2014. Distribution of PCP4 protein in the forebrain of adult mice[J]. Acta Histochemica, 116: 1056-1061. DOI:10.1016/j.acthis.2014.04.012 |
Ross J, Brodie J, Cheyne S, et al. 2015. Prionailurus bengalensis[DB/OL]. The IUCN Red List of Threatened Species 2015: e. T18146A50661611. (2014-05-20)[2021-04-02]. https://dx.doi.org/10.2305/IUCN.UK.2015-4.RLTS.T18146A50661611.en.
|
Saka T, Nishita Y, Masuda R. 2018. Low genetic variation in the MHC class Ⅱ DRB gene and MHC-linked microsatellites in endangered island populations of the leopard cat (Prionailurus bengalensis) in Japan[J]. Immunogenetics, 70: 115-124. DOI:10.1007/s00251-017-1020-8 |
Shmueli O, Horn-Saban S, Chalifa-Caspi V, et al. 2003. GeneNote: whole genome expression profiles in normal human tissues[J]. Comptes Rendus Biologies, 326: 1067-1072. DOI:10.1016/j.crvi.2003.09.012 |
Sunquist M, Sunquist F. 2002. Wild cats of the world[M]. Chicago: University of Chicago Press.
|
Spradling KD, Glenn JP, Garcia R, et al. 2013. The baboon kidney transcriptome: analysis of transcript sequence, splice variants, and abundance[J/OL]. PLoS ONE, 8(4): e57563(2013-04-23)[2021-04-02]. https://doi.org/10.1371/journal.pone.0057563.
|
Su AI, Cooke MP, Ching KA, et al. 2002. Large-scale analysis of the human and mouse transcriptomes[J]. Proceedings of the National Academy of Sciences, 99(7): 4465-4470. DOI:10.1073/pnas.012025199 |
Suzuki H, Hosoda T, Sakurai S, et al. 1994. Phylogenetic relationship between the Iriomote cat and the leopard cat, Felis bengalensis, based on the ribosomal DNA[J]. The Japanese Journal of Genetics, 69: 397-406. DOI:10.1266/jjg.69.397 |
Trapnell C, Williams B, Pertea G, et al. 2010. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 28: 511-515. DOI:10.1038/nbt.1621 |
Wang W, Kirkness EF. 2005. Short interspersed elements (SINEs) are a major source of canine genomic diversity[J]. Genome Research, 15: 1798-1808. DOI:10.1101/gr.3765505 |
Wang Z, Sun L, Guan W, et al. 2018. De novo transcriptome sequencing and analysis of male and female swimming crab (Portunus trituberculatus) reproductive systems during mating embrace (stage Ⅱ)[J/OL]. BMC Genetics, 19: 3(2018-01-03)[2021-04-02]. https://doi.org/10.1186/s12863-017-0592-5.
|
Xiong MY, Shao XN, Long Y, et al. 2016. Molecular analysis of vertebrates and plants in scats of leopard cats (Prionailurus bengalensis) in southwest China[J]. Journal of Mammalogy, 97: 1054-1064. DOI:10.1093/jmammal/gyw061 |
Yanai I, Benjamin H, Shmoish M, et al. 2005. Genome-wide midrange transcription profiles reveal expression level relationships in human tissue specification[J]. Bioinformatics, 21: 650-659. DOI:10.1093/bioinformatics/bti042 |
Ye J, Fang L, Zheng H, et al. 2006. WEGO: a web tool for plotting GO annotations[J]. Nucleic Acids Research, 34: 293-297. DOI:10.1093/nar/gkl031 |
 2021, Vol. 40
2021, Vol. 40




