扩展功能
文章信息
- 高嘉昕, 俞丹, 刘焕章
- GAO Jiaxin, YU Dan, LIU Huanzhang
- 基于cyt b基因的长江中上游大鳞马口鱼遗传多样性及谱系生物地理学过程分析
- Genetic Diversity and Phylogeography of Opsariichthys macrolepis from the Upper and Middle Yangtze River Based on Cytochrome b Sequences
- 四川动物, 2021, 40(4): 361-373
- Sichuan Journal of Zoology, 2021, 40(4): 361-373
- 10.11984/j.issn.1000-7089.20200391
-
文章历史
- 收稿日期: 2020-10-13
- 接受日期: 2021-04-12
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
大鳞马口鱼Opsariichthys macrolepis Yang et Huang隶属于鲤形目Cypriniformes鲤科Cyprinidae马口鱼属,主要分布于长江上游的干、支流中,在中游支流沅江的上游以及汉江也有分布,是一种喜栖息于流水环境的小型鱼类。杨干荣和黄宏金(1964)依据采自四川、湖北等地的标本描述了1个新种,以下咽齿2行、上下颌平整无显著凹凸的特征将其划分入










长期以来的分类混乱让人们忽视了大鳞马口鱼这一物种的存在,因此目前针对大鳞马口鱼的研究甚少,仅王雪(2019)对赤水河的大鳞马口鱼进行过资源状况、遗传多样性及种群结构方面的研究。大鳞马口鱼主要栖息于水流较急、底质为卵石或砂石的浅滩河段,是山区溪河中重要的物种,在栖居的河段通常具有较大的种群数量,具有重要的生态价值、观赏价值和一定的渔业价值。同时,大鳞马口鱼分布局限于长江中上游地区,对于探讨马口鱼类的物种分化以及生物地理学过程具有重要的意义。
近年来,线粒体DNA被大量应用于分子系统学研究,成为具有代表性的分子标记之一。线粒体细胞色素b(cyt b)基因不仅能有效评估物种和种群的遗传多样性,同时也能敏锐地检测出潜在的种群遗传分化问题(俞丹等,2019)。本研究选取长江中上游干流以及几条主要支流的13个大鳞马口鱼地理种群,对其cyt b基因进行扩增并测序,分析不同地理种群的遗传多样性、种群分化,并对其谱系地理学过程进行探讨,以期为大鳞马口鱼的遗传资源现状及保护工作提供科学依据,并探讨地质环境变化对其种群历史动态的影响。
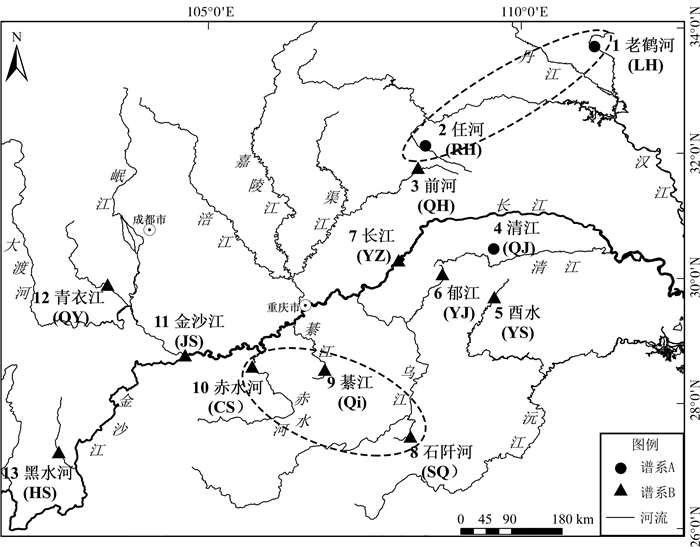
1 材料和方法 1.1 样本采集共选用414尾大鳞马口鱼样本进行分析,其中,新采集样本359尾,体长37.90~125.58 mm;其余55尾为本实验室早年采集样本,其序列已上传至GenBank(Wang et al., 2019)。样本采自河南省三门峡市卢氏县、重庆市城口县、湖北省恩施市宣恩县等13个采样点,涉及长江上游干流及汉江、清江、沅江等9条支流(表 1;图 1)。所有样本均用95%乙醇固定,保存于中国科学院水生生物研究所淡水鱼类博物馆。选取马口鱼Opsariichthys bidens和成都马口鱼Opsariichthys chengtui作为外类群,其中,马口鱼为自测序列,采自湘江,成都马口鱼序列来自GenBank(登录号:KT725244)。
| 样点编号 ID |
种群 Population |
代号 Code |
水系 River basin |
样本数量 Sample size |
体长范围 Standard length range/mm |
单倍型 Haplotype |
单倍型多样性 h |
核苷酸多样性 π |
| 1 | 老鹤河 | LH | 汉江 | 20 | 100.52~124.23 | Hap1~12 | 0.926 3±0.037 8 | 0.051 148±0.028 609 |
| 2 | 任河 | RH | 汉江 | 50 | 80.76~120.10 | Hap1, Hap5, Hap10, Hap13~22 |
0.635 9±0.077 8 | 0.028 052±0.016 453 |
| 3 | 前河 | QH | 嘉陵江 | 17 | 100.56~122.42 | Hap23~32 | 0.875 0±0.070 2 | 0.089 572±0.048 279 |
| 4 | 清江 | QJ | 清江 | 45 | 37.90~91.24 | Hap33~34 | 0.044 4±0.042 0 | 0.000 404±0.001 148 |
| 5 | 酉水 | YS | 沅江 | 44 | 38.11~100.88 | Hap35~36 | 0.045 5±0.042 9 | 0.000 413±0.001 162 |
| 6 | 郁江 | YJ | 乌江 | 35 | 53.67~84.71 | Hap24, Hap37~41 | 0.571 4±0.070 8 | 0.013 345±0.009 262 |
| 7 | 长江* | YZ | 长江 | 11 | — | Hap42~47 | 0.727 3±0.144 4 | 0.041 322±0.024 818 |
| 8 | 石阡河 | SQ | 乌江 | 43 | 84.68~124.37 | Hap24, Hap48~59 | 0.631 2±0.084 2 | 0.009 806±0.007 318 |
| 9 | 綦江* | Qi | 綦江 | 14 | — | Hap24, Hap60 | 0.263 7±0.136 0 | 0.019 181±0.012 757 |
| 10 | 赤水河* | CS | 赤水河 | 30 | — | Hap24, Hap41, Hap48, Hap61~64 |
0.549 4±0.103 4 | 0.022 842±0.014 083 |
| 11 | 金沙江 | JS | 金沙江 | 45 | 111.45~125.58 | Hap24, Hap62, Hap65~71 | 0.854 5±0.023 7 | 0.039 890±0.022 284 |
| 12 | 青衣江 | QY | 岷江 | 30 | 65.52~114.21 | Hap24, Hap67, Hap71~77 | 0.802 3±0.056 9 | 0.026 270±0.015 797 |
| 13 | 黑水河 | HS | 黑水河 | 30 | 70.75~120.76 | Hap78~79 | 0.133 0±0.081 4 | 0.004 837±0.004 614 |
| 合计 | 414 | 37.90~125.58 | Hap1~79 | 0.930 1±0.006 1 | 0.129 421±0.064 299 | |||
| 注: 样点编号: 1. 河南省三门峡市卢氏县, 2. 重庆市城口县巴山镇, 3. 重庆市城口县明通镇, 4. 湖北省恩施市白杨坪乡, 5. 湖北省恩施市宣恩县高罗乡镇, 6. 湖北省利川市忠路镇, 7. 重庆市忠县, 8. 贵州省铜仁市石阡县, 9. 贵州省遵义市松坎镇, 10. 贵州省赤水市, 11. 四川省宜宾市, 12. 四川省眉山市洪雅县, 13. 四川省凉山彝族自治州宁南县松新镇; *引自Wang等(2019)的序列 Notes: ID: 1. Lushi County, Sanmenxia City, Henan Province, 2. Bashan Town, Chengkou County, Chongqing City, 3. Mingtong Town, Chengkou County, Chongqing City, 4. Baiyangping County, Enshi City, Hubei Province, 5.Gaoluo Town, Xuan'en County, Enshi City, Hubei Province, 6. Zhonglu County, Lichuan City, Hubei Province; 7. Zhongxian County, Chongqing City, 8. Shiqian County, Tongren City, Guizhou Province, 9. Songkan County, Zunyi City, Guizhou Province, 10. Chishui City, Guizhou Province, 11. Yibin City, Sichuan Province, 12. Hongya County, Meishan City, Sichuan Province, 13. Songxin Town, Ningnan County, Liangshan Yi Autonomous Prefecture, Sichuan Province; * sequences retrieved from GenBank (Wang et al., 2019) |
||||||||
采用高盐法(Aljanabi & Martinez,1997)进行基因组DNA的提取。cyt b基因扩增和测序引物为L14724(5'-GACTTGAAAAACCACCGTTG-3')和H15915(5'-CTCCGATCTCCGGATTACAAGAC-3')。PCR反应总体积30 μL,包括:10×Buffer 3 μL,dNTPs 1.5 μL(10 mmol·L-1),引物各1 μL(10 μmol·L-1),Taq DNA聚合酶0.5 U。扩增程序为:94 ℃ 5 min;94 ℃ 45 s,54 ℃ 45 s,72 ℃ 1 min,35个循环;72 ℃ 10 min。PCR产物用1%琼脂糖凝胶电泳检测,送上海生工生物公司完成纯化和序列测定。
1.3 序列处理及分析DNA序列的比对使用Clustal X(Thompson et al., 1997),使用默认的参数设置,并在SEAVIEW (Galtier et al., 1996) 中辅以手工校正。使用MEGA 7.0 (Kumar et al., 2016)分析序列中各碱基含量及变异情况,采用Kimura双参数模型计算转换/颠换比率与遗传距离,并根据群体间的遗传距离利用邻接法(Neighbor-joining)构建聚类关系树。
遗传多样性是生物多样性的基础,是物种长期生存和演化的前提,也是评估生物资源现状的重要参数。单倍型多样性(h)和核苷酸多样性(π)是衡量一个物种群体DNA变异程度的重要指标。使用DnaSP 6.12(Rozas et al., 2017)统计种群单倍型多样性及核苷酸多样性信息,并生成单倍型序列。
在jModeltest 2.1.10(Darriba et al., 2012)中使用BIC准则选择最适合的核苷酸替换模型为GTR+I+G。系统发育分析采用最大似然法(Maximum Likelihood,ML)和贝叶斯推断(Bayesian Inferences,BI)。ML法在PhyML(Guindon et al., 2010)中进行,系统树各分支的置信度均采用1 000次自展分析(Bootstrap analysis)重复检测;BI法在MrBayes 3.2(Ronquist et al., 2012)中进行,马尔科夫链蒙特卡洛方法(Markov Chain Monte Carlo,MCMC)设置4条链运行200万代,每1 000代进行1次抽样,独立运行2次。
在BEAST 1.10(Suchard et al., 2018)中估算分化时间,由于缺乏马口鱼类的化石信息,故采用严格分子钟模型,MCMC运行1 000万代,在TreeAnnotator中生成最大谱系置信树(Maximum Clade Credibility Tree,MCC tree),估算结果取平均值及95%最高后验概率密度区间(95% highest posterior density interval,95%HPD),cyt b基因平均替代率采用1%每百万年(Zhang et al., 2008)。
在Arlequin 3.5(Excoffier & Lischer,2010)中利用分子方差分析(analysis of molecular variance,AMOVA)估算种群遗传结构和地理群体遗传变异的分布,计算种群间遗传分化指数(genetic differentiation index, FST)。为获得不同地理种群的最佳分组模式,在SAMOVA 2.0中进行空间分子方差分析(spatial analysis of molecular variance,SAMOVA)(Dupanloup et al., 2002)。对两两种群间的FST和地理距离进行Mantel检验,通过Google Earth计算任意2个采样点间的直线距离,并进行对数变换;在R中利用vegan数据包(Oksanen et al., 2008)计算相关系数r及其置信区间。遗传分化指数可在一定程度上指示种群间基因流和遗传漂变的程度,是反映群体演化历史的重要参数。FST值为0~0.05说明亚群不存在分化, 为0.05~0.15时则存在中度分化, 为0.15~0.25时存在高度分化(Wright,1965)。
在Arlequin 3.5(Tajima, 1989)中进行种群扩张检验,计算错配分布值、Tajima's D和Fu's FS值,并通过公式τ=2ut估算种群扩张发生时间(t为种群扩张以来的时间,u为序列的变异速率;u=2μk,μ为基因序列的突变率,k为分析序列的碱基数,扩张时间T=t×世代时间),进化速率采用1%每百万年。在BEAST 1.10中进行推测种群历史动态的贝叶斯天际线(Bayesian skyline plot,BSP)分析,在Tracer 1.4(Rambaut & Drummond,2007)中构图。
2 结果 2.1 cyt b基因序列变异分析共得到414条大鳞马口鱼的cyt b基因序列,比对后得到序列全长1 140 bp,包含1 030个保守位点和110个变异位点,变异位点中包含86个简约信息位点。所有cyt b序列的转换和颠换均未达到饱和,转换数明显大于颠换数,平均转换/颠换比为8.53。所有序列的碱基含量平均值为:T=29.9%、C=29.1%、A=24.2%、G=16.8%,其中A+T含量(54.1%)高于G+C(45.9%),这与其他鱼类线粒体基因A、T含量高,G、C含量低的特点相似,同时表现出强烈的低G含量的碱基组成偏向性。
2.2 单倍型多样性和核苷酸多样性在414条cyt b基因序列中共检测到79个单倍型,整体单倍型多样性为0.930 1±0.006 1,核苷酸多样性为0.129 421±0.064 299。不同地理种群的单倍型多样性和核苷酸多样性见表 1。从单倍型分布来看,老鹤河(LH,采样点1)与任河(RH,采样点2)种群有共享单倍型Hap1、Hap5和Hap10;前河(QH,采样点3)、郁江(YJ,采样点6)、石阡河(SQ,采样点8)、綦江(QJ,采样点9)、赤水河(CS,采样点10)、金沙江(JS,采样点11)和青衣江(QY,采样点12)有共享单倍型Hap24;郁江(YJ,采样点6)、石阡河(SQ,采样点8)和赤水河(CS,采样点10)有共享单倍型Hap41;赤水河(CS,采样点10)和石阡河(SQ,采样点8)有共享单倍型Hap48;赤水河(CS,采样点10)和金沙江(JS,采样点11)有共享单倍型Hap62;金沙江(JS,采样点11)和青衣江(QY,采样点12)有共享单倍型Hap67和Hap71。每个种群都有一定数量的特有单倍型,特有单倍型共70个。
2.3 单倍型的分化利用ML法和BI法构建大鳞马口鱼79个单倍型的系统发育树(图 2)。结果显示,2种方法得到的系统树拓扑结构基本一致,所有单倍型可分为2个明显的谱系:谱系A和B。其中,谱系A中的单倍型来自老鹤河、任河和清江种群;谱系B中的单倍型来自前河、酉水和郁江等其他10个种群。谱系A包含2个亚谱系,来自汉江(老鹤河、任河)和清江的单倍型各形成1个单系,其中,汉江2个支流种群组成的分支中单倍型彼此混杂;谱系B中包含多个亚谱系,其中,多数种群的单倍型彼此混杂,没有一定的地理格局。各谱系内部支持率普遍较低,且存在多歧分支。

|
| 图 2 基于大鳞马口鱼cyt b单倍型的最大似然树 Fig. 2 Maximum Likelihood tree of Opsariichthys macrolepis based on cyt b haplotypes recovered from 13 populations 节点上方数字代表分化时间(Ma);下方数字代表贝叶斯后验概率(P)和最大似然法的自展支持度(BS);仅显示大于0.5的P值和大于50的BS值 Numbers above nodes represent divergent time (Ma); numbers below nodes correspond to Bayesian posterior probability (P) and Maximum Likelihood bootstrap support values (BS); only P > 0.5 and BS > 50 are shown |
| |
对大鳞马口鱼79个单倍型进行分化时间的估算(图 2)。结果显示,大鳞马口鱼样本的最近共同祖先存在于1.34 Ma (95%HPD:1.03~1.70 Ma),这也是谱系A和B分化的时间。谱系A内部2个亚谱系约在1.10 Ma(95%HPD:0.79~1.46 Ma)开始分化;谱系B内部亚谱系的最近共同祖先存在于距今约1.04 Ma(95%HPD:0.76~1.37 Ma),几个主要亚谱系分化的时间在0.19~0.74 Ma之间。
Mantel检验表明,大鳞马口鱼种群之间遗传分化程度与地理距离不存在显著相关性(r=0.297 6,P=0.069 79),说明距离隔离模型无法很好地解释当前大鳞马口鱼种群遗传格局的成因。可以发现,本研究中部分种群地理距离较近,如任河和前河种群,清江和酉水、郁江种群,但是种群间遗传分化程度却较高;綦江与石阡河、赤水河种群地理距离较远,但却不存在种群分化。这说明地理距离并非影响大鳞马口鱼遗传格局形成的主要因素。
2.5 种群间遗传分化与种群遗传结构对大鳞马口鱼13个种群进行分子方差分析(AMOVA),结果显示遗传变异主要来源于种群间(84.5%,P<0.01);进一步对种群间进行比较分析,结果显示两两群体之间的FST范围为-0.016 24~ 0.998 27,除老鹤河和任河、赤水河和綦江、石阡河和綦江种群外,其他种群之间FST值均大于0.05(0.200 15~0.998 27,P<0.01),其中,清江种群和其他种群之间的FST值最大(0.918 59~0.998 27,P<0.01)。
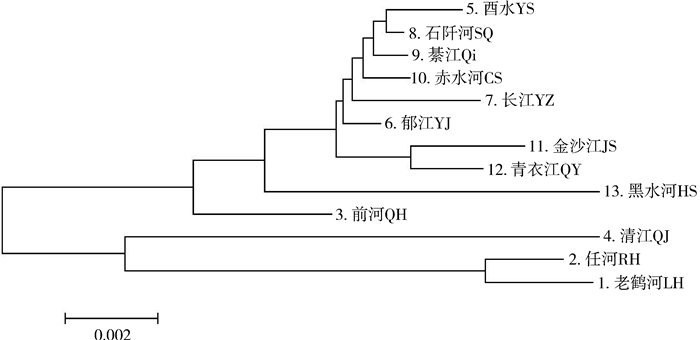
各种群之间遗传距离在0.001~0.025之间(表 2),其中,清江与黑水河种群的遗传距离最大,为0.025;石阡河与綦江种群间的最小,为0.001。利用遗传距离构建大鳞马口鱼13个地理种群的NJ树(图 3),结果显示所有种群聚为2个大分支,汉江支流老鹤河与任河种群聚为一支后再与清江种群聚类,其他长江上游及沅江上游的种群聚为另外一个大分支,与单倍型的分化非常一致。
| 1. 老鹤河LH | 2. 任河 RH |
3. 前河 QH |
4. 清江 QJ |
5. 酉水 YS |
6. 郁江 YJ |
7. 长江 YZ |
8. 石阡河SQ | 9. 綦江 Qi |
10. 赤水河CS | 11. 金沙江YS | 12. 青衣江QY | 13. 黑水河HS | |
| 1 | 0.004 | 0.021 | 0.020 | 0.023 | 0.020 | 0.023 | 0.021 | 0.022 | 0.022 | 0.024 | 0.023 | 0.024 | |
| 2 | 0.053 05 | 0.020 | 0.020 | 0.022 | 0.020 | 0.022 | 0.021 | 0.021 | 0.021 | 0.024 | 0.022 | 0.023 | |
| 3 | 0.669 08** | 0.770 46** | 0.019 | 0.008 | 0.007 | 0.009 | 0.007 | 0.007 | 0.008 | 0.010 | 0.009 | 0.013 | |
| 4 | 0.920 53** | 0.926 02** | 0.861 10** | 0.023 | 0.021 | 0.023 | 0.022 | 0.022 | 0.022 | 0.024 | 0.023 | 0.025 | |
| 5 | 0.928 98** | 0.933 72** | 0.638 74** | 0.998 27** | 0.003 | 0.005 | 0.002 | 0.003 | 0.003 | 0.007 | 0.007 | 0.011 | |
| 6 | 0.866 16** | 0.890 35** | 0.354 45** | 0.970 72** | 0.815 94** | 0.004 | 0.002 | 0.002 | 0.002 | 0.005 | 0.004 | 0.010 | |
| 7 | 0.796 69** | 0.864 54** | 0.291 91** | 0.964 54** | 0.809 16** | 0.443 61** | 0.004 | 0.004 | 0.004 | 0.007 | 0.006 | 0.012 | |
| 8 | 0.891 24** | 0.906 65** | 0.423 05** | 0.977 18** | 0.786 03** | 0.386 20** | 0.486 60** | 0.001 | 0.002 | 0.006 | 0.005 | 0.010 | |
| 9 | 0.828 80** | 0.879 01** | 0.258 10** | 0.978 60** | 0.803 02** | 0.301 36** | 0.275 27** | 0.071 69 | 0.002 | 0.006 | 0.005 | 0.010 | |
| 10 | 0.845 30** | 0.879 73** | 0.320 56** | 0.958 25** | 0.671 43** | 0.272 29** | 0.299 58** | 0.059 60 | -0.016 24 | 0.006 | 0.005 | 0.010 | |
| 11 | 0.821 69** | 0.859 05** | 0.448 51** | 0.918 59** | 0.734 61** | 0.505 45** | 0.408 30** | 0.589 09** | 0.435 13** | 0.436 46** | 0.004 | 0.013 | |
| 12 | 0.843 78** | 0.879 94** | 0.452 32** | 0.954 55** | 0.838 94** | 0.577 53** | 0.448 20** | 0.683 32** | 0.516 82** | 0.522 20** | 0.200 15** | 0.012 | |
| 13 | 0.900 20** | 0.917 35** | 0.702 55** | 0.991 63** | 0.981 51** | 0.906 74** | 0.878 09** | 0.922 68** | 0.907 62** | 0.868 53** | 0.803 95** | 0.868 64** | |
| 注Notes:* P < 0.05,** P < 0.01; 下同the same below; 下划线表示种群间没有显著分化underlines indicate populations without significant difference | |||||||||||||
|
| 图 3 基于种群间遗传距离的大鳞马口鱼13个地理种群NJ树 Fig. 3 Neighbor-joining tree of Opsariichthys macrolepis based on pairwise genetic distances among 13 populations |
| |
为进一步探究大鳞马口鱼种群的遗传结构,对所有大鳞马口鱼种群进行空间分子方差分析(SAMOVA),假定具有最大组间变异(FCT值)的划分方案为最符合自然的群体结构模式。结果显示当所有种群划分为3个组,即老鹤河和任河为一组,清江为一组,其他种群为一组时,得到的FCT值(0.743 247)最大,同时这样划分也同利用NJ法基于遗传距离构建的不同地理种群的聚类关系相吻合,说明该划分可能是最符合自然的群体结构模式。
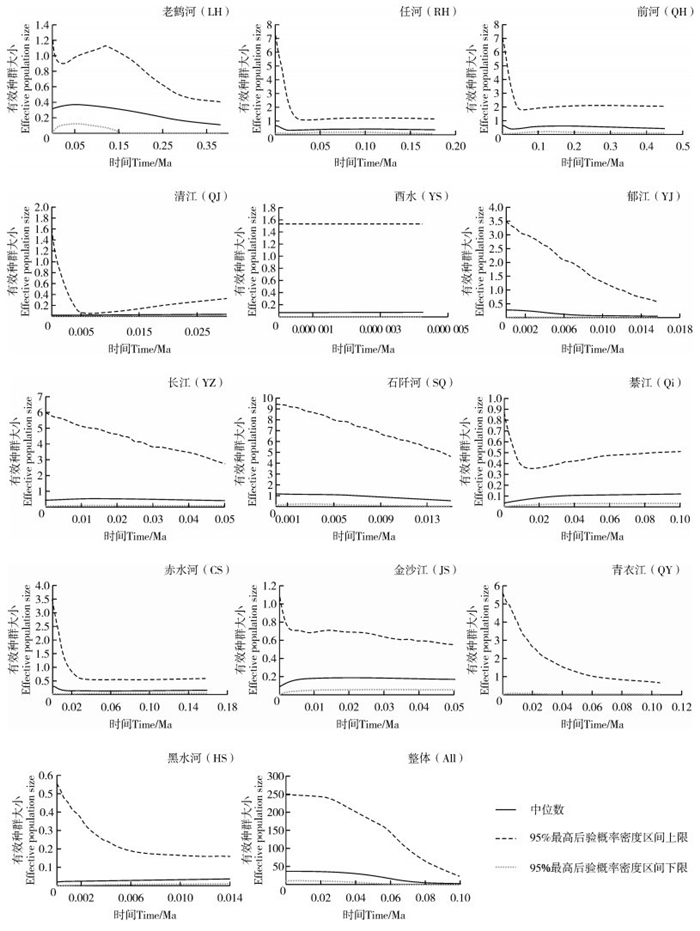
2.6 种群历史动态分析Tajima's D检验显示,任河、石阡河种群检验结果为显著的负值,检测到了种群扩张;而其他种群均符合中性进化的假设,未检测到种群扩张。Fu's FS检验显示除清江、酉水、石阡河种群外,其他种群均未检测到种群扩张。SSD检验结果显示,除前河、长江忠县段、綦江种群以外,其他种群均检测到种群扩张;而Hri检验结果显示,除长江忠县段种群以外,其他种群均检测到种群扩张;根据1%每百万年的突变速率,推算出大鳞马口鱼不同地理种群发生扩张的时间为0.005~0.236 Ma(表 3)。对所有种群进行BSP分析,得到了有效种群大小随溯祖时间变化的动态曲线(图 4)。其中,清江、酉水、长江忠县段及黑水河种群在近期处于相对稳定的状态,没有明显的种群扩张或收缩;青衣江种群在0.04 Ma左右开始扩张;前河、赤水河种群于0.02 Ma左右发生了扩张;任河、郁江、石阡河种群大致于0.01 Ma开始扩张;老鹤河、綦江、金沙江宜宾段种群分别于0.02 Ma、0.02 Ma、0.01 Ma左右经历了种群收缩。由所有个体组成的种群于0.08 Ma以来开始快速扩张,随后处于相对稳定的状态。
| 种群 Population |
Tajima's D (p) |
Fu's FS (p) |
SSD (p) |
Hri (p) |
τ | T/Ma |
| 1. 老鹤河(LH) | -1.015 | -2.184 | 0.024 | 0.088 | 6.891 | 0.151 |
| 2. 任河(RH) | -1.538* | -2.266 | 0.058 | 0.129 | 6.881 | 0.151 |
| 3. 前河(QH) | 0.946 | 0.612 | 0.074** | 0.077 | 17.194 | — |
| 4. 清江(QJ) | -1.113 | -1.548* | 0.000 | 0.832 | 3.000 | 0.066 |
| 5. 酉水(YS) | -1.115 | -1.530* | 0.000 | 0.828 | 3.000 | 0.066 |
| 6. 郁江(YJ) | 0.042 | -0.291 | 0.096 | 0.298 | 4.074 | 0.089 |
| 7. 长江(YZ) | -0.498 | 0.484 | 0.100* | 0.253* | 7.404 | — |
| 8. 石阡河(SQ) | -1.857** | -10.031** | 0.010 | 0.058 | 1.596 | 0.035 |
| 9. 綦江(Qi) | -0.608 | 4.844 | 0.099* | 0.681 | 3.000 | — |
| 10. 赤水河(CS) | -1.133 | 0.276 | 0.042 | 0.120 | 0.218 | 0.006 |
| 11. 金沙江(JS) | 0.871 | 1.701 | 0.054 | 0.068 | 10.578 | 0.236 |
| 12. 青衣江(QY) | -0.966 | -0.815 | 0.031 | 0.076 | 0.229 | 0.005 |
| 13. 黑水河(HS) | -1.238 | 1.745 | 0.026 | 0.787 | 3.000 | 0.066 |
| 合计 | -0.430 | -13.505* | 0.011 | 0.012 | 25.557 | 0.560 |
|
| 图 4 大鳞马口鱼种群动态随时间变化的BSP图 Fig. 4 Bayesian skyline plots (BSP) for population dynamics of Opsariichthys macrolepis with time |
| |
本研究414条大鳞马口鱼cyt b序列中共检测到79个单倍型,其整体单倍型多样性及核苷酸多样性分别为h=0.930 1和π=0.129 421,与台湾的须

Grant和Bowen(1998)依据32种海洋鱼类的线粒体单倍型多样性和核苷酸多样性,推测了种群演化历史的4种模式:当h和π均较低(h<0.5,π<0.5%)时,种群可能经历了瓶颈效应或奠基者效应;当h较高(h>0.5)而π较低(π<0.5%)时,种群可能正在经历瓶颈效应后的快速扩张;当h较低(h<0.5)而π较高(π>0.5%)时,可能由于彼此隔离的种群发生了二次接触,或大而稳定的群体正经历强烈的种群瓶颈;当h和π均较高(h>0.5,π>0.5%)时,则可能由于种群大而稳定并拥有较长的演化历史,亦或是发生了二次接触。
本研究中整体及绝大多数大鳞马口鱼种群均表现出h>0.5且π>0.5%,符合Grant和Bowen(1998)提出的h和π均较高的模式。大鳞马口鱼是其生境中常见的小型鱼类,通常具有较庞大且稳定的种群,而规模较大的群体往往拥有更高的遗传多样性(Frankham,1996)。这种现象也有可能是不同种群间发生二次接触的结果。二次接触造成遗传多样性增加的案例在海洋鱼类(Bay & Caley,2011)、植物(Nettel et al., 2008;Havrdová et al., 2015)中均有报道过。
3.2 长江中上游干支流大鳞马口鱼的种群遗传分化本研究中大鳞马口鱼不同种群间的遗传分化指数范围为-0.016 24~0.998 27,除老鹤河和任河,赤水河和綦江、石阡河和綦江种群不存在种群分化外,其他种群均存在不同程度的分化,说明本研究中大部分大鳞马口鱼种群间缺乏基因交流。这可能与大鳞马口鱼作为一种产沉粘性卵的小型山溪定居型鱼类,主动扩散能力较弱有关,且研究采用的多数样本来源于采样河流上游或源头的小支流,各群体之间距离较远或有山脉等地理屏障阻隔,限制了其扩散或交流。值得注意的是,老鹤河与任河群体,綦江和赤水河、石阡河群体虽相隔较远,却不存在种群分化。推测可能存在近期的地质构造事件,造成了水系的改道或河流袭夺事件的发生,并导致了鱼类的交流。同时,Mantel检验也表明,大鳞马口鱼群体之间的遗传分化程度与地理距离没有显著相关性,这进一步说明了地理距离并不是影响大鳞马口鱼遗传格局形成的主要因素。
3.3 长江中上游干支流大鳞马口鱼的谱系生物地理学过程谱系生物地理学主要依据基因谱系的空间分布,来推测近缘种间或同种种群间谱系发生过程及其历史(Avise,2000)。从大鳞马口鱼的谱系分化情况来看,所有单倍型分化为A和B 2个主要的谱系,其中, 谱系A包含老鹤河、任河和清江的单倍型,谱系B包含前河、酉水、郁江等其他几个种群的单倍型。从地理空间分布来看,谱系A、B的分化体现着东-西谱系分化格局:谱系A的采样点处于相对较东部的位置,涉及长江中游支流汉江、清江;而谱系B则处于相对较西部的位置,主要涉及长江上游干支流。应用分子钟理论估计2个谱系的分化大致发生在早更新世(1.34 Ma,95%HPD:1.03~ 1.70 Ma),在这一时期青藏高原剧烈隆起(李吉均,方小敏,1998)。高原的进一步抬升使西高东低的地势格局更为显著,而大鳞马口鱼作为一种主动扩散能力不强的小型鱼类,可能更容易受到构造运动及水系变动的影响,形成目前的地理分布格局。
值得注意的是,酉水虽隶属于长江中游沅江水系,但却与其他上游种群聚类在谱系B中。酉水等分布较东部的采样点位处我国第二、三级阶梯的过渡地带,这一区域可能拥有较为复杂的地质历史和地质环境,较活跃的地质构造事件可能导致了水系的袭夺变迁,使种群间的交流更加频繁。从遗传距离来看,酉水同乌江水系的关系更近,推测由于水系的变迁,酉水上游可能与乌江水系上游曾经存在某种联系,从而造成现今这种地理分布格局的形成。
本研究中部分种群地理距离较近,如任河和前河种群,清江和酉水、郁江种群,但是它们分别属于不同的水系,而且单倍型也属于不同的谱系:任河与清江属于谱系A,前河与郁江、酉水属于谱系B。除前河(17尾)、郁江(26尾)以外,其他地点的样本数量均≥30尾,但是没有谱系A和B的混杂。同时,在这么多的地点中都表现为没有谱系A和谱系B的混杂。从统计学的理解上说明谱系A和谱系B在目前的地点均不存在混杂。其原因可能是谱系A和谱系B较早出现分化(~1.34 Ma),此后就没有再次接触,从而形成了目前的地理种群结构。这种谱系分化格局与地理空间分布基本一致,即谱系A主要分布在较东部的位置,谱系B主要分布在较西部的位置。
从谱系内部来看,谱系A中2个亚谱系的分化代表了不同水系地理种群的分化,结合其分化时间(~1.10 Ma),推测这次分化事件同样受到了这一时期构造运动的影响。谱系B中亚谱系的分化时间为0.19~1.04 Ma,其单倍型多数没有按照地理分布聚类,且共享单倍型广泛存在,同时多数种群具有较高的单倍型多样性和核苷酸多样性水平,这意味着地质事件可能造成不同地理种群间的分化和交流,特别是这一时期经历了中更新世气候转型期(Mid-Pleistocene transition,0.8~1.2 Ma),冰期-间冰期的更替周期缩短(Chalk et al., 2017),更加频繁的水位、气候波动可能造成不同种群间的隔离或再度交流。已有研究表明,青藏高原的隆升和更新世气候动荡对淡水鱼类的遗传分化具有重要影响(Gao et al., 2012;April et al., 2013;刘思情等,2013;Chen et al., 2018)。据此推测,青藏高原的剧烈隆升以及随之而来的气候环境改变在大鳞马口鱼谱系地理格局的形成中起到了关键作用。
3.4 长江中上游干支流大鳞马口鱼的种群历史动态Tajima's D和Fu's FS是2种常用于推测种群历史动态的检验,通常以检验结果呈显著负值作为种群扩张的标志。相对而言,Fu's FS检验对种群扩张更加敏感(Fu & Li,1993)。本研究中除任河、清江、石阡河和酉水种群外,其他种群的中性检验均没有检测到种群扩张,但是不显著的SSD和Hri检验结果表示不能拒绝种群扩张模型,并估算出不同地理种群发生扩张的时间为0.005~0.236 Ma。中性检验和错配分析结果的不一致可能意味着大鳞马口鱼种群历史相对复杂。
大鳞马口鱼各地理种群的历史动态随时间变化的BSP图显示,多数种群在近期经历了一定程度的扩张。其中青衣江种群扩张时间最早,约0.04 Ma;前河、赤水河种群大致在0.02 Ma扩张;任河、郁江、石阡河种群扩张稍晚,大致在0.01 Ma。可以发现,除前河、赤水河种群外,其他种群扩张的时间刚好与历史上的暖期或间冰期的时间相对应(崔久之等,2011)。同样可以观察到,老鹤河和綦江种群均于0.02 Ma左右经历了种群收缩,这一时期刚好处于末次冰期;而金沙江宜宾段种群在末次冰期后却发生了种群收缩,推测可能与末次冰期后的一次降温事件有关(新仙女木事件,0.011 7~0.012 9 Ma)。据此推测,更新世的冰期更迭可能对大鳞马口鱼的种群历史动态产生一定影响。
3.5 长江中上游大鳞马口鱼的保护建议大鳞马口鱼通常是其栖居河段的优势种(王雪,2019),也是水生生态系统的重要组成部分,对维系整个系统结构的完整和功能的正常发挥起到了重要作用。本研究结果显示,长江中上游干流和部分支流的大鳞马口鱼种群整体遗传多样性较高,种质资源情况比较健康;老鹤河、任河和清江种群与其他种群遗传分化较大,为了防止遗传资源的丧失,在遗传资源保护管理中应作为进化显著单元予以关注和保护。
致谢: 感谢中国科学院水生生物研究所的刘飞、常涛、胡华明在样本采集方面提供的帮助;感谢陈重光先生和卫叔杨先生提供部分样本;感谢中国科学院水生生物研究所的王雪、杨萍、唐瑞在实验及数据分析方面提供的帮助。
陈宜瑜. 1982. 马口鱼类分类的重新整理[J]. 海洋与湖沼, 13(3): 293-299. |
陈义雄, 张咏青. 2005. 台湾淡水鱼类(上卷)(鲤形目)[M]. 台湾: 水产出版社.
|
崔之久, 陈艺鑫, 周力平, 等. 2011. 中国第四纪冰期历史、特征及成因探讨[J]. 第四纪研究, 31(5): 749-764. DOI:10.3969/j.issn.1001-7410.2011.05.01 |
刘思情, 唐琼英, 李小娟, 等. 2013. 基于线粒体细胞色素b基因的黑鳍鳈(Sarcocheilichthys nigripinnis)生物地理学过程分析[J]. 动物学研究, 34(5): 437-445. |
王晨帆. 2005. 台湾须鱲(鲤形目: 鲤科)的亲缘地理及形态变异之研究[D]. 台北: 国立台湾大学动物学研究所.
|
王雪. 2019. 赤水河两种代表性鱼类种群遗传结构和空间生态过程[D]. 武汉: 中国科学院水生生物研究所.
|
杨干荣, 黄宏金. 1964. Ⅰ雅罗鱼亚科[M]//伍献文. 中国鲤科鱼类志(上卷). 上海: 上海科学技术出版社.
|
俞丹, 张智, 张健, 等. 2019. 基于cyt b基因的雅鲁藏布江下游墨脱江段及察隅河墨脱裂腹鱼的遗传多样性及种群历史动态分析[J]. 水生生物学报, 43(5): 923-930. |
李吉均, 方小敏. 1998. 青藏高原隆起与环境变化研究[J]. 科学通报, 43(15): 1568-1574. |
Aljanabi SM, Martinez I. 1997. Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques[J]. Nucleic Acids Research, 25(22): 4692-4693. DOI:10.1093/nar/25.22.4692 |
April J, Hanner RH, Dion-Côté AM, et al. 2013. Glacial cycles as an allopatric speciation pump in north-eastern American freshwater fishes[J]. Molecular Ecology, 22(2): 409-422. DOI:10.1111/mec.12116 |
Ashiwa H, Hosoya K. 1998. Osteology of Zacco pachycephalus, sensu Jordan & Evermann (1903), with special reference to its systematic position[J]. Environmental Biology of Fishes, 52(1-3): 163-171. DOI:10.1023/A:1007480221190 |
Avise JC. 2000. Phylogeography: the history and formation of species[M]. Cambridge: Harvard University Press.
|
Banarescu P. 1968. Revision of the genera Zacco and Opsariichthys (Pisces, Cyprinidae)[J]. Věstník Československé Společnosti Zoologické, 32: 305-311. |
Bay LK, Caley MJ. 2011. Greater genetic diversity in spatially restricted coral reef fishes suggests secondary contact among differentiated lineages[J]. Diversity, 3(3): 483-502. DOI:10.3390/d3030483 |
Chalk TB, Hain MP, Foster GL, et al. 2017. Causes of ice age intensification across the Mid-Pleistocene transition[J]. Proceedings of the National Academy of Sciences, 114(50): 13114-13119. DOI:10.1073/pnas.1702143114 |
Chen IS, Wu JH, Huang SP. 2009. The taxonomy and phylogeny of the cyprinid genus Opsariichthys Bleeker (Teleostei: Cyprinidae) from Taiwan, with description of a new species[J]. Environmental Biology of Fishes, 86: 165-183. DOI:10.1007/s10641-009-9499-y |
Chen W, Schmidt BV, He SP. 2018. The potential colonization histories of Opsariichthys bidens (Cyprinidae) in China using Bayesian binary MCMC analysis[J]. Gene, 676: 1-8. DOI:10.1016/j.gene.2018.07.019 |
Darriba D, Taboada GL, Doallo R, et al. 2012. jModelTest 2:more models, new heuristics and parallel computing[J]. Nature Methods, 9(8): 772. |
Dupanloup I, Schneider S, Excoffier L. 2002. A simulated annealing approach to define the genetic structure of populations[J/OL]. Molecular Ecology, 11(12): 2571-2581(2014-10-09)[2020-09-14]. http://cmpg.unibe.ch/software/samova2/.
|
Excoffier L, Lischer HEL. 2010. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 10(3): 564-567. DOI:10.1111/j.1755-0998.2010.02847.x |
Frankham R. 1996. Relationship of genetic variation to population size in wildlife[J]. Conservation Biology, 10(6): 1500-1508. DOI:10.1046/j.1523-1739.1996.10061500.x |
Fu YX, Li WH. 1993. Statistical tests of neutrality of mutations[J]. Genetics, 133(3): 693-709. DOI:10.1093/genetics/133.3.693 |
Galtier N, Gouy M, Gautier C. 1996. SEAVIEW and PHYLO_WIN: two graphic tools for sequence alignment and molecular phylogeny[J]. Bioinformatics, 12(6): 543-548. DOI:10.1093/bioinformatics/12.6.543 |
Gao Y, Wang SY, Luo J, et al. 2012. Quaternary palaeo environmental oscillations drove the evolution of the Eurasian Carassius auratus complex (Cypriniformes, Cyprinidae)[J]. Journal of Biogeography, 39(12): 2264-2278. DOI:10.1111/j.1365-2699.2012.02755.x |
Grant WAS, Bowen BW. 1998. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
Guindon S, Dufayard JF, Lefort V, et al. 2010. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0[J]. Systematic Biology, 59(3): 307-321. DOI:10.1093/sysbio/syq010 |
Havrdová A, Douda J, Krak K, et al. 2015. Higher genetic diversity in recolonized areas than in refugia of Alnus glutinosa triggered by continent-wide lineage admixture[J]. Molecular Ecology, 24(18): 4759-4777. DOI:10.1111/mec.13348 |
Howes GJ. 1980. The anatomy, phylogeny and classification of the barriliine cyprinid fishes[J]. Bulletin of the British Museum (Natural History) Zoology, 37: 129-198. |
Kumar S, Stecher G, Tamura K. 2016. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
Ma GC, Tsao HS, Lu HP, et al. 2006. AFLPs congruent with morphological differentiation of Asian common minnow Zacco (Pisces: Cyprinidae) in Taiwan[J]. Zoologica Scripta, 35(4): 341-351. DOI:10.1111/j.1463-6409.2006.00232.x |
Nettel A, Dodd RS, Afzal-Rafii Z, et al. 2008. Genetic diversity enhanced by ancient introgression and secondary contact in East Pacific black mangroves[J]. Molecular Ecology, 17(11): 2680-2690. DOI:10.1111/j.1365-294X.2008.03766.x |
Oksanen J, Blanchet F, Friendly M, et al. 2008. R package for community ecologists: popular ordination methods, ecological null models & diversity analysis[DB/OL]. (2020-09-12)[2020-09-14]. https://github.com/vegandevs/vegan.
|
Rambaut A, Drummond AJ. 2007. Tracer v1.4[DB/OL]. (2008-11-29)[2020-09-14]. http://beast.bio.ed.ac.uk/Tracer.
|
Ronquist F, Teslenko M, Van Der Mark P, et al. 2012. MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space[J]. Systematic Biology, 61(3): 539-542. DOI:10.1093/sysbio/sys029 |
Rozas J, Ferrer-Mata A, Sánchez-DelBarrio JC, et al. 2017. DnaSP6:DNA sequence polymorphism analysis of large data sets[J]. Molecular Biology and Evolution, 34(12): 3299-3302. DOI:10.1093/molbev/msx248 |
Suchard MA, Lemey P, Baele G, et al. 2018. Bayesian phylogenetic and phylodynamic data integration using BEAST 1.10[J/OL]. Virus Evolution, 4(1): vey016[2020-09-01]. https://doi.org/10.1093/ve/vey016.
|
Tajima F. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 123(3): 585-595. DOI:10.1093/genetics/123.3.585 |
Thompson JD, Gibson TJ, Plewniak F, et al. 1997. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 25(24): 4876-4882. DOI:10.1093/nar/25.24.4876 |
Wang X, Liu F, Yu D, et al. 2019. Mitochondrial divergence suggests unexpected high species diversity in the opsariichthine fishes (Teleostei: Cyprinidae) and the revalidation of Opsariichthys macrolepis[J]. Ecology and Evolution, 9(5): 2664-2677. DOI:10.1002/ece3.4933 |
Wright S. 1965. The interpretation of population structure by F-statistics with special regard to systems of mating[J]. Evolution, 19: 395-420. DOI:10.1111/j.1558-5646.1965.tb01731.x |
Wu TH, Tsang LM, Chow LH, et al. 2019. Cryptic lineages and hybridization of the predaceous chub Parazacco spilurus (Actinopterygii, Cypriniformes, Xenocyprididae) in Hong Kong[J]. Hydrobiologia, 826(1): 99-111. DOI:10.1007/s10750-018-3720-y |
Zhang L, Tang QY, Liu HZ. 2008. Phylogeny and speciation of the eastern Asian cyprinid genus Sarcocheilichthys[J]. Journal of Fish Biology, 72(5): 1122-1137. DOI:10.1111/j.1095-8649.2007.01733.x |
 2021, Vol. 40
2021, Vol. 40