扩展功能
文章信息
- 浦恩念, 高子厚, 李玉琼, 邵宗体, 杜春红, 段兴德, 刘正祥
- PU Ennian, GAO Zihou, LI Yuqiong, SHAO Zongti, DU Chunhong, DUAN Xingde, LIU Zhengxiang
- 云南部分地区大绒鼠种群遗传多样性与遗传分化研究
- Genetic Diversity and Differentiation of Eothenomys miletus Populations in Yunnan Province
- 四川动物, 2021, 40(3): 254-260
- Sichuan Journal of Zoology, 2021, 40(3): 254-260
- 10.11984/j.issn.1000-7083.20200480
-
文章历史
- 收稿日期: 2020-12-30
- 接受日期: 2021-03-16
2. 云南省地方病防治所, 云南大理 671000
2. Yunnan Center for Endemic Disease Control and Prevention, Dali, Yunnan Province 671000, China
大绒鼠Eothenomys miletus隶属于仓鼠科Cricetidae绒鼠属,在云南省分布广泛(罗泽珣,2000),是云南野鼠鼠疫自然疫源地的主要储存和流行宿主动物之一。云南省特殊的地理环境和气候条件,可能导致了大绒鼠形态特征不稳定、同种异型和近缘种形态特征相似等问题:王应祥(2003)将大绒鼠分为2个地理亚种:指名亚种E. m. miletus和怒江亚种E. m. confinii;整个绒鼠属的系统关系及分类地位尚存争议(刘少英,刘洋,2005)。因此,云南不同地理位置和气候环境中分布的大绒鼠的遗传多样性与遗传分化值得研究和探讨。
动物线粒体DNA(mtDNA)分子小而稳定、母系遗传、进化速度快等特征(Birky et al., 1989),已成为遗传多样性研究的一个强有力的工具,它包含的细胞色素b(cyt b)基因是研究物种地理变异问题最可信的线粒体DNA标记之一(李楠楠等,2012)。一些学者对大绒鼠的遗传结构进行了初步研究(沐远等,2015;Mu et al., 2019),但尚未对其他区域分布的大绒鼠的遗传信息进行比较分析,本文对云南7个地区的大绒鼠cyt b基因进行检测分析,为大绒鼠乃至绒鼠属的系统研究提供分子数据。
1 材料与方法 1.1 样品的采集、鉴定和保存本研究从玉龙县、剑川县、祥云县、大理市、巧家县、禄劝县、腾冲市的7个采样点共采集到大绒鼠140只(表 1)。利用鼠夹从采样点的农田及灌木丛中捕获大绒鼠,捕获个体按形态学特征,并剥制头骨标本进行鉴定,取肌肉保存于95%乙醇中,-20 ℃保存。
| 采样点 Sampling locality |
采样时间 Sampling time |
东经 Longitude/° |
北纬 Latitude/° |
样本数量 Sample size |
| 巧家县东坪镇坪子村 | 2017年10月 | 103.047 | 27.300 | 11 |
| 玉龙县黄山镇南溪村 | 2018年8月 | 100.166 | 26.781 | 33 |
| 剑川县沙溪镇石龙村 | 2018年8月 | 99.811 | 26.378 | 24 |
| 祥云县祥城镇毛栗坡村 | 2018年9月 | 100.456 | 25.552 | 25 |
| 腾冲市荷花镇羡多村 | 2019年7月 | 98.368 | 24.920 | 10 |
| 禄劝县雪山乡丰租村 | 2019年10月 | 102.807 | 26.169 | 24 |
| 大理市下关镇吊草村 | 2019年11月 | 100.258 | 25.548 | 13 |
用肌肉标本按照QIAGEN试剂盒产品说明书步骤提取基因组DNA。cyt b的PCR扩增使用哺乳动物通用引物L14724:5'-CGAAGCTTGATATGAAAAACCATCGTTG-3',H15915R:5'-GGAATTCATCTCTCCGGTTTACAAGAC-3';扩增体系为25 μL:模板2 μL,10 μmol·L-1引物各0.5 μL,Mix 12.5 μL,ddH2O 9.5 μL。扩增条件:95 ℃ 5 min;95 ℃ 45 s,55 ℃ 1 min,72 ℃ 1 min,35个循环;72 ℃ 10 min。实验过程以水作为空白对照。扩增产物经琼脂糖凝胶电泳检测后送上海生工生物工程技术服务有限公司测序。
1.3 序列分析利用Clustal X(Thompson et al., 1997)对测序获得的有效样本序列进行比对。利用MEGA 5(Tamura et al., 2011)基于Kimura双参数距离模型计算碱基组成、转换与颠换值;以齐氏姬鼠Apodemus chevrieri(GenBank登录号:AB096821)为外群,基于类平均法(UPGMA)构建分子系统发育树,各分支的自举检验值由1 000次重复检验所得。利用Dnasp 5.0(Rozas et al., 2003)计算单倍型数目、变异位点数、简约信息位点数、单倍型多样性、核苷酸多样性和基因流(Nm)。其中,Nm < 1表明群体间可能由于遗传漂变而发生分化;Nm>1表明群体间的基因流水平较高,群体间遗传分化较小。利用PopART(Leigh & Bryant,2015)基于中连法(Median joining method)构建大绒鼠单倍型间的网络关系图。运用Arlequin 3.1(Excoffier et al., 1992)计算遗传分化系数(Fst),进行分子变异分析(AMOVA)。通过IBD 1.53(Bohonak,2002)检测地理距离与遗传距离的相关性,用Fst值表示遗传距离,地理距离采用采样点间的地球表面直线距离。
2 结果与分析 2.1 DNA的PCR扩增扩增结果经琼脂糖凝胶电泳检测未发现非特异性条带,共获得140只大绒鼠的扩增条带,部分样本扩增结果见图 1。
|
| 图 1 大绒鼠cyt b基因部分序列扩增结果 Fig. 1 Amplification of partial cyt b gene of Eothenomys miletus 1~4. 样本, M. Marker 1-4. sample, M. Marker |
| |
共获得140只大绒鼠样本cyt b基因部分序列(GenBank登录号:MW567503~MW567642),在1 027 bp的序列中,共有核苷酸变异位点128个,包含单变异位点109个和简约信息位点19个,转换和颠换(TS/TV)比为2.0,A、T、G、C平均碱基组成分别为:29%、27%、30.4%、13.6%,A+T含量显著高于G+C含量。109个变异位点共定义51种单倍型,分别命名为Hap1~Hap51。
2.3 遗传多样性和遗传结构7个采样点大绒鼠的单倍型多样性为0.711~ 0.907,核苷酸多样性为0.002 25~0.002 98,整体的单倍型多样性和核苷酸多样性分别为0.977± 0.004和0.018 45±0.001 78。所有种群均为高单倍型多样性和低核苷酸多样性的模式。Hap20为大理、祥云、剑川种群共享,Hap21、Hap22、Hap24为大理、祥云种群共享,其余47个单倍型为各个种群特有(表 2)。玉龙和剑川种群的单倍型多样性相对较高,巧家种群的核苷酸多样性最高(表 3)。
| 采样点 Sampling site |
样本数 Number of samples |
单倍型个数 Number of haplotypes |
单倍型 Haplotype |
| YL | 33 | 11 | Hap1、Hap2、Hap3、Hap4、Hap5、Hap6、Hap7、Hap8、Hap9、Hap10、Hap11 |
| LQ | 24 | 8 | Hap12、Hap13、Hap14、Hap15、Hap16、Hap17、Hap18、Hap19 |
| XY | 25 | 10 | Hap20、Hap21、Hap22、Hap23、Hap24、Hap25、Hap26、Hap27、Hap28、Hap29 |
| TC | 10 | 3 | Hap30、Hap31、Hap32 |
| JC | 24 | 11 | Hap20、Hap33、Hap34、Hap35、Hap36、Hap37、Hap38、Hap39、Hap40、Hap41、Hap42 |
| DL | 13 | 7 | Hap20、Hap21、Hap22、Hap24、Hap43、Hap44、Hap45 |
| QJ | 11 | 6 | Hap46、Hap47、Hap48、Hap49、Hap50、Hap51 |
| 注:YL. 玉龙县, JC. 剑川县, XY. 祥云县, DL. 大理市, QJ. 巧家县, LQ. 禄劝县, TC. 腾冲市; 下同 Notes: YL. Yulong County, JC.Jianchuan County, XY. Xiangyun County, DL. Dali City, QJ. Qiaojia County, LQ. Luquan County, TC. Tengchong City; the same below |
|||
| 种群 Population |
变异位点 Variable site |
单倍型多样性 Haplotype diversity |
平均核苷酸变异度 Average number of differences |
核苷酸多样性 Nucleotide diversity |
| YL | 18 | 0.907±0.023 | 4.083 | 0.003 98±0.000 28 |
| LQ | 11 | 0.859±0.038 | 2.894 | 0.002 82±0.000 40 |
| XY | 16 | 0.847±0.057 | 3.020 | 0.002 94±0.000 45 |
| TC | 6 | 0.711±0.086 | 2.311 | 0.002 25±0.000 76 |
| JC | 21 | 0.895±0.035 | 2.942 | 0.002 87±0.000 64 |
| DL | 12 | 0.885±0.064 | 3.872 | 0.003 77±0.000 57 |
| QJ | 12 | 0.891±0.063 | 5.200 | 0.005 07±0.000 56 |
种群间遗传差异分析结果显示,7个地理种群的Fst值为0.179 68~0.949 45,遗传差异均有统计学意义(P<0.001)(表 4)。分子变异分析(AMOVA)结果表明,群体间的遗传变异为84.29%,群体内的为15.71%。种群间的基因流为0.026 62~2.282 72,除玉龙、祥云、剑川、大理4个地理距离较近种群之间的Nm值大于1之外,其他均小于1,且值均较低(表 5)。
| YL | LQ | XY | TC | JC | DL | QJ | |
| YL | 260 | 154 | 265 | 48 | 143 | 270 | |
| LQ | 0.815 06* | 182 | 420 | 280 | 224 | 191 | |
| XY | 0.179 68* | 0.839 86* | 228 | 150 | 60 | 322 | |
| TC | 0.930 28* | 0.949 45* | 0.944 33* | 218 | 195 | 535 | |
| JC | 0.234 06* | 0.840 08* | 0.200 74* | 0.945 64* | 97 | 330 | |
| DL | 0.419 20* | 0.838 26* | 0.267 10* | 0.937 74* | 0.478 99* | 342 | |
| QJ | 0.893 23* | 0.903 63* | 0.905 92* | 0.929 67* | 0.907 87* | 0.886 20* | |
| 注: *种群遗传分化指数间的差异有高度统计学意义(P < 0.001) Notes: * there is an extremely significant difference between different populations (P < 0.001) |
|||||||
| 变异来源 Source of variation |
自由度 df |
平方和 Sum of squares |
方差组分 Variance components |
变异百分 率Percentage of variation/% |
| 种群间 | 6 | 1 085.999 | 9.214 26 | 84.29 |
| 种群内 | 133 | 228.329 | 1.716 76 | 15.71 |
| 总体 | 139 | 1 314.329 | 10.931 02 |
| YL | LQ | XY | TC | JC | DL | |
| LQ | 0.113 45 | |||||
| XY | 2.282 72 | 0.095 34 | ||||
| TC | 0.037 47 | 0.026 62 | 0.029 48 | |||
| JC | 1.636 20 | 0.095 18 | 1.990 78 | 0.028 74 | ||
| DL | 0.692 75 | 0.096 47 | 1.371 96 | 0.033 20 | 0.543 86 | |
| QJ | 0.059 77 | 0.053 32 | 0.051 93 | 0.037 83 | 0.050 74 | 0.064 21 |
采用UPGMA法构建的系统进化树显示,7个采样点的大绒鼠种群分化为4个地域分支(图 2),第一分支包括玉龙、祥云、剑川、大理的单倍型,第二分支为禄劝的单倍型,第三分支为巧家单倍型,第四分支为腾冲单倍型。单倍型进化网络关系图显示出与进化树相同的结果(图 3),大绒鼠已经出现了地理种群分化。
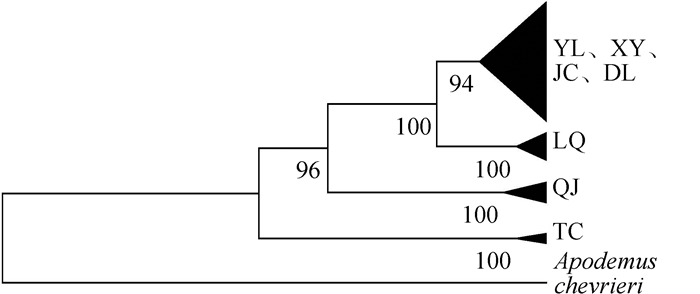
|
| 图 2 基于云南7个采样点的大绒鼠cyt b基因序列构建的单倍型UPGMA树 Fig. 2 UPGMA tree of haplotypes based on cyt b gene of Eothenomys miletus from 7 localities of Yunnan Province |
| |

|
| 图 3 基于cyt b基因序列建立的云南7个采样点的大绒鼠单倍型进化网络关系图 Fig. 3 Network relationships of haplotypes based on cyt b gene of Eothenomys miletus from 7 localities of Yunnan Province 每个实心圆代表 1个单倍型, 大小与单倍型出现的频率呈正比; 空心圆代表缺失单倍型; 每条线代表 1次突变 Each solid circlerepresents a haplotype, and its size is proportional to haplotype frequency; an open circle represents a missing haplotype; each line represents a mutation |
| |
通过IBD1.53对大绒鼠7个地理种群间的地理距离与遗传距离进行检验,结果显示,地理距离与遗传距离之间呈显著的正相关(r=0.699 7,P < 0.05,R2=0.49)。
3 讨论遗传多样性的高低与物种的适应能力、生存能力密切相关(季维智等,1999;Frankham et al., 2010),本研究中大绒鼠单倍型多样性为0.977±0.004,核苷酸多样性为0.018 45±0.001 78,多样性水平与其常同域分布的齐氏姬鼠相当(朱万龙等,2013),玉龙和剑川大绒鼠种群单倍型多样性高于其他种群,大绒鼠在这些地区小兽中的构成比也较高,说明大绒鼠在这些地区的适应能力和生存能力较强。大绒鼠种群总体表现出高单倍型多样性、低核苷酸多样性的特点,Yuan等(2012)认为高单倍型多样性、低核苷酸多样性主要与单个碱基的变异、地理种群处于扩张有关,大绒鼠种群总体表现出高单倍型多样性、低核苷酸多样性的特点可能也与这2种原因有关。
基因分化系数和基因流是衡量遗传分化的重要指标(Nei,1987),Fst值为0.15~0.25时说明遗传分化较大(Wright,1965),基于线粒体cyt b序列的AMOVA分析结果显示,大绒鼠各地理种群间遗传分化指数为0.179 68~0.949 45,表明云南地区大绒鼠各地理种群间存在明显的遗传分化。大绒鼠7个地理种群间基因流为0.026 62~2.282 72,玉龙、祥云、剑川、大理4个地理距离较近种群组合之间的Nm值大于1之外,其他种群组合间均小于1,且值均较低,说明多数种群间基因流水平贫乏。在单倍型系统进化树中,大绒鼠种群分化为4个地域分支,玉龙、祥云、剑川、大理为一分支,其他种群分别独立为一支,在实际的地理位置中,玉龙、剑川、大理、祥云的地理距离较近,而腾冲、巧家、禄劝与其他几个地区距离较远。单倍型进化网络关系图显示出与进化树相同的结果,综合研究结果表明大绒鼠已经出现了明显的种群分化。
遗传分化的程度会随着地理距离的增加而增加(Floyd et al., 2005),IBD检测结果显示,Fst与地理距离呈显著正相关,大绒鼠种群间遗传分化中49.0%的变异可以用地理隔离解释,这表明种群间地理隔离对遗传分化起重要作用,这可能与大绒鼠的迁移扩散能力较弱有关。云南气候多样,山川河流纵横,地形地势复杂,独特的生境可能造成大绒鼠种群间的地理隔离,是否因地理隔离形成独立的亚种还有待于进一步研究。
综上所述,云南地区大绒鼠高单倍型多样性、低核苷酸多样性,以及种群间高遗传分化的遗传结构特点,可能与其生物学特征和扩散能力等因素有关,同时地理距离隔离对其遗传分化起了重要作用。然而,由于本研究仅用了线粒体cyt b基因作为分子标记,可能存在局限性,欲全面了解大绒鼠的遗传多样性、遗传分化程度,还需要扩大大绒鼠采样范围,加入微卫星、核基因等标记的分析,进一步研究。
季维智, 宿兵. 1999. 遗传多样性研究的原理与方法[M]. 杭州: 浙江科学技术出版社.
|
李楠楠, 刘振生, 王正寰, 等. 2012. 基于细胞色素b基因的中国岩羊不同地理种群遗传差异分析[J]. 生态学报, 32(8): 2381-2389. |
刘少英, 刘洋. 2005. 绒鼠属(Eothenomys Miller, 1896)系统学研究概况[J]. 四川动物, 24(1): 98-103. DOI:10.3969/j.issn.1000-7083.2005.01.034 |
罗泽珣. 2000. 中国动物志 兽纲 第六卷 啮齿目 下册 仓鼠科[M]. 北京: 科学出版社.
|
沐远, 杨涛, 马壮琼, 等. 2015. 云南剑川地区大绒鼠线粒体细胞色素b和控制区遗传多样性的研究[J]. 生物学杂志, 5: 30-34. |
王应祥. 2003. 中国哺乳动物种和亚种分类名录与分布大全[M]. 北京: 中国林业出版社.
|
朱万龙, 姜文秀, 王政昆. 2013. 云南4个地区高山姬鼠线粒体细胞色素b遗传分化的研究[J]. 生物学杂志, 30(4): 19-23. DOI:10.3969/j.issn.2095-1736.2013.04.019 |
Birky CW, Fuerst P, Maruyama T. 1989. Organelle gene diversity under migration, mutation, and drift: equilibrium expectations, approach to equilibrium, effects of heteroplasmic cells, and comparison to nuclear genes[J]. Genetics, 121(3): 613-627. DOI:10.1093/genetics/121.3.613 |
Bohonak AJ. 2002. IBD (Isolation by Distance): a program for analyses of isolation by distance[J]. Journal of Heredity, (2): 153-154. |
Excoffier L, Smouse PE, Quattro JM. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: applications to human mitochondrial DNA restriction data[J]. Genetics, 131(2): 476-494. |
Floyd CH, vanVuren DH, May B. 2005. Marmots on great basin mountain tops: using genetics to test a biogeographic paradigm[J]. Ecology, 86(8): 2145-2153. DOI:10.1890/04-1227 |
Frankham R. 2010. Relationship of genetic variation to population size in wildlife[J]. Conservation Biology, 10(6): 1500-1508. |
Leigh JW, Bryant D. 2015. POPART: full-feature software for haplotype network construction[J]. Methods in Ecology and Evolution, 6(9): 1110-1116. DOI:10.1111/2041-210X.12410 |
Mu Y, Duan Y, Di Z, et al. 2019. The complete mitochondrial genome of the Yunnan red-backed vole Eothenomys miletus (Rodentia: Cricetidae) and its phylogeny[J]. Mitochondrial DNA Part B, 4(1): 1424-1425. DOI:10.1080/23802359.2019.1598801 |
Nei M. 1987. Moleculer evolutionary genetics[M]. New York: Columbia University Press.
|
Rozas J, Sánchez-DelBarrio JC, Messeguer X, et al. 2003. DnaSP, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 19(18): 2496-2497. DOI:10.1093/bioinformatics/btg359 |
Tamura K, Peterson D, Peterson N, et al. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 28(10): 2731-2739. DOI:10.1093/molbev/msr121 |
Thompson JD, Gibson TJ, Plewniak F, et al. 1997. The clustal X Windows interface: exible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 25(24): 4876-4882. DOI:10.1093/nar/25.24.4876 |
Wright S. 1965. The interpretation of population structure by F-statistics with special regard to systems of mating[J]. Evolution, 19(3): 395-420. DOI:10.1111/j.1558-5646.1965.tb01731.x |
Yuan Y, Li Q, Kong L, et al. 2012. The complete mitochondrial genome of the grand jackknife clam, Solen grandis (Bivalvia: Solenidae): a novel gene order and unusual non-coding region[J]. Molecular Biology Reports, 39(2): 1287-1292. DOI:10.1007/s11033-011-0861-8 |
 2021, Vol. 40
2021, Vol. 40




