扩展功能
文章信息
- 胥文才, 蔺杼阳, 肖雅丹, 唐瑞祥, 范振鑫, 孟杨
- XU Wencai, LIN Zhuyang, XIAO Yadan, TANG Ruixiang, FAN Zhenxin, MENG Yang
- 中华蟾蜍多组织差异转录组分析
- Differential Transcriptome Analysis in Multi-Tissue of Bufo gargarizans
- 四川动物, 2020, 39(6): 601-615
- Sichuan Journal of Zoology, 2020, 39(6): 601-615
- 10.11984/j.issn.1000-7083.20200278
-
文章历史
- 收稿日期: 2020-07-22
- 接受日期: 2020-08-24
2. 四川大学生命科学学院, 四川省濒危动物保护生物学重点实验室, 成都 610065
2. Sichuan Key Laboratory of Conservation Biology on Endangered Wildlife, College of Life Sciences, Sichuan University, Chengdu 610065, China
中华蟾蜍Bufo gargarizans隶属于无尾目Anura蟾蜍科Bufonidae蟾蜍属Bufo,是中药材蟾酥和蟾衣的药源。蟾酥是蟾蜍耳后腺分泌物,蟾衣为蟾蜍去除内脏后的表皮(国家药典委员会,2010),蟾蜍毒液是由蟾蜍耳后腺和皮肤腺不同类型腺体的混合产物组成(吴文英等,2011)。蟾酥含有丰富的化学物质,研究表明,从不同种类蟾蜍中分离得到100多种化学成分,包括多肽、类固醇、吲哚生物碱等,其中蟾蜍毒素、蟾毒配基、甾醇类等为主要成分(赵大洲等,2006;陈丽萍等,2011;辛秀兰等,2012)。在传统中药中,蟾蜍毒素被用于治疗各种疾病已有数百年的历史,现代研究(包括实验和临床试验)也揭示和支持了其中的分子机制。目前蟾酥已被广泛用作治疗心力衰歇、口腔炎、咽喉炎、咽喉肿痛、皮肤癌等(罗展雄等,2009;彭贝等,2011;杨宏梅,陈涛,2014;孙崇峰等,2018)。
转录组是特定组织或细胞在某一发育阶段或功能状态下转录的所有RNA的集合(祁云霞等,2011)。随着高通量测序技术的不断成熟,转录组高通量测序技术(RNA-Seq)在基因组学的研究中发挥了非常重要的作用,如大熊猫Ailuropoda melanoleuca(杜联明等,2014)、美洲大蠊Periplaneta americana(晋家正等,2018)、彭波半细毛羊Ovis aries(张立等,2020)等。这项技术近期已应用于中华蟾蜍研究中,如不同海拔蟾蜍基因表达的变化(Yang et al., 2017)、氟化物对蟾蜍骨骼发育的影响(Chao et al., 2018)。但多数研究都是针对不同个体的表达差异,而少有针对同一个体的不同组织,同时为了比较组织间的差异,要排除个体间的差异。本研究利用RNA-Seq对中华蟾蜍耳后腺与其他6个组织进行了转录组测序和分析,并利用多种方法对差异表达基因进行分析,探究相关基因富集的代谢通路,研究数据能支持后续对中华蟾蜍基因表达研究和药用价值的开发。
1 研究方法 1.1 数据来源中华蟾蜍个体于2018年9月捕捉于四川大学江安校区内,取其中1只雌性个体,解剖取其肝脏、脾脏、心脏、卵泡、肌肉、腹部皮肤、右耳后腺7个组织样本各2份,并迅速放于液氮中保存。样品通过冷链运输到诺禾致源公司(北京,中国)进行了总RNA的提取和RNA-Seq测序。使用高通量测序平台(Illumina NovaSeq 6000)对这7个组织的cDNA文库进行转录组测序。质控标准主要有2个限制条件:一是原始测序数据中每一条序列中的N含量不能超过这一条序列的总碱基数量的10%,如果超过则去掉这一条序列及其对应的双端序列;二是原始测序数据中每一条序列的低质量、碱基质量值小于5的碱基数量不能超过这条序列的总碱基数量的50%,如果超过则去掉这一条序列及其对应的双端序列。
1.2 转录组组装采用de novo对转录组进行从头组装,使用Trinity v2.8.3(Grabherr et al., 2011)对高质量的序列从头拼接成转录组。在使用Trinity组装时,设置只保留片段长度在300 bp以上的contig序列(Haas et al., 2013)。针对组装的转录本,在BUSCO v3.0.1(Simão et al., 2015)中以脊索动物门的保守序列为背景进行转录组的质量评估。再用hisat2 v2.1.0(Kim et al., 2019)将质控数据(clean reads)重新比对到组装好的转录本。最后使用corset v1.06(Davidson & Oshlack,2014)采用层次聚类的方法,重新筛选Unigene。
1.3 功能注释使用BLAST v2.7.0(Altschul et al., 1990)将最终得到的所有转录本同源比对到NCBI NR(RefSeq non-redundant proteins)(Pruitt et al., 2005)、Swiss-Prot、COG数据库,E值设为1e-05。用与NR数据库比对结果分析转录本的相似性以及与哪些物种共享最多的转录本,在使用BLAST进行比对的结果中,E值表明在随机的情况下,其他序列与目标序列相似度大于这条显示的序列的可能性,所以E值越小,结果越接近真实情况。再将结果导入到Blast2GO v5.2(Conesa et al., 2005)进行GO注释,根据BLAST的比对结果,匹配相应的GO功能条目,最后将结果提交到WEGO(Ye et al., 2006)网站上,对转录本按照细胞组分(cellular component)、分子功能(molecular function)和生物过程(biological process)进行功能分类统计。使用KAAS(KEGG automatic annotation server)(Moriya et al., 2007)对转录本进行KEGG通路分析。
1.4 基因差异表达分析使用Bioconductor R语言包edgeR v3.24.3(Robinson et al., 2010)和TMM对原始测序数据进行标准化,得到的结果进行差异基因表达分析,并使用Benjamini-Hochberg对所有统计检验结果的P值进行多重假设检验校正,通过控制FDR来决定P值的阈值。差异基因筛选标准为FDR < 0.05和|log2(fold change)|≥1。
使用R和python编写统计分类代码,对不同组织间的差异表达结果进行筛选得到共同上调或下调的差异表达基因。使用Bioconductor R语言包clusterProfiler v3.8.1(Yu et al., 2012)进行差异表达基因的GO功能富集分析。在进行KEGG差异表达基因功能富集时,以所鉴定出的差异表达基因序列为输入文件,使用KOBAS(Xie et al., 2011),选取和中华蟾蜍相似的同源物种为背景基因集进行KEGG通路富集分析。富集分析的P值设为0.05。
2 结果 2.1 测序结果使用RNA-Seq对中华蟾蜍7个组织进行了转录组测序,获得共计161 661 926条双端测序的原始数据,进一步质控后,共获得160 236 971条高质量的质控数据,占原始数据的99.12%,以这部分高质量的质控数据作为后面实际使用的数据(表 1)。测序数据已经提交到NCBI(GenBank登录号:PRJNA638036)。
| 样本 | 原始测序数据 | 质控数据 | 原始碱基数/G | 过滤后碱基数/G | Q20/% | Q30/% | GC/% |
| 心脏 | 23 011 535 | 22 882 350 | 6.90 | 6.86 | 96.65 | 91.31 | 46.53 |
| 肝脏 | 21 042 054 | 20 818 526 | 6.31 | 6.25 | 96.76 | 91.64 | 47.08 |
| 脾脏 | 23 039 871 | 22 827 805 | 6.91 | 6.85 | 96.63 | 91.29 | 46.72 |
| 肌肉 | 24 960 077 | 24 746 749 | 7.49 | 7.42 | 96.42 | 91.00 | 47.98 |
| 卵泡 | 22 200 340 | 22 005 856 | 6.66 | 6.60 | 96.43 | 90.91 | 47.00 |
| 腹部皮肤 | 27 029 365 | 26 791 167 | 8.11 | 8.04 | 96.59 | 91.34 | 47.54 |
| 右耳后腺 | 20 378 684 | 20 164 518 | 6.11 | 6.05 | 96.84 | 91.75 | 46.59 |
使用Trinity将7个组织样本数据全部组装,得到270 017条转录本,其中最长的36 170 bp,最短的279 bp,总长度294 515 519 bp,平均长度1 090.73 bp,整体N50的长度为1 845 bp,GC碱基数占总碱基数的45.26%,组装较为完整。
BUSCO结果显示,组装的转录本与保守序列完全比对成功的比例为87.7%,其中成功比对一次的比例为82.9%,成功比对多次的比例为4.8%,同时还有6.8%的序列为部分转录本序列比对成功,而仅有5.5%的序列没有比对上任何保守序列。对于从头组装的转录组,比对结果表明本实验组装的转录组是较为完整且准确的,可以用于后续的分析。
2.3 转录组比对使用hisat2 v2.1.0将原始测序数据与组装转录本进行比对,每个样本的比对率均高于85.00%,最高的是心脏(89.75%),最低的是右耳后腺(87.60%)(表 2)。比对率侧面反映了组装结果的正确性,在从头组装中,越高序列比对率代表了越多的序列参与了转录本的组装。最后使用corest,基于对比结果,使用层次聚类的方法将Trinity组装出来的转录本重新聚类,挑选每个类中最长的序列作为该类的代表,获得Unigene转录组,共计132 117条,用作后续的分析。
| 样本 | 比对率/% |
| 心脏 | 89.75 |
| 肝脏 | 89.64 |
| 脾脏 | 88.17 |
| 肌肉 | 88.83 |
| 卵泡 | 88.99 |
| 腹部皮肤 | 88.55 |
| 右耳后腺 | 87.60 |
将重新聚类筛选Unigene之后获得的转录组(后文称转录组)与公共数据库(包括NR、Swiss-Prot、KEGG、COG和GO)进行同源性比对注释,注释到的转录本数分别为46 744(35.38%)、34 988(26.48%)、11 871(8.99%)、11 292(8.54%)和12 320(9.33%)(表 3),其中被各数据库均注释到的转录本数为5 281(4.00%),共注释47 533(35.98%)条转录本。
| 数据库 | 注释到的转录本/条 | 占比/% |
| NR | 46 744 | 35.38 |
| Swiss-Prot | 34 988 | 26.48 |
| KEGG | 11 871 | 8.99 |
| COG | 11 292 | 8.55 |
| GO | 12 320 | 9.33 |
| 各数据库共计 | 47 533 | 35.98 |
| Total | 132 117 | 100 |
与非冗余蛋白质数据库(NR数据库)的比对结果统计发现,大量转录本注释为电子预测的蛋白质:整个转录本注释结果的E值分布反映了整体注释的结果,26.8%的E值分布在0~1e-150,8.9%在1e-150~1e-100,16.6%在1e-100~1e-50,47.7%在1e-50~1e-05。
从比对到的物种分布得出(图 1),最显著的为:热带爪蟾Xenopus tropicalis、非洲爪蟾Xenopus laevis和高山倭蛙Nanorana parkeri,反映了近缘物种在分子层面的物种相似性,而非洲爪蟾和热带爪蟾为模式物种,高比对率结果支持了后续分析的基因富集步骤中将其选为模式物种。
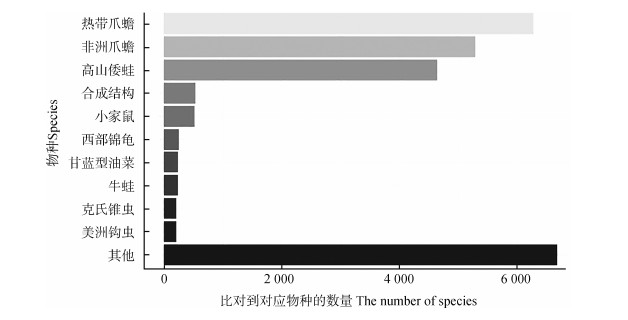
|
| 图 1 与NR数据库比对结果的物种分布 Fig. 1 Species distribution compared with the NR database |
| |
根据NR数据库的同源性比对结果,对转录本进行GO注释,12 320条转录本序列被注释,共得到24 124个GO功能条目,平均每条序列被分配到2个GO功能条目。GO功能条目主要分布在生物过程(8 272,34.28%)、分子功能(10 549,43.72%)和细胞组分(5 303,21.98%)。使用WEGO对其功能条目进行了分类(图 2)。
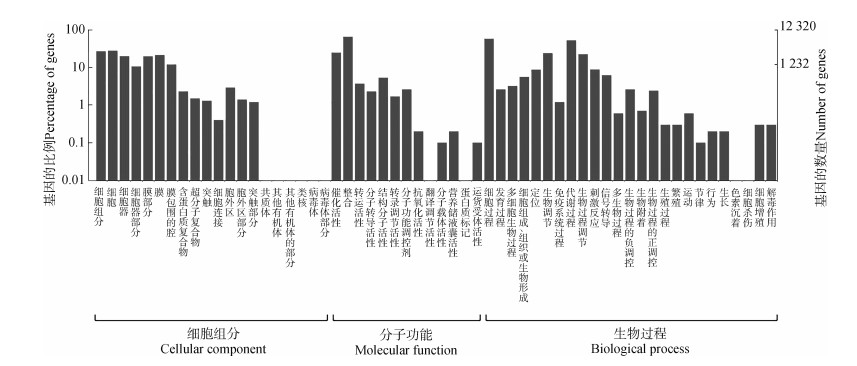
|
| 图 2 转录本分配到的GO注释分类 Fig. 2 Classification of GO annotations to which transcripts are assigned |
| |
在生物过程中,细胞过程(cellular process)和代谢过程(metabolic process)功能是主要被注释到的条目;在分子功能中,结合(binding)是最有代表性的条目;而在细胞组分中,细胞(cell)和细胞组分(cell part)最多。
2.5 差异基因分析 2.5.1 差异基因的筛选使用edgeR包依据差异表达倍数|log2(fold change)|≥1以及数据的检验值FDR < 0.05作为筛选条件,筛选得到了对应符合要求的6组差异表达基因,分别来自腹部皮肤(图 3:A)、肝脏(图 3:B)、肌肉(图 3:C)、卵泡(图 3:D)、脾脏(图 3:E)、心脏(图 3:F)与右耳后腺的比较。

|
| 图 3 差异表达基因log2倍数变化分布(红点为最显著的1 000个基因) Fig. 3 Distribution of log2 (fold change) of differentially expressed genes (red dots are the most significant 1 000 genes) FC.倍数变化fold change, CPM.每百万的数目counts per million |
| |
根据右耳后腺与其他各个组织转录组差异分析的结果,得出在右耳后腺中差异表达上调和下调的基因以及数目(表 4)。
| 样本 | 上调 | 下调 |
| 右耳后腺与腹部皮肤 | 195/9 026 | 805/7 121 |
| 右耳后腺与心脏 | 745/12 811 | 255/13 479 |
| 右耳后腺与肝脏 | 655/13 084 | 345/16 897 |
| 右耳后腺与卵泡 | 611/12 420 | 389/13 037 |
| 右耳后腺与肌肉 | 696/15 612 | 304/18 031 |
| 右耳后腺与脾脏 | 970/11 820 | 30/13 248 |
因为组织特异性的存在,不同组织之间必然存在差异表达。为了验证耳后腺组织的特异性表达,提取各个组织与右耳后腺之间的表达差异上调和下调的基因并获得彼此之前的交集,得到了对应的各个组织的差异表达基因Venn图。与其他组织相比,在右耳后腺中共同上调的基因为3 668个(图 4:左)、共同下调的基因为260个(图 4:右)。
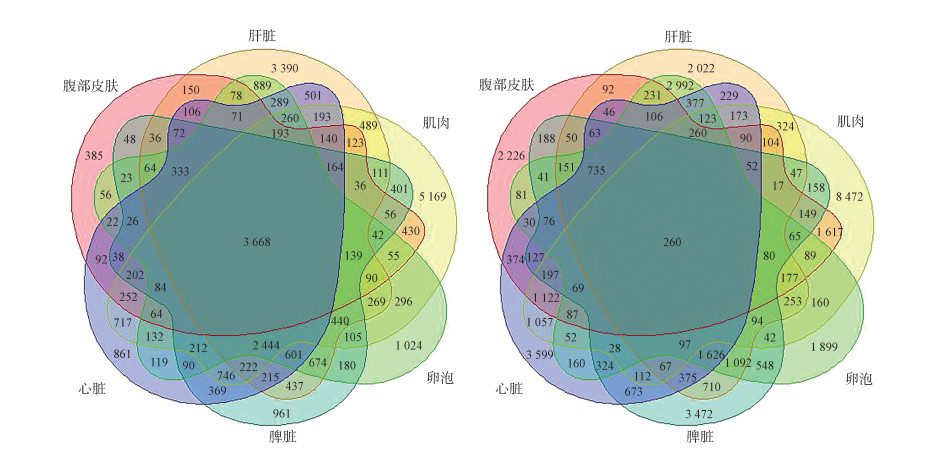
|
| 图 4 组织差异表达上调基因(左)和下调基因(右)的Venn图 Fig. 4 Venn diagrams of tissue differential expression up-regulated genes (left) and down-regulated genes (right) |
| |
为深入了解差异表达基因所起的生物学作用,针对这3 668个上调基因和260个下调基因,共计3 928个显著表达的差异基因,分别做了GO通路和KEGG通路的富集分析。
对于GO功能富集分析,以中华蟾蜍所有基因的GO注释结果为背景基因集,分别以上调差异基因和下调差异基因为目标基因集进行富集,在目标基因集中上调和下调分别有347个和42个基因有相应的GO注释,差异基因在注释到的背景基因集中覆盖率只有10%,这在非模式生物中情况较普遍。GO的富集结果以P-adjust < 0.05为筛选阈值(以BH法校正P值得到P-adjust),P-adjust < 0.05为显著表达,对于这些差异表达基因,上调基因显著富集到了78个GO条目(图 5)。

|
| 图 5 差异表达基因富集到GO功能条目及分类 Fig. 5 Enrichment of differentially expressed genes into GO function entries and classification |
| |
在分子功能中,共富集到28个条目,最显著的是肽酶抑制剂活性(GO:0030414)和肽酶调节物活性(GO:0061134),P-adjust < 1e-15,其次为酶的抑制物活性(GO:0004857,P-adjust=1.11e-13)、角质层的结构组成(GO:0042302,P-adjust=5.10e-11)、结构分子活性(GO:0005198,P-adjust=1.25e-09)等(图 6)。
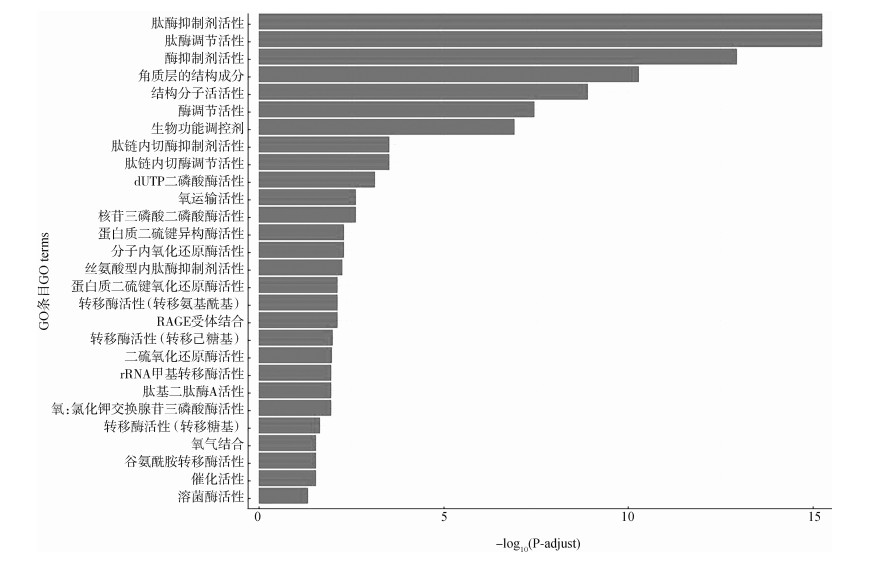
|
| 图 6 上调差异表达基因富集到分子功能GO功能条目 Fig. 6 Up-regulated differentially expressed genes enriched to molecular function GO function entry |
| |
在细胞组分中,共富集到9个条目(图 7),最显著的条目为胞外区域(GO:0005576,P-adjust=4.08e-06)、中间纤维(GO:0005882,P-adjust=4.08e-06)、中间纤维细胞骨架(GO:0045111,P-adjust=4.08e-06)、角蛋白纤维(GO:0045095,P-adjust=2.15e-05)、胶原蛋白三聚物(GO:0005581,P-adjust=2.15e-05)。
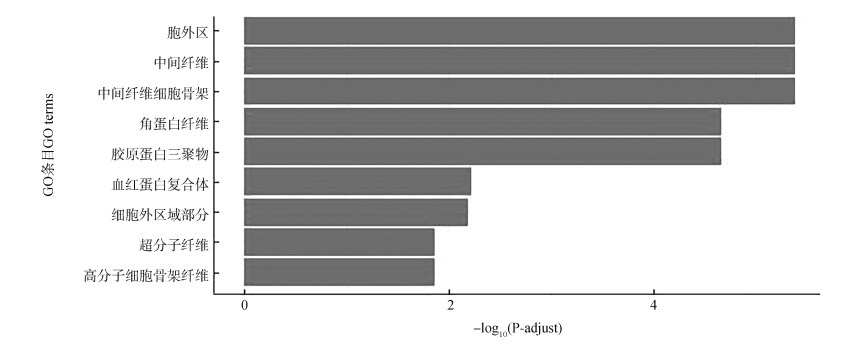
|
| 图 7 上调差异表达基因富集到细胞组分GO功能条目 Fig. 7 Up-regulated differentially expressed genes enriched into cell components GO function entry |
| |
在生物过程中,共富集到40个条目(图 8),最显著的条目中肽酶活性的负调控(GO:0010466)、蛋白水解的负调节(GO:0045861)、肽酶活性的调节(GO:0052547)P-adjust都小于1e-14,其次是水解酶活性的负调节(GO:0051346,P-adjust=3.49e-15),调节蛋白水解作用(GO:0030162,P-adjust=7.32e-15)、分子功能的负调节(GO:0044092,P-adjust=9.37e-14)、催化活性的负调节(GO:0044092,P-adjust=9.37e-14)、细胞蛋白代谢过程的负调控(GO:0043086,P-adjust=1.76e-13)、蛋白质代谢过程的负调控(GO:0051248,P-adjust=2.01e-13)。
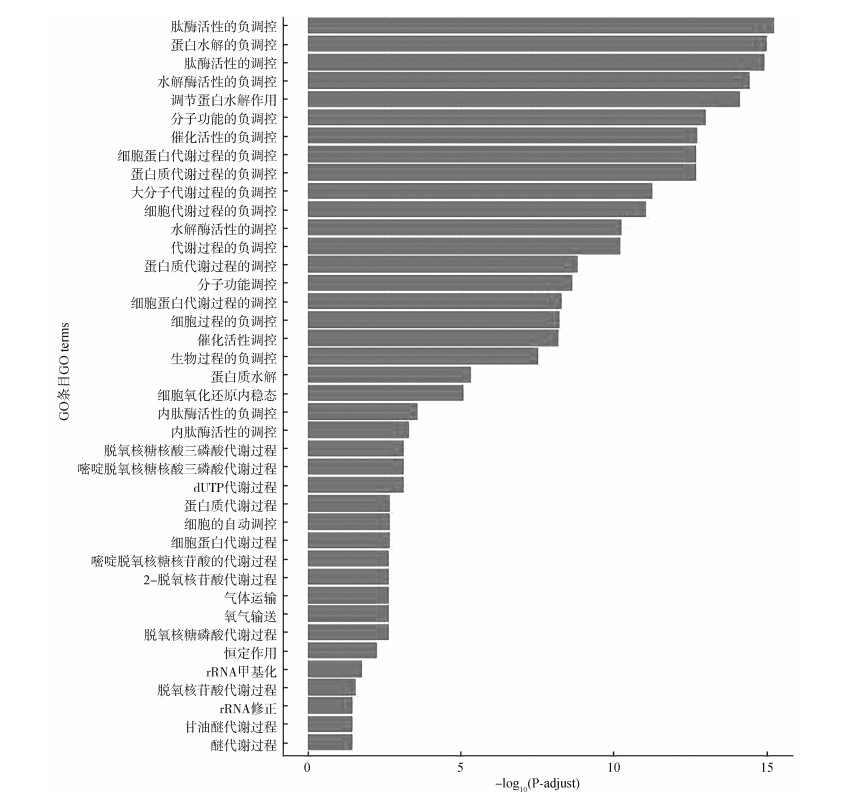
|
| 图 8 上调差异表达基因富集到生物过程GO功能条目 Fig. 8 Up-regulated differentially expressed genes enriched into biological process GO function entries |
| |
下调基因富集到3个显著的条目(图 9),均在细胞组分中,包括网格蛋白接头复合物(GO:0030131,P-adjust=0.018 770 6)、AP型膜外套接头复合物(GO:0030119,P-adjust=0.018 770 6)和网格蛋白外套(GO:0030118,P-adjust=0.020 008 4)。
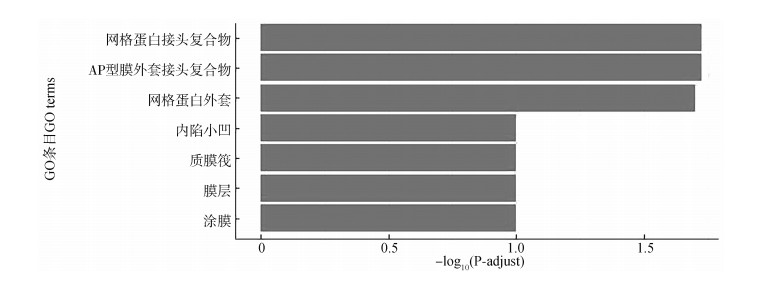
|
| 图 9 下调差异表达基因富集到细胞组分GO功能条目 Fig. 9 Down-regulated differentially expressed genes enriched into cell components GO function entry |
| |
为进一步了解差异表达基因所参与的通路,将中华蟾蜍差异表达基因进行了KEGG通路富集分析,用筛选出的差异表达基因序列,以中华蟾蜍的近缘物种非洲爪蟾和热带爪蟾的基因作为背景基因集,检测这些差异表达基因所富集的KEGG通路。
KEGG富集分析结果发现,差异表达基因显著富集到40个通路上,分为6个大类,分别是新陈代谢相关(metabolism)、人类疾病(human diseases)、生物系统(organismal systems)、环境信息过程(environment information processing)、细胞过程(celluar processes)和基因信息过程(genetic information processing)。
有大量差异表达基因富集到的通路主要为新陈代谢过程中的全局和概览通路(ko01100)、脂类代谢(ko00600)、糖的生物合成和代谢通路(ko00010);人类疾病相关的癌症通路(ko05200)、传染病通路(ko05130);生物系统中的内分泌系统相关通路(ko04915)、免疫系统相关通路(ko05321);环境信息过程中的信号转导通路(ko04012)、信号分子与相互作用(ko04060);细胞过程中的运输和分解代谢通路(ko04142);基因信息过程中的翻译相关通路(ko03010)。
针对这些基因富集到的通路,按照富集度和显著程度,挑选了其中最显著的前20个通路进行研究(图 10)。可以看到有大量差异表达基因富集到了核糖体通路上,富集程度较高的通路有肝细胞癌、肿瘤中MicroRNAs等与肿瘤相关的通路,而显著性较高的包括药物代谢、硝基甲苯降解、咖啡因代谢、鞘脂类的生物合成通路、糖磷脂生物合成以及黏蛋白型O-聚糖生物合成的相关通路。在KOBAS 3.0中,提交中华蟾蜍的差异表达基因序列,并分别以热带爪蟾和非洲爪蟾的基因作为背景基因集进行了KEGG富集(图 11、图 12),按照显著性P值排列,并筛选了P-adjust < 0.05的结果。其中显著性最高为核糖体的相关通路(P-adjust=6.56e-10)和鞘糖脂的生物合成通路(P-adjust=4.07e-06),其他富集到的包括酪氨酸、亚油酸、烟酸和烟酰胺、花生四烯酸、鞘脂类等化合物代谢合成相关通路,以及苯丙氨酸、酪氨酸和色氨酸的生物合成,缬氨酸、亮氨酸和异亮氨酸的降解通路,以及蟾酥的主要成分甾族化合物(辛秀兰等,2012)的生物合成(P-adjust=0.022 477 894)代谢通路。
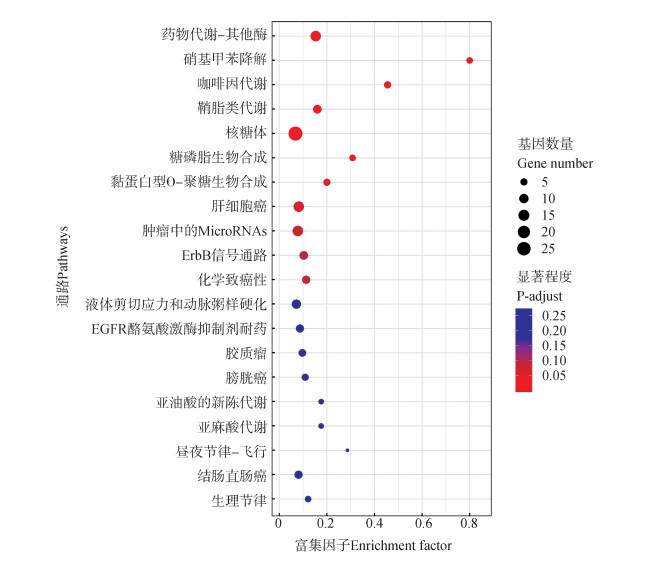
|
| 图 10 差异表达基因富集的最显著20个KEGG通路气泡图 Fig. 10 The most significant 20 KEGG pathway' bubble diagrams of differentially expressed gene enrichment |
| |
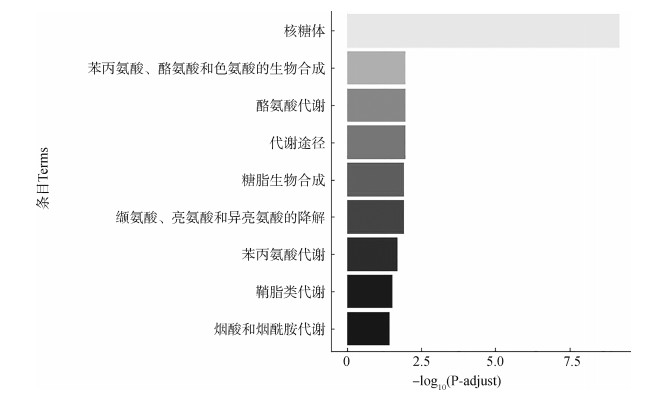
|
| 图 11 差异表达基因富集到热带爪蟾的结果 Fig. 11 Enrichment of differentially expressed genes into Xenopus tropicalis |
| |

|
| 图 12 差异表达基因富集到非洲爪蟾的结果 Fig. 12 Enrichment of differentially expressed genes into Xenopus laevis |
| |
以往研究表明了中华蟾蜍耳后腺特异分泌物——蟾酥的主要成分,并从临床医学、药学等角度肯定了其具有重要作用。本研究中使用RNA-Seq对蟾蜍多组织的转录组进行测序分析,得到了第一个由7个组织组成的较完整的中华蟾蜍转录组数据。基于此数据进行筛选获得了代表其单独基因的转录本,并对此转录组进行功能注释,完成了无参考基因组的中华蟾蜍转录组注释工作。对与NR数据库的比对结果进行了详细统计,发现大量被注释到的转录本比对到了电子注释预测的蛋白质上,这是由于目前两栖动物的研究尚未深入,有大量蛋白质的功能尚未经试验验证,目前仅能依据同源的蛋白质具有相似的功能来推断新蛋白质的潜在功能。
GO功能富集中,分子功能大致可分为2类,第一类为酶的调节相关通路,包括肽酶抑制物活性、肽酶调控分子活性、酶的抑制物活性、酶的调控分子活性、肽链内切酶调控分子活性、肽链内切酶抑制物活性、蛋白质二硫键异构酶活性等,从生物信息学的角度上验证了耳后腺组织存在大量调控和转录分泌蛋白活性及其调节物的基因的表达;第二类为其他一些组织特异性相关的通路,包括角质层的结构组成、结构分子活性、氧运输活性、用于转移糖基的转移酶活性、用于转移氨酰基的转移酶活性,这些通路来源于中华大蟾蜍耳后腺组织的特异性和对应的相关功能通路。
细胞组分多显著富集于细胞骨架、中间纤维、角蛋白纤维和胞外区域条目,而其中角蛋白纤维、中间纤维、高分子细胞骨架纤维、超分子纤维是属于同一条GO通路,这2条通路与细胞骨架有关,推测在细胞物质运输中,各类小泡和细胞器可沿着细胞骨架定向转运,因此推测在中华蟾蜍的耳后腺组织中激活了相应基因调控细胞骨架功能以更好完成其耳后腺的分泌功能。
生物过程显著富集到的通路大部分是肽酶活性的负调控、蛋白水解的负调节、肽酶活性的调节、水解酶活性的负调控、蛋白水解作用的调控、蛋白代谢过程的负调控、大分子代谢过程的负调控、细胞代谢过程的负调控、水解酶活性的调节、代谢过程的负调控,而这些通路多是与蛋白质水解、代谢相关过程的调控有关,且负调控过程居多。猜测中华蟾蜍耳后腺组织,除了需要维持着一种蛋白质大量分泌的环境,还需要储存其分泌的各类化学物质,因此虽然存在大量差异表达基因富集于蛋白质的合成、分泌与运输相关的通路,但也存在部分差异表达基因富集在维持耳后腺内分泌蛋白质稳定的相关功能,这更进一步表明中华蟾蜍耳后腺组织存在一定的机制维持这些化合物的分泌与稳定。在生物体中,酶参与药物生物转化,其代谢产物的活性通常低于母体药物或不活跃,但一些生物转化的产物可能具有增强的毒性作用,因此,生物转化一般包含“脱毒”和“中毒”的过程,决定生物体处理药物和化学物质能力的主要酶系统之一是细胞色素P450单加氧酶,其他酶系统包括脱氢酶、氧化酶、酯酶、还原酶,以及一些结合酶系统,包括葡萄糖醛酸转移酶、硫代转移酶、谷胱甘肽s-转移酶等也参与其中(Meyer et al., 1996)。这也能够解释在此次分析中,中华蟾蜍耳后腺组织的差异表达基因显著富集到了大量蛋白质合成、酶的调节相关、各类分子跨膜转运体以及各类前体物质的代谢合成通路,结果显示富集显著度最高的通路为药物代谢中的其他酶通路,其次为硝基甲苯降解,4-硝基甲苯为农药的中间体,推测这与农药污染相关,而中华蟾蜍有相应的降解硝基甲苯的适应性进化,这也与生物转化有一定的关联,环境和遗传因素导致药物代谢的个体间和个体内差异,并可能改变中毒反应和解毒反应之间的平衡。酪氨酸、亚油酸、烟酸和烟酰胺、花生四烯酸、鞘脂类等化合物作为蟾蜍分泌物蟾酥中各种物质的前体物质、修饰物质,其对应的相关代谢通路鞘糖脂的生物合成通路及核糖体相关通路出现在结果中,可以看出中华蟾蜍耳后腺作为外分泌腺体,从生物信息学分析角度上也证实其组织细胞中与代谢合成相关的基因高度活跃。特别注意的是,研究表明蟾酥的主要化学成分为各类甾族化合物,作为生物体内类固醇一类的化合物,是蟾酥的代表性成分(辛秀兰等,2012),这在差异基因富集分析中也得到一定的验证,例如,显著富集的甾类激素生物合成通路,而富集在这条通路上的基因,主要包括了羟基类固醇脱氢酶Ⅱ、细胞色素P450家族的各种基因。其中细胞色素P450作为一类末端加氧酶,能够参与生物体内的甾醇类激素合成过程(王斌,李德远,2009)。这些结果对于分析显著富集通路中差异表达基因,以及后续验证有重要意义。这些数据也为中华蟾蜍功能基因组分析和耳后腺各类化合物分泌相关研究提供了有价值的资源,有助于完善中华蟾蜍产物蟾酥、蟾衣的药用价值的研究。本研究只是一次对同一个体转录组差异的探索,可从不同个体方向进行下一阶段研究。
陈丽萍, 何丽莉, 巴云飞. 2011. 中华大蟾蜍抗菌肽对癌细胞的生长抑制作用[J]. 中国现代医生, 49(36): 24-27. |
杜联明, 李午佼, 沈福军, 等. 2014.大熊猫血液转录组分析[C].青海省动物学会.第三届中国西部动物学学术研讨会论文摘要集.
|
国家药典委员会. 2010. 中华人民共和国药典一部[M]. 北京: 中国医药科技出版社.
|
晋家正, 李午佼, 牟必琴, 等. 2018. 药用美洲大蠊全基因组测序分析[J]. 四川动物, 37(2): 121-126. |
罗展雄, 倪秉强, 郑青平, 等. 2009. 华蟾素注射液对晚期恶性肿瘤患者T淋巴细胞亚群及NK细胞的影响[J]. 现代肿瘤医学, 17(2): 340-341. |
彭贝, 巩仔鹏, 陈涛. 2011. 华蟾素注射液治疗肝癌的基础和临床研究进展[J]. 药物评价研究, 35(15): 2453-2455. |
祁云霞, 刘永斌, 荣威恒. 2011. 转录组研究新技术: RNA-Seq及其应用[J]. 遗传, 33(11): 1191-1202. |
孙崇峰, 范圣此, 罗毅, 等. 2018. 蟾酥化学成分及其人工合成的研究进展[J]. 中草药, 49(13): 3183-3192. |
王斌, 李德远. 2009. 细胞色素P450的结构与催化机理[J]. 有机化学, 29(4): 658-662. |
吴文英, 李丕鹏, 陆宇燕. 2011. 花背蟾蜍和中华蟾蜍皮肤腺体及耳后腺体的组织学研究[J]. 蛇志, 23(1): 20-25. |
辛秀兰, 张宝璟, 苏东海, 等. 2012. 中药蟾酥的药理作用研究进展[J]. 现代生物医学进展, 12(3): 588-600. |
杨宏梅, 陈涛. 2014. 华蟾素在消化系统肿瘤治疗中应用的研究进展[J]. 广东医学, 34(1): 63-66. |
张立, 普布次仁, 胡亚东, 等. 2020. 产双羔彭波半细毛羊发情期卵巢基因表达差异研究[J]. 四川动物, 39(2): 156-166. |
赵大洲, 陈继永, 秦勇, 等. 2006. 中华大蟾蜍蟾酥与蟾皮化学成分的比较分析[J]. 天津药学, 18(4): 21-24. |
Altschul SF, Gish W, Miller W, et al. 1990. Basic local alignment search tool[J]. Journal of Molecular Biology, 215(3): 403-410. |
Chao W, Zhang YH, Chai LH, et al. 2018. Transcriptomics provides mechanistic indicators of fluoride toxicology on endochondral ossification in the hind limb of Bufo gargarizans[J]. Aquatic Toxicology, 201: 138-150. DOI:10.1016/j.aquatox.2018.06.006 |
Conesa A, Götz S, García-Gómez JM, et al. 2005. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics, 21(18): 3674-3676. DOI:10.1093/bioinformatics/bti610 |
Davidson NM, Oshlack A. 2014. Corset: enabling differential gene expression analysis for de novo assembled transcriptomes[J]. Genome Biology, 15(7): 410. |
Grabherr MG, Haas BJ, Yassour M, et al. 2011. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 29(7): 644-652. DOI:10.1038/nbt.1883 |
Haas BJ, Papanicolaou A, Yassour M, et al. 2013. De novo transcript sequence reconstruction from RNA-Seq using Trinity platform for reference generation and analysis[J]. Nature Protrol, 8(8): 1494-1512. DOI:10.1038/nprot.2013.084 |
Kim D, Paggi JM, Park C, et al. 2019. Graph-based genome alignment and genotyping with HISAT2 and HISAT-genotype[J]. Nature Biotechnology, 37(8): 907-915. DOI:10.1038/s41587-019-0201-4 |
Meyer UA. 1996. Overview of enzymes of drug metabolism[J]. Journal of Pharmacokinetics and Biopharmaceutics, 24(5): 449-459. DOI:10.1007/BF02353473 |
Moriya Y, Itoh M, Okuda S, et al. 2007. KAAS: an automatic genome annotation and pathway reconstruction server[J]. Nucleic Acids Research, 35(2): 182-185. |
Pruitt KD, Tatusova T, Maglott DR. 2005. NCBI reference Sequence (RefSeq): a curated non-redundant sequence database of genomes, transcripts and proteins[J]. Nucleic Acids Research, 33(1): 501-504. |
Robinson MD, McCarthy DJ, Smyth GK. 2010. edgeR: a bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics, 26(1): 139-140. |
Simão FA, Waterhouse RM, Ioannidis P, et al. 2015. BUSCO: assessing genome assembly and annotation completeness with single-copy orthologs[J]. Bioinformatics, 31(19): 3210-3212. |
Xie C, Mao X, Huang Y, et al. 2011. KOBAS 2.0: a web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Research, 39(2): 316-322. |
Yang WZ, Qi Y, Lu B, et al. 2017. Gene expression variations in high-altitude adaptation: a case study of the Asiatic toad (Bufo gargarizans)[J/OL]. BMC Genetics, 18(1): 62[2020-05-30]. https://doi.org/10.1186/s12863-017-059-z.
|
Ye J, Fang L, Zheng H, et al. 2006. WEGO: a web tool for plotting GO annotations[J]. Nucleic Acids Research, 34(2): 293-297. |
Yu G, Wang L, Han Y, et al. 2012. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. OMICS: A Journal of Integrative Biology, 16(5): 284-287. DOI:10.1089/omi.2011.0118 |
 2020, Vol. 39
2020, Vol. 39




