扩展功能
文章信息
- 安志芳, 魏琳娜, 王志洁, 李苏华, 徐波, 李永晓, 魏莲, 魏登邦
- AN Zhifang, WEI Linna, WANG Zhijie, LI Suhua, XU Bo, LI Yongxiao, WEI Lian, WEI Dengbang
- 高原鼢鼠肝脏组织细胞周期相关基因的进化和表达
- Evolution and Expression of Liver Cell Cycle-Related Genes in Myospalax baileyi
- 四川动物, 2020, 39(1): 1-14
- Sichuan Journal of Zoology, 2020, 39(1): 1-14
- 10.11984/j.issn.1000-7083.20180349
-
文章历史
- 收稿日期: 2018-11-21
- 接受日期: 2019-11-27
2. 青海大学医学院, 西宁 810016;
3. 青海大学畜牧兽医科学院, 西宁 810016;
4. 青海大学生态环境工程学院, 西宁 810016
2. Medical College, Qinghai University, Xining 810016, China;
3. Academy of Animal Science and Veterinary Medicine, Qinghai University, Xining 810016, China;
4. College of Eco-Environmental Engineering, Qinghai University, Xining 810016, China
细胞周期是细胞生命活动的基本过程,主要分为G1、S、G2和M期(翟中和等,2000)。细胞周期的调控是通过各个时期特异的细胞周期调控因子实现的,主要包括3大类:细胞周期蛋白(cyclin),包括CyclinA、CyclinB、CyclinD、CyclinE等;细胞周期蛋白依赖性激酶(cyclin-dependent kinases,CDKs),包括CDK2、CDK4、CDK6等;细胞周期依赖性激酶抑制因子(cyclin-dependent kinases inhibitor,CDKI),包括p21、p16、p19、p27等。Cyclin是细胞周期正调控因子,CDKI是细胞周期负调控因子,CDK的活性受Cyclin正向调节,受CDKI负向调节,Cyclin会与对应的CDK结合,形成Cyclin-CDK复合物,进而决定细胞周期进程(Coats et al., 1996;Sherr & Roberts,1999;Balter & Vogel,2001)。
研究表明,低氧诱导细胞周期蛋白依赖性激酶抑制因子p16、p21、p27高表达,CyclinD1、CyclinE、CDK4、CDK2等低表达,进而引起细胞周期G1期阻滞(Krtolica et al., 1998;Gardner et al., 2001;Goda et al., 2003;Cazzalini et al., 2010;Hubbi et al., 2013)。细胞周期G1期阻滞是机体应对外界刺激的一种保护反应,可以提供充足的时间修复受损的DNA,从而避免突变基因遗传给子代细胞,避免肿瘤的发生(Kastan et al., 1991;Cazzalini et al., 2010)。低氧诱导p53下游Gadd45α、14-3-3-σ等基因的高表达以及CyclinB1等的低表达,引起细胞周期G2期阻滞(Hermeking et al., 1997;Innocente et al., 1999;Pucci et al., 2000;Hammer et al., 2007)。细胞周期G2期阻滞可以保证细胞有丝分裂过程中遗传物质分配的忠实性,保持基因组的稳定(Kastan et al., 1991;Cazzalini et al., 2010)。
地下鼠是一类终身生活在完全封闭的地下洞道中的啮齿动物,其地下洞道严重缺氧(Nevo, 1999, 2011;Nevo et al., 2001)。大量研究表明,在长期的进化过程中,地下鼠形成了一系列适应低氧的策略(Arieli & Ar,1981;Fang et al., 2014;Shao et al., 2015;Malik et al., 2016;Danial-Farran et al., 2017;Schmidt et al., 2017)。研究表明,地下鼠具有寿命长和抗肿瘤的特征,以色列鼹鼠Nannospalax galili和裸鼹鼠Heterocephalus glaber的寿命分别长达21年和30年,且在国外40多年的研究过程中,无论是野外采集的样本还是实验室养殖的样本,没有发现一例患有肿瘤的个体(Buffenstein & Jarvis,2002;Buffenstein,2008;Kim et al., 2011;Edrey et al., 2012;Tian et al., 2013)。研究发现,生活在湿润低氧的玄武岩地下洞道中的以色列鼹鼠肾脏组织中的p21基因表达水平显著高于生活在干旱高温的白垩土壤洞道中的个体(Zhao et al., 2016)。Miyawaki等(2015)的研究发现,裸鼹鼠会上调p16和p19的mRNA和蛋白水平。Fang等(2014)对以色列鼹鼠基因组和转录组的研究发现,低氧上调其脑组织中p21、p16和p19基因的表达,下调CyclinD1、CyclinG1、CyclinG2和CDK2等基因的表达。因此,地下鼠通过上调细胞周期依赖性激酶抑制因子的表达、下调细胞周期蛋白等的表达诱导细胞周期G1、G2期阻滞。
Kim等(2011)对裸鼹鼠基因组的研究发现,CyclinE1在第335号位点由丙氨酸(Ala)变异为缬氨酸(Val),该位点的变异可能与裸鼹鼠长寿抗肿瘤有关。Kim等(2011)和Miyawaki等(2015)的研究发现,裸鼹鼠中细胞周期因子p16和p19结构的变异对其功能的发挥起着重要作用。p53是一个肿瘤抑制因子,通过上调或下调其下游靶基因表达,从而调控细胞周期,进行DNA损伤修复(Agarwal et al., 1998;Vogelstein et al., 2000)。研究发现,以色列鼹鼠中p53结构的变异对其功能的发挥有重要的作用,p53在DNA结合域172位由精氨酸(Arg)突变为赖氨酸(Lys),这种变异使得p21基因高表达,细胞周期G1期阻滞(Ashur-Fabian et al., 2004;Avivi et al., 2007)。因此,地下鼠细胞周期的调控不仅与细胞周期相关基因的表达水平有关,而且与其结构的变异有关。
高原鼢鼠Myospalax baileyi是生活在青藏高原2 800~4 200 m地区的一种典型的地下鼠,其洞道内氧气的含量较同地区大气中的低20%(王祖望等,1979;樊乃昌,施银柱,1982;刘仁华,1995)。目前,在低氧条件下,关于高原鼢鼠肝脏组织中细胞周期相关基因的表达水平,以及细胞周期因子是否在长期低氧的作用下为了适应低氧环境发生了结构上变异的研究鲜见报道。因此,本文应用生物信息学方法对p53下游细胞周期相关因子p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-σ、Gadd45α、B99和CyclinB1进行了进化分析,并以SD大鼠Rattus norvegicus为对照,研究了这些基因在不同海拔(3 300 m和2 260 m)条件下高原鼢鼠肝脏组织中的表达模式。
1 材料与方法 1.1 实验动物16只高原鼢鼠捕捉于青海省西宁市湟源区宗家沟(101°17′E,36°43′N,海拔3 300 m),随机分为2组,每组8只。实验动物处理方法同An等(2018)的前期研究。第1组为高海拔组(海拔3 300 m),该组为宗家沟地区(海拔3 300 m)捕捉的样本;第2组为低海拔组(海拔2 260 m),将捕捉于宗家沟的高原鼢鼠置于西宁实验室(海拔2 260 m)饲养8 d。
16只SD大鼠购买于甘肃省兰州市[实验动物生产许可证号:SCXK(甘)2018-0002,实验动物使用许可证号:SYXK(甘)2018-0002],随机分为2组,每组8只。第1组为高海拔组(海拔3 300 m),将SD大鼠置于宗家沟地区(海拔3 300 m)饲养8 d;第2组为低海拔组(海拔2 260 m),将SD大鼠置于西宁实验室(海拔2 260 m)饲养8 d。
所有实验动物均用5%戊巴比妥钠麻醉,采集肝脏组织样品立即置于液氮中保存,采样过程中所涉及处理动物的措施均按照《实验动物管理条例(GB14923-2010)》执行。
1.2 细胞周期基因序列分析 1.2.1 序列获取从NCBI数据库下载以色列鼹鼠、大鼠、小鼠Mus musculus、橙腹草原田鼠Microtus ochrogaster、金仓鼠Mesocricetus auratus、黑线仓鼠Cricetulus griseus、突尼斯非洲跳鼠Jaculus jaculus、裸鼹鼠、达马拉鼹鼠Fukomys damarensis、豚鼠Cavia porcellus、毛丝鼠Chinchilla lanigera、智利八齿鼠Octodon degus、多纹黄鼠Ictidomys tridecemlineatus、北美鼠兔Ochotona princeps、野兔Oryctolagus cuniculus、家牛Bos taurus、牦牛Bos grunniens、绵羊Ovis aries、山羊Capra hircus、人Homo sapiens和黑猩猩Pan troglodytes的细胞周期相关基因p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-δ、Gadd45α、B99和CyclinB1的碱基及氨基酸序列。高原鼢鼠和高原鼠兔Ochotona curzoniae细胞周期相关基因的碱基序列利用Blast程序将实验室前期已测得的三代转录组数据库中全长非嵌合序列文件以及二代转录组数据库中的Trinity文件,构建本地Blast数据库,分别用以色列鼹鼠和北美鼠兔上述周期相关基因的编码区序列作为query文件进行Blast比对筛选,使用DNASTAR中的Lastergene程序(Burland,2000)拼接筛选出的基因片段,最终获得完整的编码区碱基序列,使用MEGA 7.0(Kumar et al., 2016)将所有比对以及拼接筛选出的序列进行比对,挑选与以色列鼹鼠和北美鼠兔同源性最高的一段序列作为目标基因的编码区序列,利用基因探索者软件将编码区序列翻译成氨基酸序列。
1.2.2 同源性分析选择与高原鼢鼠亲缘关系较近的2种地下鼠(以色列鼹鼠和裸鼹鼠)、大鼠、小鼠和人的p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-δ、Gadd45α、B99和CyclinB1基因编码区序列和氨基酸序列,用DNAMAN 9.0和MEGA 7.0进行同源性分析。
1.2.3 物种树构建方法从NCBI数据库中检索并下载1.2.1中包括高原鼢鼠和高原鼠兔在内的22个物种(啮齿目Rodentia 14个物种、兔形目Lagomorpha 3个物种、鲸偶蹄目Cetartiodactyla 3个物种和灵长目Primates 2个物种)的线粒体DNA全基因组序列,利用贝叶斯算法的MrBayes 3.2(Huelsenbeck & Ronquist 2001;Ronquist & Huelsenbeck,2003)构建贝叶斯系统进化树。采用PAUP 4.0(Swofford,2002)和Modeltest 2.3(Darriba et al., 2012)筛选最优模型,以赤池信息量准则(Alaike information criterion,AIC)(Bozdogan,1987;Brooks,1989)为标准进行最优模型的筛选与确定,采用马尔科夫链蒙特卡罗(Markov chain Monte Carlo,MCMC)运算(Gamerman & Lopes,2006),以随机树为起始,当运行2条和4条(1条冷链和3条热链)MCMC时,分歧频率的标准差稳定到小于0.01为止。在贝叶斯系统发育树构建的过程中,共进行106代的MCMC运算,设置每100代间隔进行一次抽样,舍弃起始老化样本数(burn-in)占总数的25%(Wang et al., 2016)。利用TreeGraph 2.7.1作图(Stöver & Müller,2010)。
1.2.4 选择压力分析分别将22个物种的p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-δ、Gadd45α、B99和CyclinB1基因编码区序列用ClustalX 1.81进行比对,比对后的结果利用MEGA 7.0进行格式转换,应用PAML 4.8中的CODEML程序(Zhang et al., 2005;Yang,2007),选用该程序中改进的基于最大似然法的分支位点模型(“test2”)对基因序列进行正向选择分析。将高原鼢鼠设为前景支,其余支系设为背景支,先用model A检测前景支中是否存在显著的正选择位点,检测标准为贝叶斯经验贝叶斯值大于0.95;再将控制文件(codeml.ctl)中的fix omega和omega值都设定为1,作为Null A进行第二次运算,提取2次运算得到的lnL值,分别记为lnL1和lnL0,计算其加倍差值2×ΔlnL。最后利用PAML 4.8中的Chi2程序,基于2×ΔlnL值计算模型的后验概率P值(df=1),当P < 0.05时,可认为此模型得到的结果较可靠。
1.2.5 趋同进化分析选用1.2.1中的22个物种,对p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-δ、Gadd45α、B99和CyclinB1基因的氨基酸序列进行地下鼠趋同进化分析。利用DNAMAN 9.0进行序列比对,使用PAML 4.8中的CODEML程序(Yang,2007)对每组蛋白序列进行了祖先序列重建。用重建后祖先位点的后验概率评估重建结果的准确性。由于祖先位点多态性会干扰后续分析,因此舍弃后验概率小于0.9的位点。应用converg 2(Zhang & Kumar,1997)检验趋同进化位点的显著性;应用MEGA 7.0中的ClustalW模式对22个物种的氨基酸序列进行比对,去掉所有空格,并按指定格式做出系统发育树,然后输入树文件和序列文件并应用Jones-Taylor-Thornton距离矩阵模型和泊松校正模型分别计算,参数使用默认值,舍弃P>0.05的结果(Zhang & Kumar,1997)。
1.2.6 变异位点对基因功能影响的评估由于SIFT数据中与高原鼢鼠亲缘关系最接近的物种为小鼠,因此从NCBI和Ensembl数据库中下载得到小鼠p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-δ、Gadd45α、B99和CyclinB1的蛋白ID,以该氨基酸序列作为query序列,采用“Sorting Tolerant From Intolerant(SIFT)”的algorithm程序评估氨基酸变异位点对该基因功能的影响,其中参数的设置使用默认值(Kumar et al., 2009)。
1.3 高原鼢鼠细胞周期基因表达水平测定利用总RNA抽提试剂盒(天根,中国)提取高原鼢鼠和SD大鼠肝脏组织总RNA,核酸蛋白含量检测仪测定A260/A280值(1.8 < A260/A280 < 2.0)及浓度(>0.4 μg·μL-1)。取1.9 μg总RNA采用First Strand cDNA Synthesis Kit(Thermo Fisher Scientific Inc.,美国)试剂盒制备cDNA。高原鼢鼠和SD大鼠基因序列的同源区利用Beacon Designer 7.7设计荧光定量特异性引物,并由南京金斯瑞生物科技有限公司进行引物合成,引物序列详见表 1。
| 引物 Primer |
引物序列 Primer sequences (5’-3’) |
| p21-F | AGGCACCATGTCCGATAA |
| p21-R | CCATGAGAGCATCACAGTC |
| CyclinD1-F | CGGATGAGAACAAGCAGAT |
| CyclinD1-R | GGTAGCAGGAGAGGAAGT |
| CDK6-F | TGTGGACCTCTGGAGTGTT |
| CDK6-R | AATGGGTTGGGCAGATTTGG |
| CyclinE-F | CTGGATGTTGGCTGCTTAGAAT |
| CyclinE-R | ACCACTGATAACCTGAGACCTT |
| CDK2-F | ATTCTGAGATTGACCAACTCTTCC |
| CDK2-R | GCTCCGTCCATCTTCATCC |
| 14-3-3-δ-F | GTGACTACTACCGCTACCT |
| 14-3-3-δ-R | ATCTCCTTCTTGCTGATGT |
| Gadd45α-F | GATCCAGACAACGTGGTC |
| Gadd45α-R | GATGTTGATGTCGTTCTCG |
| B99-F | GCACAGTTCCTCTCATCCAA |
| B99-R | TTCGCCTTCTCACGCTTC |
| CyclinB1-F | GCAGTGAGTGATGTGGATG |
| CyclinB1-R | CAGTCAATTAGGATGGCTCTC |
| β-actin-F | TCACCAACTGGGACGATATG |
| β-actin-R | GTTGGCCTTAGGGTTCAGAG |
按照Premix Ex Taq Version试剂盒(TaKaRa,日本)说明配置PCR反应体系进行PCR扩增,将产物切胶回收,用回收后的产物进行10倍梯度稀释,设定原PCR产物的浓度为1,分别稀释为1×10-1、1×10-2、1×10-3、1×10-4、1×10-5、1×10-6、1×10-7、1×10-8共8个梯度,作为标准品备用,以检测引物的扩增效率。荧光定量PCR按照SYBR©Premix Ex TaqTM Ⅱ试剂盒(TaKaRa,日本)在iQ5 Multicolor Real-Time PCR Detection System(Bio-Rad,美国)上进行。荧光定量PCR反应体系:12.5 μL SYBR Premix Ex Taq Ⅱ,10 μmol·μL-1特异引物F、R各1 μL,1 μL cDNA,加水至总体积25 μL。反应条件:95 ℃ 3 min;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 30 s,40个循环。Bio-Rad connect采集和分析数据,根据采集到的数据得到目的基因和管家基因浓度。每个样品的目的基因浓度除以其管家基因浓度,即为此样品基因校正后的相对含量(李筱等,2015;许利娜等,2015)。实验数据均以x±SD表示,采用SAS 8.2进行数据统计分析,采用独立样本t检验方法进行相对表达量的比较,P < 0.05为差异有统计学意义。
2 结果 2.1 高原鼢鼠细胞周期基因同源性比对结果同源性比对发现,高原鼢鼠细胞周期相关基因p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-σ、Gadd45α、B99和CyclinB1编码区及氨基酸序列与以色列鼹鼠同源性最高,达到90%以上(表 2)。
| 基因 Gene |
细胞周期基因(氨基酸)序列同源性Gene (Amino acid) sequence homologies of cell cycle-related genes/% | ||||
| 以色列鼹鼠 Nannospalax galili |
裸鼹鼠 Heterocephalus glaber |
大鼠 Rattus norvegicus |
小鼠 Mus musculus |
人 Homo sapiens |
|
| p21 | 92.43(95.68) | 81.33(80.00) | 81.53(85.45) | 81.10(86.50) | 78.79(78.66) |
| CyclinD1 | 95.06(97.64) | 90.99(95.59) | 89.98(95.25) | 90.65(95.25) | 90.20(93.56) |
| CDK6 | 95.92(99.08) | 92.76(97.55) | 91.95(96.63) | 91.64(96.32) | 91.95(97.55) |
| CyclinE | 94.34(96.35) | 82.49(84.22) | 88.75(91.24) | 86.81(89.54) | 81.88(82.73) |
| CDK2 | 95.96(99.33) | 91.42(97.65) | 92.53(99.33) | 93.65(99.33) | 91.64(97.99) |
| 14-3-3-σ | 93.57(97.58) | 92.37(95.56) | 92.10(97.58) | 92.37(97.58) | 92.50(97.18) |
| Gadd45α | 94.18(96.97) | 84.94(90.30) | 89.56(92.73) | 91.57(93.94) | 92.37(94.55) |
| B99 | 90.25(92.74) | 82.96(83.10) | 84.45(86.40) | 85.81(87.90) | 80.57(81.53) |
| CyclinB1 | 94.48(95.56) | 87.13(87.18) | 85.39(88.08) | 86.31(89.10) | 88.12(88.94) |
根据选取的22个物种的线粒体DNA全基因组序列构建物种进化树。DAMBE饱和度检测结果显示,指标分数(index score,ISS)低于临界分数(critical score,TSS.C)(ISS=0.685,TSS.C=0.830,P < 0.01),说明核酸替换未达到饱和,适合建树。最佳DNA进化替代模型采用with gamma-distributed rate variation across sites和a proportion of invariable sites的GTR模型。所得的贝叶斯树各支的支持率都大于85%(图 1),可以用于后续研究。
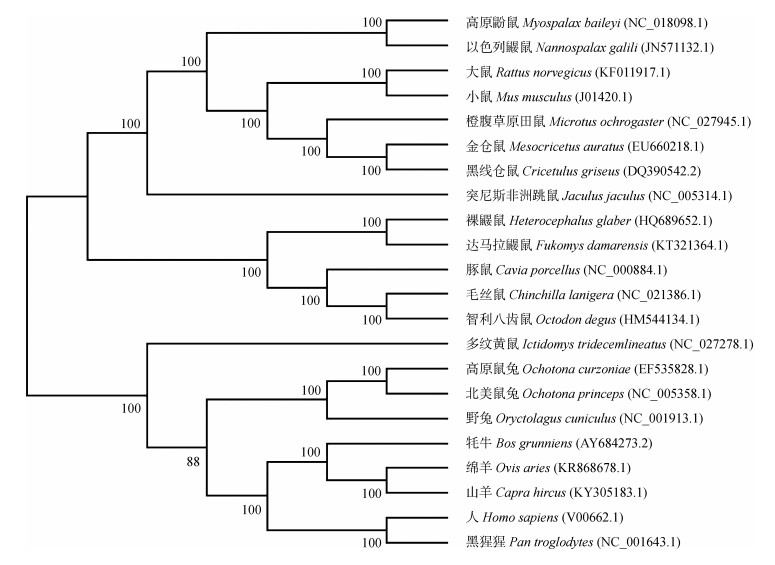
|
| 图 1 22种哺乳动物的mtDNA系统进化树 Fig. 1 Phylogenetic tree of 22 mammal species based on mtDNA |
| |
基于图 1构建的22个物种进化树,检测高原鼢鼠细胞周期相关因子p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-σ、Gadd45α、B99和CyclinB1是否具有正向选择位点。结果表明,CDK2有2个潜在的正向选择位点,分别为第330号位点的苏氨酸(Thr)和第341号位点的异亮氨酸(Ile);B99有2个潜在的正向选择位点,为第168号位点的谷氨酰胺(Gln)和第447号位点的丝氨酸(Ser);Gadd45α有2个潜在的正向选择位点,分别为第8号位点的丝氨酸(Ser)和第14号位点的赖氨酸(Lys);CyclinB1有1个潜在的正向选择位点,为第396号位点的精氨酸(Arg),但是似然比检验法显示这些位点差异不显著(2ΔlnL=0,P>0.05)(表 3)。
| 基因 Genes |
模型 Model |
参数估计 Estimate of parameters |
似然率值 Likelihood value (-lnL)a |
模型比较 Model comparison |
正向选择位点 Positively selected sites |
2倍差值(P值) 2ΔlnL (P-value) |
| p21 | Null A | p0=0.893 05,p1=0.106 95,(p2+p3=0),ω0=0.117 03,ω1=1.000 00,ω2=1.000 00 | -3 846.97 | |||
| Model A | p0=0.886 59,p1=0.107 18,(p2+p3=0.006 03),ω0=0.116 33,ω1=1.000 00,ω2=1.000 00 | -3 846.97 | Model A vs. Null A | 0 (P=1) | ||
| CyclinD1 | Null A | p0=0.983 85,p1=0.016 15,(p2+p3=0),ω0=0.026 75,ω1=1.000 00,ω2=1.000 00 | -3 665.45 | |||
| Model A | p0=0.983 85,p1=0.016 15,(p2+p3=0),ω0=0.026 75,ω1=1.000 00,ω2=1.000 00 | -3 665.45 | Model A vs. Null A | 0 (P=1) | ||
| CyclinE1 | Null A | p0=0.821 71,p1=0.178 29,(p2+p3=0),ω0=0.089 54,ω1=1.000 00,ω2=1.000 00 | -8 116.12 | |||
| Model A | p0=0.821 71,p1=0.178 29,(p2+p3=0),ω0=0.089 51,ω1=1.000 00,ω2=1.000 00 | -8 116.12 | Model A vs. Null A | 0 (P=1) | ||
| CDK6 | Null A | p0=0.961 45,p1=0.038 55,(p2+p3=0),ω0=0.023 56,ω1=1.000 00,ω2=1.000 00 | -4 241.5 | |||
| Model A | p0=0.961 45,p1=0.038 55,(p2+p3=0),ω0=0.023 56,ω1=1.000 00,ω2=1.000 00 | -4 241.5 | Model A vs. Null A | 0 (P=1) | ||
| CDK2 | Null A | p0=0.955 65,p1=0.015 96,(p2+p3=0.028 39),ω0=0.021 66,ω1=1.000 00,ω2=1.000 00 | -4 057.77 | |||
| Model A | p0=0.955 65,p1=0.015 96,(p2+p3=0.028 39),ω0=0.021 66,ω1=1.000 00,ω2=1.000 00 | -4 057.77 | Model A vs. Null A | 330 T,341 I | 0 (P=1) | |
| 14-3-3-σ | Null A | p0=0.981 46,p1=0.014 45,(p2+p3=0.004 09),ω0=0.018 25,ω1=1.000 00,ω2=1.000 00 | -3 398.51 | |||
| Model A | p0=0.981 46,p1=0.014 45,(p2+p3=0.004 09),ω0=0.018 24,ω1=1.000 00,ω2=1.000 00 | -3 398.51 | Model A vs. Null A | 0 (P=1) | ||
| B99 | Null A | p0=0.452 53,p1=0.481 07,(p2+p3=0.066 41),ω0=0.162 85,ω1=1.000 00,ω2=1.000 00 | -22 343.53 | |||
| Model A | p0=0.471 77,p1=0.503 55,(p2+p3=0.024 68),ω0=0.162 22,ω1=1.000 00,ω2=4.848 87 | -22 342.69 | Model A vs. Null A | 168 Q,447 S | 1.68 (P=0.20) | |
| Gadd45α | Null A | p0=0.882 16,p1=0.054 69,(p2+p3=0.063 16),ω0=0.025 88,ω1=1.000 00,ω2=1.000 00 | -2 285.55 | |||
| Model A | p0=0.882 16,p1=0.054 69,(p2+p3=0.063 16),ω0=0.025 88,ω1=1.000 00,ω2=1.000 00 | -2 285.55 | Model A vs. Null A | 8 S,14 K | 0 (P=1) | |
| CyclinB1 | Null A | p0=0.866 59,p1=0.133 41,(p2+p3=0),ω0=0.066 03,ω1=1.000 00,ω2=1.000 00 | -7 371.62 | |||
| Model A | p0=0.866 59,p1=0.133 41,(p2+p3=0),ω0=0.066 03,ω1=1.000 00,ω2=1.000 00 | -7 371.62 | Model A vs. Null A | 396 R | 0 (P=1) |
22个物种细胞周期相关因子p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-σ、Gadd45α、B99和CyclinB1(由于NCBI中无牦牛p21和CyclinB1基因序列,分别选用家牛对应序列:NP_001092428.1、NP_001039337.1)在地下鼠中的趋同进化分析显示,高原鼢鼠和以色列鼹鼠p21在第27号位点由谷氨酸(Glu,E)变异为丙氨酸(Ala,A)(图 2:A);CyclinD1第20、24、143和266号位点分别由苏氨酸(Thr,T)、天冬酰胺(Asn,N)、亮氨酸(Leu,L)和亮氨酸(Leu,L)变异为丝氨酸(Ser,S)、苏氨酸(Thr,T)、异亮氨酸(Ile,I)和谷氨酰胺(Gln,Q)(图 2:B);CyclinE第86、92、125、126和402号位点分别由亮氨酸(Leu,L)、苏氨酸(Thr,T)、谷氨酸(Glu,E)、赖氨酸(Lys,K)和赖氨酸(Lys,K)变异为缬氨酸(Val,V)、赖氨酸(Lys,K)、天冬氨酸(Asp,D)、天冬酰胺(Asn,N)和谷氨酰胺(Gln,Q)(图 2:C);CyclinB1第105、135和370号位点分别由缬氨酸(Val,V)、谷氨酸(Glu,E)和丝氨酸(Ser,S)变异为天冬酰胺(Asn,N)、天冬氨酸(Asp,D)和半胱氨酸(Cys,C)(图 2:D),而CDK6、CDK2、14-3-3-σ、Gadd45α和B99在高原鼢鼠和以色列鼹鼠中没有趋同进化位点。
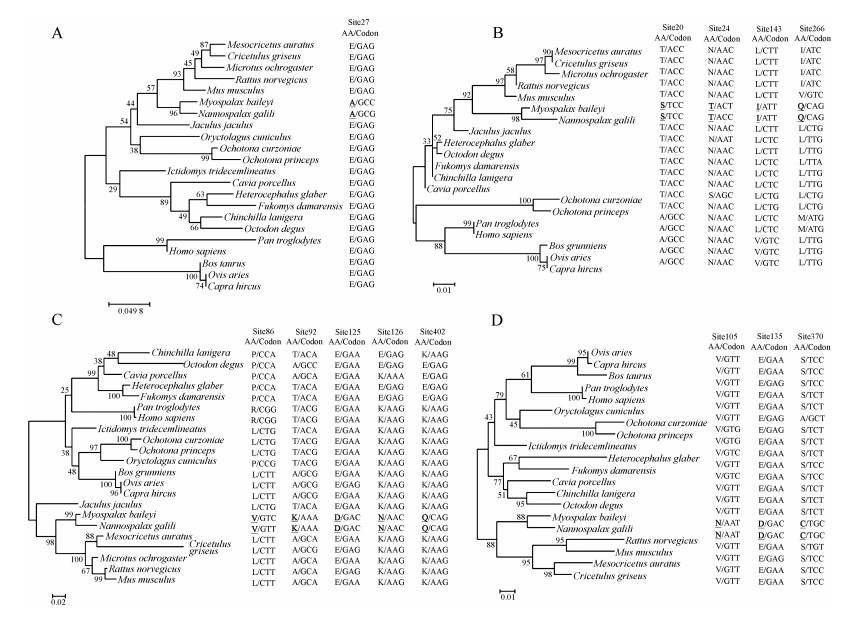
|
| 图 2 最大似然法构建的p21(A)、CyclinD1(B)、CyclinE(C)和CyclinB1(D)系统发育树和进化位点 Fig. 2 Maximum-likelihood tree of the p21(A), CyclinD1(B), CyclinE(C) and CyclinB1(D) gene sequences and the convergent sites 系统发育树后的序列分别表示氨基酸和对应的碱基序列,加粗及下划线的氨基酸表示高原鼢鼠和以色列鼹鼠共有的趋同进化位点 Amino acids and codons of convergent sites are shown in the right part of each panel, and amino acids in Myospalax baileyi and Nannospalax galili are highlighted in bold and underline |
| |
SIFT评估结果表明,高原鼢鼠与以色列鼹鼠细胞周期相关因子的趋同进化位点中,只有p21第27号位点丙氨酸(Ala,A)和CyclinB1第105号位点天冬酰胺(Asn,N)的变异对其功能有显著影响,其余变异位点对基因功能均没有影响(表 4)。
| 蛋白 Protein |
变异位点 Substitution |
SIFT分值 SIFT score |
SIFT预测结果 SIFT prediction (cutoff=0.05) |
| p21 | E27A | 0.004 | 影响 |
| CyclinD1 | T20S | 0.080 | 良性 |
| N24T | 0.190 | 良性 | |
| L143I | 0.068 | 良性 | |
| L266Q | 0.689 | 良性 | |
| CyclinE | L86V | 0.133 | 良性 |
| T92K | 0.805 | 良性 | |
| E125D | 0.208 | 良性 | |
| K126N | 0.331 | 良性 | |
| K402Q | 0.097 | 良性 | |
| CDK2 | A330T | 0.553 | 良性 |
| V341I | 0.369 | 良性 | |
| Gadd45α | A8S | 0.313 | 良性 |
| R15K | 0.575 | 良性 | |
| B99 | L168Q | 0.106 | 良性 |
| CyclinB1 | V105N | 0.025 | 影响 |
| E135D | 0.057 | 良性 | |
| S370C | 0.062 | 良性 | |
| K396R | 0.374 | 良性 |
荧光定量PCR结果表明,与低海拔条件(2 260 m)相比,在高海拔条件(3 300 m)下,高原鼢鼠肝脏组织中细胞周期基因p21的表达水平极显著高于低海拔(2 260 m)下的表达水平(P < 0.01),p21下游基因CyclinD1、CDK6、CyclinE和CDK2的表达水平显著低于低海拔(2 260 m)下的表达水平(P < 0.05),而在SD大鼠肝脏组织中上述基因的表达水平差异都没有统计学意义(P>0.05)。高原鼢鼠和SD大鼠肝脏组织中细胞周期基因14-3-3-δ、Gadd45α、B99和CyclinB1表达水平间的差异无统计学意义(P>0.05)(图 3)。在不同海拔条件下,高原鼢鼠肝脏组织中细胞周期相关基因p21、CyclinD1、CDK6、CyclinE、CDK2、14-3-3-σ、Gadd45α、B99和CyclinB1的表达水平均极显著高于SD大鼠(P < 0.01)(图 3)。

|
| 图 3 高原鼢鼠和SD大鼠肝脏组织中细胞周期基因在不同海拔条件下的mRNA表达水平 Fig. 3 Quantification of the mRNA levels of cell cycle-related genes in the liver of Myospalax baileyi and Rattus norvegicus under different altitudes * P < 0.05,**P < 0.01,ns. P>0.05 |
| |
G1期是细胞周期的第一阶段,在这时期细胞开始合成生长所需的RNA、蛋白质、糖类、脂质等,同时细胞体积显著增大,属于细胞的生长期(翟中和等,2000)。研究表明,低氧诱导细胞周期蛋白依赖性激酶抑制因子p16、p21、p27高表达,CyclinD1、CyclinE、CDK4、CDK2等低表达,进而引起细胞周期G1期阻滞(Krtolica et al., 1998;Gardner et al., 2001;Goda et al., 2003;Cazzalini et al., 2010;Hubbi et al., 2013)。细胞周期G1期阻滞是机体应对外界刺激的一种保护反应,可以提供充足的时间修复受损的DNA,从而避免突变基因遗传给子代细胞,避免肿瘤的发生(Kastan et al., 1991;Cazzalini et al., 2010)。研究表明,p21基因的高表达是引起细胞周期G1期阻滞的直接原因(Cazzalini et al., 2010)。p21作为细胞周期负调控因子,在其N端第21~26位及第49~72位的氨基酸分别与细胞周期蛋白和细胞周期蛋白依赖性激酶结合,抑制Cyclin-CDK复合物的活性,进而引起视网膜母细胞瘤蛋白不能发生磷酸化,阻止转录因子E2F的释放,使得参与细胞周期的CyclinD1、CyclinE等不能表达,阻止细胞从G1期进入S期,导致细胞周期G1期阻滞(Sherr & Roberts, 1995, 1999;Gartel & Radhakrishnan,2005)。本研究结果表明,在高原鼢鼠肝脏组织中,与G1期调控相关的基因p21表达水平随海拔的升高显著升高,p21下游基因CyclinD1、CyclinE、CDK6和CDK2的表达水平随海拔的升高显著降低,而在SD大鼠中没有差异;在高原鼢鼠和SD大鼠肝脏组织中,与G2期调控相关的基因Gadd45α、B99、14-3-3-δ和CyclinB1的表达水平随海拔改变不发生变化。因此,高原鼢鼠通过长期的低氧适应,在高海拔条件下,p21基因的高表达抑制了下游CyclinD1、CyclinE、CDK6和CDK2基因的表达,从而诱导细胞周期G1期阻滞,提供了充足的时间修复受损的DNA,保证了DNA复制的准确性。
细胞周期调控不仅与细胞周期相关基因的表达水平有关,而且与其结构的突变有关。p21是细胞周期负调控因子,主要有2个功能结构域,N端第21~26位及第49~72位的氨基酸分别与Cyclin和CDK结合,抑制Cyclin-CDK复合物的活性;C端第124~164位氨基酸与增殖细胞核抗原(proliferating cell nuclear antigen,PCNA)结合,使得PCNA不能与DNA聚合酶δ结合,抑制DNA的复制(Luo et al., 1995;Johnson & Walker,1999)。研究发现,p21第31号位点由极性不带电的丝氨酸(Ser)变异为极性带正电的精氨酸(Arg),这会使得p21蛋白结构发生变化,该位点的变异与乳腺癌、宫颈癌等的易感性有关(Lukas et al., 1997)。在人类乳腺癌中,p21第94号位点由极性带正电的精氨酸(Arg)变异为非极性的色氨酸(Trp),该位点的变异使得p21抑制CDK的能力减弱,但是与PCNA结合的能力没有发生改变(Balbín et al., 1996)。本研究的SIFT评估结果表明,高原鼢鼠p21第27号位点的变异对其功能有显著影响,该位点由极性带负电的谷氨酸(Glu)变异为非极性的丙氨酸(Ala),位于Cyclin结合域附近,其改变了该区域原本带负电的局部环境,可能引起p21对Cyclin和CDK的结合力增强,从而使得p21阻滞细胞周期的功能增强。
CyclinB1属于细胞周期蛋白,在其第201~288位有一个高度保守的细胞周期蛋白盒序列,该结构域与CDK1氨基末端的PSTAIR结构形成CyclinB1-CDK1复合物,该复合物被称为有丝分裂促进因子;CyclinB1位于第42~50位的氨基酸序列为降解盒结构,参与细胞周期蛋白的自身降解(Glotzer et al., 1991;Noble et al., 1997);此外,CyclinB1还有2个控制CyclinB1-CDK1复合物胞质-核穿梭的结构域,分别是位于第155~170位的核定位区域(nuclear localization signal,NLS)和N端第78~127位的胞质滞留结构域(cytoplasmic retention signal,CRS),NLS结构域的作用是将CyclinB1-CDK1复合物运送至细胞核内,CRS结构域是确保有丝分裂前CyclinB1-CDK1复合物在细胞质中的定位(Robbins et al., 1991;Pines & Hunter,1994)。研究表明,位于CRS结构域中丝氨酸(Ser)磷酸化位点突变为丙氨酸(Ala)时,CyclinB1活性消失,而当突变为谷氨酸(Glu)时,可以增强其活性(Li et al., 1997)。SIFT评估结果表明,高原鼢鼠CyclinB1位于CRS结构域中的第105号位点的变异对其功能有显著影响,该位点由非极性的缬氨酸(Val)变异为极性的天冬酰胺(Asn),这种变异可能会导致该点附近的静电势能发生变化,影响CyclinB1-CDK1复合物从细胞核运往细胞质,从而增强细胞周期阻滞作用。
综上所述,高原鼢鼠经过长期的低氧适应,通过上调p21基因的表达抑制下游CyclinD1、CyclinE、CDK6和CDK2基因的表达,导致细胞周期G1期阻滞,从而提供充足的时间修复受损的DNA,保证了DNA复制的准确性;同时高原鼢鼠肝脏组织中细胞周期的调控不仅与细胞周期基因的表达水平有关,而且可能与细胞周期因子p21的第27号位点和CyclinB1的第105号位点的变异有关。
樊乃昌, 施银柱. 1982. 中国鼢鼠(Eospalax)亚属分类研究[J]. 兽类学报, 2(2): 183-199. |
李筱, 魏莲, 汪洋, 等. 2015. 高原鼠兔心脏中Ldh-c基因的表达及其对无氧糖酵解水平的影响[J]. 生理学报, 67(3): 312-318. |
刘仁华. 1995. 中国鼢鼠的分类及地理区划[J]. 国土与自然资源研究, 3: 54-55. |
王祖望, 曾缙祥, 韩永才. 1979. 高原鼠兔和中华鼢鼠气体代谢的研究[J]. 动物学报, 25(1): 75-85. |
许利娜, 魏莲, 汪洋, 等. 2015. 高原鼠兔脑组织中精子特异性乳酸脱氢酶的作用[J]. 兽类学报, 35(4): 431-437. |
翟中和, 王喜忠, 丁明孝. 2000. 细胞生物学[M]. 北京: 高等教育出版社.
|
Agarwal ML, Taylor WR, Chernov MV, et al. 1998. The p53 network[J]. Journal of Biological Chemistry, 273(1): 1-4. DOI:10.1074/jbc.273.1.1 |
An ZF, Zhao K, Wei LN, et al. 2018. p53 gene cloning and response to hypoxia in the plateau zokor, Myospalax baileyi[J]. Animal Biology, 68(3): 289-308. DOI:10.1163/15707563-18000004 |
Arieli R, Ar A. 1981. Heart rate responses of the mole rat (Spalax ehrenbergi) in hypercapnic, hypoxic, and cold conditions[J]. Physiological Zoology, 54(1): 14-21. |
Ashur-Fabian O, Avivi A, Trakhtenbrot L, et al. 2004. Evolution of p53 in hypoxia-stressed Spalax mimics human tumor mutation[J]. Proceedings of the National Academy of Sciences of the United States of America, 101(33): 12236-12241. DOI:10.1073/pnas.0404998101 |
Avivi A, Ashur-Fabian O, Joel A, et al. 2007. P53 in blind subterranean mole rats-loss-of-function versus gain-of-function activities on newly cloned Spalax target genes[J]. Oncogene, 26(17): 2507-2512. DOI:10.1038/sj.onc.1210045 |
Balbín M, Hannon GJ, Pendás AM, et al. 1996. Functional analysis of a p21WAF1, CIP1, SDI1 mutant (Arg94→Trp) identified in a human breast carcinoma evidence that the mutation impairs the ability of p21 to inhibit cyclin-dependent kinases[J]. Journal of Biological Chemistry, 271(26): 15782-15786. DOI:10.1074/jbc.271.26.15782 |
Balter M, Vogel G. 2001. Cycling toward Stockholm[J]. Science, 294(5542): 502-503. DOI:10.1126/science.294.5542.502 |
Bozdogan H. 1987. Model selection and Akaike's information criterion (AIC):the general theory and its analytical extensions[J]. Psychometrika, 52(3): 345-370. DOI:10.1007/BF02294361 |
Brooks DG. 1989. Akaike information criterion statistics[J]. Technometrics, 31(2): 270-271. DOI:10.1080/00401706.1989.10488538 |
Buffenstein R, Jarvis JUM. 2002. The naked mole rat-a new record for the oldest living rodent[J/OL]. Science of Aging Knowledge Environment: Sage KE, 2002(21): pe7[2018-08-10]. https://www.sageke.sciencemag.org/cgi/content/full/2002/21/pe7. DOI: 10.1126/sageke.2002.21.pe7.
|
Buffenstein R. 2008. Negligible senescence in the longest living rodent, the naked mole-rat:insights from a successfully aging species[J]. Journal of Comparative Physiology B, 178(4): 439-445. DOI:10.1007/s00360-007-0237-5 |
Burland TG. 2000. DNASTAR's lasergene sequence analysis software[M]//Stephen M, Stephen AK. Bioinformatics methods and protocols. Totowa, NJ: Humana Press.
|
Cazzalini O, Scovassi AI, Savio M, et al. 2010. Multiple roles of the cell cycle inhibitor p21 CDKN1A in the DNA damage response[J]. Reviews in Mutation Research, 704(1): 12-20. |
Coats S, Flanagan WM, Nourse J, et al. 1996. Requirement of p27Kip1 for restriction point control of the fibroblast cell cycle[J]. Science, 272(5263): 877-880. DOI:10.1126/science.272.5263.877 |
Danial-Farran N, Nasser NJ, Beiles A, et al. 2017. Adaptive evolution of coagulation and blood properties in hypoxia tolerant Spalax in Israel[J]. Journal of Zoology, 303(3): 226-235. DOI:10.1111/jzo.12480 |
Darriba D, Taboada GL, Doallo R, et al. 2012. jModelTest 2: more models, new heuristics and parallel computing[J/OL]. Nature Methods, 9(8): 772[2018-07-02]. https://doi.org/10.1038/nmeth.2109.
|
Edrey YH, Casper D, Huchon D, et al. 2012. Sustained high levels of neuregulin-1 in the longest-lived rodents; a key determinant of rodent longevity[J]. Aging Cell, 11(2): 213-222. DOI:10.1111/j.1474-9726.2011.00772.x |
Fang XD, Nevo E, Han LJ, et al. 2014. Genome-wide adaptive complexes to underground stresses in blind mole rats Spalax[J/OL]. Nature Communications, 5: 3966[2018-09-01]. https://www.nature.com/articles/ncomms4966. DOI: 10.1038/ncomms4966.
|
Gamerman D, Lopes HF. 2006. Markov chain Monte Carlo:stochastic simulation for Bayesian inference[M]. Florida, USA: Chapman and Hall/CRC.
|
Gardner LB, Li Q, Park MS, et al. 2001. Hypoxia inhibits G1/S transition through regulation of p27 expression[J]. Journal of Biological Chemistry, 276(11): 7919-7926. DOI:10.1074/jbc.M010189200 |
Gartel AL, Radhakrishnan SK. 2005. Lost in transcription:p21 repression, mechanisms, and consequences[J]. Cancer Research, 65(10): 3980-3985. DOI:10.1158/0008-5472.CAN-04-3995 |
Glotzer M, Murray AW, Kirschner MW. 1991. Cyclin is degraded by the ubiquitin pathway[J]. Nature, 349(6305): 132. DOI:10.1038/349132a0 |
Goda N, Ryan HE, Khadivi B, et al. 2003. Hypoxia-inducible factor 1α is essential for cell cycle arrest during hypoxia[J]. Molecular and Cellular Biology, 23(1): 359-369. DOI:10.1128/MCB.23.1.359-369.2003 |
Hammer S, To KK, Yoo YG, et al. 2007. Hypoxic suppression of the cell cycle gene CDC25A in tumor cells[J]. Cell Cycle, 6(15): 1919-1926. DOI:10.4161/cc.6.15.4515 |
Hermeking H, Lengauer C, Polyak K, et al. 1997. 14-3-3σ is a p53-regulated inhibitor of G2/M progression[J]. Molecular Cell, 1(1): 3-11. DOI:10.1016/S1097-2765(00)80002-7 |
Hubbi ME, Gilkes DM, Rey S, et al. 2013. A nontranscriptional role for HIF-1α as a direct inhibitor of DNA replication[J/OL]. Science Signaling, 6(262): ra10[2018-09-01]. https://stke.sciencemag.org/content/6/262/ra10. DOI: 10.1126/scisignal.2003417.
|
Huelsenbeck JP, Ronquist F. 2001. Mrbayes:Bayesian inference of phylogenetic trees[J]. Bioinformatics, 17(8): 754-755. DOI:10.1093/bioinformatics/17.8.754 |
Innocente SA, Abrahamson JLA, Cogswell JP, et al. 1999. p53 regulates a G2 checkpoint through cyclin B1[J]. Proceedings of the National Academy of Sciences of the United States, 96(5): 2147-2152. DOI:10.1073/pnas.96.5.2147 |
Johnson DG, Walker CL. 1999. Cyclins and cell cycle checkpoints[J]. Annual Review of Pharmacology and Toxicology, 39: 295-312. DOI:10.1146/annurev.pharmtox.39.1.295 |
Kastan MB, Onyekwere O, Sidransky D, et al. 1991. Participation of p53 protein in the cellular response to DNA damage[J]. Cancer Research, 51(23 Part 1): 6304-6311. |
Kim EB, Fang X, Fushan AA, et al. 2011. Genome sequencing reveals insights into physiology and longevity of the naked mole rat[J]. Nature, 479(7372): 223-227. DOI:10.1038/nature10533 |
Krtolica A, Krucher NA, Ludlow JW. 1998. Hypoxia-induced pRB hypophosphorylation results from downregulation of CDK and upregulation of PP1 activities[J]. Oncogene, 17(18): 2295-2304. DOI:10.1038/sj.onc.1202159 |
Kumar P, Henikoff S, Ng PC. 2009. Predicting the effects of coding non-synonymous variants on protein function using the SIFT algorithm[J]. Nature Protocols, 4(7): 1073. DOI:10.1038/nprot.2009.86 |
Kumar S, Stecher G, Tamura K. 2016. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
Li J, Meyer AN, Donoghue DJ. 1997. Nuclear localization of cyclin B1 mediates its biological activity and is regulated by phosphorylation[J]. Proceedings of the National Academy of Sciences of the United States, 94(2): 502-507. DOI:10.1073/pnas.94.2.502 |
Lukas J, Groshen S, Saffari B, et al. 1997. WAF1/Cip1 gene polymorphism and expression in carcinomas of the breast, ovary, and endometrium[J]. The American Journal of Pathology, 150(1): 167. |
Luo Y, Hurwitz J, Massagué J. 1995. Cell-cycle inhibition by independent CDK and PCNA binding domains in p21Cip1[J]. Nature, 375(6527): 159-161. DOI:10.1038/375159a0 |
Malik A, Domankevich V, Lijuan H, et al. 2016. Genome maintenance and bioenergetics of the long-lived hypoxia-tolerant and cancer-resistant blind mole rat, Spalax: a cross-species analysis of brain transcriptome[J/OL]. Scientific Reports, 6: 38624[2018-08-01]. https://www.nature.com/articles/srep38624. DOI: 10.1038/srep38624.
|
Miyawaki S, Kawamura Y, Hachiya T, et al. 2015. Molecular cloning and characterization of the INK4a and ARF genes in naked mole-rat[J]. Inflammation and Regeneration, 35(1): 42-50. DOI:10.2492/inflammregen.35.042 |
Nevo E, Ivanitskaya E, Beiles A. 2001. Adaptive radiation of blind subterranean mole rats:naming and revisiting the four sibling species of the Spalax ehrenbergi superspecies in Israel:Spalax galili (2n=52), S. golani (2n=54), S. carmeli (2n=58) and S. judaei (2n=60)[M]. Leiden, Netherlands: Backhuys Publishers.
|
Nevo E. 1999. Mosaic evolution of subterranean mammals:regression, progression, and global convergence[M]. Oxford, UK: Oxford University Press.
|
Nevo E. 2011. Evolution under environmental stress at macro-and microscales[J]. Genome Biology and Evolution, 3: 1039-1052. DOI:10.1093/gbe/evr052 |
Noble MEM, Endicott JA, Brown NR, et al. 1997. The cyclin box fold:protein recognition in cell-cycle and transcription control[J]. Trends in Biochemical Sciences, 22(12): 482-487. DOI:10.1016/S0968-0004(97)01144-4 |
Pines J, Hunter T. 1994. The differential localization of human cyclins A and B is due to a cytoplasmic retention signal in cyclin B[J]. The EMBO Journal, 13(16): 3772-3781. DOI:10.1002/j.1460-2075.1994.tb06688.x |
Pucci B, Kasten M, Giordano A. 2000. Cell cycle and apoptosis[J]. Neoplasia, 2(4): 291-299. DOI:10.1038/sj.neo.7900101 |
Robbins J, Dilwortht SM, Laskey RA, et al. 1991. Two interdependent basic domains in nucleoplasmin nuclear targeting sequence:identification of a class of bipartite nuclear targeting sequence[J]. Cell, 64(3): 615-623. DOI:10.1016/0092-8674(91)90245-T |
Ronquist F, Huelsenbeck JP. 2003. MrBayes 3:Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 19(12): 1572-1574. DOI:10.1093/bioinformatics/btg180 |
Schmidt H, Malik A, Bicker A, et al. 2017. Hypoxia tolerance, longevity and cancer-resistance in the mole rat Spalax-a liver transcriptomics approach[J/OL]. Scientific Reports, 7: 14348[2018-08-01]. https://www.nature.com/articles/s41598-017-13905-z. DOI: 10.1038/s41598-017-13905-z.
|
Shao Y, Li JX, Ge RL, et al. 2015. Genetic adaptations of the plateau zokor in high-elevation burrows[J/OL]. Scientific Reports, 5: 17262[2018-08-01]. https://www.nature.com/articles/srep17262. DOI: 10.1038/srep17262.
|
Sherr CJ, Roberts JM. 1995. Inhibitors of mammalian G1 cyclin-dependent kinases[J]. Genes & Development, 9(10): 1149-1163. |
Sherr CJ, Roberts JM. 1999. CDK inhibitors:positive and negative regulators of G1-phase progression[J]. Genes & Development, 13(12): 1501-1512. |
Stöver BC, Müller KF. 2010. TreeGraph 2: combining and visualizing evidence from different phylogenetic analyses[J/OL]. BMC Bioinformatics, 11(1): 7[2018-07-30]. https://bmcbioinformatics.biomedcentral.com/articles/10.1186/1471-2105-11-7.
|
Swofford D. 2002. PAUP:phylogenetic analysis using parsimony, version 4.0 b10[M]. Sunderland: Sinauer Associates.
|
Tian X, Azpurua J, Hine C, et al. 2013. High-molecular-mass hyaluronan mediates the cancer resistance of the naked mole rat[J]. Nature, 499(7458): 346-349. DOI:10.1038/nature12234 |
Vogelstein B, Lane D, Levine AJ. 2000. Surfing the p53 network[J]. Nature, 408(6810): 307-310. DOI:10.1038/35042675 |
Wang Z, Xu S, Du K, et al. 2016. Evolution of digestive enzymes and RNASE1 provides insights into dietary switch of cetaceans[J]. Molecular Biology and Evolution, 33(12): 3144-3157. DOI:10.1093/molbev/msw191 |
Yang Z. 2007. PAML 4:phylogenetic analysis by maximum likelihood[J]. Molecular Biology and Evolution, 24(8): 1586-1591. DOI:10.1093/molbev/msm088 |
Zhang J, Kumar S. 1997. Detection of convergent and parallel evolution at the amino acid sequence level[J]. Molecular Biology and Evolution, 14(5): 527-536. DOI:10.1093/oxfordjournals.molbev.a025789 |
Zhang J, Nielsen R, Yang Z. 2005. Evaluation of an improved branch-site likelihood method for detecting positive selection at the molecular level[J]. Molecular Biology and Evolution, 22(12): 2472-2479. DOI:10.1093/molbev/msi237 |
Zhao Y, Tang JW, Yang Z, et al. 2016. Adaptive methylation regulation of p53 pathway in sympatric speciation of blind mole rats, Spalax[J]. Proceedings of the National Academy of Sciences of the United States, 113(8): 2146-2151. DOI:10.1073/pnas.1522658112 |
 2020, Vol. 39
2020, Vol. 39




