扩展功能
文章信息
- 崔昕宇, 刘宏广, 李枢强
- CUI Xinyu, LIU Hongguang, LI Shuqiang
- 基于CO Ⅰ基因探讨北京及其周边地区跳钩虾种群遗传结构
- Population Genetic Structure of Platorchestia japonica From Beijing and Its Adjacent Regions Based on CO Ⅰ Gene Sequences
- 四川动物, 2019, 38(4): 402-407
- Sichuan Journal of Zoology, 2019, 38(4): 402-407
- 10.11984/j.issn.1000-7083.20180289
-
文章历史
- 收稿日期: 2018-09-29
- 接受日期: 2019-04-11
2. 中国科学院动物进化与系统学重点实验室, 中国科学院动物研究所, 北京 100101
2. Key Laboratory of Zoological Systematics and Evolution, Institute of Zoology, Chinese Academy of Sciences, Beijing 100101, China
跳钩虾Platorchestia japonica隶属于甲壳动物亚门Crustacea端足目Amphipoda跳钩虾科Talitridae, 是唯一一种栖息在湖泊、河流沿岸的陆生钩虾, 主要分布于我国北京、河北、山西、陕西、山东, 以及日本和朝鲜等(Hou & Li, 2003)。跳钩虾具有抱卵行为, 没有自由的幼体阶段, 迁移能力较弱, 其迁移必须借助水系的连通或者水生植物的携带。研究表明, 陆生跳钩虾起源于海洋类群, 是由于中新世东亚海入侵驱动其占据陆生生境, 并在陆生生境中快速分化(Yang et al., 2013; 杨璐等, 2013)。跳钩虾的种群遗传学研究较少, Cheng等(2011)基于形态和线粒体CO Ⅰ基因序列的研究发现, 中国台湾跳钩虾与日本和中国大陆的跳钩虾存在显著分化, 可能为一个新种Platorchestia paludosus。中国内陆跳钩虾的地理格局和种群遗传结构还未见报道。
线粒体CO Ⅰ基因是一段蛋白质编码基因, 易扩增、好比对, 进化速率较快, 已被广泛用于种群水平的遗传结构和遗传多样性研究(董梦书等, 2017)。本研究通过采集北京及其周边地区的跳钩虾样品, 进行CO Ⅰ基因的序列测定, 分析其遗传结构和遗传多样性, 探讨系统发育关系及分化过程。
1 材料与方法 1.1 样品的采集、保存和鉴定跳钩虾野外采集直接用纱网沿草丛根部扫网, 将新鲜样品浸泡于95%酒精中, 当天更换酒精1次, -20 ℃长期保存。本研究共收集到北京、河北、山西、陕西、山东等地22个采集点128只跳钩虾(表 1)。
| 采样点编号 Sampling ID |
地点 Locality |
东经 East longitude/° |
北纬 North latitude/° |
样本数量 Sample size |
单倍型 Haplotype |
单倍型多样性 Haplotype diversity |
核苷酸多样性 Nucleotide diversity |
| 1 | 北京市怀柔区花木村 | 116.40 | 40.30 | 1 | Hap14 | — | — |
| 2 | 北京市圆明园 | 116.40 | 39.90 | 1 | Hap1 | — | — |
| 3 | 北京市海淀区东北旺 | 116.40 | 39.90 | 2 | Hap1 | — | — |
| 4 | 北京市海淀区玉泉山 | 116.40 | 39.90 | 1 | Hap1 | — | — |
| 5 | 北京市昌平区沙河水库 | 116.31 | 40.15 | 22 | Hap1, 31, 32, 33, 34, 35, 36, 37 | 0.697 | 0.002 55 |
| 6 | 北京市奥林匹克公园 | 116.40 | 39.92 | 27 | Hap1, 20, 26, 39, 40, 41, 42, 43 | 0.843 | 0.002 95 |
| 7 | 河北省灵寿县牛城乡 | 114.33 | 38.27 | 3 | Hap1, 2 | 0.667 | 0.001 07 |
| 8 | 河北省永年县广府镇 | 114.72 | 36.68 | 3 | Hap3, 4 | 0.667 | 0.001 07 |
| 9 | 河北省衡水市衡水湖 | 115.59 | 37.64 | 1 | Hap20 | — | — |
| 10 | 山东省聊城市东昌府区 | 116.05 | 36.38 | 3 | Hap5, 6, 7 | 1 | 0.003 21 |
| 11 | 山东省东平县老湖镇 | 116.22 | 36.05 | 3 | Hap8, 9 | 0.667 | 0.003 21 |
| 12 | 山东省滕州市西岗镇 | 116.98 | 34.97 | 3 | Hap10, 11 | 0.667 | 0.002 14 |
| 13 | 山东省微山县留庄镇 | 116.90 | 34.97 | 3 | Hap11, 12, 13 | 1 | 0.002 14 |
| 14 | 山西省长治市沁县梁家湾 | 112.68 | 36.79 | 5 | Hap21, 22 | 0.400 | 0.004 49 |
| 15 | 山西省长治市沁县景村 | 112.65 | 36.81 | 5 | Hap23 | — | — |
| 16 | 山西省长治市沁县千女湖 | 112.58 | 36.73 | 6 | Hap23, 24, 25 | 0.600 | 0.001 07 |
| 17 | 山西省大同市大同县 | 113.80 | 39.98 | 5 | Hap26 | — | — |
| 18 | 山西省临汾市霍州市 | 111.69 | 36.53 | 6 | Hap23, 27, 28, 29, 30 | 0.933 | 0.007 38 |
| 19 | 陕西省泾阳县王桥镇 | 108.58 | 34.64 | 5 | Hap1 | — | — |
| 20 | 北京市延庆区下阪泉 | 115.90 | 40.40 | 8 | Hap15, 16, 17, 18, 19 | 0.857 | 0.002 98 |
| 21 | 北京市延庆区官厅水库 | 115.90 | 40.40 | 4 | Hap4, 16, 17 | 0.833 | 0.016 85 |
| 22 | 北京市门头沟雁翅镇 | 115.86 | 40.03 | 11 | Hap16, 38 | 0.327 | 0.000 53 |
| 合计 | 128 | 43 | 0.938 | 0.011 72 | |||
取跳钩虾头部, 使用TIANamp Genomic DNA试剂盒提取总DNA, 按照说明书操作。用1%琼脂糖凝胶电泳检测DNA质量, -20 ℃保存备用。
线粒体CO Ⅰ基因扩增引物为CrustF2(5’-GGTTCTTCTCCACCAACCACAARGAYA-3’; Costa et al., 2007)和HCO2198(5’-TAAACTTCAGGGTGACCAA-AAAATCA-3’; Folmer et al., 1994)。PCR反应体系为25 μL:2.5 μL 10×buffer PCR缓冲液, 2 μL 2.5 mmol·L-1 dNTP混合液, 1 μL 10 pmol正、反向引物, 0.15 μL Taq酶(5 U·μL-1), 17.35 μL ddH2O, 1 μL DNA模板。PCR扩增反应程序为:94 ℃ 1 min; 94 ℃ 30 s, 45 ℃ 90 s, 72 ℃ 1 min, 5个循环; 94 ℃ 30 s, 51 ℃ 90 s, 72 ℃ 1 min, 35个循环; 72 ℃ 10 min。PCR产物用1%琼脂糖凝胶电泳检测, 回收产物送生工生物工程(上海)股份有限公司用ABI3730测序仪双向测序。
1.3 数据处理获得的测序峰图利用Sequencher 4.5进行正反链校对和编辑, 删除序列两端的引物区; 运用MacClade 4.0(Maddison & Maddison, 2000)进行序列比对, 并翻译成氨基酸检查是否存在假基因, 有效序列形成数据矩阵。使用MEGA 7.0.21(Kumar et al., 2016)分析碱基组成、变异位点, 并计算遗传距离(p-distance)。选取Platorchestia pacifica(CO Ⅰ基因GenBank登录号:KC578473)、Platorchestia joi(CO Ⅰ基因GenBank登录号:KC578512)为外群, 采用最大似然法(ML)和贝叶斯法(BI)构建系统发育树。ML分析用raxmlGUI 1.5b2(Silvestro & Michalak, 2012), 通过1 000次自展值检验计算支持率。BI分析用MrBayes 3.2.2(Ronquist et al., 2012), 以jModeltest 2.1.7(Posada, 2008)所得最适模型参数Nst=6, 运算400万代, 每1 000代取样1次, 去掉25%burnin, 最终用收敛后的3 000棵树构建一致树。采用DnaSP 6.12统计种群遗传多样性参数(Rozas et al., 2017), 使用Arlequin 3.5进行分子变异分析(Excoffier & Lischer, 2010)。运用PopART 1.7(Leigh & Bryant, 2015)中最小生成网络模型构建单倍型网络关系图, 分析单倍型间的进化关系。
2 结果与分析 2.1 序列分析共获取了128个跳钩虾样本CO Ⅰ基因部分序列(GenBank登录号:MK048335~MK048350、MK048352~MK048365、MK048368、MK048370~MK048371、MK048374~MK048386、MK286468~ MK286549), 序列长度为623 bp, 其中, 保守位点、变异位点和简约信息位点分别为567个、56个和38个。序列中无插入和缺失, T、C、A、G平均含量分别为38.1%、18.5%、25.3%、18.1%, A+T含量较高(63.4%); G+C在密码子第三位点的平均含量为19.5%, 明显低于第一位点和第二位点(47.9%和42.3%)。全部序列检测到43个单倍型。
2.2 系统发育关系及进化网络分析采用ML和BI法构建的系统发育树拓扑结构基本一致(图 1)。43个跳钩虾单倍型分化为2个进化支, 来自北京东部、山西、陕西、河北、山东和北京西部的1个单倍型形成一支, 来自北京西部的另外6个单倍型形成一支。
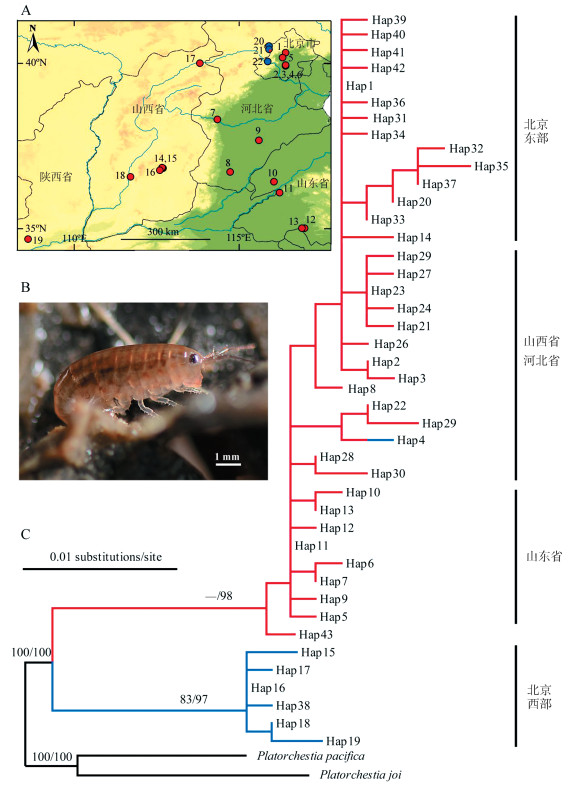
|
| 图 1 跳钩虾采样点和基于CO Ⅰ基因单倍型构建的最大似然树/贝叶斯树 Fig. 1 The sampling sites of Platorchestia japonica and the Maximum Likelihood tree/Bayesian tree based on CO Ⅰ gene haplotypes A.跳钩虾采样点, 样点编号同表 1; B.跳钩虾生态照(刘宏广摄); C.跳钩虾最大似然树和贝叶斯树, 分支上的数字分别是最大似然和贝叶斯支持率 A. Sampling sites of P. japonica, with numbers the same as those in table 1; B. Photo of P. japonica (photo by Liu Hongguang); C. Maximum Likelihood and Bayesian tree of P. japonica, numbers above the branches represent support values in ML/BI analyses, respectively |
| |
单倍型进化网络关系图显示出与系统发育树相同的结果, 跳钩虾的43个单倍型分化为2个进化支, 支系间有明显的遗传变异。北京东部及山西等地的支系包括36个单倍型, 其中Hap1为最大共享单倍型, 其他单倍型由Hap1通过一步或多步突变演化而来。北京西部支系包括6个特有单倍型, Hap16位于网络中心, 其余5个单倍型与Hap16相差1个或2个突变距离。Hap4为北京东部等地支系与北京西部支系共享单倍型。
2.3 遗传多样性及遗传结构跳钩虾整体单倍型多样性为0.938, 核苷酸多样性为0.011 72(表 1)。Hap1出现频次最高(23), 包括北京东部(包括圆明园、昌平区沙河水库、海淀区玉泉山、奥林匹克公园)、河北省灵寿县牛城乡, 以及陕西省泾阳县王桥镇共享单倍型。Hap16为北京西部3个采样点共享单倍型, 出现频次为14。
Hap4为北京市延庆区官厅水库与河北省永年县广府镇共享单倍型, 除此之外, 2个采样点各自有特有单倍型, 河北省永年县广府镇的3个样品中有2个样品共享单倍型Hap3, 北京市延庆区官厅水库的4个样品中有2个样品共享单倍型Hap16和Hap17, 2个种群间的Fst值为0.522 6。
2.4 遗传距离和遗传分化系数跳钩虾2个遗传支系间遗传距离为0.029±0.006, 支系内遗传距离均为0.004±0.001。
分子变异分析结果显示, 2个支系间的遗传变异组分为7.758 43, 种群内变异组分为1.345 71, 二者占总遗传变异组分的比率分别为85.22%、14.78%, Fst值达到0.852 19, 二者差异具有高度统计学意义(P<0.001)。

|
| 图 2 基于COI基因的跳钩虾单倍型网络图 Fig. 2 The haplotype network of Platorchestia japonica based on COI sequences 圆圈代表单倍型, 大小与单倍型频率呈正比, 颜色与图 1相同, 红色代表北京东部支系, 蓝色代表北京西部支系; 小竖线代表突变; Hap4是北京东部支系与北京西部支系的共享单倍型, 单倍型与地理分布之间没有严格对应关系 Each circle means a haplotype, and the size of circles is proportional to haplotype frequency, color annotation is the same as that in fig. 1; red. east part of Beijing, blue. west part of Beijing; each short line represents one mutation step; haplotypes from geographical populations showed no apparent distinct genetic clade |
| |
线粒体CO Ⅰ基因不受标本性别、发育阶段的限制, 在种群遗传多样性和遗传结构分析中得到广泛应用(董梦书等, 2017)。遗传多样性是物种多样性的基础, 是物种生存适应和发展进化的前提(李大命等, 2018)。跳钩虾22个采样点128只个体中共检测到43个单倍型, 单倍型多样性为0.938, 核苷酸多样性为0.011 72, 显示出较高的遗传多样性, 遗传资源丰富, 可以筛选环境敏感型表型, 用作环境毒物监测(Duan et al., 2000)。
跳钩虾地理遗传结构报道很少, 尽管Cheng等(2011)的研究发现中国台湾地区的跳钩虾与日本和中国大陆跳钩虾在形态和遗传上有显著的分化, 但随后更广泛的研究结果表明跳钩虾并没有显著的地理种群遗传结构(Yang et al., 2013)。北京西部地区大部分单倍型形成一个单系群, 但北京东部和西部并没有明显的地理阻隔, 综合北京西部的聚类以及北京东部和西部仍然存在共享单倍型, 这种聚类可能由非地理遗传差异引起, 如取样的局限性等, 北京地区跳钩虾存在东西遗传阻隔的可能性小, 北京周边地区跳钩虾是否存在地理种群遗传结构需要进一步研究。本研究中, 系统发育和单倍型网络图分析都支持跳钩虾分化为2个进化支。一支主要包括北京东部、山西、陕西、河北和山东等地的单倍型, 分支内各单倍型间存在1个或多个碱基突变; 另一支主要包括北京西部延庆、门头沟等地的单倍型, 分支内各单倍型间存在1个或2个碱基替代; 两分支存在16个碱基变异。分子变异分析显示跳钩虾两支系间Fst值(0.852 19)较高, 按照Wright(1965)认为Fst值大于0.15为高度遗传分化, 意味着检测区域的跳钩虾可能来源于2个遗传不同的地理种群。Yang等(2013)在中国大陆及台湾地区更广泛的取样基于CO Ⅰ基因的研究结果表明, 跳钩虾地理种群结构不明显, 但明显分成2个遗传不同的进化支, 这表明目前的跳钩虾可能存在2个遗传不同的种群来源。已有研究表明, 更新世冰期事件成为影响该地域动物种群遗传分化和遗传多样性的主要原因(白茜茜等, 2018), 跳钩虾在冰期是否存在2个不同的大避难所, 以及避难所的位置及冰期后的迁移扩散等还需要进一步的研究。
本研究运用线粒体CO Ⅰ基因探讨了北京及其周边地区跳钩虾的遗传多样性和遗传结构, 研究发现跳钩虾遗传多样性高, 遗传资源丰富, 跳钩虾群体存在2个遗传不同的进化支, 表明目前的跳钩虾具有2个来源。本研究在北京及周边地区采集了128个样本, 鉴定了43个CO Ⅰ基因序列单倍型, 为中国地区跳钩虾种群遗传结构的进一步研究提供了重要的基础资料。
| 白茜茜, 潘昭, 任国栋. 2018. 燕山-太行山区黑带食蚜蝇(双翅目:食蚜蝇科)谱系地理学分析[J]. 生态学杂志, 37(1): 157–163. |
| 董梦书, 杨琳, 陈祥盛, 等. 2017. 基于线粒体COⅠ基因部分序列的缅甸安小叶蝉地理种群遗传多样性研究[J]. 四川动物, 36(3): 277–283. |
| 李大命, 张彤晴, 唐晟凯, 等. 2018. 基于线粒体Cyt b基因和D-loop区序列的洪泽湖湖鲚遗传多样性分析[J]. 江苏农业科学, 46(20): 36–39. |
| 杨璐, 侯仲娥, 李枢强. 2013. 海侵驱动的生物多样性增加[J]. 动物分类学报, 38(4): 901–903. |
| Cheng YT, Nakazono K, Lin YK, et al. 2011. Cryptic diversity of the semi-terrestrial amphipod Platorchestia japonica (Tattersall, 1922)(Amphipoda:Talitrida:Talitridae) in Japan and Taiwan with description of a new species[J]. Zootaxa, 2787: 1–18. DOI:10.11646/zootaxa.2787.1.1 |
| Costa FO, de Waard JR, Boutillier J, et al. 2007. Biological identifications through DNA barcodes:the case of the Crustacea[J]. Canadian Journal of Fisheries and Aquatic Sciences, 64(2): 272–295. DOI:10.1139/f07-008 |
| Duan Y, Guttman SI, Oris JT, et al. 2000. Genotype and toxicity relationships among Hyalella azteca:Ⅱ. Acute exposure to fluoranthene-contaminated sediment[J]. Environmental Toxicology and Chemistry, 19(5): 1422–1426. DOI:10.1002/etc.5620190526 |
| Excoffier L, Lischer HE. 2010. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 10(3): 564–567. DOI:10.1111/j.1755-0998.2010.02847.x |
| Folmer O, Black M, Hoeh W, et al. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit Ⅰ from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology, 3(5): 294–299. |
| Hou Z, Li S. 2003. Terrestrial talitrid amphipods (Crustacea:Amphipoda) from China and Vietnam:studies on the collection of IZCAS[J]. Journal of Natural History, 37(20): 2441–2460. DOI:10.1080/00222930210144343 |
| Kumar S, Stecher G, Tamura K. 2016. MEGA 7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 33(7): 1870–1874. DOI:10.1093/molbev/msw054 |
| Leigh JW, Bryant D. 2015. POPART:full-feature software for haplotype network construction[J]. Methods in Ecology and Evolution, 6(9): 1110–1116. DOI:10.1111/2041-210X.12410 |
| Maddison DR, Maddison WP. 2000. MacClade 4:analysis of phylogeny and character evolution[M]. Sunderland, MA: Sinauer Associates. |
| Posada D. 2008. jModelTest:phylogenetic model averaging[J]. Molecular Biology Evolution, 25: 1253–1256. DOI:10.1093/molbev/msn083 |
| Ronquist F, Teslenko M, van der Mark P, et al. 2012. MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space[J]. Systematic Biology, 61: 539–542. DOI:10.1093/sysbio/sys029 |
| Rozas J, Ferrer-Mata A, Sánchez-DelBarrio JC, et al. 2017. DnaSP v6:DNA sequence polymorphism analysis of large datasets[J]. Molecular Biology and Evolution, 34: 3299–3302. DOI:10.1093/molbev/msx248 |
| Silvestro D, Michalak I. 2012. raxmlGUI:a graphical front-end for RAxML[J]. Organisms, Diversity & Evolution, 12: 335–337. |
| Yang L, Hou Z, Li S. 2013. Marine incursion into east Asia:a forgotten driving force of biodiversity[J]. Proceedings of the Royal Society B, 280(1757): 20122892. DOI:10.1098/rspb.2012.2892 |
| Wright S. 1965. The interpretation of population structure by F-statistics with special regard to systems of mating[J]. Evolution, 19(3): 395–420. DOI:10.1111/j.1558-5646.1965.tb01731.x |
 2019, Vol. 38
2019, Vol. 38




