扩展功能
文章信息
- 杨永刚, 张梅, 胡耀中, 赵雨, 赵大庆, 幺宝金
- YANG Yonggang, ZHANG Mei, HU Yaozhong, ZHAO Yu, ZHAO Daqing, YAO Baojin
- 基于转录组学的梅花鹿茸皮组织修复机制研究
- Study on Tissue Repair Mechanism of Sika Deer Antler Velvet Skins Based on Transcriptomics
- 四川动物, 2019, 38(4): 394-401
- Sichuan Journal of Zoology, 2019, 38(4): 394-401
- 10.11984/j.issn.1000-7083.20190005
-
文章历史
- 收稿日期: 2019-01-05
- 接受日期: 2019-04-11
2. 长春中医药大学创新实践中心, 长春 130117;
3. 长春中医药大学吉林省人参科学研究院, 长春 130117
2. Innovation Practice Center, Changchun University of Chinese Medicine, Changchun 130117, China;
3. Jilin Ginseng Academy, Changchun University of Chinese Medicine, Changchun 130117, China
鹿茸为鹿科Cervidae动物梅花鹿Cervus nippon或马鹿Cervus elaphus的雄鹿未骨化密生茸毛的幼角,为传统名贵药材,具有壮肾阳、益精血和强筋骨等功效(国家药典委员会,2015)。鹿茸具有循环再生和快速生长两大特点,其生长机制一直是研究热点。快速生长期的鹿茸每天可增加2 cm,伴随着骨化程度的不断加深和最终茸皮的脱落,进而形成彻底钙化的鹿角(Li,2012;Chu et al., 2017)。鹿茸的生长主要由鹿茸顶端的生长中心调控,在结构上主要划分为茸皮、间充质和软骨组织等(Li et al., 2002)。茸皮与鹿茸其他部位的皮肤不同,主要表现为表皮层较厚、皮脂腺较大、立毛肌和汗腺缺失、毛囊组织存在不同发育阶段及无疤痕性损伤修复等(Li & Suttie,2000;Li,2010)。但有关茸皮在鹿茸生长发育过程中的基因表达变化的研究仍然几近空白。因此,研究不同生长时期鹿茸生长中心茸皮的基因表达变化,对于揭示鹿茸再生及快速生长机制具有重要意义。同时,对于皮肤、软骨及骨组织损伤修复及相关疾病的治疗具有重要的指导意义。
近年来,高通量测序技术的不断发展促使生物学领域的研究手段发生了重大的变革,尤其是以基因组学、转录组学和蛋白质组学为代表的技术在科学研究中发挥着巨大作用(Bourgard et al., 2018)。其中,转录组测序(RNA-Seq)更是在基因表达分析研究方面起着重要作用(Hrdlickova et al., 2017)。RNA-Seq技术具有通量高、灵敏度高和成本低等特点,与基因芯片和传统测序方法相比,可以对基因表达变化进行系统研究,进而对组织或细胞在某种生理和病理条件下的生物学机制进行较为全面的分析和诠释。同时,RNA-Seq除了可以对模式生物进行转录组测序,还可以对无参考基因组的非模式生物进行从头测序(Costa-Silva et al., 2017;Sudhaga et al., 2018)。本研究采用RNA-Seq技术对快速生长期和骨化期梅花鹿鹿茸生长中心的茸皮进行高通量转录组测序,通过生物信息学分析方法对测序结果进行组装和拼接,筛选不同生长时期茸皮中的差异表达基因,这些研究结果对于揭示鹿茸生长机制及组织损伤修复研究具有重要指导意义。
1 材料与方法 1.1 材料与试剂雄性梅花鹿东北亚种Cervus nippon hortulorum(吉林省双阳鹿乡);DEPC-treated Water(AM9916,Thermo);TRIzol Reagent(15596018,Thermo);氯仿(B010 2004,北京化工);异丙醇(B0301007,北京化工);无水乙醇(B0301002,北京化工);TruSeq Stranded mRNA Library Prep Kit(RS-122-2101,Illumina)。
1.2 方法 1.2.1 不同生长时期鹿茸生长中心茸皮组织采集选取健康的4周岁雄性梅花鹿6只,分别于快速生长期(生茸后60 d)和骨化期(生茸后90 d)采集两侧鹿茸生长中心组织(距离茸尖5 cm)。按照Li等(2002)的实验方法分离鹿茸茸皮,置于纯化水中反复漂洗去除血液,切成小块后置于液氮罐中冻存。
1.2.2 总RNA提取制备及测序文库构建采用TRIzol(Invitrogen,USA)法从鹿茸生长中心茸皮中提取总RNA,通过Bioanalyzer 2100系统(Agilent Technologies,USA)考察RNA完整性。采用TruSeq Stranded mRNA试剂盒(Illumina,USA)进行测序文库构建:首先,采用带有Oligo(dT)的偶联磁珠(Life Technologies,USA)从总RNA中纯化富集mRNA,并进行mRNA片段化;以片段化的mRNA为模板,经逆转录酶和随机引物合成双链cDNA并经核糖核酸酶处理降解mRNA;最后,经末端修复和接头连接后,通过PCR扩增获得最终的测序文库。
1.2.3 文库测序和差异基因表达分析文库测序在Illumina Hiseq 2000平台(Illumina,USA)上进行,测序结果采用Illumina HCS 1.1进行数据转换和质量评价。去除载体序列和低质量序列(碱基质量值Q≤10的碱基数占整个reads的20%以上),获得clean reads,并采用Trinity(Grabherr et al., 2011)进行序列从头组装。组装数据通过BLASTX比对程序与nr(Non-redundant)和Swiss-Prot蛋白数据库比对,进行基因注释,以E<10-5为评价标准。基因表达量分析采用FPKM法(Trapnell et al., 2010)进行计算,差异基因表达分析通过DEGseq(Wang et al., 2010)进行分析,差异表达基因判定标准为:快速生长期和骨化期茸皮组织差异基因表达倍数在2倍以上[即log2(Fold change)≥1或≤-1,Fold change=骨化期FPKM/快速生长期FPKM],且假发现率(FDR)≤0.001。
1.2.4 功能分类及代谢通路富集分析针对筛选出的差异表达基因,通过Blast2GO比对到GO数据库(http://www.geneontology.org/)进行功能分类富集分析,通过KOBAS比对到KEGG数据库(http://www.genome.jp/kegg/pathway.html)进行代谢通路富集分析,采用Fisher检验和FDR校正获得Q<0.05的功能条目或代谢通路判定为显著性富集。
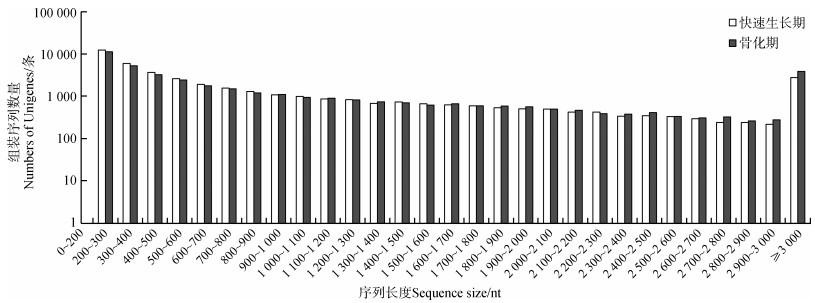
2 结果 2.1 测序和组装通过高通量RNA-Seq转录组测序,分别从快速生长期和骨化期鹿茸生长中心茸皮中获得4.42 Gb和4.45 Gb的测序数据。其中,测序raw reads分别为45 422 254条(快速生长期)和45 421 562条(骨化期),去除载体序列和低质量序列后,分别获得44 199 056条(快速生长期)和44 530 430条(骨化期)clean reads,测序质量值(Q20值)分别为98.50(快速生长期)和98.39(骨化期)。通过Trinity组装,分别获得44 352条(快速生长期)和43 194条(骨化期)组装序列(Unigenes),平均长度分别为977 nt(快速生长期)和1 135 nt(骨化期)(图 1)。
|
| 图 1 快速生长期和骨化期梅花鹿茸皮组装序列长度分布 Fig. 1 Length distribution of Unigenes of Cervus nippon antler velvet skins at rapid growth and ossification stages |
| |
通过BLASTX程序比对,将Unigenes与nr、Swiss-Prot蛋白数据库进行比对,以E<10-5为评价标准,与2个数据库同时比对上的Unigenes共有28 388条。针对这些Unigenes,采用FPKM法进行基因表达量分析,采用DEGseq进行差异基因表达分析,共筛选出5 246条差异表达基因[log2(Fold change)≥1或≤-1;FDR≤0.001]。与快速生长期比较,骨化期显著上调的差异表达基因有2 627条,显著下调的差异表达基因有2 619条。
2.3 GO功能和KEGG代谢通路富集分析将差异表达基因与GO数据库进行比对,差异表达基因显著富集的GO条目特征为:细胞组分分类主要包括细胞部分(cell part)、细胞(cell)和细胞器(organelle)等;分子功能分类主要包括结合活性(binding)和催化活性(catalytic activity)等;生物进程分类主要包括细胞进程(cellular process)、单细胞进程(single-organism process)和代谢进程(metabolic process)等(图 2)。

|
| 图 2 差异表达基因GO功能富集分析 Fig. 2 GO enrichment of differentially expressed genes (DEGs) |
| |
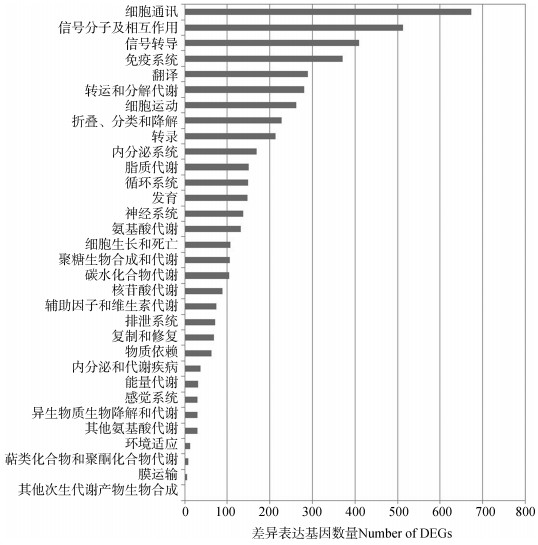
将差异表达基因与KEGG数据库进行比对,差异表达基因显著富集的代谢通路主要包括细胞通讯(cell communication)、信号分子及相互作用(signaling molecules and interaction)、信号转导(signaling transduction)和免疫系统(immune system)等(图 3)。
|
| 图 3 差异表达基因KEGG代谢通路富集分析 Fig. 3 KEGG pathway enrichment of differentially expressed genes (DEGs) |
| |
根据功能富集和代谢通路分析结果,推测这些差异表达基因主要为生长因子、转录因子和胶原类成分。因此,根据差异表达基因的注释结果,结合目前已发现和报道的这3类差异表达基因进行进一步的筛选和分析。
2.4.1 生长因子类差异表达基因在快速生长期和骨化期茸皮差异表达基因中,共筛选出7种生长因子(表 1)。与快速生长期比较,骨化期茸皮中表达量显著上调的生长因子主要包括成纤维细胞生长因子7(Fgf7)和12(Fgf12)以及血管内皮生长因子A(Vegfa);表达量显著下调的生长因子主要包括血小板衍生生长因子A(Pdgfa)、转化生长因子β3(Tgfβ3)、表皮生长因子样蛋白7(Egfl7)和血管内皮生长因子B(Vegfb)。
| GenBank登录号 | 名称 | 快速生长期 表达量 |
骨化期 表达量 |
差异倍数 |
| sp|Q5D0X0| | 成纤维细胞生长因子7 Fibroblast growth factor 7 (Fgf7) | 0.01 | 2.15 | 7.75 |
| sp|Q9MYV3| | 血管内皮生长因子A Vascular endothelial growth factor A (Vegfa) | 0.31 | 1.30 | 2.07 |
| sp|P61150| | 成纤维细胞生长因子12 Fibroblast growth factor 12 (Fgf12) | 0.95 | 2.67 | 1.49 |
| sp|Q2KJ15| | 血小板衍生生长因子A Platelet-derived growth factor subunit A (Pdgfa) | 2.41 | 0.37 | -2.70 |
| sp|P15203| | 转化生长因子β3 Transforming growth factor beta-3 (Tgfβ3) | 8.71 | 3.48 | -1.32 |
| sp|Q9UHF1| | 表皮生长因子样蛋白7 Epidermal growth factor-like protein 7 (Egfl7) | 35.40 | 15.05 | -1.23 |
| sp|Q9XS49| | 血管内皮生长因子B Vascular endothelial growth factor B (Vegfb) | 18.61 | 8.87 | -1.07 |
在快速生长期和骨化期茸皮差异表达基因中,共筛选出25种转录因子(表 2)。与快速生长期比较,骨化期茸皮中表达量显著上调的转录因子有12种(Pou2f3、Elf2、Bclaf1、Sox6、Runx1、Nfyb、Carf、Nr2f2、Elf1、Atf1、Atf6α和Bbx),表达量显著下调的有13种(Sox12、Ebf3、E4f1、Scx、Tcf4、Nr2f1、Mafk、Ubtf、Crebzf、Elf4、Tfe3、Sox4和Sox18)。
| GenBank登录号 | 名称 | 快速生长期表达量 | 骨化期表达量 | 差异倍数 |
| sp|P42571| | POU domain,class 2,transcription factor 3 (Pou2f3) | 0.01 | 2.03 | 7.67 |
| sp|Q15723| | ETS-related transcription factor Elf-2 (Elf2) | 0.01 | 0.48 | 5.58 |
| sp|Q9NYF8| | Bcl-2-associated transcription factor 1 (Bclaf1) | 0.18 | 3.38 | 4.23 |
| sp|Q5RCU4| | Transcription factor SOX-6 (Sox6) | 0.03 | 0.25 | 3.06 |
| sp|Q01196| | Runt-related transcription factor 1 (Runx1) | 1.64 | 5.85 | 1.83 |
| sp|Q32KW0| | Nuclear transcription factor Y subunit beta (Nfyb) | 0.50 | 1.77 | 1.82 |
| sp|Q58CW6| | Calcium-responsive transcription factor (Carf) | 0.52 | 1.76 | 1.76 |
| sp|Q9TTR7| | COUP transcription factor 2 (Nr2f2) | 1.87 | 4.81 | 1.36 |
| sp|A0JN51| | ETS-related transcription factor Elf-1 (Elf1) | 2.06 | 5.08 | 1.30 |
| sp|Q08DA8| | Cyclic AMP-dependent transcription factor ATF-1 (Atf1) | 1.19 | 2.90 | 1.29 |
| sp|P18850| | Cyclic AMP-dependent transcription factor ATF-6 alpha (Atf6α) | 4.60 | 9.75 | 1.08 |
| sp|Q8WY36| | HMG box transcription factor BBX (Bbx) | 1.61 | 3.25 | 1.01 |
| sp|O15370| | Transcription factor SOX-12 (Sox12) | 1.74 | 0.01 | -7.44 |
| sp|O08791| | Transcription factor COE3 (Ebf3) | 0.73 | 0.01 | -6.19 |
| sp|Q66K89| | Transcription factor E4F1 (E4f1) | 2.16 | 0.05 | -5.43 |
| sp|Q64124| | Transcription factor scleraxis (Scx) | 2.74 | 0.31 | -3.14 |
| sp|P15884| | Transcription factor 4 (Tcf4) | 1.71 | 0.26 | -2.72 |
| sp|Q60632| | COUP transcription factor 1 (Nr2f1) | 2.40 | 0.37 | -2.70 |
| sp|Q61827| | Transcription factor MafK (Mafk) | 4.71 | 1.37 | -1.78 |
| sp|P17480| | Nucleolar transcription factor 1 (Ubtf) | 5.25 | 1.58 | -1.73 |
| sp|Q9NS37| | ATF bZIP transcription factor (Crebzf) | 3.80 | 1.37 | -1.47 |
| sp|Q99607| | ETS-related transcription factor Elf-4 (Elf4) | 2.83 | 1.12 | -1.34 |
| sp|Q05B92| | Transcription factor E3 (Tfe3) | 4.30 | 1.71 | -1.33 |
| sp|Q06945| | Transcription factor SOX-4 (Sox4) | 9.25 | 3.97 | -1.22 |
| sp|P35713| | Transcription factor SOX-18 (Sox18) | 37.46 | 17.12 | -1.13 |
在快速生长期和骨化期茸皮差异表达基因中,共筛选出6种胶原分子(表 3)。与快速生长期比较,骨化期茸皮中表达量显著上调的胶原分子包括Col24a1和Col12a1;表达量显著下调的包括Col13a1、Col27a1、Col3a1和Col1a1。
| GenBank登录号 | 名称 | 快速生长期表达量 | 骨化期表达量 | 差异倍数 |
| sp|Q17RW2| | Collagen alpha-1 (ⅩⅩⅣ) chain (Col24a1) | 0.04 | 0.39 | 3.29 |
| sp|Q99715| | Collagen alpha-1 (ⅩⅡ) chain (Col12a1) | 67.65 | 232.39 | 1.78 |
| sp|Q9R1N9| | Collagen alpha-1 (ⅩⅢ) chain (Col13a1) | 1.51 | 0.35 | -2.11 |
| sp|Q5QNQ9| | Collagen alpha-1 (ⅩⅩⅦ) chain (Col27a1) | 3.87 | 1.01 | -1.94 |
| sp|P04258| | Collagen alpha-1 (Ⅲ) chain (Col3a1) | 6.47 | 2.86 | -1.18 |
| sp|P02457| | Collagen alpha-1 (Ⅰ) chain (Col1a1) | 229.29 | 104.58 | -1.13 |
组织损伤修复是涉及多种细胞和生物大分子参与的复杂的功能组织重建过程,由于人体皮肤、软骨和骨组织的再生能力差,损伤后很难修复和再生,因此,组织损伤修复一直是临床治疗上的难点(Hu et al., 2014)。鹿茸生长过程非常独特,具有循环再生和快速生长的特点,鹿茸的茸皮、软骨和骨等组织在结构和功能上与正常组织存在巨大的差别(Li et al., 2014)。因此,探寻鹿茸生长过程中不同组织基因表达变化,将有利于揭示鹿茸独特生长过程的内在分子机制,为组织损伤修复的临床研究提供理论指导。本研究针对快速生长期和骨化期鹿茸生长中心茸皮组织进行深度转录组测序,采用生物信息学分析方法对测序结果进行组装拼接和差异基因表达分析,筛选出7种生长因子、25种转录因子和6种胶原分子,这些调控因子在组织损伤修复过程中发挥重要作用。
研究结果表明,鹿茸在不同生长过程中,茸皮中基因表达水平差异显著。在快速生长过程中,茸皮生长主要由生长因子Pdgfa、Tgfβ3、Egfl7和Vegfb调控,并通过上调转录因子Sox12、Ebf3、E4f1、Scx、Tcf4、Nr2f1、Mafk、Ubtf、Crebzf、Elf4、Tfe3、Sox4和Sox18,促进胶原分子Col13a1、Col27a1、Col3a1和Col1a1的表达;而在骨化过程中,茸皮生长主要由生长因子Fgf7、Fgf12和Vegfa调控,并通过上调转录因子Pou2f3、Elf2、Bclaf1、Sox6、Runx1、Nfyb、Carf、Nr2f2、Elf1、Atf1、Atf6α和Bbx,促进胶原分子Col24a1和Col12a1的表达。
在组织损伤修复过程中,生长因子Pdgf、Egf、Vegf、Fgf和Tgf等通过调控细胞的趋化、有丝分裂、血管生成以及细胞外基质的合成,进而调控组织损伤修复(Bae et al., 2014)。研究表明,转录因子Sox4和Sox12同属于SoxC家族,在胚胎发育和组织器官形成过程中发挥重要作用,通过调控细胞的存活、增殖和分化促进组织损伤修复(Lefebvre & Bhattaram,2016;Chang & Hertz,2017;Mu et al., 2017;Kavyanifar et al., 2018)。在组织损伤修复过程中,Tgfβ通过激活并上调Scx表达,促进细胞基质分泌进而加速创伤愈合(Sakabe et al., 2018)。此外,研究也表明转录因子E4f1、Tcf4和Elf4在人和小鼠组织损伤修复过程中发挥着重要的作用(Sivina et al., 2011;Lu et al., 2012;Goguet-Rubio et al., 2016)。鹿茸每年可以采集2次,生茸后60 d可以采集1次,俗称头茬茸,鹿茸断面伤口愈合后可以再次长出鹿茸,俗称二茬茸(Suttie & Fennessy,1985;张嵩,李峰,2013)。本研究结果表明,在快速生长期,由于茸皮中促进组织损伤修复的基因显著上调,有利于鹿茸损伤后的创口愈合进而再生出新鹿茸。因此,本研究在梅花鹿茸皮中鉴定出的相关调控因子对于组织损伤修复的临床研究具有重要的指导意义。
| 国家药典委员会. 2015. 中华人民共和国药典[M]. 北京: 中国医药科技出版社. |
| 张嵩, 李峰. 2013. 不同规格鹿茸商品药材中氨基酸含量分析[J]. 中国中药杂志, 38: 1919–1923. |
| Bae IH, Park JW, Kim DY. 2014. Enhanced regenerative healing efficacy of a highly skin-permeable growth factor nanocomplex in a full-thickness excisional mouse wound model[J]. International Journal of Nanomedicine, 9: 4551–4567. |
| Bourgard C, Albrecht L, Kayano ACAV, et al. 2018. Plasmodium vivax biology:insights provided by genomics, transcriptomics and proteomics[J]. Frontiers in Celluar and Infection Microbiology, 8: 34. DOI:10.3389/fcimb.2018.00034 |
| Chang KC, Hertz J. 2017. SoxC transcription factors in retinal development and regeneration[J]. Neural Regeneration Research, 12: 1048–1051. DOI:10.4103/1673-5374.211178 |
| Chu W, Zhao H, Li J, et al. 2017. Custom-built tools for the study of deer antler biology[J]. Frontiers in Bioscience (Landmark), 22: 1622–1633. DOI:10.2741/4561 |
| Costa-Silva J, Domingues D, Lopes FM. 2017. RNA-Seq differential expression analysis:an extended review and a software tool[J]. PLoS ONE, 12: e0190152. DOI:10.1371/journal.pone.0190152 |
| Goguet-Rubio P, Seyran B, Gayte L, et al. 2016. E4F1-mediated control of pyruvate dehydrogenase activity is essential for skin homeostasis[J]. Proceedings of the National Academy of Sciences of the United States of America, 113: 11004–11009. DOI:10.1073/pnas.1602751113 |
| Grabherr MG, Haas BJ, Yassour M, et al. 2011. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 29: 644–652. DOI:10.1038/nbt.1883 |
| Hrdlickova R, Toloue M, Tian B. 2017. RNA-Seq methods for transcriptome analysis[J]. Wiley Interdisciplinary Reviews RNA, 8: e1364. DOI:10.1002/wrna.1364 |
| Hu MS, Maan ZN, Wu JC, et al. 2014. Tissue engineering and regenerative repair in wound healing[J]. Annals of Biomedical Engineering, 42: 1494–1507. DOI:10.1007/s10439-014-1010-z |
| Kavyanifar A, Turan S, Lie DC. 2018. SoxC transcription factors:multifunctional regulators of neurodevelopment[J]. Cell and Tissue Research, 371: 91–103. DOI:10.1007/s00441-017-2708-7 |
| Lefebvre V, Bhattaram P. 2016. SOXC genes and the control of skeletogenesis[J]. Current Osteoporosis Reports, 14: 32–38. DOI:10.1007/s11914-016-0296-1 |
| Li C, Clark DE, Lord EA, et al. 2002. Sampling technique to discriminate the different tissue layers of growing antler tips for gene discovery[J]. Anatomical Record, 268: 125–130. DOI:10.1002/ar.10120 |
| Li C, Suttie JM. 2000. Histological studies of pedicle skin formation and its transformation to antler velvet in red deer (Cervus elaphus)[J]. Anatomical Record, 260: 62–71. DOI:10.1002/1097-0185(20000901)260:1<62::AID-AR70>3.0.CO;2-4 |
| Li C, Zhao H, Liu Z, et al. 2014. Deer antler-a novel model for studying organ regeneration in mammals[J]. International Journal of Biochemistry & Cell Biology, 56: 111–122. |
| Li C. 2010. Exploration of the mechanism underlying neogenesis and regeneration of postnatal mammalian skin-deer antler velvet[J]. International Journal of Medical and Biological Frontiers, 16: 1–19. |
| Li C. 2012. Deer antler regeneration:a stem cell-based epimorphic process[J]. Birth Defects Research, Part C-Embryo Today:Reviews, 96: 51–62. DOI:10.1002/bdrc.21000 |
| Lu R, Qu Y, Ge J, et al. 2012. Transcription factor TCF4 maintains the properties of human corneal epithelial stem cells[J]. Stem Cells, 30: 753–761. DOI:10.1002/stem.1032 |
| Mu Z, Zhang S, He C, et al. 2017. Expression of SoxC transcription factors during zebrafish retinal and optic nerve regeneration[J]. Neuroscience Bulletin, 33(1): 53–61. DOI:10.1007/s12264-016-0073-2 |
| Sakabe T, Sakai K, Maeda T, et al. 2018. Transcription factor scleraxis vitally contributes to progenitor lineage direction in wound healing of adult tendon in mice[J]. Journal of Biological Chemistry, 293: 5766–5780. DOI:10.1074/jbc.RA118.001987 |
| Sivina M, Yamada T, Park CS, et al. 2011. The transcription factor E74-like factor controls quiescence of endothelial cells and their resistance to myeloablative treatments in bone marrow[J]. Arteriosclerosis Thrombosis and Vascular Biology, 31: 1185–1191. DOI:10.1161/ATVBAHA.111.224436 |
| Sudhagar A, Kumar G, El-Matbouli M. 2018. Transcriptome analysis based on RNA-Seq in understanding pathogenic mechanisms of diseases and the immune system of fish:a comprehensive review[J]. International Journal of Molecular Sciences, 19: E245. DOI:10.3390/ijms19010245 |
| Suttie JM, Fennessy PF. 1985. Regrowth of amputated velvet antlers with and without innervation[J]. Journal of Experimental Zoology, 234: 359–366. DOI:10.1002/jez.1402340305 |
| Trapnell C, Williams BA, Pertea G, et al. 2010. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 28: 511–515. DOI:10.1038/nbt.1621 |
| Wang L, Feng Z, Wang X, et al. 2010. DEGseq:an R package for identifying differentially expressed genes from RNA-seq data[J]. Bioinformatics, 26: 136–138. DOI:10.1093/bioinformatics/btp612 |
 2019, Vol. 38
2019, Vol. 38




