扩展功能
文章信息
- 颜岳辉, 丁雪梅, 李强, 李旭, 唐利洲
- YAN Yuehui, DING Xuemei, LI Qiang, LI Xu, TANG Lizhou
- 珠江源头入侵种波氏吻虾虎的遗传多样性分析
- Genetic Diversity of Invasive Species Rhinogobius cliffordpopei at the Source of the Pearl River
- 四川动物, 2019, 38(3): 263-270
- Sichuan Journal of Zoology, 2019, 38(3): 263-270
- 10.11984/j.issn.1000-7083.20180380
-
文章历史
- 收稿日期: 2018-12-10
- 接受日期: 2019-03-11
2. 曲靖师范学院, 云南高原生物资源保护与利用研究中心, 云南曲靖 655011
2. Center for Yunnan Plateau Biological Resources Protection and Utilization, Qujing Normal University, Qujing, Yunnan Province 655011, China
波氏吻虾虎Rhinogobius cliffordpopei隶属于鲈形目Perciformes虾虎鱼科Gobiidae虾虎鱼亚科Gobiinae吻虾虎鱼属Rhinogobius (Nichols,1925),是一类小型淡水性底栖鱼类,模式标本发现于长江水系的洞庭湖(Nichols,1925),随后入侵珠江及云南的高原湖泊(伍汉霖,钟俊生,2008;陈小勇,2013)。该物种属中国特有种,目前广泛分布于长江、黄河、珠江、辽河、钱塘江等水系的支流、湖泊和水库生境中,具有较高的经济价值(伍汉霖,钟俊生,2008;李帆,2011)。有关该物种形态特征、行为习性、食性、繁殖、营养生态位、生境选择与分化、生长和种群动态等特征已有记述(倪勇,伍汉霖,2006;伍汉霖,钟俊生,2008;王华,郭延蜀,2009;李帆,2011;Guo et al., 2012, 2013, 2014, 2016, 2017)。波氏吻虾虎线粒体基因组测定和系统发育关系的分析表明,其线粒体基因组全长16 511 bp,包括13个蛋白质编码基因,22个tRNA基因,2个rRNA基因和1个控制区,构建的系统发育树显示,波氏吻虾虎形成了一个单系群,再次验证其独立种的分类地位(Zhong et al., 2018)。除此之外,有关波氏吻虾虎群体遗传学和种群生态学方面的研究工作则未见报道,尤其是涉及该物种在入侵地的遗传多样性水平和遗传变异的研究较为缺乏。因此,从群体遗传学角度探讨波氏吻虾虎在珠江源头水体的群体遗传多样性和遗传分化程度,揭示其遗传多样性形成过程与影响成因,可以为控制该入侵种的扩散分布和生态危害提供科学依据。
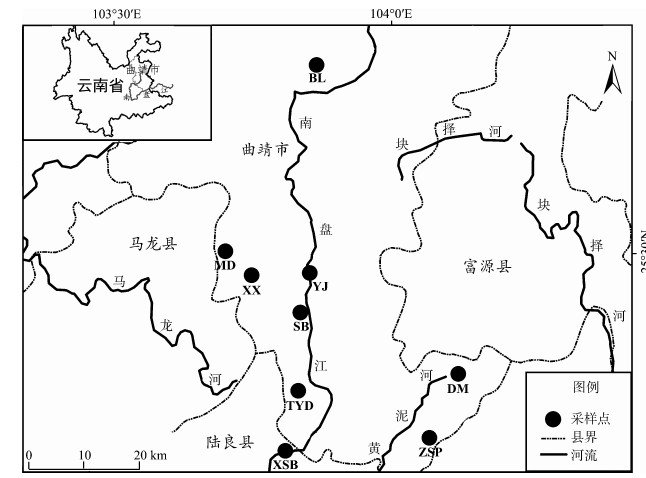
1 材料与方法 1.1 采样珠江由西江、北江、东江和珠江三角洲等组成,西江是珠江的干流,发源于云南省曲靖市沾益区境内的马雄山,南盘江是西江的第一段河流。96份波氏吻虾虎样本采自南盘江上游的云南省曲靖市的沾益区白浪水库(BL)、经济开发区面店水库(MD)、麒麟区潇湘(XX)、沿江(YJ)、三宝(SB)、独木(DM)、太阳岛(TYD)和涨水坡水库(ZSP)及陆良县响水坝(XSB) 9个自然水库(表 1,图 1)。样本用95%乙醇固定,4 ℃保存。
| 种群 Population | 东经 East longitude | 北纬 Northern latitude | 海拔 Altitude/m | 样本数 Sample size |
| 白浪BL | 103°51.904′ | 25°48.289′ | 1 988 | 12 |
| 潇湘XX | 103°45.091′ | 25°27.615′ | 1 917 | 11 |
| 面店MD | 103°42.238′ | 25°29.946′ | 1 932 | 11 |
| 沿江YJ | 103°51.457′ | 25°27.887′ | 1 835 | 10 |
| 三宝SB | 103°50.384′ | 25°24.006′ | 1 884 | 11 |
| 独木DM | 104°07.465′ | 25°18.115′ | 2 006 | 11 |
| 太阳岛TYD | 103°50.231′ | 25°16.335′ | 1 864 | 8 |
| 涨水坡ZSP | 104°04.405′ | 25°11.817′ | 2 022 | 12 |
| 响水坝XSB | 103°48.900′ | 25°10.430′ | 1 857 | 10 |
采用北京鼎国昌盛生物技术有限责任公司提供的基因组DNA提取试剂盒抽提总DNA,提取方法参考试剂盒的方案。以GenBank数据库已有波氏吻虾虎线粒体细胞色素b (cyt b)基因全序列(登录号:KX898434)为参考,由昆明硕擎生物科技有限公司设计PCR扩增引物,引物序列为F:5'-AACCACT-ACCCCAGAACCA-3'和R:5'-CTTTTCGTCATATCCGGTGCCC-3'。反应体系为50 μL:1 μL DNA母液,2 μL上、下游引物,25 μL 2×Easy Taq PCR Supermix(TRANSGEN BIOTECH),22 μL ddH2O。反应条件为:94 ℃预变性5 min;94 ℃变性30 s,57 ℃退火30 s,72 ℃延伸30 s,30个循环;72 ℃续延伸5 min,4 ℃保存。取1 μL扩增产物混合2 μL 6×Loading buffer凝胶加样缓冲液,进行琼脂糖凝胶电泳检测,凝胶成像系统观察拍照。PCR扩增产物送昆明硕擎生物科技有限公司测序。
1.3 数据分析运用Chromas 2.62(Kyte & Doolittle,1982)和Seqman(Allex,1999)进行同源排序,结合手动校对序列。运用DnaSP 5.10(Rozas & Rozas,1995)统计序列的多态性信息,分析种群的单倍型多样性、核苷酸多样性及标准差。运行MrModeltest 2.2(Darriba et al., 2012)检验核苷酸最适模型。以GenBank数据库下载的吻虾虎鱼属的子陵吻虾虎R. giurinus(R. giurinus1:KF371534、R. giurinus2:KP892753、R. giurinus3:NC_022692)、褐吻虾虎R. brunneus (R. brunneus1:NC_028435、R. brunneus2:KT601096、R. brunneus3:AB872439)、短吻红斑吻虾虎R. rubromaculatus(R. rubromaculatus1:AY137607、R. rubromaculatus2:AY137606、R. rubromaculatus3:AY137604)作为外群,利用MrBayes 3.2(Ronquist & Huelsenbeck,2003)构建波氏吻虾虎单倍型间的贝叶斯树,以HKY+G为最适模型,运算1 000万代,每100代取样一次,去掉burn-in运算开始25%的不可信区域,直到链收敛,即分裂频率平均标准差小于0.01停止运算。运行MEGA 6.0(Tamura et al., 2013)计算种群间和单倍型间遗传距离,并运用最大似然法(maximum-likelihood method)构建系统进化树。运行Network 5.0(Richards et al., 1996),利用中连法(median-joining method)构建波氏吻虾虎群体单倍型间的网络图。其次,利用Arlequin 3.5(Excoffier & Lischer,2010)的分子变异分析(AMOVA)模块(Excoffier et al., 1992)分析群体遗传结构。
2 结果 2.1 遗传多样性与种群动态波氏吻虾虎cyt b基因全序列长1 141 bp,包括1 072个保守位点,69个变异位点,其中单突变位点3个,简约信息位点66个,无插入和缺失位点。96只个体检测出cyt b单倍型5个(Hap1~Hap5,登录号:MK204741~MK204745),单倍型间的遗传距离为0.001~ 0.064,单倍型多样性(Hd)为0.359±0.059,核苷酸多样性(Nd)为0.021±0.010(表 2)。9个地理种群的76只个体共享单倍型Hap1,种群DM和ZSP共享单倍型Hap2、Hap3,种群YJ独有单倍型Hap4,种群ZSP独有单倍型Hap5(表 2)。所有地理种群中,除BL、XX、MD、SB、TYD和XSB仅有1个单倍型Hap1,未分析出多样性信息外,种群DM的Hd(0.709±0.083)和Nd(0.023±0.012)最高,种群ZSP的分别为0.561±0.154和0.017±0.009,而种群YJ的均最低,分别为0.200±0.154和0.001±0.001(表 2)。种群DM、ZSP及全部群体的中性检验显示,其Tajima's D和Fu's Fs检验均不显著(P>0.05),而种群YJ的Tajima's D为显著性负值(P<0.05),说明波氏吻虾虎的DM、ZSP种群及全部群体未检测到种群扩张,而支持YJ种群经历过近期种群扩张(表 2)。
| 种群 Population | 单倍型(个体数) Haplotype (sample size) | 单倍型多样性 Haplotype diversity (mean±sd) | 核苷酸多样性 Nucleotide diversity (mean±sd) | Tajima's D (P) | Fu's Fs (P) |
| 白浪BL | Hap1(12) | ||||
| 潇湘XX | Hap1(11) | ||||
| 面店MD | Hap1(11) | ||||
| 沿江YJ | Hap1(9)、Hap4(1) | 0.200±0.154 | 0.001±0.001 | -1.873 (0.010) | 3.338 (0.937) |
| 三宝SB | Hap1(11) | ||||
| 独木DM | Hap1(3)、Hap2(3)、Hap3(5) | 0.709±0.083 | 0.023±0.012 | 1.345 (0.944) | 14.967 (1.000) |
| 太阳岛TYD | Hap1(8) | ||||
| 涨水坡ZSP | Hap1(1)、Hap2(8)、Hap3(1)、Hap5(2) | 0.561±0.154 | 0.017±0.009 | -0.421 (0.365) | 10.848 (0.999) |
| 响水坝XSB | Hap1(10) | ||||
| 全部群体 | Hap1(76)、Hap2(11)、Hap3(6)、 Hap4(1)、Hap5(2) | 0.359±0.059 | 0.021±0.010 | 0.203 (0.654) | 29.210 (0.999) |
分子系统发育关系的贝叶斯树显示,与外群子陵吻虾虎、褐吻虾虎和短吻红斑吻虾虎分开,波氏吻虾虎的5个单倍型Hap1~Hap5以高自举值构成了一个独立的单系群,验证其独立种分类地位(图 2)。其次,波氏吻虾虎群体分化为2个系统分支,单倍型Hap2、Hap5构成的第一分支和单倍型Hap4、Hap1和Hap3构成的第二分支(图 2:左)。基于最大似然法构建的系统树分析出与贝叶斯树一致的拓扑结构。同时,单倍型间网络图同样支持波氏吻虾虎群体分化为2个系统分支(图 2:右)。
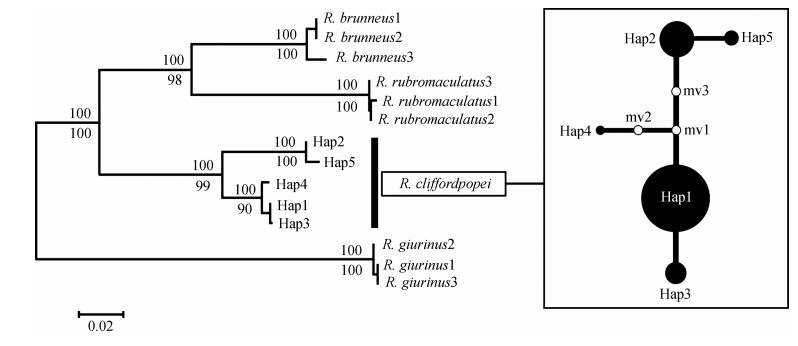
|
| 图 2 波氏吻虾虎单倍型间的贝叶斯树(左)和网络图(右) Fig. 2 The Bayesian inference tree (left) and network relationship (right) of Rhinogobius cliffordpopei haplotypes 左图中分支以上的数字是贝叶斯树的自举值;而最大似然树自举值显示在分支的下面;右图中每个实心圆代表一种单倍型,实心圆大小与单倍型频率呈正比,圆圈(mv)代表缺失单倍型,每条线条代表一步突变 In the left figure, the numbers above the branches are the bootstrap values of Bayesian inference tree; maximum-likelihood tree bootstrap values are shown under the branches; in the right figure, every solid cycle means a haplotype; the size of solid circles is proportional to haplotype frequency, the cycle (mv) represents missing haplotypes (not sampled or extinct), every line represents one mutation step |
| |
波氏吻虾虎群体遗传结构AMOVA分析结果显示(表 3),该物种整个群体种群间和种群内的遗传变异占总遗传变异组分的比率分别为62.99%和37.01%,固定指数FST为0.630(P<0.01),即种群间的遗传变异显著高于种群内,说明波氏吻虾虎群体具有显著的遗传分化格局。该物种全部群体的平均遗传距离为0.014±0.002,种群DM与ZSP、YJ之间的遗传距离分别为0.038±0.005、0.016±0.002,种群ZSP与YJ之间的遗传距离为0.048±0.006,反映出地理种群间的遗传差异程度较大。
| 遗传差异来源 Source of variation | 自由度 Degree of freedom | 平方和 Sum of squares | 遗传变异组分 Variance components | 遗传变异率 Percentage of variation/% | 固定指数 Fixation index | P |
| 种群间 | 8 | 432.204 | 4.807 15 | 62.99 | 0.630 | <0.01 |
| 种群内 | 87 | 245.723 | 2.824 40 | 37.01 | ||
| 群体 | 95 | 677.927 | 7.631 55 | 100 |
从群体遗传学和保护生物学的角度分析,遗传多样性是物种进化的物质基础,高水平的遗传多样性有利于提高外来入侵种在新生境中的适合度和定居能力,而低水平的遗传多样性则可能制约外来种对新环境的适应能力和进化潜力,因杂合度降低与近交衰退可能会导致种群灭绝(Jump et al., 2009;Crawford & Whitney,2010)。珠江源头9个波氏吻虾虎种群的96只个体具有5个单倍型,单倍型数较少,Hd为0.359±0.059,Nd为0.021 ±0.010。在珠江水系鱼类的遗传多样性研究中,分布在东江、北江、贺江、桂江和柳江的中国南方间䱻Hermibarbus medius的Hd为0.506~0.806,Nd为0.000 45~0.006 14(蓝昭军等,2016),珠江长臂Cranoglanis bouderius的分别为0.901、0.003 66(谢少林等,2016),经济鱼类西江鲫Carassius auratus的分别为0.900、0.006 7(匡天旭等,2018);珠江水系的青鱼Mylopharyngodon piceus(付晓艳,2011)、鳜鱼Siniperca chuatsi(司从利,2012)、草鱼Ctenopharyngodon idellus(朱叶,2012)、鲮鱼Cirrhinus molitorella(陈德荫,2013)、翘嘴鲌Culter Alburnus和大眼华鳊Sinibrama macrops(杨子拓,2016)、中华沙塘鳢Odontobutis sinensis(王丹,2017;王丹等,2018)等鱼类均表现出Hd高、Nd低的特点。这可能是历史种群曾发生瓶颈效应,而后伴随出现种群快速增长及积累新突变(Grant & Bowen,1998)。与此同时,珠江水系一些野生鱼类则表现出较高的遗传多样性水平,即高单倍型多样性和高核苷酸多样性,如鲇鱼Silurus asotus(徐丹丹,2013)、赤眼鳟Squaliobarbus curriculus(杨慧荣等,2013),原因可能在于此类土著物种拥有着较大而稳定的群体及长期进化历史,或者由于异域分化的谱系出现了二次交流,从而积累了丰富的核苷酸突变(Grant & Bowen,1998)。
通过与上述物种的比较,波氏吻虾虎群体的遗传多样性表现为低单倍型多样性与高核苷酸多样性,可能是该物种入侵珠江源头9个自然水库种群所在区域,群体的有效种群较低,随后经历了地理隔离种群的再次分化和高度的遗传瓶颈效应。据相关文献的记述,波氏吻虾虎成为珠江源头云南地区的入侵种,可能主要是外省随家鱼苗带入造成的(路瑞锁,宋豫秦,2003;伍汉霖,钟俊生,2008;陈小勇,2013)。家鱼苗的多次放养可能成为波氏吻虾虎入侵云南高原湖泊和水库自然种群的途径,可能因入侵地新环境的选择压力产生定居的瓶颈效应,而处于小种群状态,而水库大坝和水电站的修建导致了种群的再次隔离分化,从而出现了群体低单倍型多样性与高核苷酸多样性的特征。同时,9个自然水库种群存在的遗传多样性差异表现为,种群DM、ZSP均为高遗传多样性水平,而种群YJ则为较低的遗传多样性,其他种群未分析出遗传多样性信息。上述种群所在的水库均为饮用水或灌溉用水,筑以大坝进行保护,且均有多次家鱼苗放养或增殖放流的历史。因此,由于多次随家鱼苗引入,波氏吻虾虎的不同来源基因型个体可能在种群DM、ZSP进行了汇合,很大程度上弥补了奠基者效应或瓶颈效应造成的遗传多样性下降,反而为种内杂交创造新的单倍型或基因型提供了机会,因此2个种群保持了种群稳定和较高的遗传多样性水平(施雯等,2010)。同时,可能受到了近期奠基者事件或瓶颈效应的影响,缺乏后续有效的人为引入或不同单倍型对不同生境具有的适应性差异,种群BL、XX、MD、SB、TYD、XSB仅保持了具有唯一单倍型个体构成的种群,均为一个共同祖先繁衍形成的子代个体,种群YJ则出现了种群扩张(Tajima's D=-1.873,P=0.010)和新突变的积累而表现为低水平的遗传多样性,这些水库种群很可能面临近交衰退和种群灭绝的风险,其入侵能力将大大降低。
3.2 波氏吻虾虎群体的遗传结构淡水鱼类的生活环境因受到河流河段的限制,较容易出现地理隔离影响下的种群分化格局(郑新,2016)。构建的系统发育树和网络图表明,波氏吻虾虎种群存在一定的分化结构,5个单倍型分化为2支,单倍型Hap2、Hap5形成的第一分支(种群DM的3只个体和种群ZSP的10只个体构成),以及单倍型Hap1、Hap3、Hap4形成的第二分支(种群BL、XX、MD、YJ、SB、TYD、XSB和种群DM的8只个体、种群ZSP的2只个体构成)。AMOVA结果证实,波氏吻虾虎种群间的遗传变异率显著高于种群内,且具有较高的遗传分化系数。波氏吻虾虎在珠江源头的自然水库群体呈现出显著的遗传分化结构,亦有其他入侵种在入侵地表现出相似的遗传结构(刘红艳等,2016;秦凯波等,2017)。主要原因在于波氏吻虾虎各种群所在的自然水库生境,均建设有蓄容作用的大坝,可能提供了入侵种群相对封闭的水域条件,阻断了种群间的迁移扩散路径,种群间基因流受限,继而产生了种群间的生境隔离与遗传分化。
外来物种的适应性进化成为其入侵过程中的重要应对对策,而其遗传多样性被一致认为是影响外来物种适应和进化潜力的关键因素,成为影响入侵成功与否的重要因素之一(施雯等,2010)。本研究中,波氏吻虾虎群体具有较高的遗传多样性水平与显著的遗传结构,其丰富的核苷酸多样性来源于多次人为无意识引入而产生了种内杂交,以及水利大坝修建导致了隔离种群的遗传分化,可能成为该物种成功入侵珠江源头地区的重要原因。自20世纪60年代江南四大家鱼引入云南高原地区,无意识地将波氏吻虾虎带入了高原湖泊与水库自然生境(路瑞锁,宋豫秦,2003)。入侵种群定居过程中可能经历了奠基者事件和遗传瓶颈效应的严重影响,部分种群出现遗传多样性的降低,然而在各水库、湖泊自然种群的人为交叉放养家鱼苗的活动,可能将单一祖先起源的相同单倍型个体和不同来源的单倍型个体进行了扩散。因而,波氏吻虾虎在不同的水库自然种群呈现出遗传差异特征,具有共同起源但适应能力较强的单倍型个体入侵了所有的自然生境,有些不同来源的单倍型个体对于生境具有适应性差异,加上某些种群(如种群DM和ZSP)因人为多次引入了不同来源的单倍型而发生了汇合。故而,部分种群共享且拥有唯一单倍型或低的遗传多样性,少数种群具有较高的遗传多样性水平和种群稳定性,后者可能成为人为多次引种的汇合地,也可能成为波氏吻虾虎在珠江源头入侵的扩散起源地(施雯等,2010;刘红艳等,2016)。珠江上游地区存在因大量水利大坝建设形成的破碎化生境(陈艳等,2017;陈锋等,2018),外来物种入侵加重了生态危害。为有效保护珠江土著鱼类物种多样性与水生生物多样性,本研究建议,应该严格加强波氏吻虾虎在珠江上游地区的入侵管控,采取有效措施减少因人为放养家鱼苗和增殖放流带来的无意识扩散,尤其要重视针对波氏吻虾虎遗传多样性较高种群的监测和入侵防治,加强该物种入侵生态学的系统研究工作,并充分考虑在水利大坝旁设置土著鱼类的洄游通道。
致谢: 本研究受云南省高校云贵高原动植物多样性及生态适应性进化重点实验室、云南省高校云南特境内生菌资源的开发与利用科技创新团队的资助,在此一并予以感谢!| 陈德荫. 2013.中国南方野生鲮鱼(Cirrhinus molitorella)遗传多样性研究[D].广州: 暨南大学. http://cdmd.cnki.com.cn/Article/CDMD-10559-1013027914.htm |
| 陈锋, 雷欢, 郑海涛, 等. 2018. 珠江干流梯级开发对鱼类的影响与减缓对策[J]. 湖泊科学, 30(4): 1097–1108. |
| 陈小勇. 2013. 云南鱼类名录[J]. 动物学研究, 34(4): 281–343. |
| 陈艳, 黄晓霞, 王琇瑜. 2017. 珠江流域河流生境破碎化对鱼类物种多样性的影响[J]. 云南地理环境研究, 29(3): 62–68. DOI:10.3969/j.issn.1001-7852.2017.03.010 |
| 付晓艳. 2011.长江和珠江水系青鱼线粒体细胞色素b基因遗传多样性分析[D].广州: 暨南大学. http://cdmd.cnki.com.cn/article/cdmd-10559-1011128023.htm |
| 匡天旭, 帅方敏, 陈蔚涛, 等. 2018. 西江鲫的遗传多样性与群体结构[J]. 南方水产科学, 14(5): 29–35. |
| 蓝昭军, 范明君, 黄小林, 等. 2016. 基于线粒体Cyt b基因的中国南方唇鲭(Hermibarbus labeo)和间䱻(Hermibarbus medius)种群分化及亲缘地理研究[J]. 生态学报, 36(19): 6091–6102. |
| 李帆. 2011.钱塘江流域的吻虾虎鱼属(鲈形目: 虾虎鱼科)物种的分类与分布研究[D].上海: 复旦大学. http://d.wanfangdata.com.cn/Thesis/Y2703253 |
| 刘红艳, 李存耀, 熊飞. 2016. 入侵地和原产地太湖新银鱼群体遗传结构[J]. 水产学报, 40(10): 1521–1530. |
| 路瑞锁, 宋豫秦. 2003. 云贵高原湖泊的生物入侵原因探讨[J]. 环境保护, 8(9): 35–37. |
| 倪勇, 伍汉霖. 2006. 江苏鱼类志[M]. 北京: 中国农业出版社: 678-679. |
| 秦凯波, 仇玉萍, 郑毅. 2017. 云南8个地区食蚊鱼种群的遗传结构分析[J]. 西南林业大学学报(自然科学版), 37(4): 113–119. |
| 施雯, 耿宇鹏, 欧晓昆. 2010. 遗传多样性与外来物种的成功入侵:现状和展望[J]. 生物多样性, 18(6): 590–597. |
| 司从利. 2012.珠江水系鳜鱼的遗传多样性研究[D].广州: 暨南大学. http://cdmd.cnki.com.cn/Article/CDMD-10559-1012030876.htm |
| 王丹, 李强, 刘至治, 等. 2018. 基于线粒体Cyt b基因的中华沙塘鳢种群间遗传多样性分析[J]. 基因组学与应用生物学, 37(6): 2361–2367. |
| 王丹. 2017.长江及珠江流域的中华沙塘鳢种群遗传多样性分析[D].上海: 上海海洋大学. |
| 王华, 郭延蜀. 2009. 波氏吻鰕虎鱼的胚胎发育[J]. 四川动物, 28(2): 184–188. |
| 伍汉霖, 钟俊生. 2008. 中国动物志硬骨鱼纲鲈形目(五)虾虎鱼亚目[M]. 北京: 科学出版社: 578-580. |
| 谢少林, 吕子君, 周爱国, 等. 2016. 基于线粒体Cyt b和COⅠ基因对珠江、海南和红河长臀群体遗传结构分析[J]. 生态科学, 35(3): 65–72. |
| 徐丹丹. 2013.基于微卫星标记和线粒体Cyt b基因序列的鲇遗传多样性研究[D].重庆: 西南大学. http://cdmd.cnki.com.cn/Article/CDMD-10635-1013264327.htm |
| 杨慧荣, 刘丽, 龚世园, 等. 2013. 利用CR和Cyt b基因序列组合评价珠江和长江水系赤眼鳟的遗传多样性[J]. 华中农业大学学报, 32(4): 84–91. DOI:10.3969/j.issn.1000-2421.2013.04.016 |
| 杨子拓. 2016.珠江流域三种鲌亚科鱼类的遗传结构及遗传多样性分析[D].广州: 华南农业大学. http://cdmd.cnki.com.cn/Article/CDMD-10564-1016923436.htm |
| 郑新. 2016.中国子陵吻虾虎鱼和小黄黝鱼的种群结构及遗传多样性分析[D].上海: 上海海洋大学. http://cdmd.cnki.com.cn/Article/CDMD-10264-1016912363.htm |
| 朱叶. 2012.基于线粒体细胞色素b的3江水系草鱼群体遗传多样性分析[D].广州: 暨南大学. http://cdmd.cnki.com.cn/article/cdmd-10559-1012031210.htm |
| Allex CF, Shavlik JW. 1999. Computational methods for fast and accurate DNA fragment assembly[D]. Madison: University of Wisconsin-Madison. http://www.researchgate.net/publication/268325883_COMPUTATIONAL_METHODS_FOR_FAST_AND_ACCURATE_DNA_FRAGMENT_ASSEMBLY |
| Crawford KM, Whitney KD. 2010. Population genetic diversity influences colonization success[J]. Molecular Ecology, 19(6): 1253–1263. DOI:10.1111/mec.2010.19.issue-6 |
| Darriba D, Taboada GL, Doallo R, et al. 2012. jModele Test 2:more models, new heuristics and high-performance computing[J]. Nature Methods, 9(8): 772. |
| Excoffier L, Lischer HEL. 2010. Arlequin suitever 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 10(3): 564–567. DOI:10.1111/men.2010.10.issue-3 |
| Excoffier L, Smouse PE, Quattro JM. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J]. Genetics, 131(2): 479–491. |
| Grant WS, Bowen BW. 1998. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity, 89(5): 415–426. DOI:10.1093/jhered/89.5.415 |
| Guo ZQ, Cucherousset J, Lek S, et al. 2013. Comparative study of the reproductive biology of two congeneric and introduced goby species:implications for management strategies[J]. Hydrobiologia, 709(1): 89–99. DOI:10.1007/s10750-012-1439-8 |
| Guo ZQ, Liu JS, Lek S, et al. 2012. Habitat segregation between two congeneric and introduced goby species[J]. Fundamental and Applied Limnology, 181(3): 241–251. DOI:10.1127/1863-9135/2012/0397 |
| Guo ZQ, Liu JS, Lek S, et al. 2014. Trophic niche differences between two congeneric goby species:evidence for ontogenetic diet shift and habitat use[J]. Aquatic Biology, 20: 23–33. DOI:10.3354/ab00530 |
| Guo ZQ, Liu JS, Lek S, et al. 2016. Age, growth and population dynamics of two congeneric and invasive gobies, Rhinogobius giurinus and R. cliffordpopei (Actinopterygii, Gobiidae)in a plateau lake, southwestern China[J]. Hydrobiologia, 763(1): 69–79. DOI:10.1007/s10750-015-2361-7 |
| Guo ZQ, Liu JS, Lek S, et al. 2017. Coexisting invasive gobies reveal no evidence for temporal and trophic niche differentiation in the sublittoral habitat of Lake Erhai, China[J]. Ecology of Freshwater Fish, 26(1): 42–52. DOI:10.1111/eff.12249 |
| Jump AS, Marchant R, Penuelas J. 2009. Environmental change and the optional value of gentic diversity[J]. Trends in Plant Science, 14(1): 51–58. DOI:10.1016/j.tplants.2008.10.002 |
| Kyte J, Doolittle RF. 1982. A simple method for displaying the hydropathic character of a protein[J]. Journal of Molecular Biology, 157(1): 105–132. DOI:10.1016/0022-2836(82)90515-0 |
| Nichols JT. 1925. Some Chinese fresh-water fisher Ⅻ. A small goby from central YangTze[J]. American Museum Novitates, 185: 5. |
| Richards M, CôrteReal H, Forster P, et al. 1996. Paleolithic and neolithic lineages in the European mitochondrial gene pool[J]. American Journal of Human Genetics, 61(1): 185–203. |
| Ronquist F, Huelsenbeck JP. 2003. MrBayes 3:Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 19(21): 1572–1574. |
| Rozas J, Rozas R. 1995. DnaSP, DNA sequence polymorphism:an interactive program for estimating population genetics parameters from DNA sequence data[J]. Bioinformatics, 11(6): 621–625. DOI:10.1093/bioinformatics/11.6.621 |
| Tamura K, Stecher G, Peterson D, et al. 2013. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 44(4): 2725–2729. |
| Zhong LQ, Wang MH, Li DM, et al. 2018. Complete mitochondrial genome of freshwater goby Rhinogobius cliffordpopei (Perciformes, Gobiidae):genome characterization and phylogenetic analysis[J]. Genes & Genomics, 40(11): 1137–1148. |
 2019, Vol. 38
2019, Vol. 38