扩展功能
文章信息
- 赵遵岭, 王雪庆, 杨璞
- ZHAO Zunling, WANG Xueqing, YANG Pu
- 白蜡虫far3基因cDNA全长克隆、原核表达和酶活分析
- cDNA Cloning, Prokaryotic Expression, and Enzyme Activity Analyses of far3 Gene from Ericerus pela
- 四川动物, 2019, 38(2): 149-156
- Sichuan Journal of Zoology, 2019, 38(2): 149-156
- 10.11984/j.issn.1000-7083.20180328
-
文章历史
- 收稿日期: 2018-11-08
- 接受日期: 2019-01-15
白蜡虫Ericerus pela二龄雄虫分泌的白蜡具有隔水防热、润滑保湿、高熔点等特性,被广泛应用于食品包装、精密仪器润滑固封、医药以及化妆品等行业(何晨柳等,2010;杨璞等,2012)。白蜡主要由二十六酸二十六酯、二十八酸二十八酯组成,另有二十四酸二十四酯、三十酸三十酯等(Yang et al., 2012)。在动植物中,蜡酯主要通过酰基还原途径合成,首先由脂酰辅酶A还原酶(fatty acyl-CoA reductase,FAR)催化脂酰辅酶A还原为相应脂肪醇,之后脂肪醇与脂酰辅酶A在蜡酯合酶的作用下酯化反应成酯(Lardizabal et al., 2000;Metz et al., 2000;Yang et al., 2012;杨璞等,2012)。FAR为蜡酯合成途径关键酶之一,在生物蜡酯的合成中扮演着不可或缺的角色。
在动植物中,对人类Homo sapiens与小鼠Mus musculus(Cheng & Russell, 2004a, 2004b)、拟南芥Arabidopsis thaliana(Rowland et al., 2006;Li et al., 2008;Domergue et al., 2010)、家鸡Gallus gallus domesticus、家鹅Anser anser domesticus、仓鸮Tyto alba(Hellenbrand et al., 2011)等FAR进行了基因克隆和表达、酶活性、组织表达定位等研究,表明FAR参与泌蜡的功能与其组织表达部位一致,一些物种的FAR的最适底物与生成蜡酯的脂肪醇一致。在昆虫中,FAR的研究则主要集中在性信息素合成方面(Moto et al., 2003;Antony et al., 2009;Liénard et al., 2010;Teerawanichpan et al., 2010),而蜡酯合成的far基因相关信息很少。克隆和表达昆虫中参与蜡酯合成的far基因,对于阐述昆虫蜡酯合成机理具有重要意义。
前期对白蜡虫泌蜡高峰期转录组数据进行分析和实时定量PCR验证,筛选出了参与白蜡虫泌蜡的far候选基因far1和far3 (Yang et al., 2012)。后期获得far2基因,FAR2体外酶活实验表明可催化C28脂酰辅酶A还原为C28脂肪醇(王雪庆,2018),而白蜡虫FAR3是否可以催化白蜡其他主要成分的生成,尚不清楚。本研究通过cDNA末端快速扩增(RACE)技术扩增得到白蜡虫far3基因cDNA全长,构建原核表达载体,将far3基因在大肠杆菌Escherichia coli中进行表达,并进行质谱验证及活性分析,为后续单克隆抗体制备及FAR真核表达奠定基础。
1 实验方法 1.1 供试材料实验所用白蜡虫二龄雄虫采于云南省昆明市中国林业科学研究院资源昆虫研究所人工大棚所种女贞Ligustrum lucidum树上。
1.2 二龄雄虫总RNA提取及RACE模板的合成将白蜡虫二龄雄虫置于1.5 mL离心管中,依照Trizol试剂盒(Invitrogen,美国)操作说明提取总RNA,使用紫外分光光度计测定提取RNA的浓度以及纯度,当A260/A280为1.8~2.0时,说明RNA纯度较高。以提取的总RNA为模板,按照SMARTerTM RACE cDNA Amplification Kit User Manual(Clontech,日本)操作说明分别合成5'RACE和3'RACE模板。
1.3 RACE技术扩增白蜡虫far3基因根据已获得的far基因片段序列分别设计3'RACE和5'RACE特异性引物,并各以反转录3'RACE模板和5'RACE模板进行序列3'端和5'端的扩增,经PCR及巢式PCR获得目的条带。扩增体系为:10 μL 5×Q5 Reaction Buffer,1 μL 10 mmol·L-1 dNTPS,0.5 μL Q5 High-Fidelity DNA Polymerase(NEB,美国),2.5 μL 10 μmol·L-1 Forward Primer,2.5 μL 10 μmol·L-1 Reverse Primer,2.5 μL cDNA模板,ddH2O补足至50 μL。PCR的反应程序为98 ℃ 30 s;98 ℃ 8 s,55 ℃ 25 s,72 ℃ 1 min,32个循环;72 ℃ 2 min充分延伸。
经1%琼脂糖凝胶电泳后,在紫外凝胶成像仪上切下对应条带,按照GeneJET Gel Extraction Kit试剂盒(ThermoFisher Scientific,美国)操作说明回收胶,最后加入20 μL预热的灭菌ddH2O洗脱。然后对回收产物进行加A操作,加A体系为5 μL 10×PCR Buffer(Mg2+ plus),4 μL dNTP Mixture,0.5 μL rTaq DNA Polymerase(TaKaRa,日本),20 μL PCR回收产物,ddH2O补足至50 μL。PCR仪中72 ℃反应20 min,结束后立即置于冰上停止反应。回收加A产物操作同上所述。
取加A回收产物进行连接,连接体系如下:2.5 μL Ligase Buffer,0.5 μL T4 Ligase(Promega,上海),0.5 μL PGEM-T easy载体,1.5 μL加A回收产物,4 ℃过夜连接。将连接产物转化至大肠杆菌DH5α感受态细胞(TaKaRa,日本)中,37 ℃ 160 r·min-1培养1.5 h后涂于氨苄抗性平板进行培养,通过蓝白斑筛选获得阳性克隆,并进行菌液PCR检测,阳性克隆送生工生物工程(上海)股份有限公司测序。
1.4 序列分析使用DNAMAN对测序结果拼接获得far3基因序列全长,并推导其开放阅读框(ORF)及氨基酸序列,利用SMART(http://smart.embl-heidelberg.de/)分析FAR3序列。将其核酸序列与NCBI数据库进行比对,下载NCBI数据库中其他物种的FAR氨基酸序列(图 2),利用MEGA 5.05中Neighbour-Joining法、bootstrap置信度1 000次自导复制构建系统进化树。

|
| 图 2 不同物种FAR系统进化树 Fig. 2 Phylogenetic tree of FAR of different species |
| |
将far3基因ORF序列进行密码子优化,并加入BamH Ⅰ和Hind Ⅲ酶切位点,在其后加上增强绿色荧光蛋白基因(eGFP),交由生工生物工程(上海)股份有限公司合成。将合成的基因序列连入pET-30a载体中,重组质粒命名为pET-30a-eGFP/EpelFAR3。将重组质粒转入大肠杆菌BL21感受态细胞中,涂于Kan+抗性平板,挑选阳性克隆进行菌液PCR检测,阳性克隆送生工生物工程(上海)股份有限公司测序,测序正确的菌液加甘油后-80 ℃保存。
1.6 原核表达SDS-PAGE分析取10 μL测序成功的菌液于1 mL新鲜Kan+培养基中过夜培养,按1: 100比例将过夜培养菌液加入4 mL新鲜Kan+LB液体培养基中继续培养至OD600=0.3左右,使用浓度为0.05 mmol·L-1的异丙基硫代半乳糖苷(IPTG)诱导表达6 h后,取1 mL菌液离心收集,并加入100 μL 1×protein loading buffer(TransGen Biotech,北京)悬浮,于-80 ℃过夜,取10 μL样品经100 ℃ 5 min变性后进行10% SDS-PAGE电泳,电泳后经FastBlue蛋白快速染色液(biosharp,安徽合肥)染色清洗后在GS-900(Bio-Rad,美国)中扫描观察。
1.7 Western blot验证收集诱导表达的菌体,磷酸缓冲盐溶液(PBS)重悬浮,经超声波破碎后将上清与沉淀分别收集,沉淀为不可溶性蛋白,用2%十二烷基硫酸钠(SDS)溶解。将上清与SDS溶解沉淀蛋白分别进行10%SDS-PAGE电泳,转膜,孵育抗体,一抗为eGFP鼠抗(ABCam,美国),二抗为山羊抗小鼠IgG H & L(HRP)(ABCam,美国),洗膜后将超敏化学发光试剂(Affinity,美国)A液与B液等量混合滴于膜上,在ChemiDoc XRS仪(Bio-Rad,美国)中观察。
1.8 质谱分析诱导表达的菌体蛋白样品进行SDS-PAGE电泳,经考马斯亮蓝染色后切取目的蛋白条带于1.5 mL离心管中,并使用ddH2O漂洗2次,交由生工生物工程(上海)股份有限公司提取条带蛋白并进行LC-MSMS质谱分析。操作步骤如下:使用Nano-RPLC Buffer A溶解样品,利用Eksigent nanoLC-UltraTM 2D系统(AB SCIEX,美国)进行在线Nano-RPLC液相色谱分析,使用C18预柱(100 μm×3 cm,C18,3 μm,150 Å),以2 μL·min-1的流速上样,并以此流速冲洗脱盐10 min。采用C18反相色谱柱(75 μm×15 cm,C18,3 μm,120 Å,ChromXP Eksigent)作为分析柱,梯度洗脱程序为流动相B自5%按每3 min升高1%至35%。其中流动相A为0.1%甲酸与2%乙腈的混合液,流动相B为0.1%甲酸和98%乙腈的混合液。
质谱采用TripleTOF5600系统(AB SCIEX,美国)与纳升喷雾Ⅲ离子源(AB SCIEX,美国)结合,喷雾电压2.5 kV,气帘气压30 PSI,雾化气压5 PSI,加热器温度150 ℃,采用信息依赖的采集工作模式进行质谱扫描。利用Mascot 2.3进行数据处理,并与推导的白蜡虫FAR3氨基酸理论序列理论质谱图进行比对。
1.9 表达蛋白活性分析取诱导表达的菌液1 mL,离心收集菌体,加入100 μL 10 mmol·L-1 Tris-HCl(pH7.3)重悬浮,超声波破碎。分别以C24脂酰辅酶A、C26脂酰辅酶A、C28脂酰辅酶A以及C30脂酰辅酶A等(Avanti,美国)为底物对蛋白进行活性检测,反应体系如下:2% dimethyl sulfoxide,2.5 mmol·L-1 NADPH,脂酰辅酶A 1 μmol·L-1,10 μL表达蛋白,10 mmol·L-1 Tris-HCl(pH7.3)补足至400 μL,30 ℃反应40 min,加入400 μL氯仿停止反应并萃取反应产物,过滤。采用气相色谱法检测酶活反应结果,气相色谱(1890,惠普上海分析仪器有限公司)选用火焰电离检测器与HP-5色谱柱,1 μL上样量,程序如下:以250 ℃为色谱柱起始温度,之后以5 ℃·min-1提升至280 ℃,维持10 min。
2 结果与分析 2.1 RACE结果与序列分析RNA的A260/A280为1.92,浓度为280 ng·μL-1,可以作为RACE反转录的模板。3'RACE扩增长度为840 bp(图 1:A),5'RACE扩增长度为1 122 bp(图 1:B),与保守序列拼接后获得的基因全长为1 849 bp,命名为far3基因,其中,ORF为1 566 bp,3'末端非编码区121 bp,5'末端非编码区162 bp。DNAMAN分析结果显示,ORF编码522个氨基酸,分子量约为60.07 kDa,预测等电点为9.02。
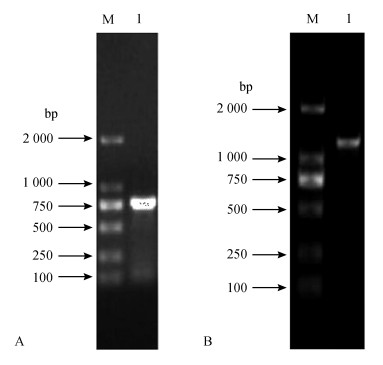
|
| 图 1 白蜡虫far3基因RACE扩增电泳图 Fig. 1 Agarose gel electrophoresis results of RACE amplification of Ericerus pela far3 gene A. 3'RACE扩增条带,B. 5'RACE扩增条带;M泳道为2 000 bp marker,1泳道为PCR扩增产物 A. 3'RACE amplification result, B. 5 'RACE amplification result; M. 2 000 bp marker, lane 1. PCR amplification product |
| |
经SMART分析,FAR3在26~297 aa处有一NAD结合结构域,496~518 aa处有一螺旋状跨膜区域,其N末端在胞内,没有预测到信号肽序列。利用MEGA 5.05构建进化树进行系统发育分析,白蜡虫FAR3与人类、小鼠、黑腹果蝇Drosophila melanogaster等物种的FAR聚为一支(图 2)。
2.2 原核表达将重组质粒pET-30a-eGFP/EpelFAR3转入大肠杆菌BL21感受态细胞,挑取阳性单克隆,采用T7引物进行PCR检测,目的基因距离上下游T7引物多出336 bp,目的条带和eGFP共有2 283 bp,总计扩增长度为2 619 bp,检测结果与之相符(图 3)。经比对,测序结果与之相符。

|
| 图 3 重组质粒pET-30a-eGFP/EpelFAR3菌液PCR检测 Fig. 3 Agarose gel electrophoresis result of bacteria liquid PCR detection of pET-30a-egfp/EpelFAR3 recombinant plasmid M泳道为5 000 bp marker,1泳道为菌液PCR扩增产物 M. 5 000 bp marker, lane 1. PCR amplification product by using bacterial liquid as template |
| |
在0.05 mmol·L-1 IPTG诱导6 h后,成功表达FAR3-eGFP融合蛋白(图 4)。

|
| 图 4 FAR3原核表达菌体总蛋白SDS-PAGE分析 Fig. 4 SDS-PAGE analysis of total proteins of bacteria expressing FAR3 M为marker,泳道1为空白对照,泳道2为诱导表达菌体蛋白样品,箭头所指条带为诱导表达的蛋白条带 M. marker, lane 1. the blank control, lane 2. the total protein of induced expression of FAR, the arrow points to a protein band which is induced expression |
| |
原核诱导表达的FAR3与eGFP融合蛋白分子量预计为86.84 kDa,经Western blot结果分析,上清与菌体沉淀中均含有FAR3-eGFP的融合表达蛋白,且目的条带位置与理论大小相符(图 5),其中,上清中含有部分目的蛋白,说明表达的该目标蛋白有一部分是可溶的。
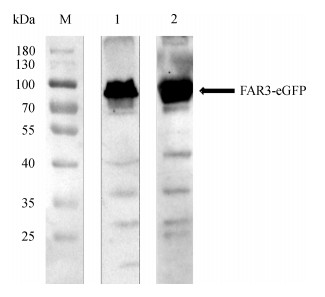
|
| 图 5 FAR3原核表达菌体裂解后上清与沉淀蛋白的Western blot验证 Fig. 5 Western blot verification of FAR3 prokaryotic expression in the supernatant and precipitation of bacteria lysate M为marker,泳道1为超声破碎离心后的上清溶液,泳道2为超声破碎离心后的菌体沉淀 M. protein marker, lane 1. supernatant after ultrasonic disruption and centrifugation, lane 2. the bacterial precipitation after ultrasonic disruption and centrifugation |
| |
经质谱检测,共有292个肽段与理论序列匹配,蛋白质分值为3 900(图 6),肽段覆盖度74%,表明原核表达的白蜡虫FAR3蛋白与理论蛋白肽段一致,目的基因表达成功。

|
| 图 6 蛋白质分值质谱分析结果 Fig. 6 Mass spectrometric analysis of protein score |
| |
经气相色谱检测,原核表达蛋白FAR3在以C24脂酰辅酶A、C26脂酰辅酶A、C28脂酰辅酶A以及C30脂酰辅酶A为底物进行活性分析时,没有产生相对应的脂肪醇(图 7)。

|
| 图 7 原核表达蛋白FAR3酶活反应产物气相色谱检测 Fig. 7 Gas chromatography detection peak map of the products from enzyme reaction of FAR3 expressed in prokaryotic cells A. C24脂肪醇标准品和以C24脂酰辅酶A为底物进行FAR3活性检测的色谱峰图,B. C26脂肪醇标准品和以C26脂酰辅酶A为底物进行FAR3活性检测的色谱峰图,C. C28脂肪醇标准品和以C28脂酰辅酶A为底物进行FAR3活性检测的色谱峰图,D. C30脂肪醇标准品和以C30脂酰辅酶A为底物进行FAR3活性检测的色谱峰图;箭头所指为标准品的色谱峰,倒三角形所指为酶活反应产物的色谱峰 A. chromatographic peak map of C24 fatty alcohol standard substance and the products of FAR3 activity enzyme reaction using C24 fatty acyl-CoA as substrate, B. chromatographic peak map of C26 fatty alcohol standard substance and the products of FAR3 activity enzyme reaction using C26 fatty acyl-CoA as substrate, C. chromatographic peak map of C28 fatty alcohol standard substance and the products of FAR3 activity enzyme reaction using C28 fatty acyl-CoA as substrate, D. chromatographic peak map of C30 fatty alcohol standard substance and the products of FAR3 activity enzyme reaction using C30 fatty acyl-CoA as substrate; the arrow points to the chromatographic peak of the standard substance, the inverted triangle points to the chromatographic peak of the FAR3 enzyme reaction products |
| |
FAR在生物体内普遍存在,但参与的生理功能不同。西蒙得木Simmondsia chinensis FAR参与西蒙得木油的合成(Kalscheuer,2006);而在家蚕Bombyx mori、欧洲玉米螟Ostrinia nubilalis以及意大利蜜蜂Apis mellifera等昆虫中鉴定到的FAR参与信息素的合成(Moto et al., 2003;Antony et al., 2009;Teerawanichpan et al., 2010)。拟南芥已鉴定的8个基因中FAR3参与根茎叶角质层蜡的形成,FAR1、FAR4、FAR5则在根内皮细胞中参与软木脂的合成(Rowland et al., 2006;Domergue et al., 2010)。研究表明,FAR的功能与组织表达部位关系很大,不同物种相同功能FAR的序列较为相似,功能相似的FAR在系统进化分析中进化关系较近(Johnson et al., 2018)。本研究在实验室前期工作的基础上通过RACE技术扩增获得了白蜡虫far3基因全长及其ORF,并对ORF编码蛋白FAR3与其他多个物种进行系统发育分析,发现白蜡虫FAR3与人类、小鼠和果蝇等参与蜡酯合成的FAR聚为一支,此外,与泌蜡不相关的白蜡虫热激蛋白单独一支,说明白蜡虫FAR3极可能在白蜡合成中起作用。
在原核诱导表达时,Zhang等(2015)发现增加IPTG浓度对形成可溶性蛋白会有不利影响,Saffarian等(2016)发现低浓度IPTG诱导表达可产生更多可溶性蛋白。因此在诱导表达FAR3-eGFP融合蛋白时选择较低的IPTG诱导浓度(0.05 mmol·L-1)。此外,诱导时间过长可能导致菌体密度过大,菌体生长至平台期,培养基中营养消耗殆尽,导致表达蛋白量不高,故设置诱导时间为6 h。实验结果表明此诱导条件能成功表达大量目的蛋白。对表达的蛋白进行质谱分析,蛋白质分值为3 900,肽段覆盖度为74%。在质谱分析中,蛋白质分值越高,质谱检测结果也更具可靠性。覆盖度越高也表明所获得肽段占理论序列比例越高,结果越可靠(Liu et al., 2004),由此可以确定所测蛋白为目的蛋白。
动植物FAR异源表达进行酶活研究时,多采用真核表达系统,如意大利蜜蜂头部分离出的FAR、拟南芥FAR5和FAR8以及小麦3种TaFAR在酵母细胞中表达成功(Teerawanichpan et al., 2010;Chacónet al., 2013;Wang et al., 2016),人与小鼠的FAR在HEK293细胞中表达成功(Cheng & Russell,2004b)。但也有真核生物FAR在大肠杆菌中表达成功的例子,如Kalscheuer等(2006)在重组大肠杆菌中共表达了西蒙得木的双功能酰基辅酶A还原酶和贝氏不动杆菌Acinetobacter baylyi ADP1的蜡酯合成酶并获得与西蒙得木相似的蜡酯;Doan等(2009)将来自于拟南芥与西蒙得木、蚕蛾、小麦及小鼠不同源的6种far基因在大肠杆菌中进行表达,其中1种far基因表达蛋白没有活性,其他5种far基因皆表达并催化生成了相应的脂肪醇产物。
本研究通过原核表达获得大量白蜡虫FAR3蛋白,对其进行活性分析。培养基上清中未检测到表达蛋白,取诱导表达菌体破碎后上清进行活性验证。另因白蜡主要成分为二十六酸二十六酯、二十八酸二十八酯,所以进行蛋白活性检测时选用C24脂酰辅酶A、C26脂酰辅酶A、C28脂酰辅酶A以及C30脂酰辅酶A为反应底物。经气相色谱检测无相应理论脂肪醇产物的生成,可能是原核表达真核蛋白的翻译后加工修饰体系不完善,不能形成正确的空间结构,影响了生物活性(郭广君等,2006),也可能是酶活反应条件不佳,催化生成产物少及萃取损失等原因导致气相色谱无结果。但白蜡虫FAR3的成功表达为抗体制备提供了抗原,后续白蜡虫FAR3将采用真核表达进行功能验证。
4 结论本研究通过RACE技术获得了far3基因ORF 1 566 bp,构建pET-30a-eGFP/EpelFAR3原核表达质粒,以0.05 mmol·L-1IPTG诱导6 h时成功表达,经Western blot及质谱分析验证表达蛋白为白蜡虫FAR3,气相色谱检测酶活反应产物无相应理论产物的生成。
| 郭广君, 吕素芳, 王荣富. 2006. 外源基因表达系统的研究进展[J]. 科学技术与工程, 6(5): 582–592. DOI:10.3969/j.issn.1671-1815.2006.05.016 |
| 何晨柳, 孔任秋, 胡晗华. 2010. 产蜡酯聚球藻PCC7002的构建[J]. 水生生物学报, 34(6): 1202–1205. |
| 王雪庆. 2018.白蜡虫泌蜡关键基因far2活性分析及ws基因dsRNA体外表达[D].北京: 中国林业科学研究院. |
| 杨璞, 徐冬丽, 陈晓鸣, 等. 2012. 蜡酯合成途径及关键酶的研究进展[J]. 中国细胞生物学学报, 34(7): 695–703. |
| Antony B, Fujii T, Moto K, et al. 2009. Pheromone-gland-specific fatty-acyl reductase in the adzuki bean borer, Ostrinia scapulalis (Lepidoptera:Crambidae)[J]. Insect Biochemistry & Molecular Biology, 39(2): 90–95. |
| Chacón MG, Fournier AE, Tran F, et al. 2013. Identification of amino acids conferring chain length substrate specificities on fatty alcohol-forming reductases FAR5 and FAR8 from Arabidopsis thaliana[J]. Journal of Biological Chemistry, 288(42): 30345–30355. DOI:10.1074/jbc.M113.499715 |
| Cheng JB, Russell DW. 2004a. Mammalian wax biosynthesis. Ⅰ. Identification of two fatty acyl-Coenzyme A reductases with different substrate specificities and tissue distributions[J]. Journal of Biological Chemistry, 279(36): 37789–37797. DOI:10.1074/jbc.M406225200 |
| Cheng JB, Russell DW. 2004b. Mammalian wax biosynthesis. Ⅱ. Expression cloning of wax synthase cDNAs encoding a member of the acyltransferase enzyme family[J]. Journal of Biological Chemistry, 279(36): 37798–37807. DOI:10.1074/jbc.M406226200 |
| Doan TT, Carlsson AS, Hamberg M, et al. 2009. Functional expression of five Arabidopsis fatty acyl-CoA reductase genes in Escherichia coli[J]. Journal of Plant Physiology, 166(8): 787–796. DOI:10.1016/j.jplph.2008.10.003 |
| Domergue F, Vishwanath SJ, Joubès J, et al. 2010. Three Arabidopsis fatty acyl-Coenzyme A reductases, FAR1, FAR4, and FAR5, generate primary fatty alcohols associatedwith suberin deposition[J]. Plant Physiology, 153(4): 1539–1554. DOI:10.1104/pp.110.158238 |
| Hellenbrand J, Biester EM, Gruber J, et al. 2011. Fatty acyl-CoA reductases of birds[J]. BMC Biochemistry, 12: 64. DOI:10.1186/1471-2091-12-64 |
| Johnson TS, Li S, Kho JR, et al. 2018. Network analysis of pseudogene-gene relationships:from pseudogene evolution to their functional potentials[J]. Pacific Symposium on Biocomputing, 23: 536–547. |
| Kalscheuer R, Stöveken T, Luftmann H, et al. 2006. Neutral lipid biosynthesis in engineered Escherichia coli:jojoba oil-like wax esters and fatty acid butyl esters[J]. Applied & Environmental Microbiology, 72(2): 1373–1379. |
| Lardizabal KD, Metz JG, Sakamoto T, et al. 2000. Purification of a jojoba embryo wax synthase, cloning of its cDNA, and production of high levels of wax in seeds of transgenic Arabidopsis[J]. Plant Physiology, 122(3): 645–655. DOI:10.1104/pp.122.3.645 |
| Li FL, Wu XM, Lam P, et al. 2008. Identification of the wax ester synthase/acyl-coenzyme A:dia-cylglycerol acyltransferase WSD1 required for stem wax ester biosynthesis in Arabidopsis[J]. Plant Physiology, 148(1): 97–107. |
| Liénard MA, Hagström AK, Lassance JM, et al. 2010. Evolution of multicomponent pheromone signals in small ermine moths involves a single fatty-acyl reductase gene[J]. Proceedings of the National Academy of Sciences of the United States of America, 107(24): 10955–10960. DOI:10.1073/pnas.1000823107 |
| Liu H, Sadygov RG, Yates JR. 2004. A model for random sampling and estimation of relative protein abundance in shotgun proteomics[J]. Analytical Chemistry, 76(14): 4193–4201. DOI:10.1021/ac0498563 |
| Metz JG, Pollard MR, Anderson L, et al. 2000. Purification of a jojoba embryo fatty acyl-coenzyme A reductase and expression of its cDNA in high erucic acid rapeseed[J]. Plant Physiology, 122(3): 635–644. DOI:10.1104/pp.122.3.635 |
| Moto K, Yoshiga T, Yamamoto M, et al. 2003. Pheromone gland-specific fatty-acyl reductase of the silkmoth, Bombyx mori[J]. Proceedings of the National Academy of Sciences of the United States of America, 100(16): 9156–9161. DOI:10.1073/pnas.1531993100 |
| Rowland O, Zheng HQ, Hepworth SR, et al. 2006. CER4 encodes an alcohol-forming fatty acyl-coenzyme A reductase involved in cuticular wax production in Arabidopsis[J]. Plant Physiology, 142(3): 866–877. DOI:10.1104/pp.106.086785 |
| Saffarian P, Peerayeh SN, Amani J, et al. 2016. Expression and purification of recombinant TAT-BoNT/A(1-448) under denaturing and native conditions[J]. Bioengineered, 7(6): 478–483. DOI:10.1080/21655979.2016.1201252 |
| Teerawanichpan P, Robertson AJ, Qiu X. 2010. A fatty acyl-CoA reductase highly expressed in the head of honey bee (Apis mellifera) involves biosynthesis of a wide range of aliphatic fatty alcohols[J]. Insect Biochemistry & Molecular Biology, 40(9): 641–649. |
| Wang M, Wang Y, Wu H, et al. 2016. Three TaFAR genes function in the biosynthesis of primary alcohols and the response to abiotic stresses in Triticum aestivum[J]. Scientific Reports, 6: 25008. DOI:10.1038/srep25008 |
| Yang P, Zhu JY, Gong ZJ, et al. 2012. Transcriptome analysis of the Chinese white wax scale Ericerus pela with focus on genes involved in wax biosynthesis[J]. PLoS ONE, 7(4): e35719. DOI:10.1371/journal.pone.0035719 |
| Zhang Z, Kuipers G, Łukasz N, et al. 2015. High-level production of membrane proteins in E. coli BL21(DE3) by omitting the inducer IPTG[J]. Microbial Cell Factories, 14: 142. DOI:10.1186/s12934-015-0328-z |
 2019, Vol. 38
2019, Vol. 38




