扩展功能
文章信息
- 张瑞, 王明晓, 安建梅
- ZHANG Rui, WANG Mingxiao, AN Jianmei
- 华北地区中华蒙潮虫(甲壳动物亚门:等足目)种群遗传多样性研究
- Population Genetic Diversity of Mongoloniscus sinensis (Dollfus, 1901)(Crustacea:Isopoda) from North China
- 四川动物, 2019, 38(2): 139-148
- Sichuan Journal of Zoology, 2019, 38(2): 139-148
- 10.11984/j.issn.1000-7083.20180305
-
文章历史
- 收稿日期: 2018-10-16
- 接受日期: 2018-12-27
中华蒙潮虫Mongoloniscus sinensis (Dollfus,1901)隶属于节肢动物门Arthropoda甲壳动物亚门Crustacea真软甲亚纲Eumalacostrace软甲纲Malacacostra囊虾总目Peracarida等足目Isopoda潮虫亚目Oniscidea缘潮虫科Agnaridae蒙潮虫属Mongoloniscus(Boyko et al., 2008),中国特有种。中华蒙潮虫的环境适应能力非常强,主要分布于山西、北京、内蒙古、吉林等地(陈国孝,2000),是我国古北界典型的温带特有种,本课题组在山东、陕西西安、辽宁、天津、河南、河北均发现该物种。师二燕(2015)基于线粒体细胞色素c氧化酶亚基Ⅰ基因(mtCO Ⅰ)对中华蒙潮虫进行了谱系地理研究,共基于26个居群(跨越5省15市26县区)进行了分析,推测该物种可能的进化路线为从北往南,且最有可能的发源地为我国东北平原。有研究表明,中华蒙潮虫还可作为环境的重金属指示物种(牛晓倩,2015;李梦雯,2016)。关于多基因联合分析中华蒙潮虫种群遗传多样性的相关研究未见报道。
分子标记以蛋白质、核酸的突变为基础,检测生物遗传结构及其变异(白玉,2007)。动物线粒体基因组遵循严格的母系遗传且分子的各部分均共享同一祖先(Wilson et al., 2010),且线粒体基因组进化速率快,是单拷贝核DNA的5~10倍,群体内变异大,群体遗传进化常用此标记(陈复生等,2003;Ito et al., 2011;Zhou et al., 2011;Li et al., 2012),也是物种生物地理研究的常用标记之一(Avise et al., 1987;徐庆刚,花保祯,2001;王兰萍等,2013)。
线粒体内不同基因的进化速率不同。NADH5氧化还原酶基因( ND5 )进化速率相当快,是种群系统发育研究中较有效的基因之一(智妍等,2008)。CO Ⅰ基因常用于种以下的系统发育研究,具有进化速率快、富含系统发育遗传信息等特点,常用于探讨物种分类及近缘种和种群遗传结构多样性等系统进化关系(邓春兴,2014;刘平等,2018)。本研究采用基因联合(CO Ⅰ+ ND5 )的方法对中国华北地区10个地理种群89只中华蒙潮虫进行群体遗传多样性分析,以期了解华北地区中华蒙潮虫种群间的遗传分化和基因交流。
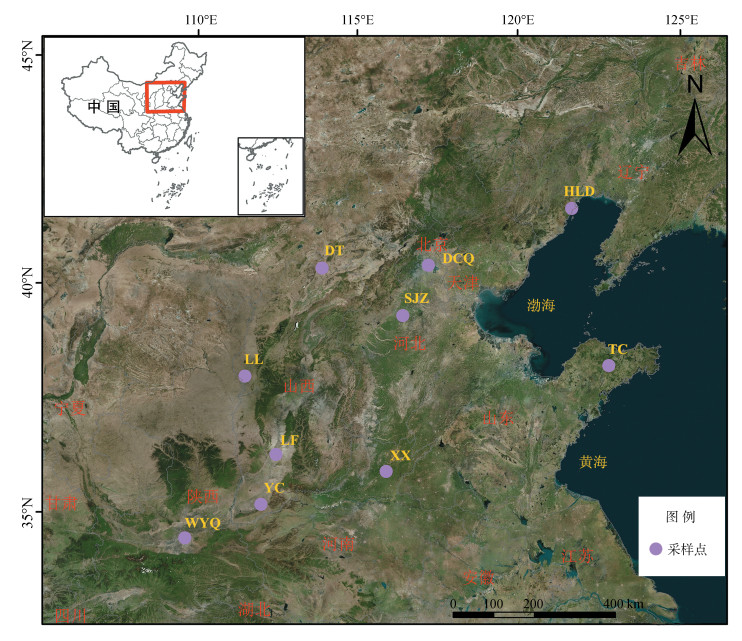
1 材料和方法 1.1 样品采集2011年4月—2018年6月采集了10个地理种群共89只中华蒙潮虫(表 1,图 1),标本浸泡于无水乙醇。
| 采样点 | 代码 | 采集时间 | 东经 | 北纬 | 样本量 |
| 北京东城区 | DCQ | 201106 | 116°24′ | 39°54′ | 6 |
| 河北石家庄 | SJZ | 201806 | 115°30′ | 38°52′ | 11 |
| 河南新乡 | XX | 201705 | 113°56′ | 35°18′ | 4 |
| 辽宁葫芦岛 | HLD | 201706 | 120°49′ | 40°42′ | 5 |
| 山西临汾 | LF | 201706 | 111°34′ | 36°05′ | 15 |
| 山西吕梁 | LL | 201707 | 110°50′ | 37°50′ | 16 |
| 山东桃村 | TC | 201706 | 121°09′ | 37°11′ | 10 |
| 山西大同 | DT | 201707 | 113°17′ | 40°04′ | 12 |
| 山西运城 | YC | 201707 | 111°04′ | 35°01′ | 5 |
| 陕西西安未央区 | WYQ | 201710 | 108°56′ | 34°22′ | 5 |
外群选用海蟑螂Ligia oceanica (Linnaeus,1767)(GenBank登录号:DQ442914)和Cylisticus convexus (De Geer,1778)(GenBank登录号:KR013002)。
1.2 DNA的提取及测序DNA的提取使用Chelex-100法(戴文申等,2007),选取绿豆大小的中华蒙潮虫胸足或腹足肌肉于0.5 mL离心管中,加纯水清洗,随后放入干净的培养皿中晾干。将晾干的组织样品放入200 μL 5%的Chelex-100离心管中,并加入5 μL 5 mg·mL-1的蛋白激酶,混匀振荡,确保肌肉组织完全浸没于Chelex-100的颗粒中。56 ℃ 3 h以上,取出振荡;100 ℃ 8 min,振荡;13 000 r·min-1离心3 min,取上清用于PCR扩增或-20 ℃保存备用。
CO Ⅰ基因引物(Folmer et al., 1994)为CO Ⅰ F:5'-GGTCAACAAATCATAAAGATATTGG-3';CO Ⅰ R:5'-TAAACTTCAGGGTGACCAAAAAATCA-3'。ND5 基因引物为877F:5'-TTTATCTTTTGGGTTCGCTA-3';1608R:5'-TAAAATTAAATCCTTGCCCTC-3',是选取中华蒙潮虫 ND5 基因全序列(GenBank登录号:MG729627)的877~1 608 bp片段(编码区序列),用Oligo 7(Rychlik,2007)加上手工调整而设计的。引物合成委托苏州金唯智生物科技有限公司完成。PCR的扩增体系为50 μL:2×HieffTM PCR Master Mix 25 μL,上、下游引物各3 μL,DNA模板7 μL,ddH2O 12 μL。
PCR在TCA0096扩增仪上进行:94 ℃ 2 min;94 ℃ 45 s,退火45 s(退火温度为CO Ⅰ基因:50 ℃;ND5 基因:45.5 ℃),68 ℃ 1 min,35个循环;72 ℃ 10 min。扩增产物用0.7%琼脂糖凝胶进行电泳检测,扩增效果良好且足量的样品原液委托苏州金唯智生物科技有限公司进行纯化及双向测序。
1.3 数据分析测序所得序列峰图用Geneious(Kearse et al., 2012)检查和筛选,经校验后进行序列双向拼接,剪切掉引物部分,并分别进行BLAST搜索以确定每个基因片段的同源性。所有序列在Geneious中排序比对。序列拼接使用SequenceMatrix,将2个基因序列合并为1个完整序列数据集,并命名为CO Ⅰ+ ND5 。利用PAUP 4.0对序列集的碱基组进行同质性检验。使用MEGA 7(Kumar et al., 2016)对获得的序列数据集计算碱基组成、保守位点、变异位点以及序列间的转换颠换比率;采用Kimura(Kimura,1980)双参数模型计算种群遗传距离。使用DnaSP 5.0(Librado & Rozas,2009)统计单倍型,计算各种群核苷酸多样性(Pi)、单倍型多样性(Hd),并构建遗传分化指数(Fst),Fst反映2个种群之间的遗传差异程度,其值与基因流(Nm)成反比,即Fst越大,基因交流程度越小;中性检验通过计算Tajima's D值和Fu's Fs值来表示种群是否经历过扩张,二者值接近0时,种群较为稳定,小于0(P < 0.05)时,种群近期经历过扩张,大于0时,种群可能出现了分化(Fu,1997)。构建单倍型岐点分布图,通过可视化曲线观察种群近期是否经历过扩张。系统发育分析分别采用MEGA 7构建最大简约(MP)树和MrBayes 3.0(Ronquist et al., 2012)构建贝叶斯(BI)树。BI树中,由于每个基因、编码基因中每个密码子位点进化速率的不同,采用Partitionfinder 1.1.1(Lanfear et al., 2012)分别对CO Ⅰ与 ND5 基因的第一、第二、第三密码子进行分区计算。
采用Arlequin 3.5(Excoffer & Lischer,2010)进行分子变异分析(AMOVA)。但所有单倍型几乎未按地理来源分支,因此按照系统发育划分出支系进行区域分组,将分布零散的运城独立为1个组。使用Network 4.1构建单倍型网络图。
2 结果与分析 2.1 序列特征中华蒙潮虫CO Ⅰ部分基因长604 bp,ND5 部分基因长615 bp,拼接后长度为1 219 bp。对数据集(CO Ⅰ + ND5 )进行统计分析,发现保守位点716个,变异位点503个(占总序列长度41.3%),其中,单一多态位点235个,简约信息位点268个。联合序列的碱基A、T、C、G平均含量分别为30.8%、41.0%、11.2%、17.0%,A+T(71.8%)明显高于C+G(28.2%),符合节肢动物门高A+T含量特点(孙红英等,2003),序列间的转换/颠换比值为2.8。
数据集(CO Ⅰ + ND5 )同质化检验结果显示,P=0.053,表明二者为同质性样品,可进行联合分析。
2.2 单倍型多样性和遗传多样性89只中华蒙潮虫线粒体联合基因序列(CO Ⅰ+ ND5 )共45种单倍型(表 2)。其中,单倍型H1[北京东城区(DCQ)、河北石家庄(SJZ)、辽宁葫芦岛(HLD)]、H16[河南新乡(XX)、山西临汾(LF)、山西吕梁(LL)]和H21[SJZ、LL、山东桃村(TC)]为3个种群间的共享单倍型;H15[LF、山西运城(YC)]、H41[XX、陕西西安未央区(WYQ)]为2个种群间的共享单倍型,其余为种群独享单倍型,表现出地理种群之间明显的遗传分化。10个种群总的Hd为0.964(DCQ仅1种单倍型,无种群内多样性体现)。其余9个地理种群中,SJZ、HLD、LL、山西大同(DT)、YC显示出较高的单倍型多样性(Hd > 0.900),但总体核苷酸多样性(0.005 6)较低。
| 种群 | 样本量 | 单倍型数 | 单倍型(个体数) | 单倍型 多样性 |
核苷酸 多样性 |
Tajima's D | Fu's Fs |
| DCQ | 6 | 1 | H1(6) | 0 | 0 | — | — |
| SJZ | 11 | 9 | H1(1)、H21(2)、H29(2)、H30(1)、H31(1)、H32(1)、H33(1)、H34(1)、H35(1) | 0.964 | 0.058 0 | -1.750 | 3.555* |
| XX | 4 | 2 | H16(3)、H41(1) | 0.500 | 0.000 4 | -0.612 | 0.172 |
| HLD | 5 | 4 | H1(1)、H12(1)、H13(1)、H14(2) | 0.900 | 0.008 2 | -0.981 | 1.603 |
| LF | 15 | 6 | H15(3)、H16(7)、H17(2)、H18(1)、H19(1)、H20(1) | 0.762 | 0.011 6 | 0.913 | 6.278 |
| LL | 16 | 9 | H16(1)、H21(3)、H22(3)、H23(1)、H24(1)、H25(1)、H26(2)、H27(1)、H28(3) | 0.917 | 0.037 3 | 0.235 | 8.076 |
| TC | 10 | 5 | H21(3)、H36(1)、H37(1)、H38(4)、H39(1) | 0.800 | 0.027 6 | -1.861* | 8.781* |
| DT | 12 | 10 | H2(1)、H3(1)、H4(2)、H5(1)、H6(1)、H7(1)、H8(1)、H9(1)、H10(1)、H11(2) | 0.970 | 0.008 2 | 0.0742 | 3.673 |
| YC | 5 | 4 | H15(2)、H43(1)、H44(1)、H45(1) | 0.900 | 0.046 1 | 1.298 | 4.641 |
| WYQ | 5 | 3 | H40(1)、H41(2)、H42(2) | 0.800 | 0.001 3 | 0.699 | 0.276 |
| 总计 | 89 | 45 | 0.964 | 0.005 6 | -1.429 | 6.499* | |
| 注:居群间共有单倍型加粗表示;* P < 0.05 Notes:bold indicates shared haplotypes;* P < 0.05 |
|||||||
WYQ、LF与XX之间的遗传距离最小(0.00~0.01),这3个种群与其他种群的遗传距离随着地理位置增大,越往北延伸遗传距离越大。DT和其他9个种群的遗传距离均较大(0.08~0.11),DCQ与HLD种群的遗传距离为0。所有种群的平均遗传距离为0.06。
Fst < 0.150的种群主要有:TC与HLD、LL与SJZ、DCQ与HLD、YC与LL和SJZ,以及SJZ与LL。LF、WYQ和XX这3个种群分别与DCQ和HLD种群的遗传分化系数较大(Fst > 0.900),基因交流水平贫乏,绝大多数种群之间的遗传分化系数为0.150~0.500(表 3)。平均Fst为0.513,平均Nm为0.24。
| 种群 | DCQ | DT | LF | WYQ | XX | YC | TC | HLD | LL | SJZ |
| DCQ | — | 0.08 | 0.07 | 0.07 | 0.07 | 0.04 | 0.02 | 0 | 0.03 | 0.04 |
| DT | 0.482 | — | 0.11 | 0.11 | 0.11 | 0.09 | 0.08 | 0.08 | 0.09 | 0.11 |
| LF | 0.914 | 0.576 | — | 0.01 | 0.01 | 0.05 | 0.08 | 0.07 | 0.06 | 0.08 |
| WYQ | 0.990 | 0.621 | 0.233 | — | 0 | 0.05 | 0.07 | 0.07 | 0.06 | 0.08 |
| XX | 0.997 | 0.626 | 0.234 | 0.160 | — | 0.05 | 0.07 | 0.07 | 0.06 | 0.08 |
| YC | 0.367 | 0.307 | 0.361 | 0.476 | 0.489 | — | 0.05 | 0.04 | 0.04 | 0.06 |
| TC | 0.163 | 0.311 | 0.725 | 0.792 | 0.799 | 0.176 | — | 0.02 | 0.04 | 0.05 |
| HLD | 0.138 | 0.420 | 0.848 | 0.925 | 0.933 | 0.275 | 0.080 | — | 0.03 | 0.04 |
| LL | 0.251 | 0.310 | 0.578 | 0.655 | 0.663 | 0.023 | 0.078 | 0.170 | — | 0.05 |
| SJZ | 0.161 | 0.297 | 0.546 | 0.607 | 0.613 | 0.036 | 0.056 | 0.113 | 0.044 | — |
仅TC种群显示出明显的种群扩张(P < 0.05),XX和HLD种群的Tajima's D虽为负值,但均不显著,未达到种群扩张(P > 0.10)。SJZ种群的Tajima's D值为不显著负值(0.05 < P < 0.10),但Fu's Fs值分化显著(表 2)。
单倍型歧点分析结果见图 2。基于10个种群的所有单倍型的岐点分布曲线未成单一峰型,表明中华蒙潮虫近期未曾经历种群扩张事件,即种群之间存在一定程度的分化。这与种群之间较大的遗传分化系数吻合。
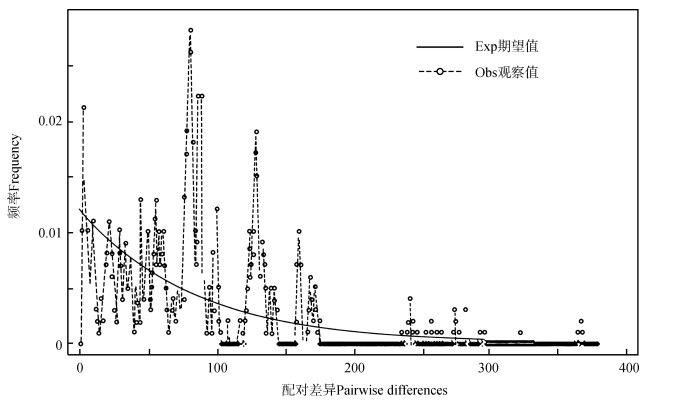
|
| 图 2 所有单倍型总体错配分布 Fig. 2 Mismatch distribution analysis for all haplotypes |
| |
Partitionfinder计算所得CO Ⅰ+ ND5 联合序列的最佳分区方案和各区最适核苷酸替代模型见表 4。
| 联合数据集 | 分区方案 | 模型 |
| CO Ⅰ+ND5 | 分区1:CO Ⅰ第一位点 | K80+G |
| 分区2:CO Ⅰ第二位点 | HKY+I | |
| 分区3:CO Ⅰ第三位点 | HKY+G | |
| 分区4:ND5第一和第二位点 | HKY+G | |
| 分区5:ND5第三位点 | HKY |
中华蒙潮虫不同地理种群的单倍型系统进化关系几乎得到了完全一致的系统发育树,仅节点支持率略有差异,这里仅显示BI树(图 3)。中华蒙潮虫个别地理种群的单倍型并没有完全按照地理来源形成明显的谱系地理结构。单倍型较好的DCQ、HLD、DT、WYQ、XX以及LF种群按地理分布进行划分,其余种群如YC、SJZ、LL以及TC种群则出现了单倍型混杂的分布情况。按照进化的先后以及不同地理种群的单倍型聚合情况大致可以分为5个主要支系;C1为DT;随之进化出的主要类群为TC和SJZ,划分为C2;C3主要为DCQ和HLD;LL虽分布零散,但绝大部分个体位于C3之后,因此划分为C4;最后演化出的是WYQ、LF及XX种群,划分为C5;YC单倍型个体在各个支系均有涉及,主要分布于C4与C5。
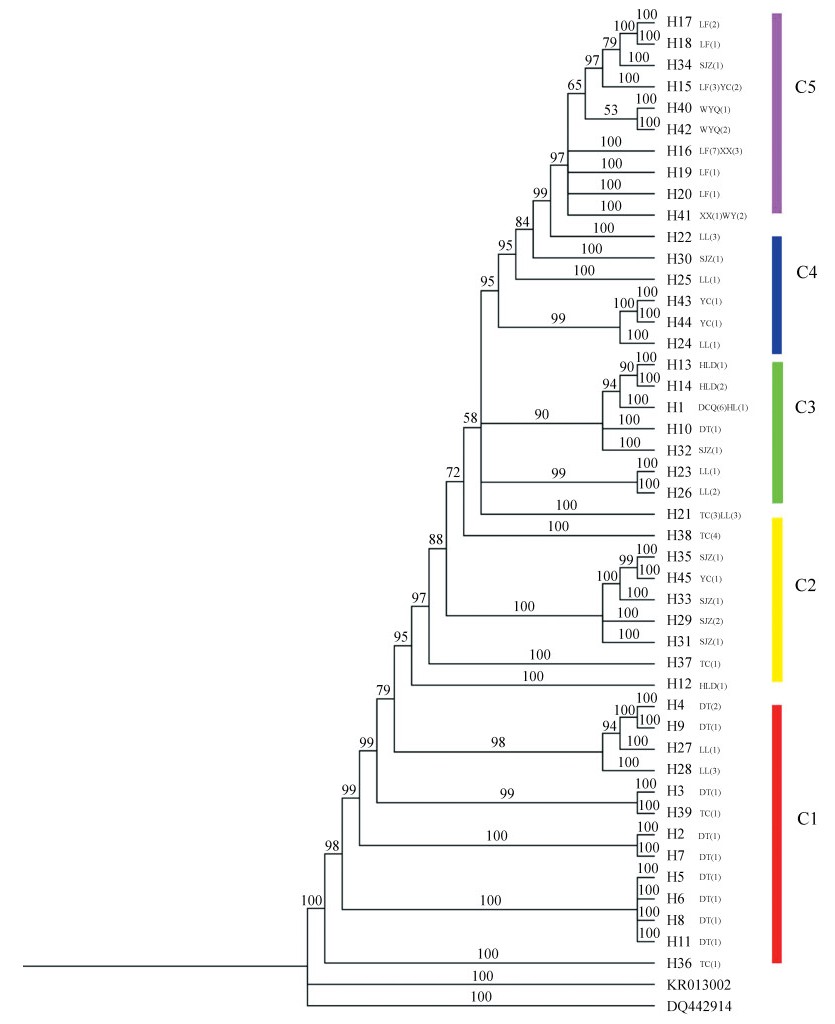
|
| 图 3 基于CO Ⅰ与 ND5 基因组合序列构建的中华蒙潮虫贝叶斯树 Fig. 3 Bayesian tree of Mongoloniscus sinensis based on CO Ⅰ and ND5 genes C1~C5代表不同演化支系 C1-C5 represent different evolutionary branches |
| |
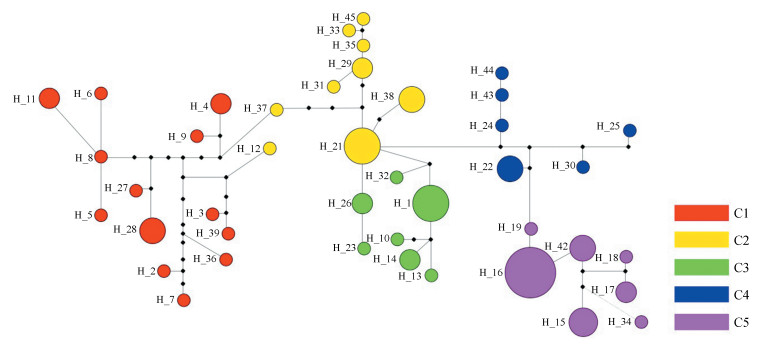
|
| 图 4 中华蒙潮虫(CO Ⅰ+ ND5 )单倍型网络图 Fig. 4 Haplotype network of Mongoloniscus sinensis based on CO Ⅰ and ND5 genes 每个圆代表一个单倍型,圆的大小与单倍型出现的频率一致;黑色小实心圆代表演化过程中可能出现的单倍型 Each circle represents a haplotype, and the size of each circle is consistent with haplotype frequency; small solid circles in black represent possible haplotypes during evolution |
| |
用Arlequin对划分后的5个组进行分子变异分析,中华蒙潮虫的分子变异在各组间以及种群内差异不大,种群内略高(53.12%)(表 5)。
| 变异来源 | df | 平方和 | 变异组分 | 变异 百分比/% |
固定指数 |
| 组间 | 5 | 1 372.343 | 18.201 41 | 48.38 | |
| 组内种群间 | 4 | 63.609 | -0.565 72 | -1.50 | |
| 种群内 | 79 | 1 578.598 | 19.982 26 | 53.12 | 0.469 |
| 总变异 | 88 | 3 014.551 | 37.617 95 |
中华蒙潮虫各个地理种群的单倍型几乎未按地理来源进行划分,这与分子系统树所得结果一致。单倍型H1、H21及H16出现的频率较高且属于2~3个种群的共享单倍型,推测其为祖先类型。LL、SJZ种群较为分散,与系统树类型一致;WYQ、LF及XX种群明显聚成一个簇群。
3 讨论遗传多样性是生态系统多样性、物种多样性和景观多样性的基础,通常遗传多样性最直接的表现形式就是决定进化潜力的遗传变异水平。对于任何一个物种而言,个体生命虽短暂,但种群或种群系统在自然界却具有特定的分布格局,这些分布格局会随着外界条件不断发生改变,因此,遗传多样性不仅包括变异水平的高低,而且包括变异的分布格局(Shen & Liu,2001)。
中华蒙潮虫属于生物圈中分布极广的节肢动物门,虽不如昆虫纲Insecta种类繁多,但也具备极强的繁殖生长能力,种群数量相对较大。该物种的研究报道十分稀少,遗传多样性的研究也非常薄弱。本研究首次联合使用CO Ⅰ及 ND5 基因序列对我国华北地区中华蒙潮虫种群遗传多样性进行了探讨。
3.1 种群遗传多样性衡量一个种群遗传多样性水平可通过Hd及Pi综合体现。本研究中,DCQ种群仅1种单倍型,该地区的遗传分化极为贫乏,结合目前城市化水平的升高导致物种栖息地破坏、生境破碎、基因交流断裂,加之北京西部是太行山余脉的西山,北部是燕山山脉的军都山,两山在南口关沟相交形成的大山弯,山脉的阻隔降低种群交流的程度。SJZ种群的Hd(0.964)和Pi(0.058 0)均显示出较高的遗传多样性;HLD、LL、DT和TC种群均具有较高的Hd(> 0.900),但Pi整体不高。一般认为,如果1个种群有较高的Hd,但核苷酸水平较低,推测可能是种群的建群者效应,即1个较小的有效种群通过内部的变异,积累了大量单倍型多态性,但却未达到积累核苷酸序列多样性的水平,由此种群迅速增长(Wright,1943)。所有种群显示出较高的Hd以及较低的Pi,据此推断华北地区中华蒙潮虫种群遗传多样性水平中等。
3.2 种群遗传结构种群遗传结构的差异是遗传多样性的重要体现,种群遗传变异决定该种群内物种进化潜力以及抵御不良环境的能力(Grant,1991)。单倍型岐点图以及中性检验显示该种群近期未经历扩张,但总体有平稳分化现象。基于AMOVA显示,中华蒙潮虫的变异主要来自种群内部,Fst(0.513)显示各个种群之间遗传分化程度明显,平均Nm(0.24)很低,考虑中华蒙潮虫是陆地爬行类物种,因此,生境限制其无法远距离活动,造成种群之间交流稀缺,这也是中华蒙潮虫种群内部不断分化的原因。
3.3 系统发育分析中华蒙潮虫作为中国古北界特有种,BI树和单倍型网络图显示,不同种群之间的单倍型并未完全按地理来源进行分布。位于系统发育起始端主要为华北偏北地区的种群,如DT、SJZ;而最后演化出来的为WYQ、LF和XX的全部个体,这也在一定程度上与师二燕(2015)的研究结果相吻合,即中华蒙潮虫的主要演化路线为从北向南,同理,与冰川影响下物种的迁移路线基本吻合。另外,TC及YC的单倍型个体较为分散。Hap36(TC)是除外群之外最早分化出的一种单倍型,考虑山东位于黄河下游,东临渤海黄海,且中华蒙潮虫的进化未经历淡水阶段,直接从海洋过渡到陆生(Broly et al., 2013),可以将Hap36看做中华蒙潮虫的原始单倍型;其次Hap39/37/38(TC)与纬度较为接近的SJZ主要支系交叉分布;基于中性检验分析,TC数据显著,近期可能经历了扩张。YC样本量虽偏少,但结合系统发育树以及单倍型分布情况,主要划分为C4与C5支系,其地理位置与演化情况几乎和研究结果相一致,推测数据对路径的演化影响不大。
本研究中,由于个别种群采集数量较少,单倍型分布零散,考虑小的样本量可能造成实验结果有一定偏差,后期将继续加大样本量,完善数据信息。近年来分子标记作为生物学领域一个强有力的工具,在遗传多样性以及生态领域均发挥重要作用,然而,当种群样本数很少或不平均的情况下,利用分子标记对遗传多样性和遗传分化进行解释就需要谨慎。样本数太大会花费很多时间、资源和经费,而样本数太小往往会导致错误的结论(王丽等,2010),因此,样本量是一个非常重要的问题。后期应加大采集量,转向南方未涉及到的区域,加大种群覆盖地区,细化中华蒙潮虫的系统演化路径以及种群分布的动态情况。
4 结语土壤中生活着丰富的生物类群,它们在自然生态系统中扮演着消费者和分解者的角色,是重要的地下生物宝库,对全球物质循环和能量流动起着不可替代的作用。然而,由于人类活动的强烈干扰,生境的不断丧失,土壤生物多样性已日趋减少,这又加剧了土地的退化。由于土壤生物与地上动植物息息相关,只有将对二者的保护结合起来,才能实现完整的全球意义上的生物多样性保护(章家恩,1999)。本研究基于线粒体CO Ⅰ和 ND5 基因的联合序列,对华北地区中华蒙潮虫遗传多样性进行了研究,发现其遗传多样性处于中等水平,主要变异来自于种群内部,个别种群的单倍型没有完全按照地理分布形成簇群;系统发育和单倍型网络图显示,基于华北地区10个种群的中华蒙潮虫的主要演化是从北向南,但存在个别单倍型的混杂分布。本研究基于多基因的联合分析与师二燕(2015)基于单基因的研究结果基本一致,这也与Avise(2009)对系统地理学归纳的第六种模式相吻合,即“即使一个单一的线粒体基因树也能提供重要的生物学见解”。后期,将扩大采集范围(由古北界向东洋界过渡),完善该物种在中国的分布地区,结合形态标记与核基因进行中华蒙潮虫演化路径的研究。
| 白玉. 2007. DNA分子标记技术及其应用[J]. 安徽农业科学, 35(24): 7422–7424. DOI:10.3969/j.issn.0517-6611.2007.24.019 |
| 陈复生, 付承玉, 汪泰初. 2003. 动物线粒体基因分子系统学研究进展[J]. 安徽农业科学, 31(4): 596–598. DOI:10.3969/j.issn.0517-6611.2003.04.039 |
| 陈国孝. 2000. 中国典型地带陆生等足类的区系研究[J]. 动物学报, 46(3): 255–264. |
| 戴文申, 徐银龙, 叶健, 等. 2007. 温度对Chelex 100法提取血斑DNA的影响[J]. 新疆警察学院学报, 27(1): 34–38. DOI:10.3969/j.issn.1672-1195.2007.01.009 |
| 邓春兴. 2014.基于COⅠ基因序列的中国东南沿海四指马鲅属鱼类的遗传多样性分析[D].广州: 暨南大学. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=D526781 |
| 李梦雯. 2016.重金属铅与锌对中华蒙潮虫(等足目: 潮虫亚目)的毒性效应研究[D].临汾: 山西师范大学. http://cdmd.cnki.com.cn/Article/CDMD-10118-1016100353.htm |
| 刘平, 任维宾, 杨瑛, 等. 2018. 角倍蚜细胞色素氧化酶(COⅠ)基因的多样性分析[J]. 生物资源, 40(1): 36–40. |
| 牛晓倩. 2015.等足目三种潮虫(甲壳动物亚门: 等足目: 潮虫亚目)对土壤重金属镉的毒性效应与回避行为研究[D].临汾: 山西师范大学. http://cdmd.cnki.com.cn/Article/CDMD-10118-1015609135.htm |
| 师二燕. 2015.中华蒙潮虫(甲壳动物亚门: 等足目)种群遗传结构和扩张历史研究[D].临汾: 山西师范大学. http://cdmd.cnki.com.cn/Article/CDMD-10118-1015609134.htm |
| 孙红英, 周开亚, 宋大祥. 2003. 节肢动物线粒体基因组与系统发生重建[J]. 动物学研究, 24(6): 467–479. DOI:10.3321/j.issn:0254-5853.2003.06.012 |
| 王兰萍, 耿荣庆, 刘忠权, 等. 2013. 草兔种群的遗传多样性与系统进化研究[J]. 西部林业科学, 42(2): 62–66. DOI:10.3969/j.issn.1672-8246.2013.02.011 |
| 王丽, 文志, 吴敏, 等. 2010.样本数与种群遗传变异[C]//厦门: 全国生物多样性保护与持续利用研讨会. http://www.wanfangdata.com.cn/details/detail.do?_type=conference&id=8492295 |
| 徐庆刚, 花保祯. 2001. 线粒体DNA在昆虫系统学研究中的应用[J]. 西北农林科技大学学报(自然科学版), 29(s1): 79–83. |
| 章家恩. 1999. 土壤生物多样性的研究内容及持续利用展望[J]. 生物多样性, 7(2): 140–144. DOI:10.3321/j.issn:1005-0094.1999.02.010 |
| 智妍, 葛振萍, 张春田. 2008. 基于线粒体ND5基因的昆虫分子系统学研究进展[J]. 沈阳师范大学学报(自然科学版), 26(3): 347–350. DOI:10.3969/j.issn.1673-5862.2008.03.027 |
| Avise JC, Arnold J, Ball RM, et al. 1987. Intraspecific phylogeography:the mitochondrial DNA bridge between population genetics and systematics[J]. Annual Review of Ecology & Systematics, 18(Ⅹ): 489–522. |
| Avise JC. 2009. Phylogeography:retrospect and prospect[J]. Journal of Biogeography, 36(1): 3–15. |
| Boyko CB, Bruce NL, Hadfield KA, et al. 2008. World marine, freshwater and terrestrial isopod crustaceans database. Mongoloniscus sinensis (Dollfus, 1901)[DB/OL]. (2016-09-07)[2018-10-15]. http://marinespecies.org/aphia.php?p=taxdetails&id=261300. |
| Broly P, Deville P, Maillet S. 2013. The origin of terrestrial isopods (Crustacea:Isopoda:Oniscidea)[J]. Evolutionary Ecology, 27(3): 461–476. DOI:10.1007/s10682-012-9625-8 |
| Excoffier L, Lischer HEL. 2010. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 10: 564–567. DOI:10.1111/men.2010.10.issue-3 |
| Folmer O, Black M, Hoeh W, et al. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit Ⅰ from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology, 3(5): 294–299. |
| Fu YX. 1997. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 147(2): 915–925. |
| Grant V. 1991. The evolutionary process. A critical study of evolutionary theory (2nd edition)[M]. New York: Columbia University Press. |
| Ito K, Nishikawa H, Shimada T, et al. 2011. Analysis of genetic variation and phylogeny of the predatory bug, Pilophorus typicus, in Japan using mitochondrial gene sequences[J]. Journal of Insect Science, 11(18): 1–13. DOI:10.1673/031.011.0118 |
| Kimura M. 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 16(2): 111–120. DOI:10.1007/BF01731581 |
| Kearse M, Moir R, Wilson A, et al. 2012. Geneious basic:an integrated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics, 28(12): 1647–1649. DOI:10.1093/bioinformatics/bts199 |
| Kumar S, Stecher G, Tamura K. 2016. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology & Evolution, 33(7): 1870–1874. |
| Librado P, Rozas J. 2009. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 25(11): 1451–1452. DOI:10.1093/bioinformatics/btp187 |
| Li N, Liu Z, Wang Z, et al. 2012. Genetic diversity based on cytochrome b gene analysis of different geographic populations of blue sheep in China[J]. Acta Ecologica Sinica, 32(8): 2381–2389. DOI:10.5846/stxb |
| Lanfear R, Calcott B, Ho SY, et al. 2012. Partitionfinder:combined selection of partitioning schemes and substitution models for phylogenetic analyses[J]. Molecular Biology and Evolution, 29(6): 1695–1701. DOI:10.1093/molbev/mss020 |
| Rychlik W. 2007. OLIGO 7 primer analysis software[J]. Methods in Molecular Biology, 402: 35–59. DOI:10.1007/978-1-59745-528-2 |
| Ronquist F, Teslenko M, Mark PVD, et al. 2012. MrBayes 3.2:efficient bayesian phylogenetic inference and model choice across a large model space[J]. Systematic Biology, 61(3): 539–542. DOI:10.1093/sysbio/sys029 |
| Shen H, Liu D. 2001. Summary of genetic diversity[J]. Journal of Biology, 18(3): 5. |
| Wright S. 1943. Isolation by distance[J]. Genetics, 28(2): 114. |
| Wilson AC, Cann RL, Carr SM, et al. 2010. Mitochondrial DNA and two perspectives on evolutionary genetics[J]. Biological Journal of the Linnean Society, 26(4): 375–400. |
| Zhou ZJ, Zhang YX, Chang YL, et al. 2011. Genetic differentiation among different geographic populations of Gampsocleis sedakovii[J]. Hereditas, 33(1): 75. |
 2019, Vol. 38
2019, Vol. 38