扩展功能
文章信息
- 姜维, 邓捷, 王启军, 赵虎, 孔飞, 张红星
- JIANG Wei, DENG Jie, WANG Qijun, ZHAO Hu, KONG Fei, ZHANG Hongxing
- 大鲵Agouti基因的克隆、表达及多态性分析
- Cloning and Polymorphism of Agouti Gene and Its Expression in Different Skin Color Phenotypes in Andrias davidianus
- 四川动物, 2019, 38(2): 121-129
- Sichuan Journal of Zoology, 2019, 38(2): 121-129
- 10.11984/j.issn.1000-7083.20180255
-
文章历史
- 收稿日期: 2018-08-21
- 接受日期: 2019-01-11
动物体色(毛色)受自然选择、性别选择、遗传漂变及群体间地理阻隔等因素的影响,呈现出丰富多变的表型,成为进化和遗传领域的研究热点。其中,两栖动物体色的多样性和变化,对于个体拟态、逃避捕食、择偶、体温调节等行为尤为重要(Wente & Phillips,2003)。两栖动物体色主要由真皮层中的3类色素细胞决定:载黑素细胞位于最下层,通过指状突起扩展到上一层色素细胞之间;中间一层为载虹素细胞,对光有反射和散射作用,产生蓝-绿色;最上面靠近表皮的一层是载黄素细胞,含蝶啶或类胡萝卜素,呈黄色。当黑色素颗粒移动到黑色素细胞的突起中时,皮肤呈现深暗色,当黑色素颗粒聚拢到细胞核周围时,皮肤颜色变浅(Obika & Bagnara,1964;Frost et al., 1984;Yasutomi & Yamada,1998)。
动物体色形成机制的研究以哺乳动物、鸟类和鱼类为主,揭示了以黑色素细胞为核心的黑色素合成和运输的分子调控系统。已经发现的参与黑色素合成的主要酶和其他分子包括:酪氨酸酶及其相关蛋白、黑皮质素受体-1(MC1R)、刺鼠信号蛋白(Agouti)、小眼畸形相关转录因子等(Hoekstra,2006)。以往主要通过生理学和行为学方面的研究来解释两栖动物独特的体色模式及其形成过程(Bagnara & Hadley,1969;Thibaudeau & Altig,2012;Rudh & Qvarnström,2013;Beukema et al., 2016;Kindermann & Hero,2016),关于两栖动物体色形成的潜在分子遗传机制知之甚少(Beukema et al., 2016)。MC1R基因突变会导致哺乳动物产生毛发白化、黄化和黑化等表型变化(Mundy & Kelly,2003;Garcia-Borron et al., 2005)。Herczeg等(2010)的研究发现MC1R基因的核苷酸突变与欧洲蛙Rana temporaria个体体色的黑化程度不相关。Woodcock等(2017)从美西钝口螈Ambystoma mexicanum的白色个体和白化病个体中分别鉴定出了相关的edn3基因突变和tyr基因142 bp的核苷酸缺失。动物黑色素合成通路十分保守,因此推测参与黑色素合成的其他基因,尤其是Agouti也可能是两栖动物体色形成的重要决定因子。Agouti是G蛋白偶联受体MC1R的天然拮抗剂,以竞争方式结合MC1R,抑制MC1R的活化,引起环腺苷酸水平下降,降低酪氨酸酶和多巴色素异构酶的活性水平,从而使黑色素的合成由合成黑色的真黑素转为合成黄色或红色的褐黑素(Suzuki et al., 1997;Ollmann & Barsh,1999)。
大鲵Andrias davidianus隶属于两栖纲Amphibia有尾目Urodela隐鳃鲵科Cryptobranchidae,是世界上现存体型最大的两栖动物(Duellman & Trueb,1986)。在漫长的进化及人工驯养过程中,大鲵形成了多种不同的体色表型,表现出丰富的多态性,使其成为研究两栖动物体色多态性形成和分子遗传机制的优良材料。本实验克隆了大鲵Agouti基因,采用实时荧光定量PCR(qPCR)方法检测其在大鲵10个组织和器官中的表达分布,并通过分析不同体色大鲵皮肤组织中Agouti基因的序列多态性和表达,探讨Agouti基因与大鲵体色之间的关系,旨在了解大鲵体色形成的分子基础,为两栖动物体色遗传机制研究提供基础资料。
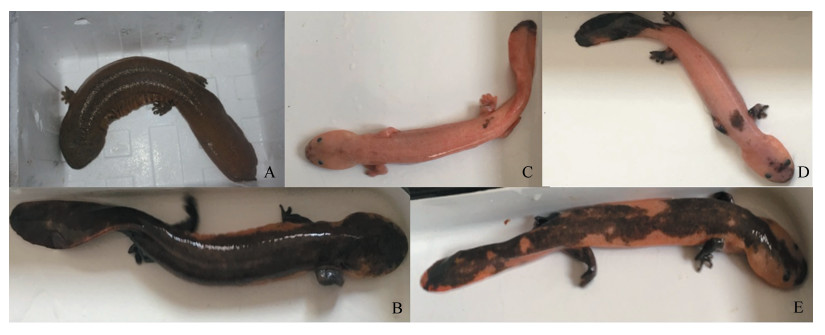
1 材料与方法 1.1 实验材料大鲵[野生动物经营利用许可证:(汉)水野经字(2015)23号]采自汉中古生大鲵有限公司:(1)选取体色为黑色的成年商品大鲵3尾(1雌2雄),处死后立即取其肌肉、皮肤、心脏、肝脏、脾、肺、胃、肠、胰腺、生殖腺(卵巢/精巢)等10个组织和器官样品,-80 ℃保存备用。(2)选取15尾大鲵,根据体色表型分为野生型(图 1:A)、白斑型(图 1:B)和白斑黑化型(图 1:C~E),于2017年10月22日手术法采集尾尖部皮肤组织约0.5 cm2,焦碳酸二乙酯(DEPC)水冲洗干净,-80 ℃保存备用。采样结束后清洗伤口并涂抹碘伏消毒液。依据采集皮肤组织的颜色分为:(1)野生型个体或白斑个体,尾部皮肤组织为深棕色-深灰色(WT;图 1:A、B),共5尾;(2)白斑黑化个体,背部皮肤为黄色,尾部皮肤组织大部分为黄色(aa;图 1:C),共3尾;(2)白斑黑化个体,背部皮肤为黄色,尾部皮肤组织为黑色(am;图 1:D),共3尾;(4)白斑黑化个体,背部及尾部皮肤组织上散布有白斑(ma;图 1:E),共4尾。
|
| 图 1 不同体色的大鲵 Fig. 1 Different body colors of Andrias davidianus A.野生型,B.白斑型,C、D、E.白斑黑化型 A. wild-type phenotype, B. albino phenotype, C-E. melanic-white spotting phenotype |
| |
取0.2 g组织,在组织研磨器中研磨匀浆后,采用Trizol一步法提取总RNA,使用1.5%琼脂糖凝胶电泳检测总RNA的完整性,以Nano Drop2000核酸检测仪测定RNA的浓度和纯度。
1.2.2 反转录用RevertAid cDNA Synthesize Kit(Fermentas,加拿大)反转录,反应体系:Total RNA 2 μg,OligodT(18)1 μL,5×buffer 2 μL,RNase抑制剂1 μL,10 mmol·L-1 dNTP mix 1 μL,反转录酶1 μL,加DEPC水至20 μL。反应程序:42 ℃ 60 min,70 ℃ 2 min。-20 ℃保存备用。
1.2.3 Agouti基因扩增及qPCR引物设计与合成参照GenBank数据库中西部锦龟Chrysemys picta bellii (登录号:XM_005295463.3)和中华鳖Pelodiscus sinensis(登录号:XM_006131706.3)的Agouti基因序列,使用PrimerQuest (http://sg.idtdna.com/Primerquest/Home/IndexPrimer)设计特异性引物用于扩增Agouti基因的cDNA序列,并设计Agouti基因qPCR引物(表 1)。
| 基因 Gene |
引物序列 Primer sequences (5'-3') |
退火温度 Tm/℃ |
产物大小 Size/bp |
| Agoutip1 | F:CGGAAGACGTGTTTCTGACTT R:TTTGACCTGATTGGAAGCTG |
53.00 | 1 068 |
| Agoutip2 | F:GAACTTCTTCCACCCATCTCAA R:GAGCCTTTCTAGGTGGAACTTT |
62.00 | 116 |
| GAPDHp3 | F:CACTGAGGAACAGGTGGTCT R:ATGGCTCATCAGGTCCACAA |
55.97 | 163 |
| 注:p1. Agouti基因克隆引物,p2. Agouti基因实时荧光定量PCR引物,p3. GAPDH基因实时荧光定量PCR引物 Notes:p1. primer pairs for Agouti gene cloning,p2. primer pairs for real-time quantitative PCR of Agouti gene,p3. primer pairs for real-time quantitative PCR of GAPDH gene |
|||
以第一链cDNA为模板,使用大鲵Agouti基因特异性引物,进行PCR扩增。PCR反应体系为25 μL:1.0 μL模板,2.5 μL 10×Taq buffer,2.5 mmol·L-1 MgCl2,2.5 mmol·L-1 dNTP,1 U TaqDNA polymerase (Fermentas,加拿大),上、下游引物各10 μmol·L-1。PCR反应程序:94 ℃ 4 min;35个循环(94 ℃ 30 s,65 ℃ 60 s,72 ℃ 120 s);72 ℃ 10 min。PCR扩增产物经1%琼脂糖凝胶电泳检测,纯化、PEMG-Teasy载体连接、转化后,选择酶切鉴定正确的质粒,送南京金斯瑞生物科技公司测序。
1.2.5 Agouti基因序列分析利用CAP3(http://biosrv.cab.unina.it/webcap3/)进行大鲵Agouti基因序列拼接;vecscreen(https://www.ncbi.nlm.nih.gov/tools/vecscreen/)去除载体序列,Open Reading Frame Finder(https://www.ncbi.nlm.nih.gov/orffinder/)推算开放阅读框和编码氨基酸序列。FeatureMap3D(http://www.cbs.dtu.dk/services/FeatureMap3D/)及SMART(http://smart.embl-heidelberg.de/)用于蛋白结构域的分析。ClustalW将大鲵Agouti氨基酸序列与GenBank中其他物种的Agouti氨基酸序列进行同源比对。
1.2.6 qPCR分析大鲵Agouti基因的组织特异性表达以大鲵肌肉、皮肤、心脏、肝脏、脾、肺、胃、胰腺、生殖腺(卵巢/精巢)、肠组织cDNA为模板,使用大鲵Agouti基因qPCR引物,进行PCR扩增。反应体系为20 μL:cDNA 1 μL,10 nmol·μL-1 dNTP 1 μL,10 μmol·L-1上、下游引物各0.75 μL,FastStart © SYBR Green qPCR Master Mix(Roche,德国)14.5 μL。PCR反应程序为:94 ℃ 1 min;40个循环(94 ℃ 18 s,62 ℃ 18 s,72 ℃ 20 s),85 ℃读板。以GAPDH作为内参基因,并以1个不加模板的反应作为空白对照,每个反应设置3次重复。使用Excel和SPSS 16.0分析数据,以Agouti基因Ct值减相应GAPDH基因Ct值,得出Agouti基因的ΔCt,使用2-ΔΔCt计算Agouti基因的相对表达量。
1.2.7 qPCR分析不同体色大鲵Agouti基因的特异性表达以WT、am、aa、ma 4种体色大鲵皮肤组织cDNA为模板,使用大鲵Agouti基因qPCR引物,进行PCR扩增。反应体系、反应程序和数据处理见1.2.6。
1.2.8 Agouti基因多态性检测分别以15尾不同体色大鲵皮肤组织cDNA为模板,使用Agouti基因特异性引物(表 1)通过PCR扩增Agouti基因,送南京金斯瑞生物科技公司测序。使用ClustalW进行序列比对,分析多态位点。
2 结果与分析 2.1 RNA检测大鲵皮肤组织总RNA电泳图显示28S条带亮度是18S亮度的2倍左右,说明提取的总RNA完整性和浓度较好,可用于下游实验。
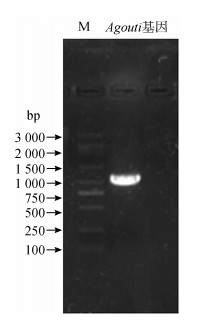
2.2 Agouti基因的全长克隆及序列分析将皮肤组织总RNA反转录为cDNA,用设计的Agouti基因引物进行PCR扩增,电泳结果显示扩增产物约1 000 bp(图 2)。经克隆、测序、拼接后得到大鲵Agouti基因cDNA全长为1 068 bp,其中编码区长399 bp,编码132个氨基酸(图 3),该序列已提交至NCBI(登录号:MH748032)。通过在线软件分析,发现大鲵Agouti基因编码的蛋白N端含1~24个氨基酸残基的信号肽,剪切位点位于第24~25位氨基酸,并在8~128位氨基酸具有保守的Agouti结构域。
|
| 图 2 Agouti基因PCR扩增产物 Fig. 2 Electrophoresis of the PCR product of Agouti gene M.Marker 5000 |
| |

|
| 图 3 大鲵Agouti基因和编码蛋白的序列 Fig. 3 The sequence of Andrias davidianus Agouti gene and the predicted amino acids |
| |
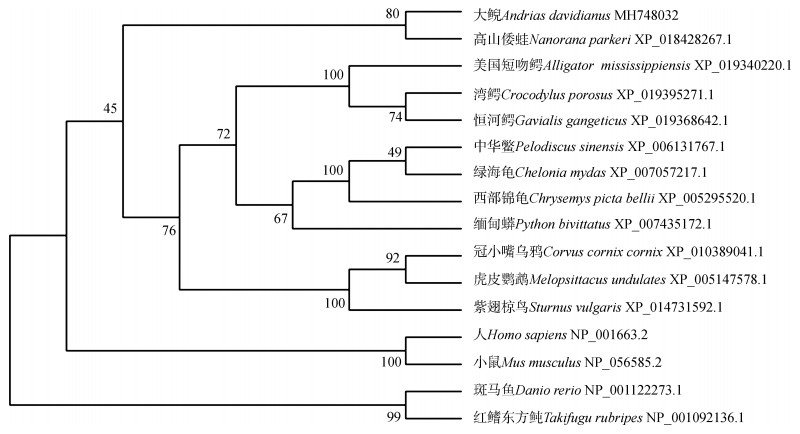
使用BioEdit的ClustalW对大鲵Agouti与其他物种氨基酸序列相似性进行多重比对(图 4),结果显示,大鲵与高山倭蛙Nanorana parkeri相似性最高,为59%,与其他爬行动物的相似性为50%~59%,与其他脊椎动物的相似性为38%~45%。采用邻接法构建蛋白系统发育树,结果显示:大鲵与高山倭蛙的Agouti进化关系最近,聚为一支,之后与美国短吻鳄Alligator mississippiensis、中华鳖等爬行动物及虎皮鹦鹉Melopsittacus undulates等鸟类聚为一个大分支,与哺乳动物和鱼类等其他脊椎动物聚为另一个大分支(图 5),这与ClustalW比对结果一致。

|
| 图 4 大鲵Agouti与其他物种同源分子的氨基酸序列比对 Fig. 4 Comparison of amino acid sequence of Agouti between Andrias davidianus and other species *、.、:分别代表 100%、75%和50%的氨基酸残基相似性 *, ., : represent 100%, 75% and 50% identity of amino acid residue, respectively |
| |
|
| 图 5 邻接法构建的Agouti氨基酸序列系统发育树 Fig. 5 Phylogenetic tree constructed by Neighbor-joining method using amino acid sequences of Agouti |
| |
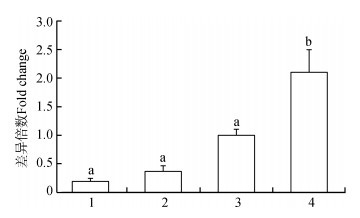
qPCR结果显示,大鲵Agouti基因在10个组织和器官中均有表达(图 6)。在不同体色大鲵的皮肤组织中也均有表达(图 7),其中,在aa型中mRNA的表达量最高,其次为WT型和尾部皮肤为am型的个体,ma型个体皮肤组织中Agouti基因的表达量最低。

|
| 图 6 Agouti基因在大鲵器官、组织中的表达分布 Fig. 6 Expression of Agouti gene in different organs and tissues of Andrias davidianus 1.心脏,2.肝脏,3.脾,4.肺,5.胃,6.肠,7.肌肉,8.性腺,9.胰腺,10.皮肤;字母不同表示差异有统计学意义(P < 0.05) 1. heart, 2. liver, 3. spleen, 4. lung, 5. stomach, 6. intestines, 7. muscle, 8. gonad, 9. pancreas, 10. skin; different letters represent there is a significant difference (P < 0.05) |
| |
|
| 图 7 Agouti基因在不同体色大鲵皮肤组织中的表达 Fig. 7 Expression of Agouti gene in skins of Andrias davidianus with different skin colors 1.黑色皮肤上散布白斑(ma型),2.尾部皮肤为黑色(am型),3.野生型(WT型),4.黄色皮肤组织(aa型);字母不同表示差异有统计学意义 1. black tail with scattered white spots (ma phenotype), 2. black tail (am phenotype), 3. grey/brown tail (wild-type phenotype), 4. yellow tail (aa phenotype); different letters represent there is a significant difference (P < 0.05) |
| |
以15尾不同体色的大鲵皮肤组织RNA为模板,扩增Agouti基因共1 068 bp的核苷酸序列,序列比对结果显示不同体色大鲵个体Agouti基因的核苷酸序列一致。
3 讨论不同物种的Agouti基因及其Agouti蛋白具有较高的同源性,人Homo sapiens、小鼠Mus musculus、牛Bos taurus、马Equus caballus和山羊Capra hircus等的Agouti基因含有4个外显子,其编码的Agouti蛋白由131~133个氨基酸残基组成(Bultman et al., 1992;Kwon et al., 1994)。本实验获得的大鲵的Agouti基因cDNA序列长1 068 bp,开放阅读框长399 bp,编码132个氨基酸,包含1个保守的Agouti功能结构域。氨基酸系统发育分析显示,大鲵和高山倭蛙聚成单独的一个分支,与人、小鼠和鱼类等其他动物分歧较大。
MC1R基因和Agouti基因是黑色素合成过程中最重要的调控基因。哺乳动物毛色(体色)的表型变化有毛色变黄或变白和黑化等类型。研究发现,这2种类型的变异主要是由MC1R基因和Agouti基因功能改变而导致的(Robbins et al., 1993)。当MC1R基因沉默或者Agouti基因过表达时,黑色素细胞内黑色素的合成转为合成褐黑素或受到较强的抑制,动物毛色(体色)呈现出黄色或者白色(Schiaffino,2010)。目前,已经鉴定出小鼠(Miller et al., 1993)、山羊(吴伟伟,2013;张天,2014)、绵羊Ovis aries(Norris & Whan,2008;Fontanesi et al., 2009)、鹌鹑Coturnix coturnix(Nadeau et al., 2008)等哺乳动物或鸟类黄色或者白色的毛色与Agouti基因的大量表达直接相关。实验发现大鲵Agouti基因mRNA在黄色皮肤组织中的表达量较野生型(深棕色-深灰色)有大幅提高,这一结果表明大鲵Agouti基因的高表达可能与载黑素细胞的消失有一定的关系。
在本实验中,约1/3的大鲵个体背部至尾部的皮肤出现黑化,这种体色上的变异在其他两栖动物中亦有报道(Herczeg et al., 2010),而毛色(肤色)黑化的个体在哺乳动物和鸟类中也普遍存在(Takeuchi et al., 1996;Theron et al., 2001;Kambe et al., 2011)。动物毛色(肤色)的深浅与细胞内黑色素的合成量呈正相关。当MC1R基因持续表达或者Agouti基因表达缺失时,细胞内大量合成黑色素,动物毛色(肤色)变深。小鼠MC1R基因核苷酸序列发生280G > A的替代,使MC1R基因持续表达,产生了黑色变异个体(Kambe et al., 2011)。在本研究中,大鲵Agouti基因mRNA在am和ma 2种黑化型个体中的表达量均低于WT的表达水平,说明大鲵体色黑化的现象可能与Agouti基因的表达量降低有关。
本实验对15尾不同体色大鲵Agouti基因编码区序列多态性进行了研究,结果发现大鲵Agouti基因编码区无突变位点。Agouti基因非转录区的多态性,能够影响马、山羊、绵羊、狐狸Vulpes vulpes等哺乳动物毛色的形成(Badaoui et al., 2011;Han et al., 2015;Jakubczak et al., 2016;Sakamoto et al., 2017)。大鲵Agouti基因的非编码区是否存在序列变异还需进一步研究。另外,本实验中大鲵Agouti基因mRNA除在皮肤中大量表达外,在胰腺、胃、肺、肝脏、肠、脾脏、生殖腺(卵巢/精巢)、心脏、肌肉中有较低水平的表达,这与人、鲤鱼Cyprinus carpio、红原鸡Gallus gallus等其他物种Agouti基因mRNA的组织表达情况类似(Dinulescu & Cone,2000;Yoshihara et al., 2012)。
本实验成功克隆了大鲵Agouti基因cDNA序列,进行了相关生物信息学分析,并对其在各组织中的表达和在不同体色个体皮肤组织中的表达进行了检测,为进一步探讨大鲵黑色素的代谢机理和体色形成机制奠定了基础。
| 吴伟伟. 2013.山羊agouti基因5'UTR变异及ASIP蛋白表达定位研究[D].保定: 河北农业大学. http://cdmd.cnki.com.cn/Article/CDMD-11920-1012331751.htm |
| 张天, 张丽英, 李详龙, 等. 2014. 不同毛色山羊皮肤组织Agouti基因mRNA及其编码ASIP差异表达研究[J]. 畜牧兽医学报, 45(12): 1932–1938. |
| Badaoui B, D'Andrea M, Pilla F, et al. 2011. Polymorphism of the goat agouti signaling protein gene and its relationship with coat color in Italian and Spanish breeds[J]. Biochemical Genetics, 49(7-8): 523–532. DOI:10.1007/s10528-011-9427-7 |
| Bagnara JT, Hadley ME. 1969. The control of bright colored pigment cells of fishes and amphibians[J]. American Zoologist, 9(2): 465–478. DOI:10.1093/icb/9.2.465 |
| Beukema W, Nicieza AG, Lourenço A, et al. 2016. Colour polymorphism in Salamandra salamandra (Amphibia:Urodela), revealed by a lack of genetic and environmental differentiation between distinct phenotypes[J]. Journal of Zoological Systematics & Evolutionary Research, 54(2): 127–136. |
| Bultman SJ, Michaud EJ, Woychik RP. 1992. Molecular characterization of the mouse agouti locus[J]. Cell, 71(7): 1195–1204. DOI:10.1016/S0092-8674(05)80067-4 |
| Dinulescu DM, Cone RD. 2000. Agouti and agouti-related protein:analogies and contrasts[J]. The Journal of Biological Chemistry, 275(10): 6695–6698. DOI:10.1074/jbc.275.10.6695 |
| Duellman WE, Trueb L. 1986. Biology of amphibians[M]. New York: McGraw-Hill Book Co. |
| Fontanesi L, Beretti F, Riggio V, et al. 2009. Copy number variation and missense mutations of the agouti signaling protein (ASIP) gene in goat breeds with different coat colors[J]. Cytogenet Genome Research, 126(4): 333–347. DOI:10.1159/000268089 |
| Frost SK, Epp LG, Robinson SJ. 1984. The pigmentary system of developing axolotls. Ⅰ. A biochemical and structural analysis of chromatophores in wild-type axolotls[J]. Journal of Embryology & Experimental Morphology, 81(1): 105. |
| Garcia-Borron JC, Sanchez-Laorden BL, Jimenez-Cervantes C. 2005. Melanocortin-1 receptor structure and functional regulation[J]. Pigment Cell Research, 18(6): 393–410. |
| Han JL, Yang M, Yue YJ, et al. 2015. Analysis of agouti signaling protein (ASIP) gene polymorphisms and association with coat color in Tibetan sheep (Ovis aries)[J]. Genetics Molecular Research, 14(1): 1200–1209. DOI:10.4238/2015.February.6.22 |
| Herczeg G, Matsuba C, Merilä J. 2010. Sequence variation in the melanocortin-1 receptor gene (Mc1r) does not explain variation in the degree of melanism in a widespread amphibian[J]. Annales Zoologici Fennici, 47(1): 37–45. DOI:10.5735/086.047.0104 |
| Hoekstra HE. 2006. Genetics, development and evolution of adaptive pigmentation in vertebrates[J]. Heredity (Edinb), 97(3): 222–234. DOI:10.1038/sj.hdy.6800861 |
| Jakubczak A, Gryzinska M, Horeckaet B, et al. 2016. Single-nucleotide polymorphism of MC1R, ASIP, and TYRP2 genes in wild and farmed foxes (Vulpes vulpes)[J]. Canadian Journal of Animal Science, 96(2): 172–179. DOI:10.1139/cjas-2015-0066 |
| Kambe Y, Tanikawa T, Matsumoto Y, et al. 2011. Origin of agouti-melanistic polymorphism in wild black rats (Rattus rattus) inferred from Mc1r gene sequences[J]. Zoological Science, 28: 560–567. DOI:10.2108/zsj.28.560 |
| Kindermann C, Hero JM. 2016. Pigment cell distribution in a rapid colour changing amphibian (Litoria wilcoxii)[J]. Zoomorphology, 135(2): 1–7. |
| Kwon HY, Bultman SJ, Löffler C, et al. 1994. Molecular structure and chromosomal mapping of the human homolog of the agouti gene[J]. Proceedings of the National Academy of Sciences of the United States of America, 91(21): 9760–9764. DOI:10.1073/pnas.91.21.9760 |
| Miller MW, Duhl DM, Vrieling H, et al. 1993. Cloning of the mouse agouti gene predicts a secreted protein ubiquitously expressed in mice carrying the lethal yellow mutation[J]. Genes & Development, 7(3): 454–467. |
| Mundy NI, Kelly J. 2003. Evolution of a pigmentation gene, the melanocortin-1 receptor, in primates[J]. American Journal of Physical Anthropology, 121(1): 67–80. DOI:10.1002/ajpa.10169 |
| Nadeau NJ, Minvielle F, Ito S, et al. 2008. Characterization of Japanese quail yellow as a genomic deletion upstream of the avian homolog of the mammalian ASIP (agouti) gene[J]. Genetics, 178(2): 777–786. DOI:10.1534/genetics.107.077073 |
| Norris BJ, Whan VA. 2008. A gene duplication affecting expression of the ovine ASIP gene is responsible for white and black sheep[J]. Genome Research, 18(8): 1282–1293. DOI:10.1101/gr.072090.107 |
| Obika M, Bagnara JT. 1964. Pteridines as pigments in amphibians[J]. Science, 143(3605): 485–487. DOI:10.1126/science.143.3605.485 |
| Ollmann MM, Barsh GS. 1999. Down-regulation of melanocortin receptor signaling mediated by the amino terminus of agouti protein in Xenopus melanophore[J]. The Journal of Biological Chemistry, 274(22): 15837–15846. DOI:10.1074/jbc.274.22.15837 |
| Robbins LS, Nadeau JH, Johnson KR, et al. 1993. Pigmentation phenotypes of variant extension locus alleles result from point mutations that alter MSH receptor function[J]. Cell, 72: 827–834. DOI:10.1016/0092-8674(93)90572-8 |
| Rudh A, Qvarnström A. 2013. Adaptive colouration in amphibians[J]. Seminars in Cell & Developmental Biology, 24(6-7): 553–561. |
| Sakamoto T, Fawcett JA, Innan H. 2017. Evaluating the potential roles of the gray and extension loci in the coat coloration of thoroughbred racing horses[J]. Journal of Equine Science, 28(2): 61–65. DOI:10.1294/jes.28.61 |
| Schiaffino MV. 2010. Signaling pathways in melanosome biogenesis and pathology[J]. The International Journal of Biochemistry & Cell Biology, 42(7): 1094–1104. |
| Suzuki I, Tada A, Ollmann MM, et al. 1997. Agouti signaling protein inhibits melanogenesis and the response of human melanocytes to α-melanotropin[J]. Journal of Investigative Dermatology, 108(6): 838–842. DOI:10.1111/1523-1747.ep12292572 |
| Takeuchi S, Suzuki H, Yabuuchi M, et al. 1996. A possible involvement of melanocortin 1-receptor in regulating feather color pigmentation in the chicken[J]. Biochimica et Biophysica Acta, 1308: 164–168. DOI:10.1016/0167-4781(96)00100-5 |
| Theron E, Hawkins K, Bermingham E, et al. 2001. The molecular basis of an avian plumage polymorphism in the wild:a melanocortin-1-receptor point mutation is perfectly associated with the melanic plumage morph of the bananaquit, Coereba flaveola[J]. Current Biology, 11: 550–557. DOI:10.1016/S0960-9822(01)00158-0 |
| Thibaudeau G, Altig R. 2012. Coloration of anuran tadpoles (Amphibia):development, dynamics, function, and hypotheses[J]. Journal of Mass Spectrometry, 48(1): 1–15. |
| Wente WH, Phillips JB. 2003. Fixed green and brown color morphs and a novel color-changing morph of the Pacific tree frog Hyla regilla[J]. American Naturalist, 162(4): 461–473. DOI:10.1086/378253 |
| Woodcock MR, Vaughn-Wolfe J, Elias A, et al. 2017. Identification of mutant genes and introgressed tiger salamander DNA in the laboratory axolotl, Ambystoma mexicanum[J]. Scientific Reports, 7(1): 1–10. |
| Yasutomi M, Yamada S. 1998. Formation of the dermal chromatophore unit (DCU) in the tree frog Hyla arborea[J]. Pigment Cell Research, 11(4): 198–205. DOI:10.1111/pcr.1998.11.issue-4 |
| Yoshihara C, Fukao A, Ando K, et al. 2012. Elaborate color patterns of individual chicken feathers may be formed by the agouti signaling protein[J]. General and Comparative Endocrinology, 175: 495–499. DOI:10.1016/j.ygcen.2011.12.009 |
 2019, Vol. 38
2019, Vol. 38




