扩展功能
文章信息
- 龚雄, 刘金龙, 周天和, 宋琪, 郭宪光
- GONG Xiong, LIU Jinlong, ZHOU Tianhe, SONG Qi, GUO Xianguang
- 新疆博乐和伊犁河谷敏麻蜥的种下分类地位探讨
- Taxonomical Status of Eremias arguta from Bole City and Ili River Valley, Xinjiang Uyghur Autonomous Region, China
- 四川动物, 2018, 37(4): 387-399
- Sichuan Journal of Zoology, 2018, 37(4): 387-399
- 10.11984/j.issn.1000-7083.20180009
-
文章历史
- 收稿日期: 2018-01-07
- 接受日期: 2018-04-26
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
2017年6月,在新疆野外考察中于博乐市连霍高速过四台路段的戈壁生境(81.692 29°E,44.615 63°N,海拔1 324 m)采集到30余号麻蜥属Eremias标本,经鉴定均为敏麻蜥Eremias arguta。然而,其外部形态特征与准噶尔盆地记录分布的敏麻蜥东方亚种E. a. potanini差异较大,而与伊犁河谷敏麻蜥种群的形态特征相似。经查阅相关文献(赵尔宓等,1999;戴鑫等,2006),博乐虽有敏麻蜥的记载,但一直缺乏对其种下分类地位的探讨。长期以来,多数国内学者认为准噶尔盆地的敏麻蜥为东方亚种,伊犁河谷的为指名亚种E. a. arguta(赵尔宓等,1999)。近年来,Poyarkov等(2014)基于线粒体细胞色素b(Cyt b)基因的研究发现,哈萨克斯坦伊犁河谷的敏麻蜥为一独立进化的支系,是一个未描述的新亚种Eremias arguta ssp.。然而,他们的研究并未涵盖中国的标本,中国伊犁河谷敏麻蜥的种下分类地位仍不清楚。因此,本研究拟联合形态学和分子系统学的方法对新疆博乐和伊犁河谷的敏麻蜥种群进行种下分类识别和比较研究,旨在探讨其分类地位,为深入研究亚洲内陆干旱区敏麻蜥的谱系地理结构和演化历史积累有价值的资料。
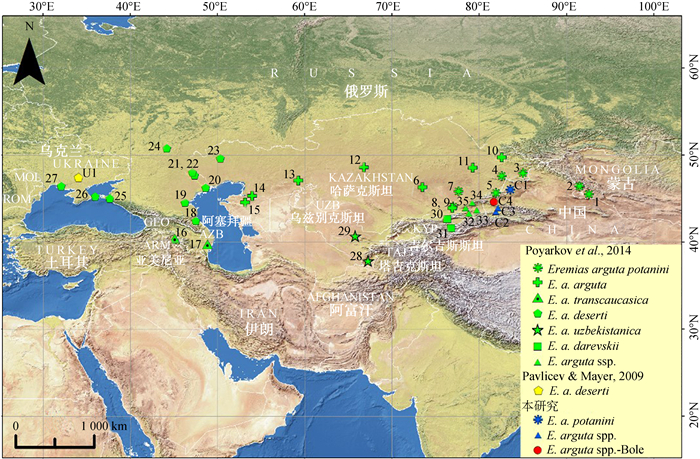
1 材料和方法 1.1 样品来源用于分子系统发育分析的敏麻蜥样品共28号,包括从新疆博乐随机选取的10号样品,以及用于比较的采自新疆托里县、伊宁县和巩留县的各6号样品。标本经75%乙醇固定后,保存于中国科学院成都生物研究所两栖爬行动物标本馆。所有肝脏组织样品均固定于95%乙醇,于-20 ℃保存。从GenBank下载Pavlicev和Mayer(2009)以及Poyarkov等(2014)发表的敏麻蜥Cyt b基因序列共61条,用于分子系统发育分析。样品采样点的分布见图 1,所有样品和序列的信息见表 1。
| 亚种 Subspecies |
样点号 Site ID |
采样地点 Locality |
标本数 n |
标本编号 Specimen ID |
单倍型 Haplotype type |
GenBank登录号 GenBank accession number |
来源 References |
| 东方亚种 E. a. potanini |
1 | Bulgan Somon,Mongolia | 2 | R-12604 | Hap20 | KM977908~09 | § |
| 2 | Dzhungar Gov,Mongolia | 1 | R-12459 | Hap21 | KM977910 | § | |
| 3 | Aigyrkum,Kazakhstan | 2 | R-12417 | Hap22 | KM977911~12 | § | |
| 4 | Oichilik,Kazakhstan | 2 | R-12420 | Hap23 | KM977913~14 | § | |
| 5 | Zhalanashkol,Kazakhstan | 1 | R-12415 | Hap24 | KM977915 | § | |
| 7 | Ainabulak,Kazakhstan | 2 | R-12489 | Hap28~29 | KM977919、 KM977960 |
§ | |
| 8 | Kerbulak,Kazakhstan | 1 | R-12414 | Hap30 | KM977920 | § | |
| C1 | 中国新疆维吾尔自治区托里县 | 6 | Guo2271~76 | Hap1~3 | MG745404~09 | 本研究 | |
| 指名亚种 E. a. arguta |
6 | W coast of Balkhash,Kazakhstan | 3 | R-12488 | Hap25~27 | KM977916~18 | § |
| 9 | Shingeldy,Kazakhstan | 1 | R-12419 | Hap31 | KM977921 | § | |
| 10 | Chernyi Irtysh,Kazakhstan | 1 | R-12416 | Hap32 | KM977970 | § | |
| 11 | Kopa,Kazakhstan | 1 | R-12412 | Hap33 | KM977922 | § | |
| 12 | Dzhezkazgan,Kazakhstan | 2 | — | Hap34 | KM977923~24 | § | |
| 13 | Baikodam,Kazakhstan | 2 | R-12424 | Hap35~36 | KM977925~26 | § | |
| 14 | N Ustyurt,Ozektyk,Kazakhstan | 2 | R-12409 | Hap36 | KM977927~28 | § | |
| 15 | NW Ustyurt,Kazakhstan | 1 | R-11981 | Hap32 | KM977969 | § | |
| 外高加索亚种 E. a. transcaucasica |
16 | Noraduz,Sevan Lake,Armenia | 1 | R-12237 | Hap37 | KM977929 | § |
| 17 | Azerbaijan | 1 | R-12473 | Hap38 | KM977930 | § | |
| 西方亚种 E. a. deserti |
18 | Papas,Russia | 1 | R-12455 | Hap39 | KM977931 | § |
| 19 | Chechen Island,Russia | 2 | R-12072 | Hap40 | KM977932~33 | § | |
| 20 | Akhtuba River,Russia | 2 | — | Hap41 | KM977936~37 | § | |
| 21 | Bogdo Mt.,Russia | 2 | R-12433 | Hap42 | KM977940~41 | § | |
| 22 | Baskunchak Lake,Russia | 2 | — | Hap43 | KM977938~39 | § | |
| 23 | Manat,Kazakhstan | 2 | R-12224 | Hap44~45 | KM977942~43 | § | |
| 24 | Dikovka Village,Russia | 2 | — | Hap44 | KM977934~35 | § | |
| 25 | Taman,Russia | 3 | — | Hap46 | KM977944~46 | § | |
| 26 | Kerch,Ukraine | 3 | — | Hap47 | KM977947~49 | § | |
| 27 | Golaya Pristan,Ukraine | 2 | R-12256 | Hap48 | KM977950~51 | § | |
| U1 | Ukraine | 1 | — | Hap19 | GQ142115 | # | |
| 乌兹别克斯坦亚种 E. a. uzbekistanica |
28 | Kyzylcha,Uzbekistan | 1 | R-12249 | Hap49 | KM977953 | § |
| 29 | Aidarkul Lake,Uzbekistan | 1 | R-11698 | Hap50 | KM977952 | § | |
| 伊塞克湖亚种 E. a. darevskii |
30 | Turu-Aigyr,Kyrgyzstan | 2 | R-12490 | Hap51 | KM977954~55 | § |
| 31 | AK-Olen,Kyrgyzstan | 4 | R-12943 | Hap51 | KM977956~59 | § | |
| 伊犁河谷支系 E. arguta ssp. |
32 | Kokpek,Kazakhstan | 2 | R-12410 | Hap52 | KM977964~65 | § |
| 33 | Kainar,Kazakhstan | 1 | R-12425 | Hap52 | KM977963 | § | |
| 34 | Konyrolen,Kazakhstan | 1 | R-12426 | Hap53 | KM977961 | § | |
| 35 | Konyrolen environs,Kazakhstan | 1 | Kn-1 | Hap54 | KM977962 | § | |
| C2 | 中国新疆维吾尔自治区巩留县 | 6 | Guo4731~34、 Guo4737~38 |
Hap4~8 | MG745410~15 | 本研究 | |
| C3 | 中国新疆维吾尔自治区伊宁县 | 6 | Guo4158~63 | Hap9~13 | MG745416~21 | 本研究 | |
| C4 | 中国新疆维吾尔自治区博乐市 | 10 | Guo6420~29 | Hap14~18 | MG745422~31 | 本研究 | |
| 注Notes:# Pavlicev & Mayer,2009;§ Poyarkov et al.,2014 | |||||||
鉴于本研究侧重于探讨敏麻蜥亚种的分类地位,而不是种群内形态变异,故从博乐采集的标本中仅选取了雌、雄成体各2号作为代表用于形态测量。按照性状选取原则,并参考戴鑫等(2006)对麻蜥属的形态学研究和刘金龙等(2014)对虫纹麻蜥Eremias vermiculata的形态描述,选取了13个常规性状进行测量和计数:头体长(snout-vent length,SVL)、尾长(tail length,TL)、头长(head length,HL)、头宽(head width,HW)、头高(head depth,HD)、前肢长(foreleg length,FLL)、后肢长(hindleg length,HLL)、腹鳞行数(number of ventral scales in a longitudinal row across belly,VS)、最长横向腹鳞数(number of ventral scales in the longest line,NVLL)、股孔数(number of femoral pores,FPRS)、股孔间鳞数(number of scales between the femoral pores,VBFP)、喉鳞数(gulars,G)和尾部鳞数(number of caudal scales,CDA)。数显游标卡尺(精确到0.1 mm)测量标本的可量性状。
1.2.2 基因组DNA提取、PCR扩增及测序取约20 mg肝脏组织,采用核酸纯化试剂盒(EasyPure Genomic DNA kit EE101-01,北京全式金生物技术有限公司),按照说明书操作步骤提取基因组DNA。用超微量紫外检测仪(NanoDrop 2000C,Thermo Scientific)和琼脂糖凝胶电泳检测DNA浓度和纯度。最后,将提取的总DNA置于-20 ℃保存备用。
PCR扩增参考Burbrink等(2000)和Zhao等(2011)使用的Cyt b引物,结合实际扩增效果对引物的个别碱基进行了优化和调整,最终使用改良后的引物:Cyt bF:5’-AACCGCCGTTGTTATTCAAC-3’和Cyt bR-A:5’-TGGTTTACAAAACCAGGGCTTTAGGTT-3’。所有引物均由成都擎科梓熙生物技术有限公司合成。PCR反应Mix采用成都擎科梓熙生物技术有限公司TSINGKE Master Mix (blue)。PCR反应体系为20 μL:2×TSINGKE Master Mix (blue) 10 μL,ddH2O 8 μL,引物各0.5 μL,DNA模板1 μL。PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s,42 ℃退火30 s,72 ℃延伸80 s,35个循环;72 ℃终末延伸10 min。每次PCR扩增均设置阴性对照,以确保实验过程不受外源DNA的影响。取PCR产物4 μL于1%琼脂糖凝胶电泳检测,阳性PCR产物送成都擎科梓熙生物技术有限公司进行双向测序。测序引物为:Cyt bF1-A:5’-GGCTCAAACAACCCAACAGGAC-3’和Cyt bR1-A:5’-TGCGGGGGTAAAA-TTTTC-3’。
1.2.3 数据分析首先使用DNAStar(DNAStar Inc.,Madison,WI,USA)中的SeqmanⅡ查看所测得的DNA序列峰图并进行正反链校对和编辑,手工去除序列两端的引物,获得Cyt b基因全序列1 143 bp。将获得的序列在NCBI中用BLAST(Altschul et al., 1997)进行序列同源性搜索,确保所得的序列是目标基因。在Seaview 4.2.5(Gouy et al., 2010)中进行序列多重比对,并辅以手工校正。在MEGA7(Kumar et al., 2016)中进行序列组成和特征分析,并用脊椎动物线粒体遗传密码子将DNA序列翻译成氨基酸,以确认蛋白编码基因是否具有功能。在序列中没有发现提前终止密码子,提示所获得的序列不是细胞核线粒体假基因拷贝(Song et al., 2008)。整合从GenBank下载的序列,共计89条序列一起分析。使用DnaSP 5.0(Librado & Rozas,2009)识别单倍型,再用所有的单倍型序列以及外群序列重建系统发育树。
根据Poyarkov等(2014)对敏麻蜥系统发育的研究,以快步麻蜥Eremias velox(GenBank登录号:KF999319)和中介麻蜥Eremias intermedia(GenBank登录号:MG745432)为外群,并以草甸蜥蜴Darevskia praticola(GenBank登录号:KM977968)赋根。采用最大简约法(maximum parsiomony,MP)、最大似然法(maximum likelihood,ML)和贝叶斯推断法(Bayesian inference,BI)构建单倍型的系统发育树。在PAUP*4.0b10(Swofford,2003)中进行最大简约分析,采用启发式搜索(heuristic search)、10次随机序列加入重复、树二等分再连接选项(tree-bisection reconnection),通过Bootstrap方法经1 000次叠加重复估计节点支持率(bootstrap support,BP)。使用RAxMLHPC 8.1.20(Stamatakis,2006)进行最大似然分析,对密码子一、二、三位点进行分割,选择GTR+G为核苷酸替代模型,对每个分区单独进行模型参数推断,通过Bootstrap方法经1 000次叠加重复估计节点支持率。采用Partitionfinder 2.1.1(Lanfear et al., 2017)基于贝叶斯信息标准(Bayesian information criterion;Schwarz,1978)选取用于BI分析的最优核苷酸替代模型,在MrBayes 3.2(Ronquist et al., 2012)中重建BI树。密码子一、二、三位点分别采用K80+G、HKY+I、GTR+G模型。起始树为随机树,以3条热链、1条冷链的马尔可夫链蒙特卡罗(Markov Chain Monte Carlo)方法运行2 000万代,每1 000代抽样1次,最初5 000棵树作为老化样本(burn-in)被舍弃,余下的15 001棵树用于估算50%多数一致树,分支节点的可信度以贝叶斯后验概率(posterior probability,PP)表示。
采用贝叶斯因子(Bayes factor,BF;Kass & Raftery,1995)检验相互竞争的系统发育假设。2个不同假设H0和H1的贝叶斯因子等于H0和H1边际似然值(marginal likelihood)的比值。由于边际似然值很难直接计算,可用调和平均数(harmonic-mean)来代替(Newton & Raftery,1994)。采用Suchard等(2001)改进的方法,在Tracer 1.5.0(Rambaut & Drummond,2009)中估算调和平均数。因此,BF可表示为2种不同系统发育假设之间调和平均数的比值。本研究检验了5种关于敏麻蜥伊犁河谷种群的分类地位的相关假设:(1)伊犁河谷支系E.arguta ssp.位于进化树的基础位置,即与其余支系(亚种)形成姐妹群关系;(2)伊犁河谷支系属于指名亚种(赵尔宓等,1999);(3)伊犁河谷支系属于东方亚种(Szczerbak et al., 1993);(4)伊犁河谷支系属于乌兹别克斯坦亚种E.a.uzbekistanica(Chernov,1934);(5)指名亚种和东方亚种聚成一支,但各自均不形成单系(Poyarkov et al., 2014)。将这些假设均设定为“硬性”约束,在MrBayes 3.2中分别进行贝叶斯系统发育分析,然后分别和“非约束”的贝叶斯系统发育树(零假设)进行比较。如果2lnBF≥10,则认为有很强的证据(显著)拒绝备择假说(Kass & Raftery,1995)。
2 结果 2.1 形态学比较 2.1.1 形态特征标本外观见图 2,外形度量见表 2。体型粗壮而稍平扁,尾部短粗,尾长不超过头体长的1.5倍;头短宽,颞部稍膨大;吻圆钝,吻部背面有一纵沟从吻端向后延伸至额鳞中部;吻端有1枚大吻鳞,后接2枚左右上鼻鳞;左右鼻孔各围有鼻鳞3枚;额鼻鳞1枚;前额鳞2枚;额鳞1枚;大形眶上鳞2枚;额顶鳞2枚;顶鳞2枚;顶间鳞1枚,较小;无枕鳞;眶下鳞不楔入上唇鳞之间;颈部与头宽相等;趾外侧无栉缘;尾部第9环或第10环鳞片数大于18枚;尾形短圆,尾部鳞片呈棱状,平滑或轻微向后凸起,有的鳞末端钝尖;指(趾)短,指(趾)长的顺序均为4-3-2-5-1,爪细尖,呈灰白色;大腿两侧的股孔列均未到达膝关节处,其间距至少有1~2枚鳞片;两侧股孔列于肛门前,相距较窄,其间隔7~9枚细鳞;腹鳞行数37~38,腹部一横列鳞16~17枚;背部黑灰色,有8~9纵列醒目的黑缘白斑,呈不规则分布,繁殖季节雄性成体在背部侧面会呈现出明亮的蓝色眼点,雄性亚成体则呈淡黄色偏绿色的眼点;而繁殖季节时的雌性成体背部侧面为白色眼点,亚成体背部侧面有明亮的黄色眼点。

|
| 图 2 新疆博乐敏麻蜥成体,雌性(左)、雄性(右,显示体侧蓝绿色眼点)(龚雄摄) Fig. 2 General view of adult Eremias arguta in Bole city, Xinjiang Uyghur Autonomous Region, China, female (left) and male (right, with bluish-greenish ocelli)(photo by GONG Xiong) |
| |
| 性状 Characteristics |
性别Sex(标本编号Specimen ID) | ||||
| 雄性Male (Guo6424) |
雄性Male (Guo6425) |
雌性Female (Guo6426) |
雌性Female (Guo6429) |
||
| 头体长SVL/mm | 55.93 | 54.08 | 53.95 | 49.09 | |
| 尾长TL/mm | 66.26 | 55.18 | 57.44 | 45.71 | |
| 头长HL/mm | 15.22 | 13.65 | 13.16 | 12.12 | |
| 头宽HW/mm | 9.57 | 9.48 | 8.41 | 7.59 | |
| 头高HD/mm | 7.94 | 6.99 | 6.06 | 5.37 | |
| 前肢长FLL/mm | 19.36 | 19.29 | 18.13 | 17.46 | |
| 后肢长HLL/mm | 30.67 | 30.57 | 27.56 | 26.09 | |
| 腹鳞行数VS/行 | 37 | 38 | 38 | 38 | |
| 最长横向腹鳞数NVLL/枚 | 17 | 16 | 16 | 17 | |
| 股孔数FPRS | 左Left | 12 | 9 | 12 | 10 |
| 右Right | 12 | 10 | 11 | 11 | |
| 股孔间鳞数VBFP/枚 | 9 | 8 | 7 | 8 | |
| 喉鳞数G/枚 | 29 | 30 | 31 | 30 | |
| 尾部鳞数CDA | 第9环9th tail ring | 29 | 27 | 28 | 26 |
| 第10环10th tail ring | 27 | 28 | 27 | 27 | |
敏麻蜥分布区东缘3个亚种或支系的形态特征比较见表 3。东方亚种的体型更为粗壮,成体个体更大,尾部更粗短,背面灰黄色,饰有6~9行纵列斑带,为黑缘白斑横断及由此联结所成的横纹,但是呈更大的拉伸和横断的延长,大腿两侧的股孔列均未到达膝关节处,中间有1~2枚鳞片,2列股孔数多为9~11枚;指名亚种背斑为不规则的黑色横断的斑点和条纹,而伊犁河谷敏麻蜥多为纵列的黑缘白斑(Szczerbak, 1974, 2003;赵尔宓等,1999)。于博乐采集的敏麻蜥,外部形态特征和度量数据与准噶尔盆地分布的东方亚种相比,在体型、背斑和股孔数等方面差别很大;与指名亚种相比,主要为背斑差异和在繁殖季节体侧出现了特殊的眼点,而与伊犁河谷分布的敏麻蜥的描述相符(Dujsebayeva et al., 2007)。
| 亚种或支系 Subspecies or lineages |
性别 Sex |
形态特征Morphological characteristics | ||||
| 头体长 SVL/mm |
股孔数 FPRS |
背部颜色 Color of the back |
背斑特征 Types of the dorsum drawing pattern |
体侧眼点 Ocelli on lateral body |
||
| 东方亚种b E. a. potanini |
♂ | 62~75 | 9~10 | 灰黄色 | 黑缘白斑横断及由此联结所成的横纹,呈更大拉伸和横断的延长 | 无 |
| ♀ | 68.5~72.0 | 9~11 | ||||
| 指名亚种a,c E. a. arguta |
♂/♀ | 34.15~85.10 | 5~12 | 黑黄色 | 黑缘白斑联结形成黑色横断的斑点条纹 | 无 |
| 伊犁河谷支系b E. arguta ssp. |
♂ | 50~59 | 9~13 | 灰黑色 | 8~9纵列醒目的黑缘白斑,呈不规则分布 | 雄性成体有明亮的蓝绿色眼点;亚成体为淡黄色偏绿色眼点 |
| ♀ | 51~67 | 9~11 | 雌性成体有白色眼点;亚成体为明亮黄色眼点 | |||
| 注:a Szczerbak等,1993;b 赵尔宓等,1999;c Szczerbak,2003 Note:a Szczerbak et al., 1993;b Zhao et al., 1999;c Szczerbak,2003 |
||||||
新疆博乐发现的敏麻蜥栖息地为天山以北一片荒漠戈壁(图 3),生境单调,地表土壤主要为沙土砾石,动物种类较少,除同栖分布的沙蜥和偶见的天山黄鼠Citellus relictus和蛇类的洞穴外,几乎无其他脊椎动物。分布的步甲科Carabidae和拟步甲科Tenebrionidae甲虫、蜘蛛、黑蚂蚁和一些蝴蝶的幼虫等为敏麻蜥食物来源。主要植被包括北疆锦鸡儿Caragana camilli-schneideri、芨芨草Achnatherum splendens和一些低矮的草本植物。敏麻蜥主要在低矮的北疆锦鸡儿丛中及其周围活动,其洞穴也大多分布在较密的北疆锦鸡儿丛和一些较大的岩石底部。

|
| 图 3 新疆博乐敏麻蜥采集地生境(龚雄摄) Fig. 3 Biotope of Eremias arguta in Bole city, Xinjiang Uyghur Autonomous Region, China (photo by GONG Xiong) |
| |
PCR扩增获得敏麻蜥Cyt b序列(1 143 bp)28条,识别出18个单倍型;从GenBank下载Pavlicev和Mayer(2009)发表的同源序列1条(1 143 bp)和Poyarkov等(2014)发表的同源序列60条(778 bp)。联合3条外群序列比对生成的数据矩阵,识别出57个单倍型,1 143个同源位点中有变异位点446个,简约信息位点296个,单一多态位点150个,转换与颠换之比平均为3.15。
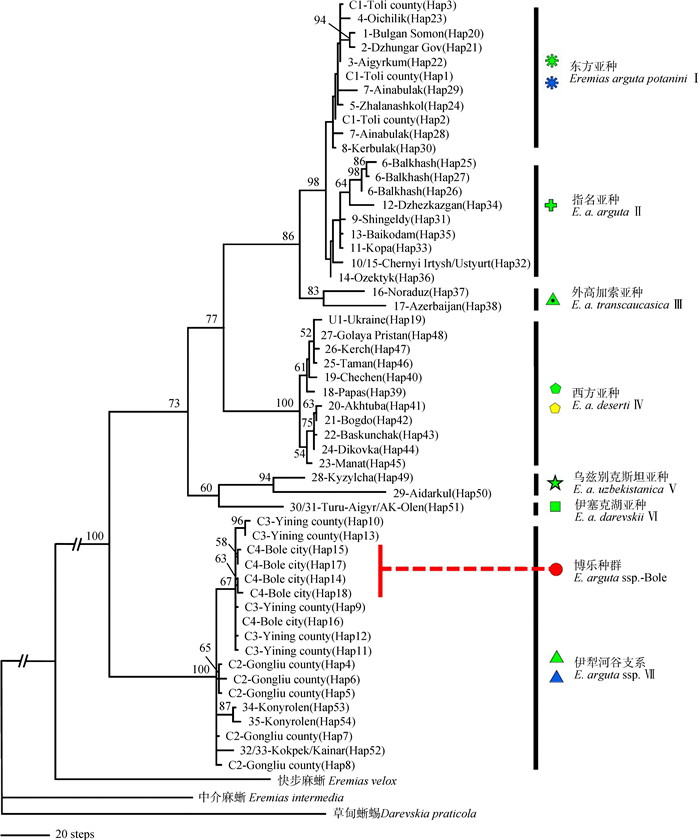
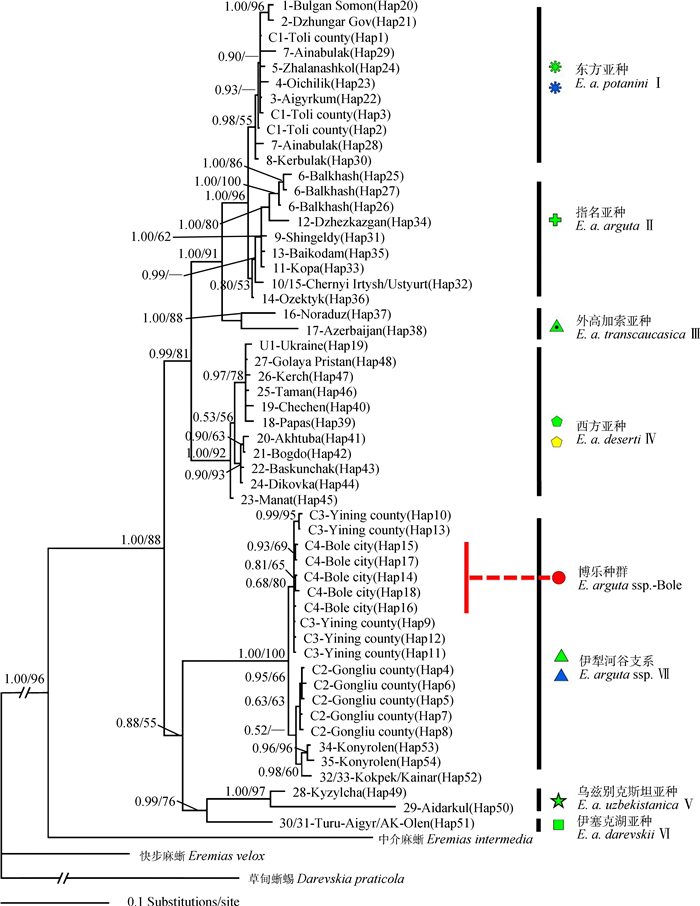
采用3种方法所得到的系统发育树的拓扑结构基本一致,特别是ML树和BI树的拓扑结构几乎完全一致(图 4、图 5)。3种方法所得到的进化树均支持敏麻蜥形成一个单系(MP/ML/BI:100%/88%/1.00),并识别出7个明显的分支(Clade Ⅰ~Ⅶ),分别与基于形态划分的亚种相对应:敏麻蜥东方亚种、指名亚种、外高加索亚种E. a. transcaucasica、西方亚种E. a. deserti、乌兹别克斯坦亚种、伊塞克湖亚种E. a. darevskii和伊犁河谷支系。其中,采自博乐的敏麻蜥E. arguta ssp. -Bole的单倍型序列均聚在伊犁河谷支系所在的CladeⅦ(MP/ML/BI:100%/100%/1.00);且与准噶尔盆地分布的东方亚种和哈萨克斯坦中部分布的指名亚种没有构成姐妹群,二者亲缘关系较远。
|
| 图 4 基于线粒体Cyt b基因序列采用PAUP*4.0b10重建的最大简约严格一致树 Fig. 4 Maximum parsiomony strict consensus tree based on Cyt b gene sequences using PAUP*4.0b10 节点上的数值为1 000次重复抽样分析所得到的自展支持率(仅显示>50%);树长=922,一致性指数=0.637,留存指数=0.856,尺度化一致性指数=0.545 Numbers above the nodes represent bootstrap support with 1 000 pseudoreplicates (only shown BP>50%); tree length=922, consistercy index=0.637, retention index=0.856, rescaled consistercy=0.545 |
| |
|
| 图 5 基于线粒体Cyt b基因序列采用MrBayes 3.2进行分区贝叶斯分析所得的50%多数一致树 Fig. 5 Cyt b 50% majority-rule consensus tree inferred from partitioned Bayesian inference by using MrBayes 3.2 节点上的数值代表贝叶斯后验概率/最大似然自展支持率(仅显示PP>0.50或BP>50%),—表示节点的自展支持率小于50%或此节点不存在 Bayesian posterior probabilities (>0.50) and maximum likelihood bootstrap values (>50%) are shown, respectively; — represents node with bootstrap value lower than 50% or node not existed |
| |
MP分析得到了1 001棵同等简约树,树长(tree length)=922,一致性指数(consisteney index,CI)=0.637,留存指数(retention index,RI)=0.856,尺度化一致性指数(rescaled consistency,RC)=0.545。其中,Clade Ⅰ(东方亚种)与Clade Ⅱ(指名亚种)聚成一支(BP=98%),但二者各自形成单系的支持率均很低(BP < 50%);Clade Ⅶ(伊犁河谷支系)位于树的基础位置,与其余支系形成姐妹群关系,但支持率不高(BP=73%)。BI树和ML树中,Clade Ⅰ与Clade Ⅱ聚成一支(PP/BP=1.00/96%),二者形成交互单系(Clade Ⅰ:PP/BP=0.98/53%,Clade Ⅱ:PP/BP=0.80/53%);Clade Ⅴ与Clade Ⅵ聚成一支(PP/BP=0.99/76%),再和Clade Ⅶ形成姐妹群关系,尽管支持率不高(PP/BP=0.88/55%)。
2.4 贝叶斯因子检验采用贝叶斯因子检验几种相互竞争的拓扑结构的结果见表 4。伊犁河谷支系属于指名亚种或东方亚种或乌兹别克斯坦亚种的3种备择假设均被本研究的分子数据极强地拒绝;而伊犁河谷支系位于系统发育树的基础位置,即与其余支系形成姐妹群关系的假设没有被显著拒绝。本研究的MP树(图 4)与BI树(图 5)间的差异并不显著(2lnBF=5.94 < 10)。指名亚种和东方亚种不形成交互单系的假设(Poyarkva et al., 2014),与本研究BI树差异不显著(2lnBF=8.184 < 10),没有被很强的证据拒绝。
| 系统发育关系假设 Alternative phylogenetic hypotheses |
约束结构描述 Description of constraint |
边际似然值 Marginal likelihood |
贝叶斯因子 Bayes factor |
|
| H0 | 贝叶斯树(图 5) | -5 473.693 | ||
| H1 | 伊犁河谷支系与其余支系形成姐妹群(图 4) | Clade (Ⅰ,Ⅱ,Ⅲ,Ⅳ,Ⅴ,Ⅵ) | -5 476.663 | 5.940 |
| H2 | 伊犁河谷支系属于指名亚种(赵尔宓等,1999) | Clade (Ⅱ+Ⅶ) | -5 513.279 | 79.172 |
| H3 | 伊犁河谷支系属于东方亚种(Szczerbak et al., 1993) | Clade (Ⅰ+Ⅶ) | -5 512.500 | 77.614 |
| H4 | 伊犁河谷支系属于乌兹别克斯坦亚种(Chernov,1934) | Clade (Ⅴ+Ⅶ) | -5 478.741 | 10.096 |
| H5 | 指名亚种和东方亚种聚成一支,但各自均不形成单系 (Poyarkov et al., 2014) |
Clade (Ⅰ+Ⅱ) | -5 477.785 | 8.184 |
敏麻蜥为麻蜥属中分布范围最广的物种之一,其分布区横贯欧亚大陆,包括欧洲黑海地区的罗马尼亚到俄罗斯的北高加索,外高加索(格鲁吉亚,亚美尼亚,阿塞拜疆)的东部,中亚五国以及向东一直到中国新疆西北部(伊犁河谷、准噶尔盆地)和蒙古国的西部戈壁地区等(Szczerbak, 1974, 2003;赵尔宓等,1999;Sindaco et al., 2008)。敏麻蜥为典型的耐干旱物种,因其分布广泛,生境异质性高,地理种群变异很大,导致种下分类复杂。传统的形态学研究将敏麻蜥分为敏麻蜥指名亚种、西方亚种、外高加索亚种、乌兹别克斯坦亚种、伊塞克湖亚种和东方亚种6个亚种(Szczerbak, 1974, 2003;Sindaco et al., 2008;郭宪光等,2010)。
3.1 博乐种群的分类地位根据《中国动物志爬行纲(第二卷)有鳞目蜥蜴亚目》(赵尔宓等,1999)记载,新疆博乐有敏麻蜥分布,但志书并未对博乐种群进行分类讨论,其种下分类地位不明确。戴鑫等(2006)对中国8种麻蜥开展了多变量形态比较,其中敏麻蜥包括博乐的2号标本,未提及其亚种地位。本研究重新检视了这2号标本:采集号625134(馆藏号CIB3531)、625135(馆藏号CIB3532),标签上记录分别于1958年8月15日和29日采自博乐城郊草坝。这2号标本经福尔马林固定,由于年代相对较久远,一些关键特征不易辨识,难于鉴定到亚种,馆藏记录仅鉴定至种级水平,推测编写志书时,没有进一步讨论其种下分类地位。志书在相关部分将中国的敏麻蜥归为指名亚种(分布于尼勒克、新源、伊宁等地)和东方亚种(塔城-额敏地区自西向东直到富蕴-青河一带)。
然而,此次在博乐采集的敏麻蜥标本,其形态特征与准噶尔盆地分布的东方亚种相比,在体型、背斑和股孔数等方面差别很大;与指名亚种相比,主要为背斑差异和在繁殖季节出现了特殊的眼点,而与伊犁河谷地区的敏麻蜥描述(Dujsebayeva et al., 2007)相符。在生境方面,此次博乐敏麻蜥的采集地生境与赵尔宓等(1999)描述的伊犁河谷敏麻蜥生境和Dujsebayeva等(2007)在哈萨克斯坦东南部伊犁河谷地区发现的敏麻蜥生境相似。而敏麻蜥东方亚种的栖息地主要为干枯河床的砾石堆下(赵尔宓等,1999),与此次博乐敏麻蜥的采集地生境有较大区别。基于Cyt b基因的分子系统发育分析表明,MP、ML和BI树均一致地支持其与伊犁河谷样本聚成一支。因此,此次于新疆博乐发现的敏麻蜥应划分为伊犁河谷支系,隶属于敏麻蜥中未描述的一个新亚种(Poyarkov et al., 2014)。这一发现暗示,阿拉套山、科尔古琴山和博罗科努山可能并未对伊犁河谷和博乐的敏麻蜥种群的基因流构成明显的地理阻隔。这是敏麻蜥伊犁河谷支系在准噶尔盆地有分布的新纪录,推测准噶尔盆地还有更多的分布点。此外,有趣的是,博乐的敏麻蜥种群似乎与东方亚种的分布区相连,伊犁河谷支系与东方亚种是否有同域分布的种群,有待进一步考察研究。
3.2 伊犁河谷支系的系统发育位置长期以来,伊犁河谷敏麻蜥种群的分类地位主要有4种观点:Chernov(1934)认为属于乌兹别克斯坦亚种;Yakovleva(1964)认为属于乌兹别克斯坦亚种和指名亚种中间类型;Sczcerbak等(1993)将其修订为东方亚种;赵尔宓等(1999)将其划为指名亚种。Dujsebayeva等(2007)发现,伊犁河谷敏麻蜥雄性成体在繁殖季节体侧有醒目的蓝色眼点,而敏麻蜥其他亚种均无这一特征,这暗示了伊犁河谷的敏麻蜥种群可能处于一个独特的分类地位。Orlova等(2012)通过多变量判别分析,发现伊犁河谷的敏麻蜥种群聚成单独的一簇,可与其他几个亚种分开。Guo等(2011)基于16S rRNA基因片段探讨了麻蜥属部分物种的分子系统发育和分歧时间,但并未涉及敏麻蜥种内关系的探讨。Poyarkov等(2014)基于Cyt b基因片段,首次从分子上发现伊犁河谷种群不是指名亚种、乌兹别克斯坦亚种或东方亚种间的过渡形式,而是形成一个独立的支系,与其余支系形成姐妹群关系,尽管支持率不高(MP/ML/BI:71%/65%/0.93),可能是一个未描述的新亚种。遗憾的是,Poyarkov等(2014)的研究未包含中国分布敏麻蜥的代表样本。
本研究在Poyarkov等(2014)的基础上,增加中国新疆博乐、伊宁和巩留的敏麻蜥种群以及塔城盆地(托里县)东方亚种种群的代表样品。重建的MP树重现了Poyarkov等(2014)关于伊犁河谷支系的系统发育位置,即位于树的基础位置,与其余支系形成姐妹群关系(BP=73%);而ML和BI树中,伊犁河谷支系与乌兹别克斯坦亚种和伊塞克湖亚种形成的分支构成姐妹群关系(ML/BI:0.88/55%)。可见,伊犁河谷支系的系统发育位置尚未解决,下一步研究有必要增加样品和利用多基因位点来检验这2种相互竞争的假设,并在整合分类学(Padial et al., 2010)研究的基础上,来描述这一新分类亚种单元。
3.3 东方亚种的有效性Orlova等(2012)发现敏麻蜥整个分布范围的东部,背部从斑点状的“arguta”-type到带状的“potanini”-type表现出由西至东的渐变。Poyarkov等(2014)发现,敏麻蜥指名亚种和东方亚种聚成一支,但二者并未形成交互单系,东方亚种散布在指名亚种形成的支系中。但本研究的MP、ML和BI树均支持这2个亚种形成交互单系,尽管MP树的自展支持率低于50%。通过BF检验(2lnBF=8.184 < 10),发现Poyarkov等(2014)的假设不能被当前的Cyt b基因数据很强地拒绝。当然,如果放宽标准,按传统的6 < 2lnBF < 10来理解(Kass & Raferty,1995),还是有强的证据拒绝“指名亚种和东方亚种不形成交互单系”的假设。也就是说,本研究中,东方亚种的有效性得到了分子数据一定程度上的支持。因此,综合形态数据的结果,东方亚种是否有效尚不确定,有必要增加更多的数据,引入整合分类学的方法(Padial et al., 2010;Torstrom et al., 2014)。鉴于对生物多样性的认识和保护目的,建议当前保留此亚种,不宜将其视为指名亚种的次定同物异名。
致谢: 四川大学华西基础医学与法医学院陈达丽协助新疆伊犁河谷的标本采集,何金蕾和陈晗在标本整理过程中提供了帮助,审稿专家对稿件提出了宝贵意见,在此一并致谢!| 戴鑫, 陈彬, 张正卫, 等. 2006. 中国八种麻蜥(蜥蜴科, 麻蜥属)形态学研究[J]. 动物分类学报, 31(4): 697–708. |
| 郭宪光, 陈达丽, 万宏富, 等. 2010. 麻蜥属Eremias的系统学研究进展[J]. 四川动物, 29(4): 665–672. |
| 刘金龙, 原秀云, 周天和, 等. 2014. 新疆天山以北发现爬行动物虫纹麻蜥[J]. 动物学杂志, 49(3): 423–427. |
| 赵尔宓, 赵肯堂, 周开亚, 等. 1999. 中国动物志爬行纲(第二卷)有鳞目蜥蜴亚目[M]. 北京: 科学出版社: 226-228. |
| Altschul SF, Madden TL, Schaffer AA, et al. 1997. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J]. Nucleic Acids Research, 25(17): 3389–3402. DOI:10.1093/nar/25.17.3389 |
| Burbrink FT, Lawson R, Slowinski JB. 2000. Mitochondrial DNA phylogeography of the polytypic north American rat snake (Elaphe obsoleta):a critique of the subspecies concept[J]. Evolution, 54(6): 2107–2118. DOI:10.1111/evo.2000.54.issue-6 |
| Chernov SA. 1934. On the subspecies and distribution of the steppe racerunner[J]. Proceedings of the USSR Academy of Sciences, Biological Series, 3(8-9): 666–668. |
| Dujsebayeva ТN, Belyalov OV, Orlova VF, et al. 2007. Unusual find of the steppe-runner, Eremias arguta (Pallas, 1773) with blue spots in southeast of Kazakhstan[J]. Terra, 2007: 118–121. |
| Gouy M, Guindon S, Gascuel O. 2010. SeaView version 4:a multiplatform graphical user interface for sequence alignment and phylogenetic tree building[J]. Molecular Biology & Evolution, 27(2): 221–224. |
| Guo X, Dai X, Chen D, et al. 2011. Phylogeny and divergence times of some racerunner lizards (Lacertidae:Eremias) inferred from mitochondrial 16S rRNA gene segments[J]. Molecular Phylogenetics & Evolution, 61(2): 400–412. |
| Kass RE, Raferty AE. 1995. Bayes factors[J]. Journal of the American Statistical Association, 90(430): 773–795. DOI:10.1080/01621459.1995.10476572 |
| Kumar S, Stecher G, Tamura K. 2016. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology & Evolution, 33(7): 1870–1874. |
| Lanfear R, Frandsen PB, Wright AM, et al. 2017. PartitionFinder 2:new methods for selecting partitioned models of evolution for molecular and morphological phylogenetic analyses[J]. Molecular Biology & Evolution, 34(3): 772–773. |
| Librado P, Rozas J. 2009. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 25(11): 1451–1452. DOI:10.1093/bioinformatics/btp187 |
| Newton MA, Raftery AE. 1994. Approximate Bayesian inference with the weighted likelihood bootstrap[J]. Journal of the Royal Statistical Society, Series B:Methodological, 56(1): 3–48. |
| Orlova VF, Chirikova MA, Pavlinov NJ. 2012. Steppe racerunner, Eremias arguta (Pallas, 1773) (Sauria, Lacertidae) in the eastern part of its range:morphological variability and taxonomic status of populations[J]. Zoological Journal, 91(11): 1366–1376. |
| Padial JM, Miralles A, De la Riva I, et al. 2010. The integrative future of taxonomy[J]. Frontiers in Zoology, 7(1): 1–14. DOI:10.1186/1742-9994-7-1 |
| Pavlicev M, Mayer W. 2009. Fast radiation of the subfamily Lacertinae (Reptilia:Lacertidae):history or methodical artefact?[J]. Molecular Phylogenetics & Evolution, 52(3): 727–734. |
| Poyarkov NJ, Orlova VF, Chirikova MA. 2014. The mitochondrial phylogeography and intraspecific taxonomy of the steppe racerunner, Eremias arguta (Pallas) (Lacertidae:Sauria, Reptilia), reflects biogeographic patterns in middle Asia[J]. Zootaxa, 3895(2): 208–223. DOI:10.11646/zootaxa.3895.2 |
| Rambaut A, Drummond AJ. 2009. Tracer MCMC trace analysis tool, version v1. 5. 0 2003-2009[M]. Computer program distributed by the authors. [2017-12-10]. http://tree.bio.ed.ac.uk/software/tracer/. |
| Ronquist F, Teslenko M, van Der Mark P, et al. 2012. MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space[J]. Systematic Biology, 61(3): 539–542. DOI:10.1093/sysbio/sys029 |
| Schwarz G. 1978. Estimating the dimension of a model[J]. Annals of Statistics, 6(2): 461–464. DOI:10.1214/aos/1176344136 |
| Sindaco R, Jeremenko VK, Venchi A. 2008. The reptiles of the western Palearctic:annotated checklist and distributional Atlas of the turtles, crocodiles, amphisbaenians and lizards of Europe, North Africa, Middle East and Central Asia[M]. Latina: Edizioni Belvedere: 228-362. |
| Song H, Buhay JE, Whiting MF, et al. 2008. Many species in one:DNA barcoding overestimates the number of species when nuclear mitochondrial pseudogenes are coamplified[J]. Proceedings of the National Academy of Sciences of the United States of America, 105(36): 13486–13491. DOI:10.1073/pnas.0803076105 |
| Stamatakis A. 2006. RAxML-VI-HPC:maximum likelihood-based phylogenetic analyses with thousands of taxa and mixed models[J]. Bioinformatics, 22(21): 2688–2690. DOI:10.1093/bioinformatics/btl446 |
| Suchard MA, Weiss RE, Sinsheimer JS. 2001. Bayesian selection of continuous-time Markov chain evolutionary models[J]. Molecular Biology & Evolution, 18(6): 1001–1013. |
| Swofford DL. 2003. PAUP*:phylogenetic analysis using parsimony (*and other methods), version 4.0b10[M]. Sunderland, Massachusetts: Sinauer Associates. |
| Szczerbak NN, Neruchev VV, Okulova NM, et al. 1993. Taxonomy, geographic variation and interspecific structure[M]//Szczerbak NN. Eremias arguta. Kiev: Naukova Dumka: 22-34 (in Russian). |
| Szczerbak NN. 1974. Yashchurki Palearktiki (Eremias lizards of the Palearctic)[M]. Kiev: Naukova Dumka: 146-180 (in Russian). |
| Szczerbak NN. 2003. Guide to the reptiles of the eastern Palearctic[M]. Malabar, Florida: Krieger Publishing Company: 79-80. |
| Torstrom SM, Pangle KL, Swanson BJ. 2014. Shedding subspecies:the influence of genetics on reptile subspecies taxonomy[J]. Molecular Phylogenetics & Evolution, 76: 134–143. |
| Yakovleva ID. 1964. Reptiles of Kirgizia[M]. Frunze: Academy of Sciences of the Kirghiz SSR Press: 138-167 (in Russian). |
| Zhao Q, Liu H, Luo L, et al. 2011. Comparative population genetics and phylogeography of two lacertid lizards (Eremias argus and E. brenchleyi) from China[J]. Molecular Phylogenetics & Evolution, 58(3): 478–491. |
 2018, Vol. 37
2018, Vol. 37