扩展功能
文章信息
- 陈熙, 王斌, 罗旭
- CHEN Xi, WANG Bin, LUO Xu
- 白腹鹭COⅠ和Cyt b基因序列分析及鹭属系统发育研究
- Gene Sequences of COⅠ and Cyt b of Ardea insignis and Molecular Phylogenetic Analysis of Genus Ardea
- 四川动物, 2018, 37(3): 268-273
- Sichuan Journal of Zoology, 2018, 37(3): 268-273
- 10.11984/j.issn.1000-7083.20170353
-
文章历史
- 收稿日期: 2017-11-10
- 接受日期: 2018-03-13
2. 云南高黎贡山国家级自然保护区泸水管护分局, 云南泸水 673100
2. Lushui Management Bureau of Gaoligongshan National Nature Reserve, Lushui, Yunnan Province 673100, China
白腹鹭Ardea insignis隶属于鹭科Ardeidae鹭属Ardea(Gill & Donsker, 2014), 是一种体型较大的涉禽, 高达127 cm, 整体呈灰色, 分布于尼泊尔、不丹、印度东北部和缅甸北部, 我国云南西部(Hoyo et al., 1992;韩联宪等, 2015)。自1994年, 白腹鹭被世界自然保护联盟(IUCN)列为濒危(EN)物种, 后升级为极危(CR)物种, 全球数量不足500只(IUCN, 2016)。在《中国脊椎动物红色名录》中, 白腹鹭的现状是缺乏数据(DD)(蒋志刚等, 2016)。对于该物种, 不论是野外分布、种群现状, 还是系统分类、分子生物学研究, 均近于空白。
关于鹭类的系统分类研究历来不多, 或者分类取样十分有限(张保卫等, 2002;常青等, 2003;张国萍等, 2005)。鹭属内的物种归属也一直存在争议。比如, 大白鹭A. alba和中白鹭A. intermedia长期以来被置于白鹭属Egretta(郑作新, 1964, 1994;Hermann et al., 1972;Ben et al., 1975), 但Sibley和Monoroe(1990)根据DNA杂交研究结果, 将大白鹭归入大白鹭属Casmerodius, 中白鹭则独立成中白鹭属Mesophoyx。但在国际鸟类联合会(International Ornithologists Committee, IOC)的鸟类名录和目前国内普遍采用的鸟类分类系统中, 大白鹭和中白鹭都置于鹭属(郑光美, 2017)。对于白腹鹭, 由于缺乏研究, 中国鸟类分类系统一直沿用Peters的分类方式, 将其置于鹭属(Peters, 1945;郑作新, 1964;郑光美, 2017), 但尚无分子系统学的验证。
本研究根据IOC的分类系统, 对鹭属鸟类进行系统发育分析。分子标记采用线粒体DNA中的细胞色素b(cytochrome b, Cyt b)基因和细胞色素C氧化酶亚基Ⅰ(cytochrome oxidase subunit Ⅰ, COⅠ)基因, 它们都被广泛运用于鸟类分类与系统发育关系的研究(Mindell, 1997;Weibel & Moore, 2002;Hebert et al., 2004;Webb & Moore, 2005)。选择鹭属内7种作为内群, 选择鹭科中与鹭属亲缘关系近且有相应基因序列信息的其他6属7种作为外群, 构建系统发育树。
1 材料和方法 1.1 材料2014年, 在云南泸水发现的1只白腹鹭是该物种在中国分布的第一条确切记录(韩联宪等, 2015)。该个体于云南野生动物园进行救助, 饲养过程中死亡, 我们获得少量实验样品。其余物种的Cyt b和CO Ⅰ 基因序列均来自美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)(表 1)。
| 属Genus | 物种Species | 登录号 | |
| Cyt b基因 | CO Ⅰ基因 | ||
| 鹭属Ardea | 草鹭A. purpurea | KJ941160 | AB842574 |
| 大白鹭A. alba | GU346975 | AB842563 | |
| 大蓝鹭A. herodias | KX534434 | DQ432748 | |
| 白腹鹭A. insignis | 本研究 | 本研究 | |
| 黑冠白颈鹭A. cocoi | — | KM896243 | |
| 苍鹭A. cinerea | AY567914 | GU571740 | |
| 中白鹭A. intermedia | FJ499022 | HM804921 | |
| 啸鹭属Syrigma | 啸鹭S. sibilatrix | AF193820 | FJ028369 |
| 美洲夜鹭属Nyctanassa | 黄冠夜鹭N. violacea | EU166979 | DQ433047 |
| 波斑鹭属Zebrilus | 波斑鹭Z. undulatus | AF193834 | JN802112 |
| 牛背鹭属Bubulcus | 西方牛背鹭B. ibis | DQ485898 | AB842593 |
| 虎鹭属Tigrisoma | 横纹虎鹭T. fasciatum | EU166980 | JN802047 |
| 栗虎鹭T. lineatum | AF193831 | FJ028443 | |
麻  属Botaurus
属Botaurus |
大麻 B. stellaris B. stellaris |
AY465748 | AB842589 |
取-80 ℃保存的肌肉样品提取DNA。具体操作参考生工生物工程(上海)股份有限公司的Ezup柱式动物细胞/组织基因组DNA抽提试剂盒中使用说明书的标准抽提步骤。提取DNA后经琼脂糖凝胶电泳, 在凝胶成像仪下观察提取结果, 若出现明显条带且与目的条带长度相近, 则说明已成功提取出样品的总DNA, -20 ℃储存备用。
PCR反应体系为25 μL:5 μL DNA模板+12.5 μL Mix+5.5 μL ddH2O+2 μL引物(上下游引物各1 μL), 其中DNA模板和引物的密度均为10-5 mol·L-1。反应在Applied Biosystems型PCR仪中进行。CO Ⅰ 基因片段扩增选用的引物对为BirdF1(TTCTCCAACCACAAAGACATTGGCAC)和BirdR1(ACGTGGGAGATAATTCCAAATCCTG)(Kerr et al., 2007)。PCR反应条件为:94 ℃ 4 min;94 ℃1 min, 48 ℃ 1 min, 72 ℃ 1 min, 5个循环;94 ℃1 min, 55 ℃ 1 min, 72 ℃ 1 min, 35个循环;72 ℃7 min。Cyt b基因片段扩增选用的引物对为H16067(AGCCTTCAATCTTTGGCTTACAAG)和L14731(AATTGCATCCCACTTAATCGA)(Saetre et al., 2001)。PCR反应条件为:94 ℃ 4 min;94 ℃ 1 min, 48 ℃ 1 min, 72 ℃ 1.5 min, 35个循环;72 ℃ 7 min。在凝胶成像仪下确认PCR产物是目标条带后, 送生工生物工程(上海)股份有限公司进行双向测序。
1.3 DNA序列数据整理及分析将测序结果用SEQMAN进行拼接并辅以人工校正。结合序列图(abI格式文件), 剪去两端信号较杂乱的序列, 再查看2条链的重叠区, 即在序列的内部进行人工校正。一般情况下, 为保证序列的准确性, 重叠区域的碱基至少≥50 bp(黄原, 1998)。将拼接校正过的DNA序列在NCBI上进行BLAST相似性搜索, 以确保获得的序列为目的基因片段。
运用Clustal W(Thompson et al., 1997)将所有物种的序列进行比对, 再运用MEGA6.0对已经比对好的序列计算碱基组成情况、保守位点、变异位点、简约信息位点等, 用Kimura 2-parameter(K2P)法计算遗传距离。选取除鹭属之外的其他6属7种为外群, 将2段基因合并, 应用MrBayes3.1(Huelsenbeck & Ronquist, 2003)构建贝叶斯树, 运行4个马尔可夫链, 共运行5 000 000代, 至平均标准离差(sd)=0.004 315(只有当sd<0.01时说明达到收敛, 才可结束运行)。在FigTree 1.4.2中查看和修饰系统树。
2 研究结果 2.1 白腹鹭基因序列特征白腹鹭Cyt b基因序列共有1 163个位点(表 2)。碱基含量中G最高, C最低, A+T(51.7%)高于G+C(48.3%)。嘧啶含量(39.7%)明显低于嘌呤含量(60.3%)。第一位点和第二位点的A+T均高于G+C, 第三位点的A+T(48.0%)低于G+C(52.0%)。在密码子的不同位点, 碱基频率有不同偏倚, 第二位点上4种碱基含量较接近, 偏倚程度最小;第三位点上G含量增加到46.8%, C含量只有5.2%, 偏倚最突出。
白腹鹭CO Ⅰ 基因序列共有722个位点(表 2)。C含量最高, G含量最低。A+T(52.5%)高于G+C(47.5%)。嘧啶含量(57.0%)高于嘌呤含量(43.0%)。第一位点的A+T(41.5%)低于G+C(58.5%), 第二位点和第三位点的A+T均高于G+C。第一位点上碱基含量比较平均;第二位点上T高达41.0%, A只有14.9%;第三位点上A含量最高, G最少, 碱基偏倚现象在第三位点最明显。
| 位点Locus | Cyt b/COⅠ | ||||
| 数量Number | T/% | C/% | A/% | G/% | |
| 第一位点 | 388/241 | 22.2/17.0 | 11.3/28.2 | 38.4/24.5 | 28.1/30.3 |
| 第二位点 | 388/241 | 25.8/41.0 | 21.9/28.7 | 26.0/14.9 | 31.7/15.4 |
| 第三位点 | 387/240 | 32.8/18.8 | 5.2/37.4 | 15.2/41.3 | 46.8/2.5 |
| 合计 | 1 163/722 | 26.9/25.6 | 12.8/31.4 | 24.8/26.9 | 35.5/16.1 |
本研究获得白腹鹭的Cyt b基因片段, 合并来自NCBI的另外5种鹭属鸟类的同源区序列(NCBI中未查询到黑冠白颈鹭A. cocoi的Cyt b基因信息)。经过比对排列后共有822个位点。除了中白鹭A+ T的含量(49.3%)低于G+C的含量(50.7%)外, 其余5种的A+T的含量均高于G+C的含量(表 3)。保守位点650个, 可变位点172个, 简约信息位点47个, 单变异位点125个。物种间序列位点变异表现为转换多于颠换, R(转换/颠换)的平均值为3.46。
本研究获得白腹鹭的CO Ⅰ 基因片段, 合并来自NCBI的另外6种鹭属鸟类的同源区序列, 经过比对排列后共有599个位点。A+T的含量均高于G+C的含量(表 3)。保守位点485个, 可变位点111个, 简约信息位点60个, 单变异位点51个。物种间序列位点变异表现为转换多于颠换, R(转换/颠换)的平均值为10.04。
基于CO Ⅰ 基因计算鹭属各物种间的遗传距离, 白腹鹭与草鹭A. purpurea的遗传距离最小(0.062), 与大白鹭的遗传距离最大(0.093)(表 4)。
| 物种Species | Cyt b/COⅠ | |
| A+T/% | G+C/% | |
| 白腹鹭Ardea insignis | 51.5/52.0 | 48.5/48.0 |
| 大白鹭Ardea alba | 53.1/52.8 | 46.9/47.2 |
| 苍鹭Ardea cinerea | 50.2/52.4 | 49.8/47.6 |
| 中白鹭Ardea intermedia | 49.3/52.0 | 50.7/48.0 |
| 草鹭Ardea purpurea | 50.7/51.8 | 49.3/48.2 |
| 大蓝鹭Ardea herodias | 50.8/52.2 | 49.2/47.8 |
| 黑冠白颈鹭Ardea cocoi | —/52.2 | —/47.8 |
| 1 | 2 | 3 | 4 | 5 | 6 | |
| 1.苍鹭Ardea cinerea | ||||||
| 2.草鹭Ardea purpurea | 0.091 | |||||
| 3.大蓝鹭Ardea herodias | 0.019 | 0.096 | ||||
| 4.大白鹭Ardea alba | 0.129 | 0.103 | 0.125 | |||
| 5.白腹鹭Ardea insignis | 0.083 | 0.062 | 0.082 | 0.093 | ||
| 6.黑冠白颈鹭Ardea cocoi | 0.021 | 0.091 | 0.019 | 0.119 | 0.077 | |
| 7.中白鹭Ardea intermedia | 0.119 | 0.095 | 0.117 | 0.087 | 0.083 | 0.115 |
基于CO Ⅰ 和Cyt b基因片段构建贝叶斯系统发育树显示(图 1), 鹭属鸟类构成单系(支持率96%)。鹭属7种鸟类中, 最先分化的是大白鹭, 其次是白腹鹭、草鹭。苍鹭A. cinerea、大蓝鹭A. herodias、中白鹭、黑冠白颈鹭聚为一支(支持率97%)。白腹鹭位于鹭属分支内, 单独聚为一支, 无姐妹类群。
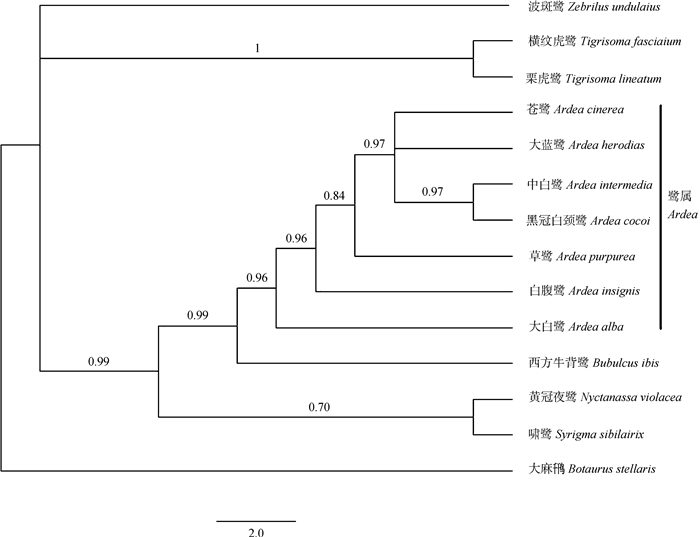
|
| 图 1 基于Cyt b+CO Ⅰ 基因序列构建的鹭科14种鸟类的贝叶斯树 Fig. 1 Bayes tree of 14 species from family Ardeidae based on Cyt b and CO Ⅰ genes |
| |
线粒体基因序列的碱基组成研究较多, 对74种鸟类线粒体基因组序列的分析表明, 密码子第二位的G、C含量波动较小, 第三位的波动幅度大, 这与第三位的C、T含量变化有关(高英凯等, 2009)。白腹鹭Cyt b和CO Ⅰ 基因序列的嘧啶和嘌呤含量有明显差异, 同样是由第三位点上G、C含量变化引起, 这与第三位点的变异通常不改变氨基酸编码、受选择压力小有关(钟东等, 2002)。
在鸟类线粒体基因序列中, 碱基组成存在一定的偏向性是普遍存在的现象(Arnaiz-Villena et al., 2001;Webb & Moore, 2005)。偏倚情况可能因物种不同而不同, 对24种雀类的研究表明, Cyt b基因序列密码子第一位点出现的频率相似, 第二位点G的频率明显低于其他3种碱基, 第三位点偏倚G、T(Arnaiz-Villena et al., 2001)。在本研究中, Cyt b基因序列的第一、二位点均无明显偏倚, 但第三位点明显偏倚G, 在该位点上的碱基偏倚与上述雀类研究相似。白腹鹭CO Ⅰ 基因序列的碱基偏倚和Cyt b基因不同, 第一位点没有明显的偏倚, 第二位点偏倚T, 第三位点偏倚A。
3.2 白腹鹭的系统分类地位根据本文结果, 鹭属7种聚为一支, 该节点有较高的支持率(96%), 支持IOC分类系统中把白腹鹭、大白鹭、中白鹭均放入鹭属中的处理(Gill & Donsker, 2014), 这与目前国内最新的鸟类分类系统相同(郑光美, 2017)。在鹭属中, 白腹鹭是较早分化的物种, 遗传距离分析表明和白腹鹭亲缘关系最近的是草鹭。然而, Rasmussen和Anderton(2005)提及大嘴鹭Ardea sumatrana外部形态特征和白腹鹭最为接近, 但大嘴鹭的物种地位尚不明确, 抑或是白腹鹭的某些地理种群产生了形态分化, 因此, 白腹鹭的系统分类地位还需进一步研究确认。
| 常青, 张保卫, 金宏. 2003. 从12s RNA基因序列推测鹭类13种鸟类的系统发生关系[J]. 动物学报, 49(2): 205–210. |
| 高英凯, 苗正旺, 苏小茜, 等. 2009. 74种鸟类线粒体基因组碱基组成及特征分析[J]. 云南农业大学学报, 24(1): 51–58. |
| 韩联宪, 何臣相, 王斌, 等. 2015. 云南发现白腹鹭[J]. 四川动物, 34(2): 281. |
| 黄原. 2012. 分子系统发生学[M]. 北京: 科学出版社. |
| 蒋志刚, 江建平, 王跃招, 等. 2016. 中国脊椎动物红色名录[J]. 生物多样性, 24(5): 500–551. DOI:10.17520/biods.2016076 |
| 张保卫, 常青, 魏辅文. 2002. 鹭科鸟类分类及系统学研究进展[J]. 动物学杂志, 37(3): 84–88. |
| 张国萍, 王蔚, 朱世杰, 等. 2005. 鹳形目12种鸟类核c-mos基因和线粒体12s rRNA基因序列分析及其系统发生研究[J]. 四川动物, 24(4): 500–506. |
| 郑光美. 2017. 中国鸟类分类与分布名录(第三版)[M]. 北京: 科学出版社. |
| 郑作新. 1964. 中国鸟类系统检索[M]. 北京: 科学出版社: 9-12. |
| 郑作新. 1994. 中国鸟类种和亚种分类名录大全[M]. 北京: 科学出版社: 9-11. |
| 钟东, 赵贵军, 张振书, 等. 2002. 基因组内碱基分布整体均衡与局部不均衡的研究进展[J]. 遗传, 24(3): 351–355. |
| Arnaiz-Villena A, Guillén J, Ruiz-del-Valle V, et al. 2001. Phylogeography of crossbills, bullfinches, grosbeaks, and rosefinches[J]. Cellular and Molecular Life Sciences, 58(8): 1159–1166. DOI:10.1007/PL00000930 |
| Ben FK, Edward CD, Martin WW. 1975. A field guide to the birds of south-east Asia[M]. Boston: Houghton Mifflin Press: 41-47. |
| Gill F, Donsker D. 2014. IOC world bird list (v 4. 3)[EB/OL]. [2017-11-07]. DOI: 10.14344/IOC.ML.4.3. |
| Hebert PDN, Stoeckle MY, Zemlak TS, et al. 2004. Identification of birds through DNA barcodes[J]. PLoS Biology, 2(10): 1657–1663. |
| Hermann H, Richard F, John P. 1972. The birds of Britain and Europe with north African and the middle East[M]. London: Collins Pres: 34-39. |
| Hoyo JD, Elliott A, Sargatal J. 1992. Handbook of the birds of the world vol. 5[M]. Spain: Lynx Edicions Press: 407. |
| Huelsenbeck JP, Ronquist F. 2003. MrBayes:Bayesian inference of phylogenetic trees[J]. Bioinformatics, 17: 754–755. |
| IUCN. 2016. The IUCN red list of threatened species[EB/OL]. (2016-10-01)[2017-11-07]. http://www.iucnredlist.org. |
| Kerr KCR, Stoeckle MY, Dove CJ, et al. 2007. Comprehensive DNA barcode coverage of north American birds[J]. Molecular Ecology Notes, 7(4): 535–543. DOI:10.1111/j.1471-8286.2007.01670.x |
| Mindell DP. 1997. Avian molecular evolution and systematics[M]. San Diego: Academic Press: 84-86. |
| Peters JL. 1945. Checklist of birds of the world vol. 5[M]. Cambridge, MA: Harvard University Press. |
| Rasmussen PC, Anderton JC. 2005. Birds of south Asia: the ripley guide[M]. Washington, Barcelona: Smithsonian Institution and Lynx Edicions. |
| Saetre GP, Borge T, Lindell J, et al. 2001. Speciation, introgressive hybridization and nonlinear rate of molecular evolution in flycatchers[J]. Molecular Ecology, 10(3): 737–749. |
| Sibley CG, Monoroe BLJ. 1990. Distribution and taxonomy of birds of the world[M]. New Haven & London: Yale University Press: 302-310. |
| Thompson JD, Gibson TJ, Plewniak F, et al. 1997. The Clustal X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Resreach, 24: 4876–4882. |
| Webb DM, Moore WS. 2005. A phylogenetic analysis of woodpeckers and their allies using 12S, Cyt b and COⅠ nucleotide sequences (Class Aves; Order Piciformes)[J]. Molecular Phylogenetics Evolution, 36: 233–248. DOI:10.1016/j.ympev.2005.03.015 |
| Weibel AC, Moore WS. 2002. Molecular phylogeny of a cosmopolitan group of woodpeckers (Genus Picoides) based on COⅠ and Cyt b mitochondrial gene sequences[J]. Molecular Phylogenetics Evolution, 22: 65–75. DOI:10.1006/mpev.2001.1010 |
 2018, Vol. 37
2018, Vol. 37




