扩展功能
文章信息
- 韦筱媚, 刘凡, 颜琳妙, 潘亚姐, 张月云, 赵成坚, 徐永莉, 李力, 黄勇
- WEI Xiaomei, LIU Fan, YAN Linmiao, PAN Yajie, ZHANG Yueyun, ZHAO Chengjian, XU Yongli, LI Li, HUANG Yong
- 基于线粒体基因的广西变色树蜥种群分子系统关系与遗传多样性
- Phylogenetic Relationship and Genetic Diversity of Calotes versicolor in Guangxi
- 四川动物, 2017, 36(5): 519-530
- Sichuan Journal of Zoology, 2017, 36(5): 519-530
- 10.11984/j.issn.1000-7083.20170067
-
文章历史
- 收稿日期: 2017-03-03
- 接受日期: 2017-06-20
2. 广西动力技工学校, 南宁 530023;
3. 广西师范学院, 南宁 530001
2. Guangxi Dongli Mechanic School, Nanning 530023, China;
3. Guangxi Teachers Education University, Nanning 530001, China
变色树蜥Calotes versicolor又叫东方园蜥,隶属于爬行纲Reptilia有鳞目Squamata蜥蜴亚目Lacertilia鬣蜥科Agamidae树蜥属Calotes,分布范围非常广,从阿曼、南亚、东南亚到中国,多栖息于热带和亚热带地区,常见于林下、山坡草丛、坟地、河边、路旁、住宅附近的草丛或树干上,分布海拔为80~2 000 m(Günther,1864;Boulenger,1912;Smith,1935;赵尓宓等,1999;Radder,2006)。这些充分反映了变色树蜥分布区域有很高的环境异质性,不同种群可能受到不同的选择压力从而产生分化。
变色树蜥形态的差异微乎其微,易被忽略;若不借助分子手段,基本难以区分。Zug等(2006)利用线粒体DNA序列重建缅甸变色树蜥复合体的系统发育关系,支持树蜥属物种构成单系,节点支持率为100%,其中普通树蜥C. calotes、屯温树蜥C. htunwini、实皆树蜥C. irawadi嵌在变色树蜥之中,并未形成交互单系。通过形态测量和主成分分析,进一步发现有2个新种(C. htunwini和C. irawadi)同域分布。而在之前,通常认为这2个新种均为“变色树蜥”。这表明常见物种“变色树蜥C.versicolor”是物种复合体,可能还有隐存种。
Huang等(2013)对海南岛分布的变色树蜥进行系统发育和谱系地理学分析,结果表明变色树蜥的演化历史非常复杂,存在2个显著的遗传谱系。分子钟估算表明变色树蜥起源于0.26 Ma前(95% CI=0.05~0.61 Ma),2个遗传谱系的分化时间分别为0.05 Ma和0.13 Ma。然而该报道的变色树蜥样品主要采自海南岛,涉及的大陆(如广西)样品(仅1个种群3个个体)较少,因此有必要进一步扩大采样,重建其系统发育关系,以期能更详细阐述该物种系统关系和揭示隐存种多样性。线粒体DNA由于其严格的母系遗传、进化速度快、高拷贝性等特有的优点而被广泛应用于种间或种内研究(Brown et al., 1979;Shearer et al., 2002),其中蛋白质编码基因(如ND2) 进化速率较快,适合物种内遗传变异研究(Poulakakis et al., 2003),涉及的物种包括爬行类(Heulin et al., 1999;Poulakakis et al., 2003)和其他脊椎动物(Hirota et al., 2004;Brown et al., 2007)、无脊椎动物(Horn et al., 2006)。因此,本研究通过采集在广西分布的变色树蜥样品(15个种群共90个个体),对其线粒体ND2-tRNA基因序列进行测定,分析种群的遗传多样性、结构等,探讨其系统发育关系及进化过程,以期揭示隐存种。
1 材料与方法 1.1 材料2012—2015年收集了分布于广西的变色树蜥样品,共15个种群90个个体(表 1)。标本在野外编号后,取肝脏或肌肉组织置于装有95%乙醇溶液的组织管保存。
| 种群编号 Population ID |
采样地点 Locality |
东经 East longitude |
北纬 North latitude |
样本大小 Sample size |
单倍型分布 Haplotypes |
遗传多样性 Genetic diversity |
中性检测 Neutrality test |
碱基错配分布 Mismatch distribution |
||||||
| N | h | π | Fu's Fs | Tajima's D | SSD | HRI | ||||||||
| 1 | 广西药用植物园 | 108°22′37.34″ | 22°51′35.82″ | 2 | Hap65(1),92(1) | 2 | 1.000 0 | 0.005 780 | 2.079 44 | 0.000 00 | 0.000 00 | 0.000 00 | ||
| 2 | 陆川县米场镇 | 110°16′33.07″ | 22°24′2.67″ | 1 | Hap93(1) | 1 | n.a. | n.a. | n.a. | n.a. | n.a. | n.a. | ||
| 3 | 南宁市吴圩镇 | 108°14′25.94″ | 22°39′34.59″ | 1 | Hap94(1) | 1 | n.a. | n.a. | n.a. | n.a. | n.a. | n.a. | ||
| 4 | 涠洲岛 | 109°8′21.54″ | 21°4′0.18″ | 21 | Hap95(7),96(1),97(2),98(1),99(1),100(3),101(1),104(1),116(1),117(1),118(1),119(1) | 12 | 0.881 0 | 0.005 239 | -0.000 24 | -1.438 66 | 0.038 86 | 0.045 87 | ||
| 5 | 北海市银海区 | 109°4′42.25″ | 21°28′05.51″ | 12 | Hap65(1),102(7),103(1),120(1),121(1),122(1) | 6 | 0.681 8 | 0.004 283 | 1.828 37 | -1.829 63 | 0.069 03 | 0.128 33 | ||
| 6 | 钦州市钦南区 | 108°39′13.74″ | 21°58′51.43″ | 8 | Hap102(1),105(2),106(1),107(1),108(1),109(1),115(1) | 7 | 0.964 3 | 0.005 869 | -0.605 75 | -0.652 09 | 0.071 40 | 0.167 09 | ||
| 7 | 防城港市港口区 | 110°18′4.94″ | 21°38′7.92″ | 7 | Hap102(2),110(1),111(4) | 3 | 0.666 7 | 0.000 549 | -0.437 69 | -0.274 92 | 0.050 44 | 0.292 52 | ||
| 8 | 南宁市五塘镇 | 108°33′20.03″ | 21°56′43.12″ | 3 | Hap65(1),102(1),112(1) | 3 | 1.000 0 | 0.000 480 | -1.216 40 | 0.000 00 | 0.164 34 | 0.666 67 | ||
| 9 | 浦北县文明村 | 109°41′59.28″ | 22°24′42.84″ | 4 | Hap113(1),114(3) | 2 | 0.500 0 | 0.000 360 | 0.171 85 | -0.612 37 | 0.021 94 | 0.250 00 | ||
| 10 | 浦北县高田村 | 109°41′24.72″ | 22°22′50.16″ | 2 | Hap113(1),114(1) | 2 | 1.000 0 | 0.000 721 | 0.000 00 | 0.000 00 | 0.000 00 | 0.000 00 | ||
| 11 | 天等县天等镇 | 107°9′0.47″ | 23°5′15.03″ | 3 | Hap123(1),124(1),125(1) | 3 | 1.000 0 | 0.002 405 | 0.308 30 | 0.000 00 | 0.171 54 | 0.444 44 | ||
| 12 | 上林县云储村 | 108°31′37.93″ | 23°31′11.87″ | 5 | Hap126(1),127(1),128(1),129(1),130(1) | 5 | 1.000 0 | 0.003 462 | -0.874 53 | -1.205 39 | 0.089 10 | 0.180 00 | ||
| 13 | 武鸣县大明山 | 108°20′33.21″ | 23°31′35.54″ | 5 | Hap102(3),131(2) | 2 | 0.600 0 | 0.000 865 | 2.428 55 | 1.458 84 | 0.302 50 | 0.880 00 | ||
| 14 | 隆安县丁当镇 | 107°58′33.75″ | 23°7′49.40″ | 15 | Hap65(1),102(5),132(6),133(2),134(1) | 5 | 0.752 4 | 0.000 755 | 0.585 32 | 1.850 31 | 0.057 12 | 0.130 07 | ||
| 15 | 凭祥市布关 | 106°47′31.76″ | 22°4′27.99″ | 1 | Hap135(1) | 1 | n.a. | n.a. | n.a. | n.a. | n.a. | n.a. | ||
| 所有种群 | 90 | 45 | 0.941 8 | 0.005 500 | -22.382 15*** | -2.0613 6** | 0.003 85 | 0.007 58 | ||||||
| 注:**P<0.01,***P<0.001;单倍型后括号中数字为每一种群中该单倍型的个体数目; n.a.该项数据无法计算或不可用。 Notes:**P<0.01,***P<0.001; numbers in the parentheses following the haplotype are the numbers of individuals for each population; n.a.data not applicable. |
||||||||||||||
基因组DNA的提取采用Ezup柱式动物基因组DNA抽提试剂盒[生工生物工程(上海)股份有限公司]。基因序列片段通过PCR扩增所得,扩增所用引物为L3705和H5162(Huang et al., 2013)。PCR反应的总体积为50 μL,用大约100 ng的基因组DNA作为模板,反应体系包括:10×Ex-Taq Buffer(不含Mg2+)5 μL,dNTPs 4 μL (2.5 mmol·L-1),引物各2 μL (1 mmol·mL-1),Ex-Taq DNA聚合酶(5 U·mL-1) 0.3 μL,加灭菌双蒸水至50 μL。PCR反应条件为:95 ℃预变性3 min;94 ℃变性35 s,58~61 ℃退火45 s,72 ℃延伸1 min,共35个循环;最后72 ℃延伸8 min。将获得的PCR产物送生工生物工程(上海)股份有限公司进行双向测序测通,测序引物和PCR扩增引物一致。
1.2.2 数据分析首先用BioEdit 7.0.9.0(Hall,1999)对所获得的核苷酸序列进行多重比对,并辅以手工校正。在MEGA 6.0(Tamura et al., 2013)中用脊椎动物线粒体遗传密码子把所有ND2蛋白质编码基因片段翻译为氨基酸,以确认蛋白质编码基因片段是否具有功能。在序列中没有发现终止密码子,表明所获得的序列是线粒体而不是核基因拷贝。然后用DnaSP 5.0(Librado & Rozas,2009)对所有序列或每个种群计算单倍型数、单倍型多样性(h)和核苷酸多样性(π)(Nei et al., 1975)。
为了构建更加全面的变色树蜥系统进化树,从GenBank上下载了相关类群的ND2-tRNA基因片段(表 2),特别是将Huang等(2013)发表的变色树蜥个体(共212个个体91个单倍型,Hap1~Hap91) 的序列全部下载一起分析(GenBank登录号为KC875609~KC875820;表 2)。基于单倍型序列数据,利用jmodeltest 0.1(Posada & Crandall,1998)分析序列最佳的碱基替换模型,得到基于贝叶斯信息准则标准的最佳替换模型TPM+I+G,然后分别采用最大简约法(Maximum parsimony,MP)、贝叶斯法(Bayesian inference method,BI)和最大似然法(Maximum likelihood method,ML)构建分子系统发育树。MP分析在PAUP* 4.0b10(Swofford,2003)中进行,系统树的分支置信度采用自引导法进行1 000次重复检测。BI用MrBayes 3.2(Ronquist et al., 2011),以随机树为起始树,替代模型参数Nst=6,马尔科夫链的蒙特卡洛方法设置为4条链同时运行1×106代,3条热链1条冷链,每1 000代对系统树进行抽样,最终得到10 001棵系统发育树,舍弃前面的5 000棵树,构建一致树。ML采用GTR模型在RAxML 7.0.4(Stamatakis et al., 2008;http://phylo-bench.vital-it.ch/raxml-bb/index.php)上完成运算。
| 变异来源 Source of variation |
自由度 df |
平方和 Sum of squares |
变异组分 Variance components |
变异百分数/% Percentage of variation |
| 种群间 | 13 | 127.845 | 1.222 09 | 29.35 |
| 种群内 | 71 | 208.873 | 2.941 87 | 70.65 |
| 总计 | 84 | 336.718 | 4.163 96 |
为了检测不同种群间的基因交流情况,采用分子方差分析(AMOVA)方法分析不同种群间的遗传分化程度(Fst),在Arlequin 3.5(Excoffier & Lischer,2010)中完成。利用Network 5.0(Bandelt et al., 1999)中的Median-Joining模型构建单倍型之间的网络关系。
为检测所采集种群的历史种群动态,采用Arlequin 3.5(Excoffier & Lischer,2010)估计Tajima's D值(Tajima,1989)、Fu's Fs值(Fu,1997),以及碱基的错配分布。此外,使用基于溯祖模拟的方法——贝叶斯轮廓图(Bayesian skyline plot)(Drummond et al., 2005)进行种群历史动态分析,在BEAST 1.8.4中完成(Drummond & Rambaut,2007)。目前未见树蜥属的化石记录及相关地质事件报道,故使用Huang等(2013)分析所得进化速率每位点每百万年5.87%。碱基替换模型选择GTR模型,每次运行2×107,舍去10%的老化样本,每隔1 000代对系统树抽样。最后在Tracer 1.5(Rambaut & Drummond,2007)的Bayesian Skyline Reconstruction中构建种群历史动态图,各项参数的Effective sample size均大于200。
2 结果 2.1 序列多态性分析本研究共测序和收集了15个种群90个变色树蜥的序列,共1 390 bp。序列中包括全部ND2基因序列(共1 023 bp)、部分ND2上游和下游的tRNA序列(共367 bp)。ND2基因内部没有发现终止密码子,ND2全序列的终止密码子相同,插入和缺失都发生在tRNA部分。序列中A、T、C、G碱基平均含量分别为35.0%、23.8%、29.0%、12.2%,表现出明显的反G偏倚,同时A+T的含量(58.9%)高于G+C的含量(41.1%),这都与脊椎动物线粒体DNA的特点一致。变异位点86个,其中简约信息位点49个。
2.2 不同地理种群遗传差异分析各采样点单倍型的分布情况及遗传多样性参数见表 1。90条序列共定义了45种单倍型,其中6个地理种群(北海市银海区、钦州市钦南区、防城港市港口区、南宁市五塘镇、武鸣县大明山和隆安县丁当镇)的个体共享1个单倍型(单倍型102,占总单倍型数的2.2%),4个地理种群(广西药用植物园、北海市银海区、南宁市五塘镇和隆安县丁当镇)的个体共享1个单倍型(单倍型65,占总单倍型数的2.2%),3个地理种群(北海市银海区、南宁市五塘镇和隆安县丁当镇)的个体共享2个单倍型(单倍型65和102,占总单倍型数的4.4%),2个地理种群(浦北县文明村和浦北县高田村)的个体共享2个单倍型(单倍型113和114,占总单倍型数的4.4%);剩下地理种群的个体均为私有单倍型,占总单倍型数的91.1%。
单倍型分布范围从1种到12种,其中涠洲岛地理、种群的单倍型最多,有12种,最多7个个体共享1个单倍型。h值从0.500 0到1.000 0,π值从0.000 360到0.005 869。钦州市钦南区种群的核苷酸多样性最高,而浦北县文明村种群的最低。
对种群间遗传分化程度的研究表明,大部分种群间遗传分化指数Fst值均较高且差异有统计学意义,遗传分化程度都比较高。此外,对所有种群进行AMOVA分析,总体上,遗传变异主要来自于种群内(70.65%),种群间的遗传变异相对较小(29.35%)(表 2)。
2.3 单倍型系统进化分析及网络分析变色树蜥单倍型间的系统发育关系见图 1、图 2和图 3。以表 3的物种为外群,3种分析方法得到的系统发育树的拓扑结构有差异(所有单倍型构建系统发育树可向作者邮件索取),但都显示本文所研究的变色树蜥种群和GenBank下载的变色树蜥序列聚成一单系,支持率为100%(MP)、100%(ML)和1.00(BI)。此外,本研究的变色树蜥种群的拓扑结构并不能聚成单系,各种群间不存在明显的谱系结构。值得一提的是,涠洲岛种群的所有个体并不能构成单系。在MP和BI法构建的系统发育树中,涠洲岛(5个单倍型)、北海市银海区(1个单倍型)、钦州市钦南区(1个单倍型)、上林县云储村(1个单倍型)和广西药用植物园(1个单倍型)种群构建的拓扑结构一致,支持率分别是100%和0.89。这个拓扑结构在MP树和BI树中分别和其他种群单倍型组成的姐妹群也不一致,MP树和在陆川县、吴圩镇和天等镇采集到的个体组成姐妹群(支持率为100%),BI树中仅和凭祥市采集的个体组成姐妹群(支持率为0.55)。然而这些单倍型在ML构建的系统发育树中是多系。此外,在涠洲岛采样的其他个体(7个单倍型)和北海市银海区(1个单倍型)、钦州市钦南区(1个单倍型)在MP树聚成单系(支持率为100%),而在BI树和ML树没有得到解析,呈梳齿状排列。
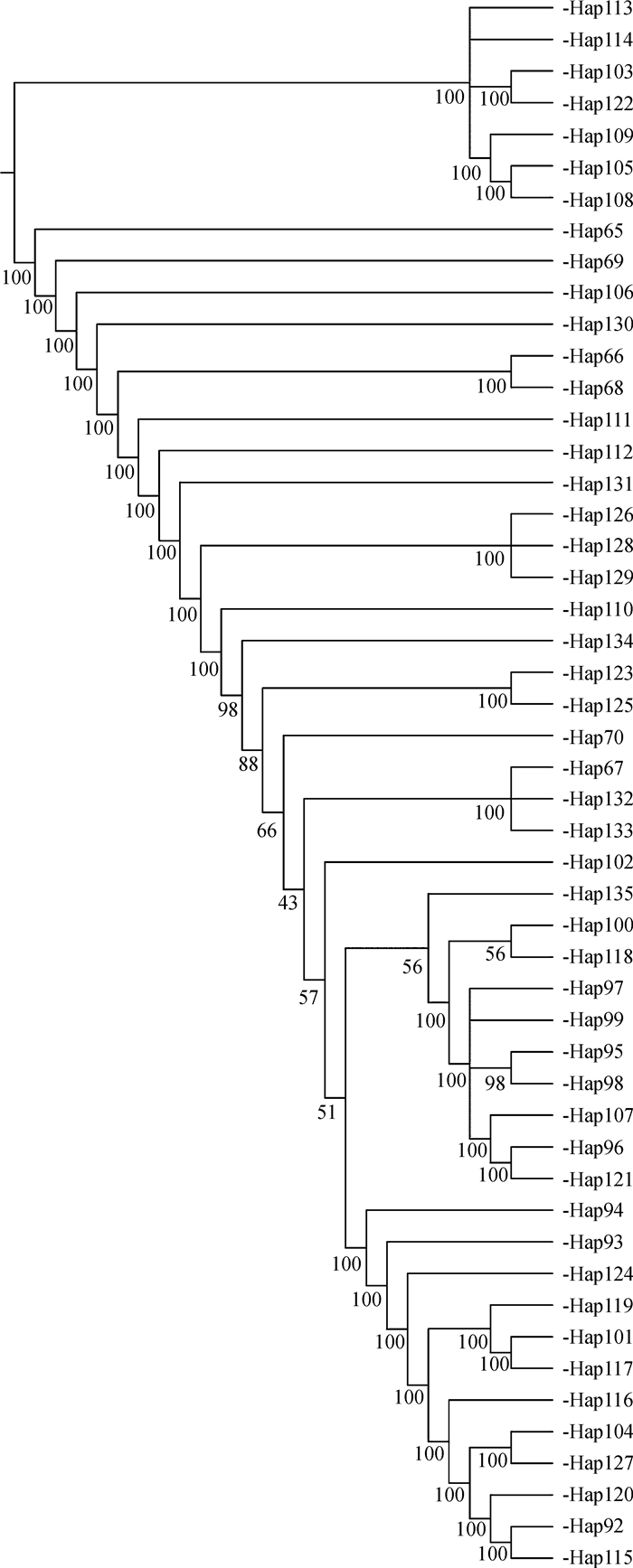
|
| 图 1 基于单倍型构建的变色树蜥不同地理种群的最大简约树 Fig. 1 Maximum parsimony tree of each Calotes versicolor population based on haplotype 节点下的数值代表支持率;Hap表示单倍型,其后的序号为单倍型的编号,见表 1和表 3;下同。 Values below the node represent support values; Hap denotes haplotype, subsequent number means haplotype code following Table 1 and Table 3; the same below. |
| |
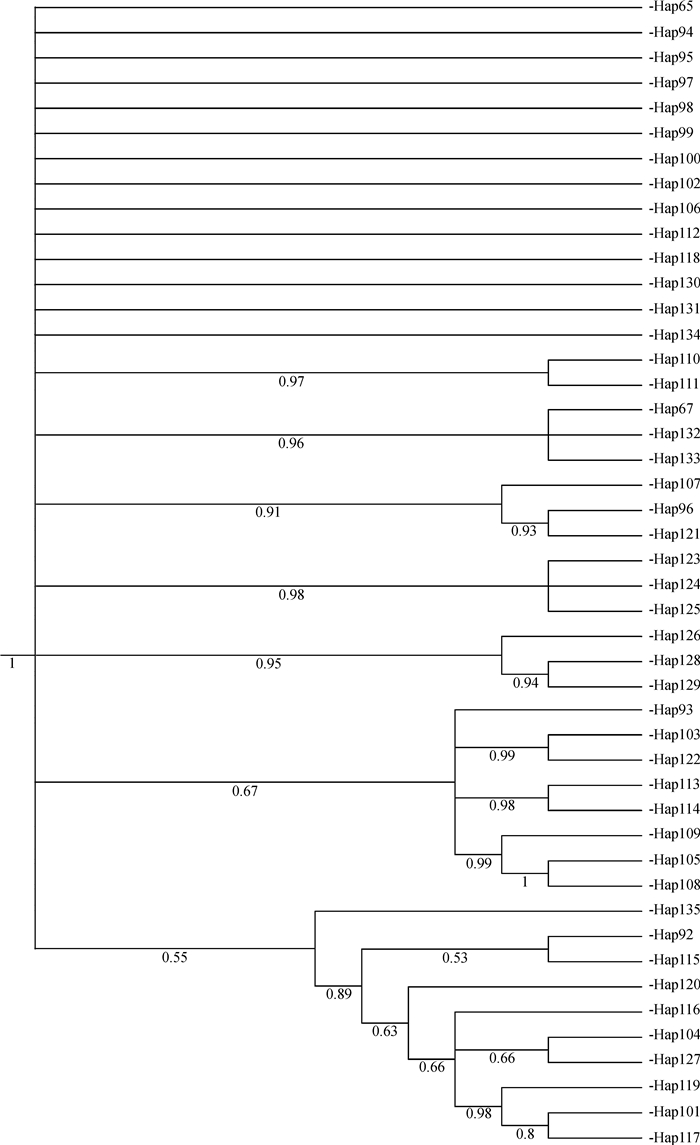
|
| 图 2 基于单倍型构建的变色树蜥不同地理种群的贝叶斯树 Fig. 2 Bayesian inferences (BI) tree of each Calotes versicolor population based on haplotype |
| |

|
| 图 3 基于单倍型构建的变色树蜥不同地理种群的最大似然树 Fig. 3 Maximum likelihood method (ML) tree of each Calotes versicolor population based on haplotype |
| |
| 物种Species | GenBank登录号Accession number |
| 长棘蜥Acanthosaura armata | AB266452 |
| 棕背树蜥Calotes emma | DQ289460 |
| 普通树蜥Calotes calotes | AF128482 |
| 斯里兰卡树蜥Calotes ceylonensis | AF128483 |
| 滑头树蜥Calotes liocephalus | AF128484 |
| 滑鳞树蜥Calotes liolepis | AF128485 |
| 白唇树蜥Calotes mystaceus | AF128487,AF128488 |
| 黑唇树蜥Calotes nigrilabris | AF128486 |
| 钦山树蜥Calotes chincollium | DQ289458,DQ289459 |
| 屯温树蜥Calotes htunwini | DQ289461,DQ289462,DQ289463,DQ289464 |
| 实皆树蜥Calotes irawadi | DQ289465,DQ289466,DQ289467,DQ289468 |
| 绿背树蜥Calotes jerdoni | GQ502783 |
| 变色树蜥Calotes versicolor | CAS230481,CAS206551,CAS204991,CAS222606,CAS205008,USNMGZ35831,USNMGZ35815,USNMGZ35783,USNMGZ35987,KC875609~KC875820 (Hap1~Hap91) |
单倍型进化网络关系图的结果也显示各采样种群间不存在明显的谱系结构,不存在原始单倍型和进化中心,并且只有2个单倍型有种群间共享。单倍型间相差一步或多步的突变距离彼此相连,部分单倍型之间通过缺失的中间单倍型相互连接(图 4)。
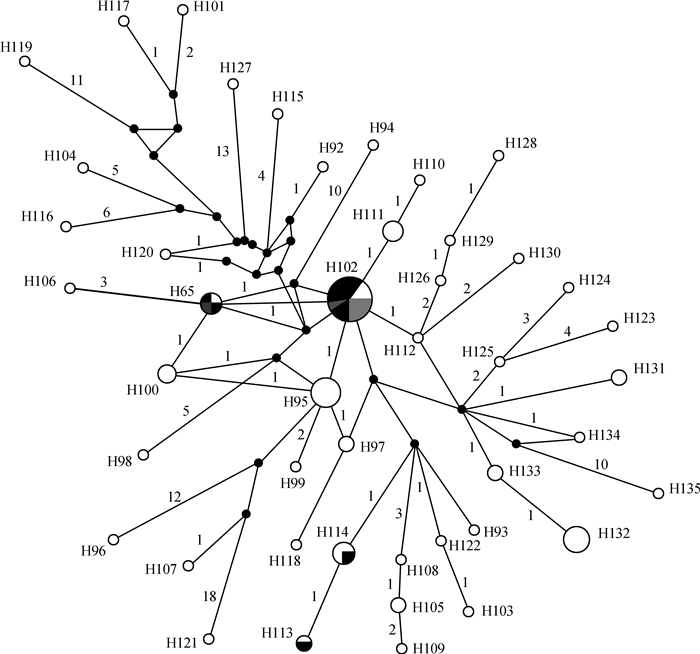
|
| 图 4 变色树蜥不同地理种群的单倍型网络图 Fig. 4 The haplotype network of each Calotes versicolor population 有数字的圆圈代表单倍型,黑色实点代表没有采到的单倍型;单倍型圆圈中灰色、黑色和白色部分分别代表不同的地理来源;每个圆圈的大小代表相应单倍型个体数的比例。 Numbers represent haplotype number and black dots representing unsampled haplotypes; the size of each circle represents the corresponding haplotype in proportion to its frequency; gray, black and white portions in the haplotype circle represent the geographic origin of the Calotes versicolor respectively; each mutation step is shown as a short line connecting neighboring haplotypes and numbers of mutations between haplotypes near branches. |
| |
Tajima's D和Fu's Fs中性检验及错配分布分析表明,所检测的地理种群均没有偏离中性假说。Tajima's D和Fu's Fs值虽出现负值,但差异均无统计学意义(P>0.05)。此外,各个地理种群中的平方差总和(SSD)和Harpending's参差不齐指数(HRI)的差异均无统计学意义(P>0.05)(表 1)。另外,分析更大范围内的种群,即把所有地理种群视为同一种群进一步分析表明,Tajima's D和Fu's Fs值均是负值且差异有高度统计学意义,错配分布呈单峰型(表 1,图 5)。
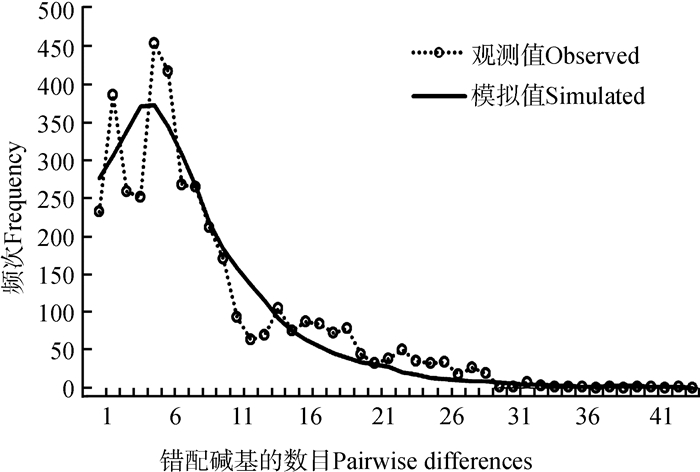
|
| 图 5 变色树蜥种群碱基错配分布图 Fig. 5 Mismatch distribution of overall Calotes versicolor populations |
| |
整个变色树蜥的有效种群大小约在0.05 Ma前都呈现扩张趋势,且平均θ值达到最高值,为0.07,接着呈现出比较稳定的趋势(图 6)。
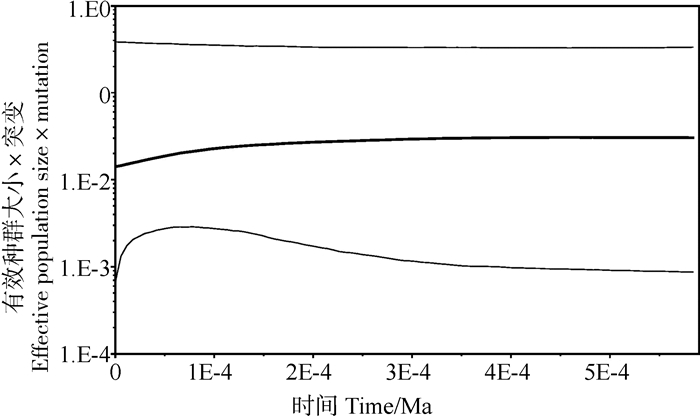
|
| 图 6 变色树蜥种群贝叶斯轮廓图 Fig. 6 A Bayesian skyline plot representing the historical demographic trend of each group of overall Calotes versicolor populations 中间的黑色线是有效种群大小的中值估计,而灰色线是95%的区间。 The black central line shows the meadian estimates for effective population size, while gray lines represent the 95% interval. |
| |
Wright等(1965)认为Fst值在0~0.05表明种群间没有遗传分化,0.05~0.15为中度遗传分化,大于0.15为高度遗传分化。本研究中,除少数种群外(约占所有种群间的组合数量20%),大部分种群间的Fst值均大于0.15,意味着大部分种群间存在高度遗传分化。从同一个地点采样的地理种群的私有单倍型出现频率很高,种群间共享单倍型极少,这意味着种群间的基因流有限,不同变色树蜥地理种群可能正进行着相对独立地理隔离内遗传分化。这与Huang等(2013)主要分析的结果一致。
值得注意的是,分布在涠洲岛的变色树蜥种群和其他种群(除与南宁市五塘镇的种群外)的Fst值均大于0.15,且差异有统计学意义(除分别与南宁市五塘镇、浦北县高田村和凭祥市布关的种群外)。此外,涠洲岛种群的单倍型也没有和其他种群共享。这些结果表明,涠洲岛种群正和邻近大陆的其他种群在分子水平上发生遗传分化,这意味着可能存在隐存种,需要在形态上进一步研究。DNA分子序列经历着迅速而连续的变化,而生物的形态进化需要长时间不断与环境相互作用的积累才能有显著的改变。因为绝大多数DNA序列变化的信息并不传递到蛋白体上,因而对生物的表型没有多大的影响(王元青,1991)。这种分子进化和形态进化不同步的现象在很多物种如楔齿蜥Sphenodon punctatus (Hay et al., 2008)上都存在。
3.2 变色树蜥种群的分子系统关系通过MP、BI和ML构建的单倍型系统发育树揭示了变色树蜥地理种群并没有出现明显的遗传分支,且在地理上也没有严格的分布范围和分化,这可能是种群间存在渐渗杂交或物种间近期的分化导致不完全的谱系筛选,这均是自然界普遍发生的模式(Salzburger et al., 2002;Nosil,2008)。3种方法构建的系统发育树的拓扑结构不完全一致,没有完全解决采集样品的系统关系问题,这可能是构建系统发育树的方法不同导致。各种建树方法均有优劣(Holder & Lewis,2003),在实际分析中,往往需要联合使用不同的建树方法以获得最佳分析结果。如果不同的方法构建进化树所得到的拓扑结构类似,且支持率均较高,则得到的结果较为可靠。
此外,所选基因长度的代表性有限和受谱系随机分选的影响,提供的系统发育信息不足以解析全面的系统关系也会导致拓扑结构的不一致。ND2-tRNA的基因片段作为分子标记在许多爬行动物重建系统发育关系中使用,并且能有效解决种间、种内的系统发育关系(Macey et al., 2000;Townsend et al., 2004;Zug et al., 2006;Schulte II & Moreno-Roark,2010)。然而本研究在种群内进行分析,可能由于所选的基因序列在进化速率上无法解析它们的近缘关系。这可以通过增加样本量或增加序列长度和基因位点(如核基因位点)来解决。如Huang等(2013)通过测序变色树蜥的ND2、COX1以及它们之间的tRNA片段共2 663 bp,能够有效地发现变色树蜥种群间分化为2个遗传谱系,而本研究只分析了ND2-tRNA片段,无法得到和Huang等(2013)一致的2个遗传谱系。
由于长期地理隔离,同一物种的不同地理种群可能会产生遗传分化(Duncan et al., 2016)。涠洲岛位于广西沿海大陆架之上,距离广西北海市约36海里,是我国地质年龄最年轻的火山岛,约在250万年前到7 000年前发生了数百次基性火山喷发。直到晚更新世后期,涠洲岛才完全露出海面。种群遗传结构分析表明,涠洲岛的变色树蜥种群正和邻近大陆的其他种群在分子水平上发生遗传分化,这可能是地理隔离导致,然而在单倍型系统发育中,涠洲岛变色树蜥种群的单倍型并没有全部聚成一支,其中5个单倍型和其他种群(北海市银海区、钦州市钦南区、上林县云储村和广西药用植物园)的单倍型聚在一起,这可能是因为共同的祖先种群扩散和进化,表明这些种群间近期存在基因交流,也意味着涠洲岛种群内部存在遗传分化,因此需要更多的样本和基因位点作进一步分析。此外,北海市银海区、钦州市钦南区、防城港市港口区、南宁市五塘镇、武鸣县大明山和隆安县丁当镇这些地理种群共享1个单倍型,虽然这些地理种群相距很远,但也表明它们很可能来自共同的祖先,同时也是一种能够适应环境选择的单倍型。
3.3 变色树蜥的种群历史一般来讲,经历扩张的种群,其碱基错配分布呈单峰,Fu's Fs或者Tajima's D为负值且统计显著(Rogers & Harpending,1992);而核苷酸错配分布呈多峰或双峰型的种群具有较强的种群遗传结构,或正在经历种群规模下降,而不规则的曲线则提示种群经历了广泛的扩散(Excoffier et al., 1992;Rogers & Harpending,1992;Excofıer & Schneider,1999)。多峰型也可能是种群受到迁移扩散、分化和(或)经历了种群收缩(Marjoram & Donnelly,1994;Ray et al., 2003)。在单独对每个地理种群进行碱基错配分析时,并没有发现Fu's Fs或者Tajima's D为负值且呈显著性。当把所有的采样种群合并在一起分析时,Tajima's D和Fu's Fs值均是负值且差异有高度统计学意义,错配分布呈单峰型,说明整个种群在历史上发生过快速扩张。此外,SSD及HRI的统计检验不显著表明不能拒绝群体扩张的假说,即符合原来种群扩张假说(Excoffier & Lischer,2010)。
与碱基错配分析方法不同,贝叶斯轮廓图分析可以展示各个时期的历史动态变化,当然也可以显示种群扩张或者下降、瓶颈等发生的时间。所有变色树蜥合并在一起的贝叶斯轮廓图分析表明,变色树蜥在距今0.05 Ma前存在种群扩张现象。此外,遗传多样性也能为种群动态历史分析提供支持。种群遗传多样性低被认为可能是由于瓶颈效应、建群效应和遗传漂变(Lehmann et al., 1998;Hedrick,1999)。总体变色树蜥种群的单倍型多样性较高(0.941 8),而核苷酸多样性相对较低(0.005 5),可能是在某段时间内有效种群数量的减少所致(Nei et al., 1975)。
| 王元青. 1991. 分子进化与形态进化是相互矛盾还是协调的?[J]. 古脊椎动物学报, 29(1): 80–81. |
| 赵尓宓, 赵肯堂, 周开亚, 等. 1999. 中国动物志[M]. 北京: 科学出版社: 38. |
| Bandelt HJ, Forster P, Rohl A. 1999. Median-joining networks for inferring intraspecific phylogenies[J]. Molecular Biology and Evolution, 16(1): 37–48. DOI:10.1093/oxfordjournals.molbev.a026036 |
| Boulenger GA. 1912.A vertebrate fauna of the Malay Peninsula from the Isthamus of Kra to Singapore, including the adjacent islands[M]//Robinson HC, Boulenger GA. Reptilia and Batrachia. London:Taylor and Francis. |
| Brown WM, George M Jr., Wilson AC. 1979. Rapid evolution of mitochondrial DNA[J]. Proceedings of the National Academy of USA, 76: 1967–1971. DOI:10.1073/pnas.76.4.1967 |
| Brown JW, Van Coeverden de Groot PJ, Birt TP, et al. 2007. Appraisal of the consequences of the DDT-indueed bottle neck on the level and geographic distribution of neutral genetic variation in Canadian peregrine falcons, Falco peregrinus[J]. Molecular Ecology, 16(2): 327–343. DOI:10.1111/j.1365-294X.2007.03151.x |
| Drummond AJ, Rambaut A. 2007. BEAST:Bayesian evolutionary analysis by sampling trees[J]. BMC Evolutionary Biology, 7(1): 214. DOI:10.1186/1471-2148-7-214 |
| Drummond AJ, Rambaut A, Shapiro B, et al. 2005. Bayesian coalescent inference of past population dynamics from molecular sequences[J]. Molecular Biology and Evolution, 22(5): 1185–1192. DOI:10.1093/molbev/msi103 |
| Duncan C, Worth J, Jordan G, et al. 2016. Genetic differentiation in spite of high gene flow in the dominant rainforest tree of southeastern Australia, Nothofagus cunninghamii[J]. Heredity, 116(1): 99–106. DOI:10.1038/hdy.2015.77 |
| Excoffier L, Lischer HE. 2010. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 10(3): 564–567. DOI:10.1111/men.2010.10.issue-3 |
| Excofıer L, Schneider S. 1999. Why hunter-gatherer populations do not show sign of Pleistocene demographic expansions[J]. Proceedings of the National Academy of USA, 96(19): 10597–10602. DOI:10.1073/pnas.96.19.10597 |
| Excoffier L, Smouse P, Quattro J. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J]. Genetics, 131(2): 479–491. |
| Fu YX. 1997. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 147(2): 915–925. |
| Günther A. 1864. The reptiles of British India[M]. London: Taylor and Francis: ⅹⅹⅶ+452. |
| Hall TA. 1999. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 41(41): 95–98. |
| Hay JM, Subramanian S, Millar CD, et al. 2008. Rapid molecular evolution in a living fossil[J]. Trends in Genetics, 24(3): 106–109. DOI:10.1016/j.tig.2007.12.002 |
| Hedrick PW. 1999. Perspective:highly variable loci and their interpretation in evolution and conservation[J]. Evolution, 53(2): 313–318. DOI:10.1111/evo.1999.53.issue-2 |
| Heulin B, Surget-Groba Y, Guiller A, et al. 1999. Comparisons of mitoehondrial DNA (mtDNA) sequenees (16S rRNA gene) between oviparous and viviparous strains of Lacertavivi vivipara:a preliminary study[J]. Molecular Ecology, 8(10): 1627–1631. DOI:10.1046/j.1365-294x.1999.00746.x |
| Hirota T, Hirohata T, Mashima H, et al. 2004. Population structure of the large Japanese field mouse Apodemus speciosus (Rodentia:Muridae) in suburban landscape based on mitoehondrial D-loop sequences[J]. Molecular Ecology, 13(11): 3275–3282. DOI:10.1111/j.1365-294X.2004.02324.x |
| Huang Y, Guo X, Ho YWS, et al. 2013. Diversification and demography of the Oriental garden lizard (Calotes versicolor) in Hainan Island and the adjacent mainland[J]. PLoS ONE, 8(6): e64754. DOI:10.1371/journal.pone.0064754 |
| Holder MT, Lewis PO. 2003. Phylogeny estimation:traditional and Bayesian approaches[J]. Nature Reviews Genetics, 4(4): 275–284. DOI:10.1038/nrg1044 |
| Horn A, Roux-Morabito G, Lieutier F, et al. 2006. Phylogeographic strueture and past history of the circum-Mediterranean species Tomicus destruens Woll. (ColeoPtera:Seolytinae)[J]. Molecular Ecology, 15(6): 1603–1615. DOI:10.1111/j.1365-294X.2006.02872.x |
| Lehmann T, Hawley WA, Grebert H, et al. 1998. The effective population size of Anopheles gambiae in Kenya:implications for population structure[J]. Molecular Biology and Evolution, 15(3): 264–276. DOI:10.1093/oxfordjournals.molbev.a025923 |
| Librado P, Rozas J. 2009. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 25(11): 1451–1452. DOI:10.1093/bioinformatics/btp187 |
| Macey Jr., Schulte JA, Larson A, et al. 2000. Evaluating trans-tethys migration:an example using acrodont lizard phylogenetics[J]. Systematic Biology, 49(2): 233–256. DOI:10.1093/sysbio/49.2.233 |
| Marjoram P, Donnelly P. 1994. Pairwise comparisons of mitochondrial DNA sequences in subdivided populations and implications for early human evolution[J]. Genetics, 136(2): 673–683. |
| Nei M, Maruyama T, Chakraborty R. 1975. The bottleneck effect and genetic variability in populations[J]. Evolution, 29(1): 1–10. DOI:10.1111/evo.1975.29.issue-1 |
| Nosil P. 2008. Speciation with gene flow could be common[J]. Molecular Ecology, 17(9): 2103–2106. DOI:10.1111/mec.2008.17.issue-9 |
| Posada D, Crandall KA. 1998. Modeltest:testing the model of DNA substitution[J]. Bioinformatics, 14(9): 817–818. DOI:10.1093/bioinformatics/14.9.817 |
| Poulakakis N, Lymberakis P, Antoniou A, et al. 2003. Molecular phylogeny and biogeography of the wall-lizard Podarcis erhardii (Squamata:Lacertidae)[J]. Molecular Phylogenetics and Evolution, 28(1): 38–46. DOI:10.1016/S1055-7903(03)00037-X |
| Radder RS. 2006. An overview of geographic variation in the life history traits of the tropical agamid lizard, Calotes versicolor[J]. Current Science, 91(10): 1354–1363. |
| Ray N, Currat M, Excoffier L. 2003. Intra-deme molecular diversity in spatially expanding populations[J]. Molecular Biology and Evolution, 20(1): 76–86. DOI:10.1093/molbev/msg009 |
| Rambaut A, Drummond A. 2007. Tracer. MCMCtrace analysis tool version v1.5.0, 2003-2009[M]. Oxford, UK: University of Oxford. |
| Rogers AR, Harpending H. 1992. Population growth makes waves in the distribution of pairwise genetic differences[J]. Molecular Biology and Evolution, 9(3): 552–569. |
| Ronquist F, Teslenko M, van der Mark P, et al. 2011. MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space[J]. Systematic Biology, 61(3): 539–542. |
| Salzburger W, Baric S, Sturmbauer C. 2002. Speciation via introgressive hybridization ineast African cichlids[J]. Molecular Ecology, 11(3): 619–625. DOI:10.1046/j.0962-1083.2001.01438.x |
| Schulte II JA, Moreno-Roark F. 2010. Live birth among iguanian lizards predates Pliocene-Pleistocene glaciations[J]. Biology Letter, 6(6): 216–218. |
| Shearer TL, van Oppen MJH, Romano S, et al. 2002. Slow mitochondrial DNA sequence evolution in the Anthozoa (Cnidaria)[J]. Molecular Ecology, 11(12): 2475–2487. DOI:10.1046/j.1365-294X.2002.01652.x |
| Smith MA. 1935. The fauna of British India, including Ceylon and Burma. Reptiles and Amphibia, Vol. Ⅱ. Sauria[M]. London: Taylor and Francis: 40. |
| Stamatakis A, Hoover P, Rougemont J. 2008. A rapid bootstrap algorithm for the RAxML web-servers[J]. Systematic Biology, 75(5): 758–771. |
| Swofford DL. 2003. PAUP* phylogenetic analysis using Parsimony (*and other methods), version 4.0b10[M]. Sunderland: Massachusetts:Sinauer Associates. |
| Tamura K, Stecher G, Peterson D, et al. 2013. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 30(12): 2725–2729. DOI:10.1093/molbev/mst197 |
| Tajima F. 1989. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 123(3): 585–595. |
| Townsend T, Larson A, Louis EJ, et al. 2004. Molecular phylogenetics of Squamata:the position of snakes, amphisbaenians, and dibamids, and the root of the squamate tree[J]. Systematic Biology, 53(5): 735–757. DOI:10.1080/10635150490522340 |
| Wright S. 1965. The interpretation of population structure by F-statistics with special regard to systems of mating[J]. Evolution, 19(3): 395–420. DOI:10.1111/evo.1965.19.issue-3 |
| Zug GR, Brown HHK, Schulte II JA, et al. 2006. Systematics of the garden lizards, Calotes versicolor group (Reptilia, Squamata, Agamidae), in Myanmar:central dry zone populations[J]. Proceedings of the California Academy of Sciences, 57(2): 35–68. |
 2017, Vol. 36
2017, Vol. 36




