扩展功能
文章信息
- 史博洋, 李媛媛, 王儒晓, 曲江勇, 郭承华, 刘传林, 侯建海, 房辉, 刘楚童
- SHI Boyang, LI Yuanyuan, WANG Ruxiao, QU Jiangyong, GUO Chenghua, LIU Chuanlin, HOU Jianhai, FANG Hui, LIU Chutong
- 基于线粒体16S rRNA探讨渤海海区矮拟帽贝的种群生态遗传
- Population Ecological Genetics of Patelloida pygmaea Based on Mitochondrial 16S rRNA Fragment Sequences in the Bohai Sea
- 四川动物, 2017, 36(4): 386-391
- Sichuan Journal of Zoology, 2017, 36(4): 386-391
- 10.11984/j.issn.1000-7083.20170013
-
文章历史
- 收稿日期: 2017-01-12
- 接受日期: 2017-03-28
2. 海南热带海洋学院, 海南两栖爬行动物研究重点实验室, 海南三亚 572000
2. Hainan Key Laboratory for Herpetological Research, Hainan Tropical Ocean University, Sanya, Hainan Province 572000, China
环境因子在物种的进化过程中发挥着重要作用,物种的分布、行为、生理和遗传结构通常都会受到环境因子的影响,探究环境因子与生物多样性的相关性是生态遗传研究的重要内容之一(Scheiner,1993)。通常种群占据不同地理环境并呈现梯度性遗传变化,这种结果主要受自然选择与环境变迁影响(Jin & Liu,2008),遗传结构分化和自然环境变迁之间的相互作用是目前生态地理学关注的焦点。同类物种生活在不同环境下(温度、降水、日照等)的地理种群遗传多样性存在地理变异,如荒漠沙蜥Phrynocephalus przewalskii的遗传多样性与纬度、降水量显著正相关,大石鸡Alectoris magna、雉鸡甘肃亚种Phasianus colchicus strauchi的遗传多样性与温度呈显著负相关,以及同种生物因环境而产生的形态改变影响基因频率等(Michod,1981;Nevo,1981;曲江勇,刘迺发,2012)。环境对生物的影响(环境的演替、种群遗传结构的多变)伴随着表型与基因型的变化继而改变种群结构,环境促进演替使种群遗传分化趋于平衡稳定(Slatkin,1987)。
分子标记作为研究种群遗传结构和地理分化的重要工具,目前已广泛用于解决动物生态遗传等问题(Wayne & Robert,2004)。动物线粒体DNA具有分子量小、进化速度快、结构简单、表现为母系遗传等特点,非常适于动物群体遗传学和系统进化研究(Canapa et al., 2000),其中16S rRNA基因进化速率适中,适合种群水平的遗传多样性及遗传结构研究(Honda et al., 2002)。
矮拟帽贝Patelloida pygmaea隶属软体动物门Mollusca腹足纲Gastropoda原始腹足目Archaeogastropoda笠贝科Acmaeidae(Surhone et al., 2010),中国习见种类,沿岸、岛屿均有分布。矮拟帽贝遍布整个潮间带(海岸带以上到浅层潮下带),栖息附着在大型水生植物、激浪区礁岩、珊瑚藻及其他贝壳等表面,主要摄食藻类与浮游植物(Lindberg,1990;Takenori & Takashi,1993;Kirkendale & Meyer,2004;Nakano & Ozawa,2007)。目前矮拟帽贝研究主要集中在系统学和分类学等领域,包括种水平鉴定、形态变异、遗传多样性等(Nakai et al., 2006;Gonzálezwevar et al., 2010),种群系统地理研究较少。国内研究重点多为具有较大经济价值的贝类,分布广泛但无经济价值的潮间带贝类的系统地理格局受历史因素、现今环境和生态因素的影响,其遗传进化、系统地理分布格局未得到深入研究(Ni et al., 2012)。
本研究以矮拟帽贝线粒体16S rRNA部分序列,探究矮拟帽贝的生态遗传,揭示种群遗传多样性随环境因子的变异规律,为中国矮拟帽贝种群遗传研究提供理论依据。
1 材料与方法 1.1 样品采集、DNA提取及序列获取矮拟帽贝采样点见图 1,大陆沿岸种群包括大连、庄河、营口、锦州、葫芦岛、秦皇岛、莱州、蓬莱、烟台;岛屿种群包括长岛、砣矶岛、大钦岛、南隍城岛、北隍城岛。采集后测量个体的壳长、壳宽、壳高、壳周长、长斜边、短斜边、壁厚等;返回实验室称量壳干重、计算头端角度与尾端角度(表 1),无水乙醇保存备用。
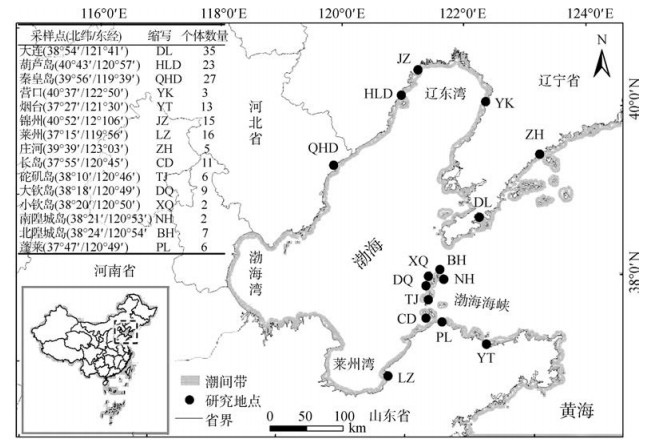
|
| 图 1 矮拟帽贝地理种群采集点 Fig. 1 The sampling sites of Patelloida pygmaea in the Bohai Sea |
| |
| 种群 Population |
壳长 Length /mm |
壳宽 Width /mm |
壳高 Height /mm |
壳周长 Perimeter /cm |
短斜边 Short hypo- tenuse/mm |
长斜边 Long hypo- tenuse/mm |
头端角度 Headend angle/° |
尾端角度 End angle/° |
干重 Dry mass/g |
壁厚Wall thickness /mm |
| 大连 | 13.63±2.67 | 10.96±2.31 | 6.14±0.97 | 3.92±0.82 | 7.77±1.40 | 10.63±1.80 | 50.91±5.40 | 34.67±5.08 | 0.25±0.13 | 0.53±0.13 |
| 葫芦岛 | 9.89±1.93 | 8.14±1.87 | 5.09±1.21 | 2.95±0.66 | 6.03±1.48 | 8.08±1.57 | 54.67±4.18 | 37.28±4.69 | 0.15±0.11 | 0.54±0.16 |
| 秦皇岛 | 11.04±1.24 | 8.82±1.05 | 5.02±0.69 | 3.26±0.35 | 6.13±0.77 | 8.89±1.15 | 53.09±4.83 | 33.65±4.44 | 0.14±0.06 | 0.51±0.12 |
| 营口 | 9.48±0.82 | 7.85±0.90 | 4.75±0.68 | 2.89±0.35 | 5.73±0.96 | 7.68±0.82 | 53.77±3.90 | 36.89±4.18 | 0.10±0.04 | 0.50±0.11 |
| 烟台 | 12.37±1.03 | 9.91±0.93 | 5.43±0.93 | 3.91±0.30 | 7.07±1.04 | 9.40±0.76 | 48.86±4.57 | 34.48±5.21 | 0.20±0.09 | 0.55±0.13 |
| 锦州 | 11.47±1.57 | 9.36±0.98 | 5.24±0.50 | 3.42±0.37 | 6.38±0.53 | 9.17±1.42 | 52.52±4.35 | 34.25±5.45 | 0.18±0.07 | 0.62±0.12 |
| 莱州 | 10.37±1.23 | 8.39±1.04 | 5.02±0.58 | 3.15±0.52 | 6.09±0.59 | 8.30±1.03 | 52.59±4.96 | 36.33±6.57 | 0.15±0.04 | 0.49±0.07 |
| 庄河 | 7.07±0.53 | 5.75±0.55 | 3.01±0.50 | 1.84±0.12 | 3.57±0.45 | 4.97±0.47 | 39.17±9.07 | 27.10±7.23 | 0.03±0.01 | 0.48±0.10 |
| 长岛 | 10.90±1.25 | 8.80±1.01 | 5.37±0.73 | 3.35±0.36 | 6.39±0.78 | 8.56±1.23 | 51.44±5.47 | 35.81±4.22 | 0.20±0.07 | 0.71±0.15 |
| 砣矶岛 | 12.16±1.22 | 9.63±1.11 | 5.14±0.68 | 3.71±0.38 | 5.98±0.88 | 9.27±1.09 | 47.25±5.61 | 28.51±5.33 | 0.18±0.07 | 0.62±0.11 |
| 大钦岛 | 13.42±1.33 | 10.66±1.32 | 6.41±0.67 | 4.13±0.44 | 7.11±0.60 | 10.34±1.96 | 49.27±10.90 | 30.97±3.74 | 0.23±0.12 | 0.54±0.09 |
| 小钦岛 | 20.97±3.21 | 17.33±2.71 | 8.43±0.96 | 6.44±0.90 | 11.17±1.66 | 15.64±1.97 | 47.22±4.74 | 31.42±2.88 | 0.58±0.27 | 0.71±0.19 |
| 南隍城岛 | 16.57±1.11 | 13.49±1.02 | 8.00±0.66 | 5.46±0.36 | 9.57±0.90 | 13.40±0.75 | 53.83±3.77 | 35.31±4.42 | 0.63±0.11 | 0.80±0.11 |
| 北隍城岛 | 14.84±1.49 | 12.05±1.37 | 7.25±0.96 | 4.39±0.44 | 8.97±1.40 | 11.55±0.78 | 51.08±2.92 | 37.06±4.95 | 0.38±0.12 | 0.78±0.12 |
| 蓬莱 | 9.92±1.43 | 8.52±1.31 | 4.52±0.65 | 3.03±0.46 | 5.74±0.93 | 7.52±1.09 | 48.91±4.20 | 34.89±2.40 | 0.12±0.06 | 0.45±0.10 |
DNA提取:DNeasy©组织 & 血液试剂盒(GTIN: 04053228006077,Lot:151046088,GIAGEN,德国)。PCR扩增:选用16S rRNA通用引物(华大基因合成),16Sar-L:5′-CGCCTGTTTATCAAAAACAT-3′;16SbrH:5′-CCGGTCTGAACTCAGATCACGT-3′(Folmer et al., 1994),PCR扩增反应体系(25 μL):12.5 μL Taq MIX(0.625 U Taq酶、0.2 mmol·L-1 dNTP、1.5 mmol·L-1 MgCl2;Lot:A3701A,TaKaRa,中国大连);0.15 μL (100 μM)上下游引物、1 μL DNA模板(80~100 ng)。PCR反应条件设置:94 ℃预变性2 min;94 ℃变性30 s,49 ℃退火40 s,72 ℃延伸50 s,35个循环;72 ℃延伸5 min。PCR结束后取4 μL于1%琼脂糖凝胶电泳检测(1×TBE、5 V/cm恒压、Goldenview染色)(琼脂糖,Lot:142075,BIOWEST,西班牙;Goldenview,Lot:OHO9RL,济南力戈科技有限公司)。PCR产物送北京六合华大公司进行ABI PRIMS 3730双向测序。
1.2 环境因子选取经度、纬度、年均温度、温度变异系数、年均降水量、降水量变异系数、年均日照时数、日照率作为环境因子(表 2),从国家气象数据共享服务网获取10个地点近30年气象数据,并计算气温、降水量变异系数(砣矶岛、大钦岛、小钦岛、北隍城岛、南隍城岛等地区气象站因缺少相关数据,未被国家气象数据共享服务网收录)。
| 地点Site | 年平均气温 Annual average temperature/℃ |
气温变异系数 Temperature variation coefficient/% |
年平均降水量 Annual average precipitation/mm |
降水量变异系数 Precipitation variation coefficient/% |
日照时数 Sunshine hours/h |
日照百分率 Sunshine percentage/% |
| 锦州 | 9.9±0.7 | 4.51 | 568.60±157.6 | 29.09 | 2 647.9 | 59.7 |
| 葫芦岛 | 9.7±0.7 | 4.00 | 579.85±147.4 | 30.72 | 2 716.8 | 61.4 |
| 营口 | 9.1±0.6 | 4.00 | 581.15±166.4 | 26.92 | 2 661.6 | 60.1 |
| 秦皇岛 | 11.1±0.6 | 4.58 | 600.21±138.2 | 25.71 | 2 536.1 | 57.2 |
| 庄河 | 9.3±0.5 | 6.59 | 736.10±156.5 | 28.62 | 2 429.7 | 54.8 |
| 大连 | 11.3±0.6 | 6.70 | 579.75±170.0 | 25.42 | 2 670.5 | 60.1 |
| 烟台 | 13.1±0.6 | 7.07 | 588.60±151.3 | 27.72 | 2 550.8 | 57.7 |
| 莱州 | 13.3±0.6 | 5.38 | 603.65±175.6 | 21.26 | 2 781.9 | 62.7 |
| 长岛 | 12.5±0.5 | 5.31 | 544.60±167.3 | 29.32 | 2 668.7 | 60.1 |
| 蓬莱 | 12.5±0.5 | 5.59 | 608.00±163.7 | 23.02 | 2 594.4 | 58.4 |
双向测序结果用Conting进行拼接,通过CLUSTAL_X 1.83进行序列比对,单倍型用DnaSP v5统计(Librado & Rozas,2009)。通过ARLEQUIN 3.5计算群体多样性指数:单倍型多样性(h)、核苷酸多样度(π)、序列平均核苷酸差异数(k)、扩张系数(Tau)、有效种群大小(θ)、种群分化系数(Fst);AMOVA分析(Excoffier & Lischer,2010);通过MEGA7计算遗传距离(D)与序列变异(S)(Kumar et al., 2016);样品所测壳长、壳宽、壳高、壳周长、短斜边、长斜边、头端角度、尾端角度、干重、壁厚等形态数据用平均数±标准差表示,形态变异采用主成分分析与相关性分析,以上统计分析均在SPSS 19.0(Arbuckle,2010)完成。
2 结果 2.1 序列变异与遗传多样性180个矮拟帽贝样本的mtDNA 16S rRNA部分序列中,共发现了264个多态位点,碱基组成为C(18.12%)、T(27.74%)、A(33.07%)、G(21.07%),转换/颠换比为1.34(缺失位点少于5%),界定32种单倍型。总体单倍型多样性为0.835 1,核苷酸多样性为0.449,核苷酸两两差异度为118.533。除庄河、蓬莱外,其他地理种群单倍型多样性较高(表 3);大陆沿岸各种群的核苷酸多样性、遗传距离与序列变异整体高于岛屿种群,总体核苷酸多样性差异较大(π大陆沿岸=0.197 8、π岛屿=0.107 3),扩张系数与种群遗传多样性表明种群存在近期扩张(总体CI 95%:Tau=426.109、θ0=0、θ1=inf);大陆沿岸种群与岛屿种群Fst=0.099(P<0.001);AMOVA分析可知变异主要来自种群内(66.93%,P<0.001)(表 4)。
| 地点Site | 单倍型数量n | 遗传距离D | 序列变异S | 单倍型多样性h | 核苷酸多样性π | 平均两两差异数k | 扩张系数Tau |
| 大连 | 4 | 0.170 | 0.219 | 0.856(0.040) | 0.179 3(0.087 7) | 109.573(48.193) | 0.004 |
| 葫芦岛 | 2 | 0.149 | 0.194 | 0.486(0.105) | 0.155 7(0.077 5) | 94.648(42.229) | — |
| 秦皇岛 | 3 | 0.114 | 0.147 | 0.821(0.043) | 0.121 3(0.060 1) | 74.006(32.990) | 6.850 |
| 营口 | 2 | 0.077 | 0.085 | 1.000(0.272) | 0.071 1(0.053 8) | 37.333(22.667) | 398.930 |
| 烟台 | 5 | 0.101 | 0.122 | 0.808(0.113) | 0.122 5(0.063 5) | 75.205(34.681) | 429.000 |
| 锦州 | 3 | 0.173 | 0.225 | 0.562(0.095) | 0.181 0(0.092 4) | 110.067(50.113) | — |
| 莱州 | 4 | 0.180 | 0.235 | 0.800(0.068) | 0.185 5(0.094 2) | 112.758(51.131) | 287.707 |
| 庄河 | 1 | — | — | — | — | — | — |
| 长岛 | 5 | 0.080 | 0.101 | 0.782(0.093) | 0.091 5(0.048 4) | 56.109(26.311) | 1.938 |
| 砣矶岛 | 2 | 0.071 | 0.079 | 0.600(0.129) | 0.099 8(0.058 3) | 61.200(30.948) | — |
| 大钦岛 | 1 | 0.047 | 0.052 | 0.917(0.073) | 0.066 0(0.036 0) | 40.472(19.453) | 2.436 |
| 小钦岛 | 1 | 0.004 | 0.004 | 1.000(0.500) | 0.003 3(0.004 1) | 2.000(1.7320 | — |
| 南城隍岛 | 2 | 0.000 | 0.000 | 1.000(0.500) | 0.001 6(0.002 3) | 1.000(1.000) | — |
| 北隍城岛 | 2 | 0.077 | 0.081 | 0.810(0.130) | 0.100 0(0.056 5) | 61.333(30.261) | 9.373 |
| 蓬莱 | 1 | — | — | — | — | — | — |
| 变异来源 Source of variation |
自由度df | 平方和 Sum of squares |
方差分量 Variance components |
变异百分比 Percentage of variation |
P值 P value |
| 种群间Among populations | 14 | 3 819.561 | 20.220 | 33.07 | <0.001 |
| 种群内Within populations | 165 | 6 751.600 | 40.919 | 66.93 | <0.001 |
所有环境因子经正态分布检验,结果显示均为正态分布,通过Person相关性检验(表 5),发现矮拟帽贝种群遗传距离、序列变异、核苷酸多样性与日照时数、日照百分率呈显著正相关(P<0.05);其他数据间无显著相关性(P>0.05)。主成分分析显示,壳长、壳宽为变异关键因子(贡献值:74.6%、18.4%),壳长、壳宽与环境因子相关性分析结果表明:壳长、壳宽与年均降水量呈显著负相关(r=-0.703/-0.728,P=0.023/0.017)。
| 经度LO | 纬度LA | 年平均 气温AAT |
气温变异系 TVC |
年平均 降水AAP |
降水量变异 系数PVC |
日照时数SU | 日照 百分率SP |
|
| 遗传距离D | 0.330 | 0.465 | 0.093 | -0.233 | -0.557 | -0.029 | 0.686* | 0.692* |
| 序列变异S | 0.299 | 0.443 | 0.101 | -0.227 | -0.537 | -0.036 | 0.681* | 0.686* |
| 单倍型多样性h | 0.362 | 0.357 | 0.156 | -0.200 | -0.654 | -0.038 | 0.483 | 0.493 |
| 核苷酸多样性π | 0.302 | 0.434 | 0.149 | -0.176 | -0.570 | -0.005 | 0.655* | 0.662* |
| 壳长 | 0.361 | 0.244 | 0.454 | -0.223 | -0.703* | -0.123 | 0.355 | 0.350 |
| 壳宽 | 0.376 | 0.247 | 0.459 | -0.194 | -0.728* | -0.155 | 0.381 | 0.375 |
| 注Note: * P<0.05. | ||||||||
对矮拟帽贝15个种群的线粒体16S rRNA基因序列研究表明,各地理种群较高的单倍型多样性(0.486~1.000) 和较低的核苷酸多样性(0.001 6~ 0.185 5),符合Grant和Bowen(1998)提出的海洋类生物较高的单倍型多样性和较低的核苷酸多样性模式,该类型种群可能经历过历史扩张事件(Bowen et al., 2001),Tau值与θ值也表明该物种存在近期历史扩张;大陆沿岸种群与岛屿种群遗传差异显著,大陆沿岸种群遗传多样性整体高于岛屿种群,且分化显著,暗示存在显著的陆岛遗传模式,大陆沿岸种群更易扩张与进化,推测大陆沿岸种群更为古老,岛屿种群基因频率受陆地基因流影响显著(Finston & Peck,1995;曲若竹等,2004)。
气候因子是影响生物进化的重要因子,其中温度和降水量则是影响陆生动物进化的2个最重要生态因子,对外温动物与内温动物均具有显著影响,且气候对不同地区的同类物种的影响程度也不同。渤海不同地理种群矮拟帽贝遗传结构研究结果表明,16S rRNA序列的遗传距离、序列变异、核苷酸多样性与日照时数、日照百分率呈显著正相关,说明日照是影响矮拟帽贝遗传多样性的重要因素,日照时数越大,遗传多样性越高。温度是影响贝类遗传变异的关键环境因子(Kim et al., 2009),海表温度作为海水热力状况的物理量,受太阳辐射影响显著(Joos & Spahni,2008),推测矮拟帽贝作为潮间带生物,栖息地以岩石海床为主,日照改变海面温度继而影响种群遗传多样性,且环境越不稳定,遗传多样性越低,与海洋无脊椎动物生态研究结果基本一致(Monari et al., 2007;Yu et al., 2009)。
形态变异主成分(壳长、壳宽)与环境因子相关性分析表明,降水量是影响形态变异的主要因素,与遗传存在差异的主要原因是自然选择导致物种基因遗传与形态变异的模式不同,即选择压力对不同的类型作用不同(Corre et al., 2002)。形态量度和其他环境因子的相关性趋势与遗传分析一致,说明表型通常是受基因控制和影响,自然选择对表型作用实际上是作用于基因或基因型,即环境因素可以通过影响个体表型进而影响基因型频率(Huang et al., 2007)。稳定环境中,较小的自然选择压力使不同的基因型被最大限度地保留,因此遗传多样性较高;在气候不稳定区域,种群保留适应多变环境的基因型而在选择压力下淘汰其他基因型,遗传多样性会降低。
| 曲江勇, 刘迺发. 2012. 环境因子对雉鸡甘肃亚种种群遗传多样性的影响[J]. 四川动物, 31(4): 518–523. |
| 曲若竹, 侯林, 吕红丽, 等. 2004. 群体遗传结构中的基因流[J]. 遗传, 26(3): 377–382. |
| Arbuckle JL. 2010. IBM SPSS Amos 19 user's guide[M]. Chicago:Amos Development Corporation. |
| Bowen BW, Bass AL, Rocha LA, et al. 2001. Phylogeography of the trumpetfishes(Aulostomus):ring species complex on a global scale[J]. Evolution, 55(5): 1029–1039. DOI:10.1554/0014-3820(2001)055[1029:POTTAR]2.0.CO;2 |
| Canapa A, Barucca M, Caputo V, et al. 2000. A molecular analysis of the systematics of three Antarctic bivalves[J]. Bolletino di Zoologia, 67(sup1): 127–132. |
| Corre VL, Roux F, Reboud X. 2002. DNA polymorphism at the FRIGIDA gene in Arabidopsis thaliana:extensive nonsynonymous variation is consistent with local selection for flowering time[J]. Molecular Biology & Evolution, 19(8): 1261–1271. |
| Excoffier L, Lischer HEL. 2010. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 10(3): 564–567. DOI:10.1111/men.2010.10.issue-3 |
| Finston TL, Peck SB. 1995. Population structure and gene flow in Stomion:a species swarm of flightless beetles of the Galápagos Islands[J]. Heredity An International Journal of Genetics, 75(4): 390–397. |
| Folmer O, Black M, Hoeh W, et al. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J]. Molecular Marine Biology & Biotechnology, 3(5): 294–299. |
| Gonzálezwevar CA, Nakano T, Cañete JI, et al. 2010. Molecular phylogeny and historical biogeography of Nacella (Patellogastropoda:Nacellidae) in the Southern Ocean[J]. Molecular Phylogenetics & Evolution, 56(1): 115–124. |
| Grant W, Bowen BW. 1998. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity, 89(5): 415–426. DOI:10.1093/jhered/89.5.415 |
| Honda M, Yasukawa Y, Ren H, et al. 2002. Phylogenetic relationships of the Asian box turtles of the genus Cuora sensu lato (Reptilia:Bataguridae) inferred from mitochondrial DNA sequences[J]. Zoologicalence, 19(11): 1305–1312. |
| Huang Z, Liu N, Luo S, et al. 2007. Ecological genetics of rusty-necklaced partridge (Alectoris magna):environmental factors and population genetic variability correlations[J]. Korean Journal of Genetics, 29(2): 115–120. |
| Jin YT, Liu NF. 2008. Ecological genetics of Phrynocephalus vlangalii on the North Tibetan (Qinghai) Plateau:correlation between environmental factors and population genetic variability[J]. Biochemical Genetics, 46(9-10): 598–604. DOI:10.1007/s10528-008-9174-6 |
| Joos F, Spahni R. 2008. Rates of change in natural and anthropogenic radiative forcing over the past 20, 000 years[J]. Proceedings of the National Academy of Sciences of the United States of America, 105(5): 1425–1430. DOI:10.1073/pnas.0707386105 |
| Kim M, Ahn IY, Cheon J, et al. 2009. Molecular cloning and thermal stress-induced expression of a pi-class glutathione S-transferase (GST) in the Antarctic bivalve Laternula elliptica[J]. Comparative Biochemistry & Physiology Part A Molecular & Integrative Physiology, 152(2): 207–213. |
| Kirkendale LA, Meyer CP. 2004. Phylogeography of the Patelloida profunda group (Gastropoda:Lottiidae):diversification in a dispersal-driven marine system[J]. Molecular Ecology, 13(9): 2749–2762. DOI:10.1111/mec.2004.13.issue-9 |
| Kumar S, Stecher G, Tamura K. 2016. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology & Evolution, 33(7): 1870. |
| Librado P, Rozas J. 2009. DnaSP v5[J]. Bioinformatics, 25(11): 1451–1452. DOI:10.1093/bioinformatics/btp187 |
| Lindberg DR. 1990. Systematics of Potamacmaea fluvia tilis (Blanford):a brackish water patellogastropod (Patelloidinae:Lottiidae)[J]. Journal of Molluscan Studies, 56(2): 309–316. DOI:10.1093/mollus/56.2.309 |
| Michod RE. 1981. Ecological genetics[J]. Science, 212(4491): 154–155. |
| Monari M, Matozzo V, Foschi J, et al. 2007. Effects of high temperatures on functional responses of haemocytes in the clam Chamelea gallina[J]. Fish & Shellfish Immunology, 22(1-2): 98–114. |
| Nakai S, Miura O, Maki M, et al. 2006. Morphological and habitat divergence in the intertidal limpet Patelloida pygmaea[J]. Marine Biology, 149(3): 515–523. DOI:10.1007/s00227-005-0241-9 |
| Nakano T, Ozawa T. 2007. Worldwide phylogeography of limpets of the order Patellogastropoda:molecular, morphological and palaeontological evidence[J]. Journal of Molluscan Studies, 73(1): 79–99. DOI:10.1093/mollus/eym001 |
| Nevo E. 1981. Genetic variation and climatic selection in the lizard Agama stellio in Israel and Sinai[J]. Theoretical and Applied Genetics, 60(6): 369–380. |
| Ni G, Li Q, Kong L, et al. 2012. Phylogeography of the bivalve Tegillarca granosa in coastal China:implications for management and conservation[J]. Marine Ecology Progress, 452: 119–130. DOI:10.3354/meps09624 |
| Scheiner SM. 1993. Genetics and evolution of phenotypic plasticity[J]. Annual Review of Ecology & Systematics, 24(1): 35–68. |
| Slatkin M. 1987. Gene flow and the geographic structure of natural populations[J]. Science, 236(4803): 787. DOI:10.1126/science.3576198 |
| Surhone LM, Tennoe MT, Henssonow SF. 2010.Patelloida pygmaea[M]. Saarbrücken:Betascript Publishing. |
| Takenori S, Takashi O. 1993. Anatomy and systematic position of Yayoiacmea, a new genus for Japanese tiny limpet "Collisella" oyamai Habe, 1955 (Gastropoda:Lottiidae)[J]. The Japanese Journal of Malacology, 52: 193–209. |
| Wayne, Robert K. 2004. Molecular markers, natural history and evolution[M]. Stamford:Sinauer Associates:25-86. |
| Yu JH, Song JH, Choi MC, et al. 2009. Effects of water temperature change on immune function in surf clams, Mactra veneriformis (Bivalvia:Mactridae)[J]. Journal of Invertebrate Pathology, 102(1): 30–35. DOI:10.1016/j.jip.2009.06.002 |
 2017, Vol. 36
2017, Vol. 36




