扩展功能
文章信息
- 李蓉, 郑雨田, 李春青, 陈艳艳, 杨振升, 陈善元, 肖蘅
- LI Rong, ZHENG Yutian, LI Chunqing, CHEN Yanyan, YANG Zhensheng, CHEN Shanyuan, XIAO Heng
- 基于线粒体COI基因序列的壮真蝎与普洱真蝎的分子鉴定
- Molecular Identification of Euscorpiops validus and E. puerensis Based on Mitochondrial COI Gene Sequences
- 四川动物, 2017, 36(2): 139-144
- Sichuan Journal of Zoology, 2017, 36(2): 139-144
- 10.11984/j.issn.1000-7083.20160305
-
文章历史
- 收稿日期: 2016-11-07
- 接受日期: 2017-01-03
壮真蝎Euscorpiops validus和普洱真蝎E. puerensis均属节肢动物门Arthropoda螯肢动物亚门Chelicerata蛛形纲Arachnida蝎目Scorpiones真蝎科Euscorpiidae真蝎属Euscorpiops,为真蝎属中等大小的物种 (Kovařík et al., 2015)。壮真蝎分布于中国云南省蒙自市;普洱真蝎俗名山螃蟹,分布于中国云南省普洱市翠云区和澜沧县 (Di et al., 2011)。壮真蝎与普洱真蝎为发现较晚的真蝎属物种,二者分布的纬度基本相同,气候相似,均属于亚热带山地季风气候。壮真蝎与普洱真蝎的传统分类主要依靠触肢螯长宽比,但二者属于昼伏夜出的物种,获取样本相对困难,在样本采集过程中会导致触肢螯、步足、尾部等重要体征脱落,用游标卡尺对触肢螯长、触肢螯宽、头胸甲长、腿节长、膝节宽等进行测量时,工作较为繁琐,且容易产生人为误差。此外,传统分类学家对壮真蝎与普洱真蝎的物种描述尚未统一,Di等 (2011)研究发现壮真蝎与普洱真蝎的触肢螯长宽比分别为2.9~3.2与2.7;杨振升 (2012)分析发现壮真蝎与普洱真蝎的触肢螯长宽比分别为2.7~3.7与3.1。因此,仅依靠传统的形态学特征对二者进行准确区分,存在一定的局限性。近年来,随着分子生物学技术的发展,分子鉴定技术为解决物种分类难题提供了有效手段。
加拿大分类学家Hebert于2003年首次提出了DNA条形码的概念,即将线粒体细胞色素氧化酶亚基Ⅰ (COI) 基因的一段长度约648 bp的片段作为物种鉴定的基础片段 (Hebert et al., 2003a)。对动物界11个门13 320个物种的研究结果显示,COI基因序列间的差异能够对动物界所有物种进行有效鉴定 (Hebert et al., 2003b)。由于DNA条形码技术无需依赖形态分类就能够对所研究的物种进行准确辨别,不仅操作简单迅速 (彭居俐等,2008),而且减少了物种鉴别的模糊性,因此被广泛应用。Yamashita和Rhoads (2013)基于线粒体COI基因对刺尾蝎属Centruroides的C. vittatus与C. pantheriensis构建系统发育树并进行聚类分析,结果同形态学分类结果吻合。Barrett和Hebert (2005)利用线粒体COI基因对168种蜘蛛及其他35种蛛形纲物种 (美洲沙漠木蝎Centruroides vittatus、土耳其斯坦叶螨Tetrany chus turkestani、美洲大革蜱Dermacentor variabilis等) 的系统发育关系和遗传距离的DNA条形码分析发现,COI基因能很好地实现对蜘蛛及其他蛛形纲物种的有效鉴定。何静超等 (2016)对小五台山10属25种蟹蛛样本进行划分及DNA条形码分子鉴定分析发现,其划分及鉴定结果同形态学分类方法得出的结果一致。Talal等 (2015)利用COI及其他基因片段对中东金蝎Scorpio maurus的S. maurus fuscus和S. maurus palmatus 2个亚种进行形态特征、系统发育及遗传差异分析发现,中东金蝎是一个复合种,与先前的分类观点相符,并证明了S. fuscus、S. kruglovi、S. palmatus、S. propinquus物种分类修订的合理性。
目前,基于线粒体COI基因对壮真蝎与普洱真蝎的DNA条形码进行物种鉴定的相关研究还未见报道,因此COI基因能否对壮真蝎与普洱真蝎进行有效鉴定还未曾得知。鉴于此,本研究首次对形态相似的壮真蝎与普洱真蝎共24个个体进行基因组总DNA提取、PCR扩增及序列测定,并对二者的遗传距离、系统发育和单倍型网络图进行分析,以期为二者及其他蝎类物种的鉴定提供分子水平的参考依据。
1 材料与方法 1.1 材料来源壮真蝎 (编号:HHMZ) 与普洱真蝎 (编号:PELC) 分别采自云南省红河州蒙自市和普洱市澜沧县的潮湿混交林石块、土堆或废墟中,总计24个样本。采样点的经纬度分别为103.0°E,22.9°N和99.9°E,22.5°N,采集后使用数码相机记录样本的形态特征,选取较为完整的样本进行形态鉴定后,保存于75%乙醇中,标本存放于云南大学生命科学学院动物学实验室。
1.2 DNA提取取样本蝎腹部肌肉组织约30 mg,将组织剪碎用ddH2O处理为细胞悬液 (处理2次),按照血液/细胞/组织基因组提取试剂盒 (TIANGEN,北京) 说明书的步骤提取样本蝎基因组总DNA,提取后,使用NanoDrop 2000超微量分光光度计及琼脂糖凝胶电泳检测技术进行DNA浓度和纯度检测,检测后将提取的总DNA置于-20 ℃保存备用。
1.3 PCR扩增及序列测定PCR扩增所用引物为COI基因通用引物LCO1490(5'-GGTCAACAAATCATAAAGATATTGG-3'),HCO2198(5'-TAAACTTCAGGGTGACCAAAAAATCA-3') (Folmer et al., 1994),所有引物均由上海捷瑞生物工程技术有限公司合成。PCR扩增体系为50 μL,包括10×Buffer 5 μL,25 mM MgCl2 5 μL,5 U/μL Taq DNA聚合酶0.15 μL,2.5 mM dNTP4 μL,10 μM正反引物各1 μL,DNA模板2 μL,ddH2O 31.85 μL。PCR反应程序为:95 ℃预变性4 min;95 ℃变性1 min,40 ℃退火1 min,72 ℃延伸1 min,35个循环;循环结束后,72 ℃延伸5 min。每次PCR扩增均设置阴性对照,以确保实验过程不受外源DNA的污染。扩增结束后,1%琼脂糖凝胶电泳检测。将检测合格的PCR产物送北京六合华大基因科技有限公司广州分公司进行双向测序。
1.4 数据分析将2种蝎获得的测序峰图利用DNASTAR (Burland,2000) 中的SeqMan (Swindell & Plasterer,1997) 进行正反链校对和编辑,手动去除序列两端的引物区,获得有效片段的样本序列,用MegAlign (Clewley & Arnold,1997) 进行排序后,利用Clustal X (Jeanmougin et al., 1998) 进行多序列比对,采用DnaSP v5(Librado & Rozas,2009) 计算信息位点、变异位点、单倍型数,通过BioEdit (Hall,1999) 计算单倍型序列间的相似度,利用MEGA 6.06(Tamura et al., 2013) 分析碱基组成、单倍型间变异位点,并基于Kimura-2-parameter (K2P) 模型进行遗传距离分析及采用邻接法 (Neighbor-Joining) 构建系统发育树,系统发育树的节点支持率采用自举值进行估计,重复检测1 000次 (Felsenstein,1985)。单倍型网络图采用POPART (Leigh & Bryant,2015) 构建。
2 结果与分析 2.1 基于COI基因的序列分析经COI基因通用引物PCR扩增、序列测定,共获得24条DNA序列,经人工校对后,获得有效序列长度均为660 bp,所测COI基因序列中未发现碱基的插入或缺失。壮真蝎15个样本的A、T、C、G平均含量分别为21.5%、45.4%、14.0%、19.1%,其中A+T含量 (66.9%) 大于G+C含量 (33.1%);15条序列共检测到1个信息位点,占序列总长度的0.15%,4个变异位点,4个单倍型 (HAP1~HAP4),单倍型之间的相似度为99.3%~99.8%。普洱真蝎9个样本的A、T、C、G平均含量分别为23.1%、42.5%、15.2%、19.2%,其中A+T含量 (65.6%) 大于G+C含量 (34.4%),9条序列共检测到2个信息位点,占序列总长度的0.30%,3个变异位点,4个单倍型 (HAP5~HAP8),单倍型之间的相似度为99.6%~99.8%。壮真蝎与普洱真蝎种间相似度为90.1%~90.6%(表 1),2种蝎共8个单倍型中的变异位点数为68,其中61个位点为二者的稳定差异位点,可作为壮真蝎与普洱真蝎物种鉴定的诊断位点 (图 1)。
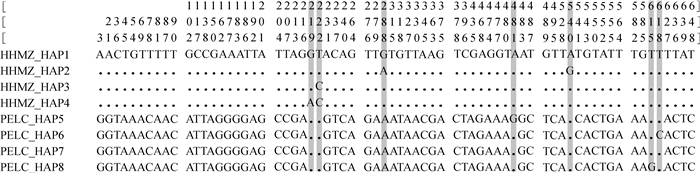
|
| 图 1 基于COI基因序列的壮真蝎与普洱真蝎单倍型之间的变异位点 Fig. 1 Variable sites of haplotypes from Euscorpiops validus and E. puerensis based on COI gene sequences HAP1~HAP4为壮真蝎的单倍型,HAP5~HAP8为普洱真蝎的单倍型;阴影部分为非稳定变异位点。 HAP1-HAP4 are the haplotypes of E. validus, HAP5-HAP8 are the haplotypes of E. puerensis; the shaded regions are non-stable variable sites. |
| |
| HAP1 | HAP2 | HAP3 | HAP4 | HAP5 | HAP6 | HAP7 | HAP8 | |
| HAP1 | 0.003 | 0.002 | 0.003 | 0.103 | 0.103 | 0.101 | 0.103 | |
| HAP2 | 0.996 | 0.005 | 0.006 | 0.103 | 0.103 | 0.101 | 0.103 | |
| HAP3 | 0.998 | 0.995 | 0.002 | 0.105 | 0.105 | 0.103 | 0.105 | |
| HAP4 | 0.996 | 0.993 | 0.998 | 0.107 | 0.107 | 0.105 | 0.106 | |
| HAP5 | 0.904 | 0.904 | 0.903 | 0.901 | 0.003 | 0.002 | 0.002 | |
| HAP6 | 0.904 | 0.904 | 0.903 | 0.901 | 0.996 | 0.002 | 0.003 | |
| HAP7 | 0.906 | 0.906 | 0.904 | 0.903 | 0.998 | 0.998 | 0.002 | |
| HAP8 | 0.904 | 0.904 | 0.903 | 0.901 | 0.996 | 0.996 | 0.998 | |
| 注:对角线上方为单倍型间遗传距离,对角线下方为单倍型间相似度;HAP1~HAP4为壮真蝎的单倍型,HAP5~HAP8为普洱真蝎的单倍型。 Notes:The genetic distances between haplotypes are above the diagonal,and the similarities between haplotypes are below the diagonal; HAP1-HAP4 are the haplotypes of E. validus,HAP5-HAP8 are the haplotypes of E. puerensis. | ||||||||
基于COI基因,利用MEGA 6.06中的双参数模型计算壮真蝎与普洱真蝎的遗传距离。结果显示,壮真蝎4个单倍型 (HAP1~HAP4) 间的遗传距离为0.002~0.006,平均遗传距离为0.004 0;普洱真蝎4个单倍型 (HAP5~HAP8) 间的遗传距离为0.002~0.003,平均遗传距离为0.002 3;壮真蝎与普洱真蝎单倍型间的遗传距离为0.101~0.107,种间平均遗传距离为0.103 9(表 1)。壮真蝎与普洱真蝎种间平均遗传距离显著高于种内平均遗传距离,为种内平均遗传距离的34.6倍。
2.3 系统发育关系与单倍型网络图分析以E. flavicaudis (GenBank登录号:KF548117、JN018212、HM418267) 的3条序列为外群,结合本研究的壮真蝎与普洱真蝎单倍型序列,采用邻接法构建系统发育树 (图 2)。结果显示,二者的单倍型序列都各自聚为独立的单系枝,且具有较高的分枝自举值 (100%)。为进一步了解二者单倍型之间的关系,利用POPART的中间结合法 (Median-joining) 构建了2种蝎的单倍型网络图 (图 3)。结果显示,8个单倍型可分为2大类群,普洱真蝎的单倍型为聚类A,壮真蝎的单倍型为聚类B,无共享单倍型。且壮真蝎的单倍型HAP2与普洱真蝎的单倍型HAP7之间的突变步数高达62步。
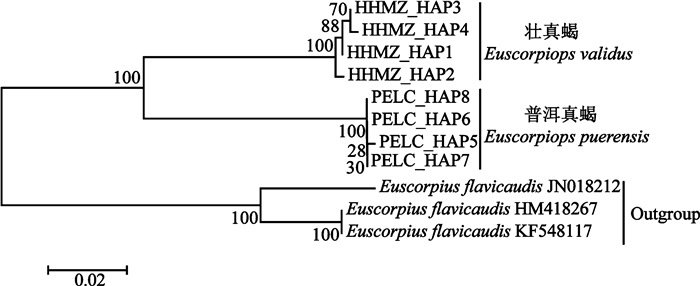
|
| 图 2 基于COI基因序列构建的壮真蝎和普洱真蝎的系统发育树 Fig. 2 Phylogenetic tree of Euscorpiops validus and E. puerensis based on COI gene sequences using Neighbor-Joining method |
| |
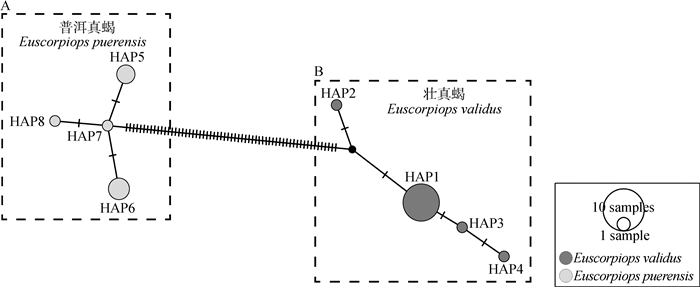
|
| 图 3 基于COI基因序列构建的壮真蝎与普洱真蝎的单倍型网络图 Fig. 3 The haplotype network of Euscorpiops validus and E. puerensis based on COI gene sequences |
| |
线粒体基因序列中稳定的变异位点或核苷酸诊断位点可作为物种划分的鉴别位点。朱振华等 (2005)基于线粒体Cyt b基因对6种果实蝇进行序列比对发现,基因片段的30个变异位点具有较高的稳定性,可作为果实蝇物种分子鉴定的依据。王康等 (2016)通过比较沙果小食心虫Grapholita dimorpha Komai和梨小食心虫G. molesta Busck的线粒体COI基因与CO Ⅱ基因序列发现,COI基因序列中存在30个稳定变异位点,CO Ⅱ基因序列中存在26个稳定变异位点,并以此作为2种食心虫鉴定的参考依据。Li等 (2011)对棉铃虫Helicoverpa armigera和烟夜蛾H. assulta COI基因序列分析,发现独特的17对核苷酸诊断位点可对二者进行区分。本研究发现,壮真蝎与普洱真蝎COI基因序列间存在61个稳定差异核苷酸位点,这些变异位点可作为壮真蝎与普洱真蝎分子鉴定的依据。
线粒体DNA序列的差异可作为不同物种划分的依据 (Wiens & Penkrot,2002;Hebert et al., 2003a)。例如,Avise和Walker (1999)基于线粒体Cyt b基因序列对252种脊椎动物研究发现,90%的脊椎动物显示出超过2%的序列差异。Hebert等 (2003a)基于COI基因序列对200种鳞翅目Lepidoptera昆虫分析发现,COI基因3%的序列差异可用于鳞翅目昆虫的物种鉴定。Barrett和Hebert (2005)对203种蛛形纲物种COI基因序列研究发现,COI基因4%的序列差异可作为鉴定不同物种的标准。本研究中壮真蝎与普洱真蝎的种间平均序列差异为9.6%,同Barrett和Hebert (2005)的研究结果一致。
种内、种间遗传距离是进行物种鉴别的主要标准。本研究基于COI基因对壮真蝎和普洱真蝎的遗传距离进行分析,发现两者的种内平均遗传距离分别为0.004 0、0.002 3,同Hebert等 (2003b)对动物界11个动物门13 320个物种进行分析得出的种内遗传距离大多小于1%,很少大于2%的结论相符。壮真蝎与普洱真蝎的种间平均遗传距离 (0.103 9) 显著高于种内平均遗传距离 (0.003 0),为种内平均遗传距离的34.6倍,符合种间平均遗传距离大于种内平均遗传距离10倍以上的原理 (Hebert et al., 2004),且种间遗传距离和种内遗传距离无重叠区域,符合对物种进行有效性鉴定的检验标准 (Hebert et al., 2003a;Aliabadian et al., 2009)。因此,基于线粒体COI基因的壮真蝎与普洱真蝎的遗传距离分析表明,COI基因可用于2种蝎的分子鉴定。
系统发育分析显示,壮真蝎与普洱真蝎的单倍型序列都各自聚为单系枝,且具有较高的分枝自举值 (100%),与遗传距离分析得出的结论一致。单倍型网络图分析发现所有单倍型明显分为2大类群,整个单倍型网络图无共享单倍型,且种间的单倍型突变步数高达62步。以上鉴定结果同传统分类结论吻合 (Di et al., 2010a, 2010b)。综上所述,基于线粒体COI基因可实现对壮真蝎与普洱真蝎物种的分子鉴定。
本研究首次利用COI基因的部分片段序列对真蝎属的壮真蝎与普洱真蝎进行分子鉴定,初步证明了DNA条形码对壮真蝎与普洱真蝎物种鉴定的有效性。鉴于本实验的研究利用了COI基因的部分序列片段,与全长序列相比,包含的信息量有限,且只运用了线粒体COI基因进行了分析,难免存在一定的局限性。为此,今后需要考虑全长序列的分析及结合其他核基因或运用下一代测序技术开发更加稳定准确的分子鉴定的基因组标记,以此来验证采用分子手段对壮真蝎与普洱真蝎物种鉴定的有效性。
致谢: 感谢罗康、罗正荣、唐天强、黄顺福在样品采集中给予的支持和帮助,感谢杨阳、沈灵、饶峻瑜在实验技术方面给予的指导与帮助。| 何静超, 胡岚岚, 郭晨辉, 等. 2016. 小五台山蟹蛛DNA条形码分子鉴定[J]. 河北大学学报 (自然科学版), 36(3): 286–292. |
| 彭居俐, 王绪桢, 何舜平. 2008. DNA条形码技术的研究进展及其应用[J]. 水生生物学报, 32(6): 916–919. |
| 王康, 李玉婷, 郑燕, 等. 2016. 基于线粒体COI和COⅡ基因的沙果小食心虫与梨小食心虫的分子鉴定[J]. 西北农林科技大学学报 (自然科学版), 44(2): 156–164. |
| 杨振升. 2012. 云南省蝎类资源分布、部分种类再记述和形态差异研究[D]. 昆明: 云南大学. |
| 朱振华, 叶辉, 张智英. 2005. 基于mtDNA Cytb的六种果实蝇的分子鉴定 (双翅目:实蝇科)[J]. 昆虫学报, 48(3): 386–390. |
| Aliabadian M, Kaboli M, Nijman V, et al. 2009. Molecular identification of birds:performance of distance-based DNA barcoding in three genes to delimit parapatric species[J]. PLoS ONE, 4(1): e4119. DOI:10.1371/journal.pone.0004119 |
| Avise JC, Walker D. 1999. Species realities and numbers in sexual vertebrates:perspectives from an asexually transmitted genome[J]. Proceedings of the National Academy of Sciences, 96(3): 992–995. DOI:10.1073/pnas.96.3.992 |
| Barrett RDH, Hebert PDN. 2005. Identifying spiders through DNA barcodes[J]. Canadian Journal of Zoology, 83(3): 481–491. DOI:10.1139/z05-024 |
| Burland TG. 2000. DNASTAR's Lasergene sequence analysis software[J]. Methods in Molecular Biology, 132: 71–91. |
| Clewley JP, Arnold C. 1997. MEGALIGN. The multiple alignment module of LASERGENE[J]. Methods in Molecular Biology, 70(70): 119–129. |
| Di Z, He Y, Wu Y, et al. 2011. The scorpions of Yunnan (China):updated identification key, new record and redescriptions of Euscorpiops kubani and E. shidian (Arachnida, Scorpiones)[J]. Zookeys, 30(82): 1–33. |
| Di ZY, Cao ZJ, Wu YL, et al. 2010a. A new species of the genus Euscorpiops Vachon, 1980 (Scorpiones:Euscorpiidae, Scorpiopinae) from Yunnan, China[J]. Zootaxa, 2361(1): 39–48. |
| Di ZY, Wu YL, Cao ZJ, et al. 2010b. A catalogue of the genus Euscorpiops Vachon, 1980 (Scorpiones:Euscorpiidae, Scorpiopinae) from China, with description of a new species[J]. Zootaxa, 2361(2477): 49–61. |
| Felsenstein J. 1985. Confidence limits on phylogenies:an approach using the bootstrap[J]. Evolution, 39(4): 783–791. DOI:10.2307/2408678 |
| Folmer O, Black M, Hoeh W, et al. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit Ⅰ from diverse metazoan invertebrates[J]. Molecular Marine Biology & Biotechnology, 3(5): 294–299. |
| Hall TA. 1999. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 41(41): 95–98. |
| Hebert PDN, Cywinska A, Ball SL, et al. 2003a. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society B Biological Sciences, 270(1512): 313–321. DOI:10.1098/rspb.2002.2218 |
| Hebert PDN, Ratnasingham S, de Waard JR. 2003b. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings of the Royal Society B Biological Sciences, 270(suppl_1): S96–S99. DOI:10.1098/rsbl.2003.0025 |
| Hebert PDN, Stoeckle MY, Zemlak TS, et al. 2004. Identification of birds through DNA barcodes[J]. PLoS Biology, 2(10): e312. DOI:10.1371/journal.pbio.0020312 |
| Jeanmougin F, Thompson JD, Gouy M, et al. 1998. Multiple sequence alignment with Clustal X[J]. Trends in Biochemical Sciences, 23(10): 403–405. DOI:10.1016/S0968-0004(98)01285-7 |
| Kovařík F, KošuličO, Št'áhlavskyý F, et al. 2015. Two new species of Euscorpiops Vachon, 1980 from Thailand and Myanmar (Scorpiones:Euscorpiidae:Scorpiopinae)[J]. Annales Zoologici, 65(1): 109–122. DOI:10.3161/00034541ANZ2015.65.1.009 |
| Leigh JW, Bryant D. 2015. Popart:full-feature software for haplotype network construction[J]. Methods in Ecology & Evolution, 6(9): 1110–1116. |
| Li QQ, Li DY, Ye H, et al. 2011. Using COI gene sequence to barcode two morphologically alike species:the cotton bollworm and the oriental tobacco budworm (Lepidoptera:Noctuidae)[J]. Molecular Biology Reports, 38(8): 5107–5113. DOI:10.1007/s11033-010-0658-1 |
| Librado P, Rozas J. 2009. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 25(11): 1451–1452. DOI:10.1093/bioinformatics/btp187 |
| Swindell SR, Plasterer TN. 1997. SEQMAN. Contig assembly[J]. Methods in Molecular Biology, 70(70): 75–89. |
| Talal S, Tesler I, Sivan J, et al. 2015. Scorpion speciation in the Holy Land:multilocus phylogeography corroborates diagnostic differences in morphology and burrowing behavior among Scorpio subspecies and justifies recognition as phylogenetic, ecological and biological species[J]. Molecular Phylogenetics & Evolution, 91: 226–237. |
| Tamura K, Stecher G, Peterson D, et al. 2013. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 30(12): 2725–2729. DOI:10.1093/molbev/mst197 |
| Wiens JJ, Penkrot TA. 2002. Delimiting species using DNA and morphological variation and discordant species limits in spiny lizards (Sceloporus)[J]. Systematic Biology, 51(51): 69–91. |
| Yamashita T, Rhoads DD. 2013. Species delimitation and morphological divergence in the scorpion Centruroides vittatus (Say, 1821):insights from phylogeography[J]. PLoS ONE, 8(7): e68282. DOI:10.1371/journal.pone.0068282 |
 2017, Vol. 36
2017, Vol. 36




