扩展功能
文章信息
- 伍仕鑫, 徐传飞, 孙磊, 赵旺生, 蔡欣
- WU Shixin, XU Chuanfei, SUN Lei, ZHAO Wangsheng, CAI Xin
- 长链非编码RNA在哺乳动物精子发生中的功能研究进展
- Progresses of Research on the Function of LncRNA Involved in Mammalian Spermatogenesis
- 四川动物, 2016, 36(1): 114-120
- Sichuan Journal of Zoology, 2016, 36(1): 114-120
- 10.11984/j.issn.1000-7083.20160171
-
文章历史
- 收稿日期: 2016-06-22
- 接受日期: 2016-10-19
长链非编码RNA (lncRNA)占非编码RNA (ncRNA)的80%,多由RNA聚合酶Ⅱ转录形成(Gong & Maqual,2011),其碱基组成从200 nt到100 000 nt不等(Brosnan & Voinnet,2009)。LncRNA位于细胞核内或胞浆中,转录水平低于蛋白质编码基因,不参与蛋白质编码。其以RNA形式在多个层面(表观遗传调控、转录调控以及转录后调控等)上调控基因的表达水平(Mercer et al., 2009)。然而,越来越多的证据表明,lncRNAs不是基因组的“暗物质”,它们在各种生物过程中发挥重要的监管作用,包括X染色体失活、基因组印记、细胞分化、细胞凋亡、干细胞多能性、大脑发育、视网膜发育、核运输、热休克反应以及基因组重排(Mattick, 2011a, 2011b)。在对若干来源于多个小鼠组织的全长cDNA文库进行分析后发现,大量的lncRNA基因呈现出组织特异性的表达(Ravasi et al., 2006),随后有研究者利用原位杂交技术在小鼠脑组织中鉴定出了大量表达水平与特定细胞类型相关的lncRNAs (Mercer et al., 2008)。很多lncRNAs在染色体上具有特异的基因座位,表明它们的转录受到动态调控,这种调控具有明显的细胞特异性(Guttman et al., 2009)。
目前对雄性生殖细胞发育中lncRNA的研究日渐增加,但是在精子发生中只有少数lncRNAs,如Tsx、HongrES2、Mrhl和Spga-lncRNA被报道(Ganesan & Rao,2008)。LncRNAs的表达谱具有组织或细胞特异性(Rasmussen et al., 2000;Park et al., 2002;Prasanth et al., 2005;Pandey et al., 2008),而这在2005年于模式动物中才有以组织和细胞特异性方式表达的大量lncRNAs被报道(Inagaki et al., 2005),因此在雄性生殖细胞发育中只鉴定出少量的lncRNAs (Ganesan & Rao,2008)。
1 LncRNA来源及其作用机制 1.1 LncRNA的起源大多数ncRNA受到低程度的进化限制,只有小部分在不同物种之间表现出序列保守性(Kutter et al., 2012)。与蛋白质编码基因不同,ncRNA基因不形成大的同源家族。除转位因子外,ncRNA的序列都不具有相似性。LncRNA有以下几种产生方式:(1)一个蛋白质编码基因发生一次框插入,转变为一个与先前编码序列合并的有功能的lncRNA。参与细胞发育过程中X染色体失活的X染色体特异性失活转录物(X inactivation-specific transcript,Xist)是这种生成方式的有力佐证(柏庆然,宋旭,2010);(2)在染色质重排之后,导致2个原本距离较远的非转录片段串联到一起,产生一个含多个外显子的ncRNA。犬科Canidae动物睾丸中有一种跨越2个区域并被数十兆核苷酸分开的ncRNA,而在其他真兽类哺乳动物中仍保留有祖先染色体的结构,这也是对染色体重排后产生ncRNA这一推测的有力佐证;(3)非编码基因通过反转录转座作用复制,产生一个有功能的非编码反义基因,或者产生一个没有功能的非编码反转录假基因;(4) ncRNA内部某段序列的重复复制,产生了具有相邻重复序列的lncRNA。如在Kcnq1ot1和Xist转录本的5’端发现了重复序列;(5)转座元件插入基因组中产生一个有功能的lncRNA。如RNABC1(brain cytoplasmic RNA 1)和BC200(brain cytoplasmic RNA 200-nucleotide)这2类ncRNA就源于不同的转座元件(Ponting et al., 2009)。
1.2 LncRNA的基本功能LncRNA可在多个层面调控基因的表达水平,主要在表观遗传调控、转录调控及转录后调控3个层面上调控基因的表达。随着对lncRNA研究的深入,许多新的与基因调控相关的lncRNA被发现,而多年的研究也已经证实lncRNA与表观遗传调控的关系十分密切(Probst et al., 2009)。在表观遗传水平,lncRNA可通过DNA甲基化、组蛋白修饰和染色质重构等机制影响基因的表达(Shi et al., 2013)。分别与基因组印记和X染色体失活相关的H19和Xist则是参与表观遗传调控中最先被研究的lncRNAs (Yang & Kuroda,2007)。转录自同源异型框基因C (homeobox C,HOXC)位点的同源异型框基因反义基因间RNA (HOX antisense intergenic RNA,HOTAIR)能够通过反式调节作用募集多梳抑制复合体2(polycomb repressive complex 2,PRC2)并将其定位到HOXD位点,进而诱导HOXD基因座上长达40 kb的转录本发生表观遗传沉默(Jonkers et al., 2008)。在转录水平,lncRNA可以通过干扰邻近基因的表达、与DNA碱基互补形成复合物、与转录因子或RNA聚合酶Ⅱ相互作用以及作为内源性竞争性RNA (competing endogenous RNA,ceRNA)调控靶基因的转录(Bonasio & Shiekhattar,2014;Ma et al., 2014)。如DNA损伤信号诱导人类的细胞周期蛋白D1(cyclin D1,Ccnd1)启动子上游一段lncRNA的表达,进而调节RNA结合蛋白TLS的活性,随后TLS抑制环磷腺苷效应元件结合蛋白(cAMP responsive element-binding protein,CREB)的表达,使Ccnd1基因的表达沉默(Wang et al., 2008)。在转录后水平,lncRNA可与microRNA (miRNA)结合抑制miRNA的功能,或者调控mRNA的可变剪接,也可与mRNA互补配对形成双链RNA,改变其稳定性(Yoon et al., 2013)。如具有MRE转录本的lncRNA在人和鼠的骨骼肌分化过程中可以竞争结合miRNA并调控miRNA及靶基因的表达(Cesana et al., 2011)。
2 LncRNA在精子发生中的调控功能精子发生是一个复杂而精巧的过程,精原干细胞(spermatogonial stem cells,SSCs)在曲细精管基底膜增殖并连续分化成各种生精细胞,包括精原细胞、精母细胞、精子细胞和释放到曲细精管管腔的成熟精子。成熟精子的形成依赖于精确的转录程序,并涉及复杂的基因表达调控过程,包括表观遗传调控、转录调控以及转录后调控等,其中任何一个环节出错都可能导致生精障碍。生精障碍是雄性不育的主要原因,目前雄性不育占全世界诊断不孕病例的一半以上,所以哺乳动物成功的精子发生在生殖发育以及物种延续中具有重要作用。最近发现的lncRNAs作为正常机体和疾病发生的关键调节因子,可为解析雄性生殖细胞发育的分子调控机制提供新的线索。
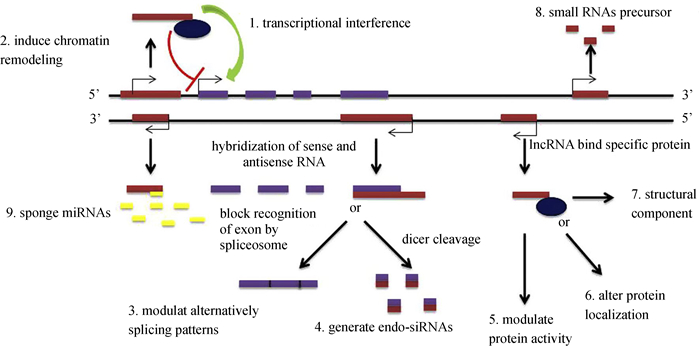
|
| 图 1 LncRNAs的细胞生物学功能示意图(Shi et al., 2013) Fig. 1 Paradigm for the cellular functions of lncRNAs (Shi et al., 2013) |
| |
LncRNA作为新一类重要调控因子,在精子发生过程中的相关研究较少,目前只有少数被功能注释和深入研究,在雄性生殖细胞发育中仍有许多潜在的lncRNAs有待鉴定。一些lncRNAs由高通量的方法鉴定,其他的由从头测序(de novo sequencing)及其功能注释鉴定。在精子发生中这些已被鉴定的lncRNA表达模式在图 2中予以阐明(Luk et al., 2014)。对这些lncRNA调控作用的解析将为精子发生相关的新lncRNA的发现和鉴定提供参考。
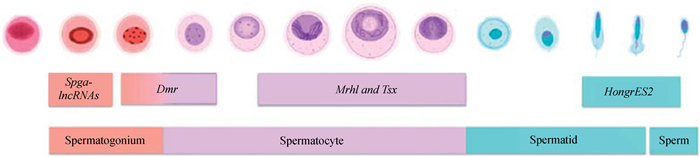
|
| 图 2 在精子发生中的功能特性lncRNAs,其表达模式与特定的雄性生殖细胞一同标示(Luk et al., 2014) Fig. 2 Functionally characterized lncRNAs in spermatogenesis, whose expression pattern is indicated alongside with the specific male germ cell (Luk et al., 2014) |
| |
原始生殖细胞随着雄性动物的出生转化为生殖母细胞,在迁移到生精小管基膜部后分化为SSCs。SSCs位于睾丸曲细精管靠近基底膜处,是雄性生殖腺内的一类原始精原细胞,其既能自我更新、维持自身群体恒定,又能定向分化产生精母细胞,SSCs代表了雄性生殖干细胞(male germline stem cells,mGSCs)有分化为各种雄性生殖细胞的能力。精子发生的整体过程非常复杂,涉及很多基因的时序性表达调控,而lncRNAs能够调控基因的表达,具有非常广泛的生物学功能。目前国内外研究主要集中在SSCs的分子标记、体外培养、增殖分化信号通路、移植和多能性等方面;到目前为止,关于lncRNA对精原干细胞增殖和分化的研究还较少。近几年,lncRNA高通量测序及其数据库的建立为研究lncRNA的功能提供了很好的平台,其中lncRNA数据库提供了目前已研究的、在真核动物体内发挥功能的lncRNAs的综合信息。
自我更新和分化是干细胞的2个关键特性,而这些特性是通过转录因子Oct4、Sox2和Nanog等的精确转录调节机制维持。最近发现通过调节这些干性维持基因的表达可以阐明几个lncRNAs在维持干细胞干性中的作用。通过全基因组筛选结合小鼠胚胎干细胞(mouse embryonic stem cells,mESCs)全转录组数据分析以及mESCs转录因子Oct4和Nanog基因组位点的染色质免疫共沉淀定位,在Oct4和Nanog活跃的结合位点附近鉴定出4种高保守的lncRNA编码基因,即AK 005651、AK028326、AK043754和AK141205,其中AK028326和AK141205直接与Oct4和Nanog的调控相关。这些lncRNA转录本被敲除或过表达会导致Oct 4、Nanog和其他细胞谱系特定基因的表达水平以及mESCs多向分化潜能的改变(Sheik et al., 2010)。考虑到lncRNAs在ESCs自我更新、多向分化潜能和分化过程中普遍发挥作用,而且ESCs和GSCs中lncRNAs分子水平的作用非常相似,可以预测在GSCs发生分化并获得ESC样干性特征的过程中可能也受到lncRNAs的调控。
AK011429是一个被确认为细胞周期蛋白D2(cyclin D2,Ccnd2)的外显子正义转录本。AK011429与Ccnd2的表达谱呈正相关。而Ccnd2在精原细胞中高度表达,并在SSCs自我更新中扮演着一个重要的角色(Lee et al., 2009)。AK080917是一个在成年睾丸中下调的lncRNA,被认定为锌指蛋白的内含子4和5与Zbtb 16基因之间序列的正义转录本。Zbtb 16在精原干细胞中高水平表达,是SSCs干性维持所需的关键基因(Costoya et al., 2004)。AK080917和Zbtb16之间表达的正相关性表明两者的功能或调控可能具有相关性。AK052984是在成年鼠睾丸中高度表达的lncRNA,来源于Hoxc 4和Smug1基因间的序列。Hoxc4对于精原干细胞自我更新是必需的,且在新生儿睾丸中高度表达(Schmidt et al., 2009)。AK052984的表达与Hoxc 4的表达呈负相关,因此,可以推测AK052984可能会影响Hoxc4的表达。
2.2 LncRNA对精原细胞分化的影响有研究者通过整合SAGE数据(Lee et al., 2006, 2009)和全基因组嵌合微阵列数据(Lee et al., 2012a)来鉴定lncRNA,此种方法已经用于研究3种关键生精细胞(A型精原细胞、粗线期精母细胞、圆形精子细胞)转录组差异表达的编码基因。Lee等(2012b)利用上述方法分别在A型精原细胞、粗线期精母细胞、圆形精子细胞中鉴定出特异性表达的50个、35个和24个单外显子lncRNAs,通过qPCR、northern blot和RACEs进一步验证了精原细胞中2个名为Spga-lncRNA1和Spga-lncRNA2的特异性lncRNA在体外细胞模型中表现出的明显分化抑制作用,表明它们可能对于精原细胞干性的维持具有重要的作用。
与Dsx-and Mab3-related transcription factor-1(Dmrt1)有关的基因Dmr (Dmrt 1 -related)是睾丸特异功能lncRNA中一个有代表性的lncRNA。它在无意中被发现,研究者试图克隆Dmrt 1基因,却在其3'链中间区段发现了一个意想不到的具有特定序列的新型RNA异构体(Zhang et al., 2010)。通过基因定位表明此神秘的异构体实际是由5号染色体上一个基因编码的,因此命名为Dmr,而Dmrt 1是在19号染色体上的基因。换句话说,Dmr能够形成一个反式剪接RNA异构体Dmrt 1。这种嵌合mRNA的形成干扰Dmrt 1的编码区和取代Dmrt1的3’-UTR,均导致正常Dmrt1蛋白质的下调。由于Dmrt1蛋白质是一个重要的转录因子,可以通过上调转录因子spermatogenesis-and oogenesis-specific basic helix-loop-helix 1(Sohlh1)以维持支持因子的周期性表达来促进精原细胞发育,也可以通过抑制维甲酸(retinoic acid,RA)依赖性的stimulated by retinoic acid gene 8(Stra8)因子的表达,从而间接抑制RA的转录以阻止精原细胞过早减数分裂(Ottolenghi et al., 2000, Agbor et al., 2013),此外,通过特异性敲除支持细胞中的Dmrt 1基因,发现Dmrt1基因能够通过影响支持细胞的成熟而影响精子发生(Agbor et al., 2013)。所以Drm对Dmrt 1基因的抑制也可能参与到生殖细胞发育的有丝分裂和减数分裂之间的转换中。
当Nishant等(2004)在研究小鼠8号染色体新型减数分裂重组热点区域内一个17.2 kb的片段时,鉴定出减数分裂重组热点基因座(meiotic recombination hot spot locus,Mrhl) RNA。Mrhl是一个位于细胞核的2.4 kb单外显子lncRNA,由小鼠基因组编码,并在睾丸、肝、脾、肾脏中表达(Nishant et al., 2004)。随后的研究表明Mrhl RNA可能通过2种分子机制调控精子发生。一是通过Drosha分裂Mrhl形成一个80 nt的RNA中间体。证据表明这些RNAs都位于GC1精原细胞系的细胞核内,暗示其与染色质可能存在互作的关系(Ganesan & Rao,2008)。除此之外,Mrhl RNA是第一个显示调节Wnt信号的lncRNA,而Wnt信号在调节哺乳动物精子发生过程中起着非常重要的作用(Kerr et al., 2014)。Mrhl可以通过与p68互作在小鼠精原细胞的Wnt信号中发挥负调控作用。在GC-1 Spg细胞系中降低Mrhl RNA的表达,能导致一些与细胞黏附、细胞信号转导和细胞发育及分化相关基因表达的紊乱,这其中大多是Wnt信号通路中的基因(Arun et al., 2012)。然而,这个lncRNA对精子发生的调控作用仍需要通过基因敲除动物模型来进一步研究。
2.3 LncRNA对精母细胞减数分裂的影响减数分裂作为雄性生殖细胞增殖过程中的一种特殊分裂方式,在精子发生中受到调节因子的严格调控。在减数分裂期间,染色体会经历包装、配对和重组等一系列的结构变化,而染色体域经由甲基化的异染色质化是这些过程的必要条件。G9A是一个在体细胞常染色质区域参与赖氨酸残基9单一二甲基化作用的组蛋白甲基转移酶,已被证明与雄性减数分裂有关。敲除G9a基因后会导致胚胎在妊娠中期死亡,而且G9a基因敲除小鼠的雄性不育可归因于初级精母细胞在减数分裂粗线期发生的阻滞(Tachibana et al., 2007)。如Kcnq1ot1和Air等lncRNAs被发现与G9a存在交互作用,通过染色质修饰机制来调节多个基因的转录沉默。Kcnq1ot1是小鼠胎盘中一个源于父本等位基因表达的90 kb lncRNA,与G9a和PRC2互作,通过招募多聚复合物有效地在其转录位点形成顺式抑制结构域(Pandey et al., 2008)。Air转录单位在小鼠Igf2r (insulin-like growth factor 2 receptor)基因第二内含子启动,并招募G9a到其目标启动子使基因沉默(Nagano et al., 2008)。LncRNA和G9a在小鼠生精细胞减数分裂过程中的互作机制是基于染色质重塑器(chromatin remodelling machinery)的募集与转录抑制。一些已知单倍体雄性生殖细胞的lncRNAs在成年睾丸组织中上调表达,例如Aldoart2、Speer9-ps1;而一些印记lncRNAs下调表达,如H19、Meg3和Airn等。早期的研究发现著名的父本印记基因H19的甲基化在精子发生过程中呈连续性,小鼠H19的甲基化始于胚胎发育的E15.5期到E18.5期,在出生后的粗线期精母细胞中则被完全甲基化(Davis et al., 1999)。
Testis-specific x-linked (Tsx)以前被认为是一个位于染色体失活中心的蛋白质编码基因,但随后被证明是一个lncRNA。人和小鼠的Tsx基因在外显子1、3、4、5和6区域具有非常明显的高度同源性(Sado & Brockdorff,2013)。Tsx突变雄性小鼠由于粗线期精母细胞的特异性凋亡导致其睾丸体积明显比野生型小,表明Tsx参与了雄性生殖细胞的发育,与此同时,Tsx在粗线期精母细胞中特异性表达,而不是在精原细胞或圆精细胞期间,暗示其在生殖细胞减数分裂中具有一定的调控作用(Anguera et al., 2011)。TUNEL分析显示,小鼠Tsx基因敲除后,粗线期精母细胞凋亡的比例异常高,表明这些lncRNAs在减数分裂进程中至关重要,但其具体作用机制还有待进一步研究。
AK077193是联会复合体蛋白2(synaptonemal complex protein 2,Sycp2)的外显子反义转录本,而Sycp2对于精母细胞特异性基因在雄性减数分裂期间的联会复合体装配和染色体联会是必需的(Yang et al., 2006)。数据显示AK077193在成年睾丸中上调表达,且AK077193和Sycp2之间的表达呈正相关(Sun et al., 2013)。因此,可以推测AK077193有可能调节Sycp2的表达。
2.4 LncRNA对精子成熟的影响HongrES2是一个1 588 bp的lncRNA,在附睾尾区特异性表达(Ni et al., 2011)。HongrES2作为一个1.6 kb mRNA-like的前体,可在大鼠附睾中产生一个新的类似microRNA的小RNA (mil-HongrES2)。附睾特异性羧酸酯酶7(carboxylesterase 7)在HongrES2的3’末端有100%同源的cDNA序列,其蛋白产物可以通过mil-HongrES2由HongrES2引起下调,进而影响胆固醇酯酶的活性,膜的流动性随精子膜中胆固醇/磷脂类的比例降低而增强,有利于精子的获能(Zhang et al., 2009),由此mil-HongrES2能够参与精子在附睾中的成熟过程。此外,通过整体酪氨酸磷酸化水平分析表明,mil-HongrES2的过度表达会导致精子获能的弱化(Ni et al., 2011),这表明在附睾中正常的精子成熟过程中,内源性低水平表达的HongrES2具有关键的调控作用。
Narcolepsy candidate-region 1 genes (NLC1-C)是一种仅在精原细胞和早期精母细胞表达的基因间lncRNA,主要表达于细胞质,其在精子的成熟阻滞(maturation arrest,MA)患者中的表达量降低,但在细胞核中的表达上调。微阵列分析技术可见健康对照者与MA患者的睾丸lncRNA表达谱之间存在明显差异(Lü et al., 2015)。NLC1-C在细胞质的过表达能够促进细胞增殖,若被敲除,则细胞增殖受到抑制并加速凋亡。此外,NLC1-C能够与位于核仁素的RBD结构域结合共同抑制miR-320a和miR-383的转录,而miR-320a和miR-383在加工成熟后则能直接抑制NLC1-C的作用以此调节细胞的存活。由此细胞核内NLC1-C的高表达可以加强对miR-320a和miR-383的转录抑制,进而导致精原细胞和初级精母细胞的过度增殖和成熟阻滞(Lü et al., 2015)。
2.5 LncRNA与细胞凋亡AK005744是一个源于spermatogenesis associated 17(Spata 17)的内含子6和7区域之间的内含子反义转录本,在睾丸中特异表达和在成年睾丸中上调;而Spata 17是参与雄性生殖细胞凋亡的睾丸特异性基因,并在成年睾丸中高表达(Deng et al., 2005;Nie et al., 2013)。AK005744和Spata 17表达谱的正相关性表明它们之间可能有一定的调控关系。LncRNA033862是一个位于Gfra1内的GDNF调控反义lncRNA转录本,主要在小鼠睾丸的精原细胞中表达。LncRNA033862通过RNA-DNA相互作用和介导GDNF受体基因Gfra 1的转录激活来调节SSCs的存活及增殖(Li et al., 2016)。
3 总结与展望近年来,大量研究表明lncRNAs在哺乳动物精子发生过程中具有重要的调控作用,包括精原细胞增殖、精母细胞减数分裂和圆形精子的形态变化等一系列过程。虽然在精子发生的特定阶段有少数lncRNAs的调控功能已得到确定,但还有很大一部分与精子发生相关的lncRNAs及其具体的调控功能有待研究。鉴于lncRNAs的重要调节作用,基于生物信息学方法对lncRNAs的功能注释和预测将是研究lncRNAs功能的主要手段之一。目前关于lncRNAs的功能注释方法主要包括基于共表达、miRNA调节、蛋白质结合、表观遗传修饰和基于ceRNA调节的方法。这些方法在一定程度上对lncRNAs的功能研究起到推动作用。LncRNAs在雄性生殖细胞研究中作为一个有吸引力的研究方向,随着高通量测序、强大的生物信息学分析、ontology数据库和蛋白质RNA结合模型的应用,更多的功能性lncRNAs可以被鉴定。因此,未来的研究鉴定基因组中以前的“暗物质”lncRNAs,必定会在精子发生过程中获取更多与lncRNAs相关的研究成果。与此同时,在生精障碍的情况下尽快鉴定出更多与此相关的lncRNAs,将有助于男性不育诊断技术和相关治疗药物的研发。
| 柏庆然, 宋旭. 2010. 长非编码RNA的作用机制及在肿瘤发生发展中的意义[J]. 生命科学, 22(7): 641–648. |
| Agbor VA, Tao S, Lei N, et al. 2013. A Wt1-Dmrt1 transgene restores DMRT1 to sertoli cells of Dmrt1-/- testes:a novel model of DMRT1-deficient germ cells[J]. Biology of Reproduction, 88(2): 51. DOI:10.1095/biolreprod.112.103135 |
| Anguera MC, Ma W, Clift D, et al. 2011. Tsx produces a long noncoding RNA and has general functions in the germline, stem cells, and brain[J]. PLoS Genetics, 7(9): e1002248. DOI:10.1371/journal.pgen.1002248 |
| Arun G, Akhade VS, Donakonda S, et al. 2012. mrhl RNA, a long noncoding RNA, negatively regulates Wnt signaling through its protein partner Ddx5/p68 in mouse spermatogonial cells[J]. Molecular and Cellular Biology, 32(15): 3140–3152. DOI:10.1128/MCB.00006-12 |
| Bonasio R, Shiekhattar R. 2014. Regulation of transcription by long noncoding RNAs[J]. Annual Review of Genetics, 48: 433–455. DOI:10.1146/annurev-genet-120213-092323 |
| Brosnan CA, Voinnet O. 2009. The long and the short of noncoding RNAs[J]. Current Opinion in Cell Biology, 21(3): 416–425. DOI:10.1016/j.ceb.2009.04.001 |
| Cesana M, Cacchiarelli D, Legnini I, et al. 2011. A long non-coding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J]. Cell, 147(2): 358–369. DOI:10.1016/j.cell.2011.09.028 |
| Costoya JA, Hobbs RM, Barna M, et al. 2004. Essential role of Plzf in maintenance of spermatogonial stem cells[J]. Nature Genetics, 36(6): 653–659. DOI:10.1038/ng1367 |
| Davis TL, Trasler JM, Moss SB, et al. 1999. Acquisition of the H19 methylation imprint occurs differentially on the parental alleles during spermatogenesis[J]. Genomics, 58(1): 18–28. DOI:10.1006/geno.1999.5813 |
| Deng Y, Nie DS, Wang J, et al. 2005. Molecular cloning of MSRG-11 gene related to apoptosis of mouse spermatogenic cells[J]. Acta Biochimica Biophysica Sinica, 37(3): 159–166. DOI:10.1093/abbs/37.3.159 |
| Ganesan G, Rao SM. 2008. A novel noncoding RNA processed by Drosha is restricted to nucleus in mouse[J]. RNA, 14(7): 1399–1410. DOI:10.1261/rna.838308 |
| Gong C, Maquat LE. 2011. lncRNAs transactivate STAU1-mediated mRNA decay by duplexing with 3'UTRs via Alu elements[J]. Nature, 470(7333): 284–288. DOI:10.1038/nature09701 |
| Guttman M, Amit I, Garber M, et al. 2009. Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals[J]. Nature, 458(7235): 223–227. DOI:10.1038/nature07672 |
| Inagaki S, Numata K, Kondo T, et al. 2005. Identification and expression analysis of putative mRNA-like non-coding RNA in Drosophila[J]. Genes to Cells, 10(12): 1163–1173. DOI:10.1111/j.1365-2443.2005.00910.x |
| Jonkers I, Monkhorst K, Rentmeester E, et al. 2008. Xist RNA is confined to the nuclear territory of the silenced X chromosome throughout the cell cycle[J]. Molecular and Cellular Biology, 28(18): 5583–5594. DOI:10.1128/MCB.02269-07 |
| Kerr GE, Young JC, Horvay K, et al. 2014. Regulated Wnt/beta-catenin signaling sustains adult spermatogenesis in mice[J]. Biology of Reproduction, 90(1): 1–12. DOI:10.1095/biolreprod.113.115535 |
| Kutter C, Watt S, Stefflova K, et al. 2012. Rapid turnover of long noncoding RNAs and the evolution of gene expression[J]. PLoS Genetics, 8(7): e1002841. DOI:10.1371/journal.pgen.1002841 |
| Lee JY, Kanatsu-Shinohara M, Morimoto H, et al. 2009. Genetic reconstruction of mouse spermatogonial stem cell self-renewal in vitro by Ras-cyclin D2 activation[J]. Cell Stem Cell, 5(1): 76–86. DOI:10.1016/j.stem.2009.04.020 |
| Lee TL, Alba D, Baxendale V, et al. 2006. Application of transcriptional and biological network analyses in mouse germ-cell transcriptomees[J]. Genomics, 88(1): 18–33. DOI:10.1016/j.ygeno.2006.03.008 |
| Lee TL, Cheung HH, Claus J, et al. 2009. GermSAGE:a comprehensive SAGE database for transcript discovery on male germ cell development[J]. Nucleic Acids Research, 37(Database issue): D891–D897. |
| Lee TL, Rennert OM, Chan WY. 2012a. Revealing the transcriptome landscape of mouse spermatogonial cells by tiling microarray[J]. Methods in Molecular Biology, 825: 75–92. DOI:10.1007/978-1-61779-436-0 |
| Lee TL, Xiao A, Rennert OM. 2012b. Identification of novel long noncoding RNA transcripts in male germ cells[J]. Methods in Molecular Biology, 825: 105–114. DOI:10.1007/978-1-61779-436-0 |
| Li L, Wang M, Wang M, et al. 2016. A long non-coding RNA interacts with Gfra1 and maintains survival of mouse spermatogonial stem cells[J]. Cell Death & Disease, 7: e2140. DOI:10.1038/cddis.2016.24 |
| Lü M, Tian H, Cao YX, et al. 2015. Downregulation of miR-320a/383-sponge-like long non-coding RNA NLC1-C (narcolepsy candidate-region 1 genes) is associated with male infertility and promotes testicular embryonal carcinoma cell proliferation[J]. Cell Death & Disease, 6(11): e1960. DOI:10.1038/cddis.2015.267 |
| Luk AC, Chan WY, Rennert OM, et al. 2014. Long noncoding RNAs in spermatogenesis:insights from recent high-throughput transcriptome studies[J]. Reproduction, 147(5): R131–R141. DOI:10.1530/REP-13-0594 |
| Ma X, Shao C, Jin Y, et al. 2014. Long non-coding RNAs:a novel endogenous source for the generation of Dicer-like 1-dependent small RNAs in Arabidopsis thaliana[J]. RNA Biology, 11(4): 373–390. DOI:10.4161/rna.28725 |
| Mattick JS. 2011a. Long noncoding RNAs in cell and developmental biology[J]. Seminars in Cell & Developmental Biology, 22(4): 327. |
| Mattick JS. 2011b. The central role of RNA in human development and cognition[J]. FEBS Letters, 585(11): 1600–1616. DOI:10.1016/j.febslet.2011.05.001 |
| Mercer TR, Dinger ME, Mattick JS. 2009. Long non-coding RNAs:insights into functions[J]. Nature Reviews Genetics, 10(3): 155–159. DOI:10.1038/nrg2521 |
| Mercer TR, Dinger ME, Sunkin SM, et al. 2008. Specific expression of long noncoding RNAs in the mouse brain[J]. Proceedings of the National Academy of Sciences of the United States of America, 105(2): 716–721. DOI:10.1073/pnas.0706729105 |
| Nagano T, Mitchell JA, Sanz LA, et al. 2008. The Air noncoding RNA epigenetically silences transcription by targeting G9a to chromatin[J]. Science, 322(5908): 1717–1720. DOI:10.1126/science.1163802 |
| Ni MJ, Hu ZH, Liu Q, et al. 2011. Identification and characterization of a novel non-coding RNA involved in sperm maturation[J]. PLoS ONE, 6(10): e26053. DOI:10.1371/journal.pone.0026053 |
| Nie DS, Liu Y, Juan H, et al. 2013. Overexpression of human SPATA17 protein induces germ cell apoptosis in transgenic male mice[J]. Molecular Biology Reports, 40(2): 1905–1910. DOI:10.1007/s11033-012-2246-z |
| Nishant KT, Ravishankar H, Rao MR. 2004. Characterization of a mouse recombination hot spot locus encoding a novel non-protein-coding RNA[J]. Molecular and Cellular Biology, 24(12): 5620–5634. DOI:10.1128/MCB.24.12.5620-5634.2004 |
| Ottolenghi C, Veitia R, Barbieri M, et al. 2000. The human doublesex-related gene, DMRT2, is homologous to a gene involved in somitogenesis and encodes a potential bicistronic transcript[J]. Genomics, 64(2): 179–186. DOI:10.1006/geno.2000.6120 |
| Pandey RR, Mondal T, Mohammad F, et al. 2008. Kcnqlotl antisense noncoding RNA mediates lineage-specific transcriptional silencing through chromatin-level regulation[J]. Molecular Cell, 32(2): 232–246. DOI:10.1016/j.molcel.2008.08.022 |
| Park Y, Kelley RL, Oh H, et al. 2002. Extent of chromatin spreading determined by roX RNA recruitment of MSL proteins[J]. Science, 298(5598): 1620–1623. DOI:10.1126/science.1076686 |
| Ponting CP, Oliver PL, Reik W. 2009. Evolution and functions of long noncoding RNAs[J]. Cell, 136(4): 629–641. DOI:10.1016/j.cell.2009.02.006 |
| Prasanth KV, Prasanth SG, Xuan Z, et al. 2005. Regulating gene expression through RNA nuclear retention[J]. Cell, 123(2): 249–263. DOI:10.1016/j.cell.2005.08.033 |
| Probst AV, Dunleavy E, Almouzni G. 2009. Epigenetic inheritance during the cell cycle[J]. Nature Reviews Molecular Cell Biology, 10(3): 192–206. DOI:10.1038/nrm2640 |
| Rasmussen TP, Mastrangelo MA, Eden A, et al. 2000. Dynamic relocalization of histone MacroH2A1 from centrosomes to inactive X chromosomes during X inactivation[J]. The Journal of Cell Biology, 150(5): 1189–1198. DOI:10.1083/jcb.150.5.1189 |
| Ravasi T, Suzuki H, Pang KC, et al. 2006. Experimental validation of the regulated expression of large numbers of non-coding RNAs from the mouse genome[J]. Genome Research, 16(1): 11–19. |
| Sado T, Brockdorff N. 2013. Advances in understanding chromosome silencing by the long non-coding RNA Xist[J]. Philosophical Transactions of the Royal Society of London Series B-Biologic, 368(1609): 20110325. |
| Schmidt JA, Avarbock MR, Tobias JW, et al. 2009. Identification of glial cell line-derived neurotrophic factor-regulated genes important for spermatogonial stem cell self-renewal in the rat[J]. Biology of Reproduction, 81(1): 56–66. DOI:10.1095/biolreprod.108.075358 |
| Sheik Mohamed J, Gaughwin PM, Lim B, et al. 2010. Conserved long noncoding RNAs transcriptionally regulated by Oct4 and Nanog modulate pluripotency in mouse embryonic stem cells[J]. RNA, 16(2): 324–337. DOI:10.1261/rna.1441510 |
| Shi X, Sun M, Liu H, et al. 2013. Long non-coding RNAs:a new frontier in the study of human diseases[J]. Cancer Letters, 339(2): 159–166. DOI:10.1016/j.canlet.2013.06.013 |
| Sun J, Lin Y, Wu J. 2013. Long non-coding RNA expression profiling of mouse testis during postnatal development[J]. PLoS ONE, 8(10): e75750. DOI:10.1371/journal.pone.0075750 |
| Tachibana M, Nozaki M, Takeda N, et al. 2007. Functional dynamics of H3K9 methylation during meiotic prophase progression[J]. The EMBO Journal, 26(14): 3346–3359. DOI:10.1038/sj.emboj.7601767 |
| Wang X, Arai S, Song X, et al. 2008. Induced ncRNAs allosterically modify RNA-binding proteins in cis to inhibit transcription[J]. Nature, 454(7200): 126–130. DOI:10.1038/nature06992 |
| Yang F, De La Fuente R, Leu NA, et al. 2006. Mouse SYCP2 is required for synaptonemal complex assembly and chromosomal synapsis during male meiosis[J]. The Journal of Cell Biology, 173(4): 497–507. DOI:10.1083/jcb.200603063 |
| Yang PK, Kuroda MI. 2007. Noncoding RNAs and intranuclear positioning in monoallelic gene expression[J]. Cell, 128(4): 777–786. DOI:10.1016/j.cell.2007.01.032 |
| Yoon JH, Abdelmohsen K, Gorospe M. 2013. Posttranscriptional gene regulation by long noncoding RNA[J]. Journal of Molecular Biology, 425(19): 3723–3730. DOI:10.1016/j.jmb.2012.11.024 |
| Zhang L, Liu Q, Zhou Y, et al. 2009. Baculo-expression and enzymatic characterization of CES7 esterase[J]. Acta Biochimica et Biophysica Sinica, 41(9): 731–736. DOI:10.1093/abbs/gmp061 |
| Zhang L, Lu H, Xin D, et al. 2010. A novel ncRNA gene from mouse chromosome 5 trans-splices with Dmrt1 on chromosome 19[J]. Biochemical and Biophysical Research Communications, 400(4): 696–700. DOI:10.1016/j.bbrc.2010.08.130 |
 2016, Vol. 36
2016, Vol. 36




