


2. 山西中医学院中药学院,山西 晋中 030600
2. Dept of Chinese Medicine, Shanxi University of TCM, Jinzhong Shanxi 030600,China
胃癌是世界上最常见的恶性肿瘤之一,其致死率在肿瘤相关疾病中排第2位。胃癌在东亚尤其高发,病例总数约占全球的50%,我国胃癌的死亡率超过世界平均水平的两倍,其高死亡率则主要由于早期诊断困难及缺乏有效的干预手段。胃癌的发病机制复杂,包括遗传因素、环境因素等多个方面,胃癌的发病机制尤其是诊断及预后标志物研究受到越来越多学者的重视。
现有的研究已经证实,胃癌的发生常常与转录异常有关,这种异常不限于蛋白编码RNA(mRNA)水平的异常,也包括基因组中非编码RNA(non-coding RNAs,ncRNAs)调控能力的异常,包含长链非编码RNA(long non-coding RNAs,lncRNAs)、假基因、microRNAs(miRNAs)等[1]。大量研究已经证实[2],miRNAs广泛参与肿瘤细胞的生物进程,在包括胃癌在内的多种肿瘤疾病中发现miRNAs异常表达;其可以通过序列互补与靶基因3′端非编码区域(3′UTRs)的miRNA反应原件(miRNA response elements,MREs)结合,使靶基因降解或抑制其翻译,进而抑制靶基因的水平。miRNA作为潜在的肿瘤早期诊断或预后标志物,对开发新的肿瘤治疗策略有积极的意义[3-7]。同时,细胞内有一类内源性RNA,它们的3′UTRs包含相同的MREs,可以通过MREs竞争性结合相同的miRNAs,进而调节各自的表达水平[8],发挥重要的转录后调控作用,调节肿瘤进程,称之为竞争性内源RNA(competing endogenous RNAs,ceRNAs)。
1 ceRNA假说“ceRNA”不是一种RNA,而是一种全新的基因表达调控模式,是RNA的一种作用方式,其实,在ceRNA假说提出之前,竞争性RNA这一概念就已经进入人们的视野。2007年,Ebert等[9]通过人工合成得到miRNA的抑制剂,称为miRNA海绵(miRNA sponge),随着越来越多的实验验证及内源性miRNA海绵的发现,ceRNA的概念在2011年被正式提出[10-13]。ceRNA概念的提出,将基因的相互作用模式由“miRNAs→RNAs”变成了“RNAs→miRNAs→RNAs”。阐明这种新的基因相互作用方式将为肿瘤形成的机制研究以及抗肿瘤治疗手段的发展提供新的视角。
迄今为止,已有多项研究支持ceRNA假说。如PTEN是重要的抑癌基因,在多种人肿瘤中异常改变,在黑色素瘤、前列腺癌和胶质母细胞瘤中都已经发现了PTEN的ceRNAs[14-16]。这些研究结果证实了细胞内ceRNA调控网络的存在,且广泛参与肿瘤血管生成、侵袭、转移等多个进程[17-18]。ceRNAs网络的平衡一旦被打破,就可能导致疾病的发生,包括肿瘤。
理论上,任何包含miRNA结合位点的RNA都可能是ceRNAs,包括假基因、mRNAs、lncRNAs等。而在众多的可以作为ceRNA调节肿瘤进程的RNA中,lncRNAs有着重要地位。
2 lncRNA简介 2.1 lncRNA的功能lncRNAs是一类长度大于200 nt的非编码RNA,参与了染色质修饰、转录激活等多种重要的调控过程,可作为信号分子、桥梁、向导、诱饵等与其他ncRNAs、mRNA、蛋白质及基因组DNA交流,参与多种肿瘤进程[19-22]。最近的研究显示,与正常细胞相比,lncRNAs在肿瘤细胞中常常处于“解除管制”状态,说明lncRNAs是潜在的肿瘤生物标志物;同时,过表达或下调肿瘤细胞中特定的lncRNA,常常可以触发凋亡或使肿瘤细胞对诱导凋亡的治疗敏感,即lncRNAs可作为某些类型肿瘤的治疗靶点,如LUNAR1可以作为T细胞急性淋巴母细胞性白血病的分子标志物和潜在治疗靶标[23-24]。此外,lncRNAs还可调节肿瘤转移相关的信号通路、参与肿瘤的迁移[25]、耐药[26],如lncRNA MRUL (MDR-related and upregulated lncRNA)可促进耐阿霉素胃癌细胞SGC7901/ADR中ABCB1 (ATP-binding cassette,subfamily B,member 1)基因的表达,是逆转胃癌细胞多药耐药表型的潜在靶点。
2.2 lncRNA与miRNA的相互作用机制lncRNA既可以被miRNA调控,也可以调控miRNA的表达。例如,Xu等[27]研究发现,lncRNA-AC130710是miR-129-5P的靶基因,其表达水平受miR-129-5P的调控。肿瘤抑制因子7(tumour suppressor candidate 7,TUSC7)在胃癌样本中低表达,是胃癌患者无病生存率(disease-free survival ,DFS)和疾病特异性生存率(disease-specific survival ,DSS)独立的预后指标,在体内外均可抑制肿瘤细胞生长。TUSC7上有一个miR-23b结合位点,可通过转录后调控机制抑制miR-23b表达[28]。
lncRNA调控miRNA的作用机制主要有3个:① lncRNA能与miRNA竞争性结合靶基因mRNA的3′-UTR,从而对miRNA的负向调控机制进行抑制;② 有些lncRNA是miRNA的前体,通过细胞核中的Drosha裂解、细胞质中Dicer切割,产生成熟的miRNA,调控靶基因的表达而发挥功能;③ 部分lncRNA能发挥内源性“miRNA海绵”的功能,与mRNA竞争性结合miRNA的MREs,进而达到抑制miRNA表达及其对靶基因的负向调控,即ceRNA机制[29-31](Fig 1)。
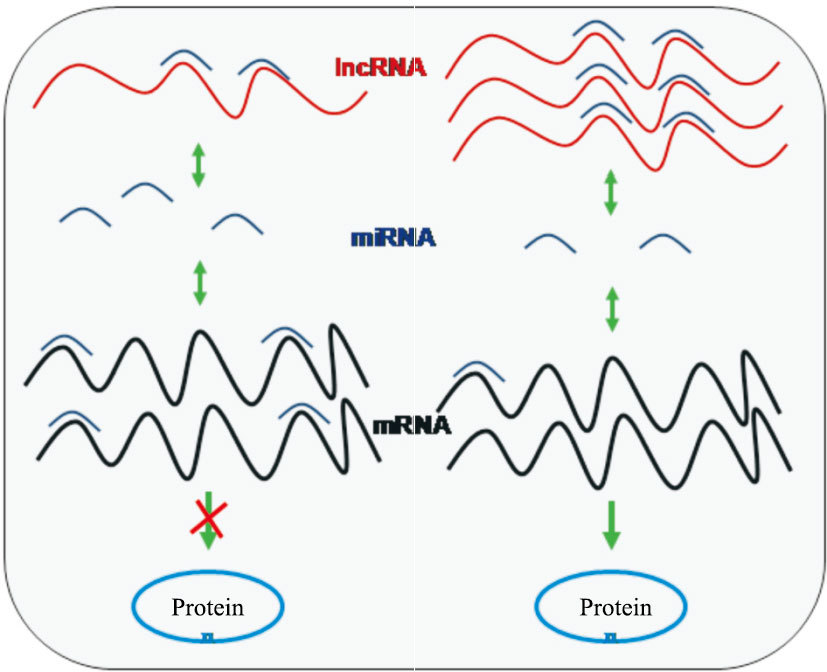
|
| 图 1 The regulation mechanism of lncRNA on target gene expression as ceRNA |
越来越多的研究表明,lncRNA的异常表达与胃癌的发生发展密切相关,其作用涉及多种分子机制。有些lncRNAs可以与DNA、RNA和蛋白质相互作用,参与胃癌的发生和发展。如MALAT1、GHET1和TINCR可通过碱基互补配对与mRNAs结合或与RNA结合蛋白(RNA binding proteins,RBPs)形成复合物,介导mRNA的稳定性和拼接;还有些lncRNAs,如ANRIL、GACAT3、H19、MEG3和TUSC7通过与miRNAs结合发挥生物学作用;ANRIL、fendrr、H19、HOTAIR、MALAT1和PVT1,可以通过招募组蛋白修饰物,抑制靶基因的顺式或反式转录;CCAT1、GAPLINC、GAS5、H19、MEG3和TUSC7分别通过调节肿瘤抑制因子p53、癌基因c-myc发挥致癌或抑癌作用[32-33]。近年来,多项研究证据表明,lncRNA与miRNA及其靶基因之间内源性竞争的调控模式与胃癌的发展进程密切相关,如bc032469、GAPLINC和HOTAIR等[34-35]。这些lncRNAs在胃癌调控网络中有些作为癌基因,有些作为抑癌基因,参与胃癌的进程。
3.1 癌基因近年来,已经发现4个lncRNA可作为癌基因,以内源性竞争机制参与调控胃癌进程,包括GAPLINC(gastric adenocarcinoma predictive long intergenic noncoding RNA)、HOTAIR(Hox transcript antisense intergenic RNA)、BC032469和H19。
Hu等[36]的研究发现,lncRNA GAPLINC在胃癌组织中高表达,通过与CD44竞争性结合miR-311-3P的结合位点,削弱miR-311-3P对CD44的抑制作用,上调CD44的表达水平,进而调控细胞的迁移路径和增殖进程,即作为胃癌中miR-311-3P的分子诱饵及癌基因CD44的ceRNA,与胃癌患者的预后明显负相关。Pan等[37]发现,最初因其参与原发性乳腺癌以及乳腺癌转移而被熟知,可促进肿瘤侵袭和转移,与结直肠癌、肝细胞癌、胰腺癌以及胃肠道肿瘤的恶性程度和预后不良正相关[38]的HOTAIR,在胃癌组织中的表达水平显著高于癌旁组织,HOTAIR通过与胃癌细胞内miR-331-3P上的人上皮细胞生长因子受体2(HER2)miRNA反应元件占位结合,使HER2与miR-331-3P的结合减少,抑制了miR-331-3P对HER2的降解作用,间接上调了细胞中HER2的表达水平,进而调控胃癌细胞的增殖、迁移等进程[39]。HOTAIR的表达水平与瘤体积大小、病理分期以及转移情况正相关,与胃癌患者的生存率负相关;在体外可促进胃癌细胞增殖、转移及浸润。
Lyu等[40]采用lncRNA芯片对3对胃癌及癌旁组织样本进行筛选,发现lncRNA BC032469在胃癌组织中明显高表达,约为癌旁组织中的20倍,在58对样本中进行验证,发现其异常高表达与胃癌患者瘤体积大,分化程度低以及预后不良相关,采用RNAi技术下调BC032469可明显抑制肿瘤细胞在体内外的增殖,而共表达人端粒酶逆转录酶(human telomerase reverse transcriptase,hTERT)则可取消BC032469下调介导的细胞增殖抑制。进一步研究其机制,发现BC032469可作为ceRNA直接结合miR-1207-5p,进而上调miR-1207-5p的靶基因hTERT的表达。hTERT是端粒末端转移酶的限速亚基,可直接决定端粒末端转移酶的活性。多项研究已经证实,端粒末端转移酶及hTERT的活化与多种恶性肿瘤的发生发展相关,hTERT在胃癌中的表达水平与肿瘤体积、浸润深度、淋巴结转移、病理等级以及临床分期呈正相关,是潜在的肿瘤治疗靶标。因此BC032469有望成为胃癌的治疗靶标和预后不良的生物标志物。
已有研究发现,ZEB1是miR-141的靶基因,在上皮间质转移(epithelial to mesenchymal transition,EMT)进程中发挥重要作用,而H19和miR-141在胃癌细胞和组织中表达呈负相关,且H19可以序列特异性形式与miR-141结合,H19 siRNA可降低ZEB1表达,H19过表达增加ZEB1的表达,即H19可通过抑制miR-141对靶基因ZEB1的降解作用,调控ZEB1的表达[41]。此外,H19在肝细胞癌、结肠癌、膀胱癌、乳腺癌[42]等多种肿瘤中高表达,可促进多种恶性肿瘤的发生。
从这些研究我们发现,基于ceRNA机制,作为促癌基因调控胃癌进程的lncRNAs均在胃癌组织中明显高表达,通过与胃癌发展进程明显相关的癌基因(mRNA),包括HER2、hTERT等,竞争性结合相同的miRNA,拮抗miRNA对其靶基因HER2等的抑制作用,间接上调癌基因mRNA的表达水平,促进胃癌的发生发展。这些lncRNAs因其调控的靶基因作用不同,涉及胃癌进程的多个方面,包括胃癌细胞的增殖、迁移、浸润;瘤体积大小、病理分期;胃癌患者的预后、生存率等,可作为胃癌患者预后不良的生物标志物。研究也提示我们,是否可以采用基因干扰的手段,外源性导入目标lncRNA的抑制序列,间接下调HER2等癌基因的表达,使lncRNA成为胃癌潜在的治疗靶标。
3.2 抑癌基因lncRNA除了可以作为ceRNA促进胃癌的发展进程,也可以作为ceRNA在胃癌进程中发挥抑癌基因的作用,下调靶基因mRNA的水平,进而抑制其蛋白质的表达,如FER1L4和MEG3。Xia等[43-44]发现,lncRNA-FER1L4是PTEN mRNA的竞争性内源RNA。研究显示,胃癌组织中的lncRNA-FER1L4表达明显低于癌旁组织3倍以上,且与miR-106a-5p间存在直接相互作用,miR-106a-5p过表达可降低lncRNA-FER1L4和PTEN mRNA的表达水平,siRNA-FER1L4可下调PTEN的mRNA和蛋白质水平,同时上调miR-106a-5p的水平。同时,发现下调lncRNA-FER1L4导致细胞增殖加快与其促进细胞从G0/G1期进入S期有关。PTEN是一种抑癌基因,其功能障碍在肿瘤发病机制中扮演重要角色,PTEN作为磷脂磷酸酶,可催化第二信使磷脂酰基醇3(PtdIns(3,4,5)P3,PIP3)水解,抑制PI3K/AKT信号通路的激活,进而调控细胞增殖和代谢[45]。Peng等[46]发现母系印记基因(maternally expressed gene3,MEG3)在胃癌患者及胃癌细胞系中低表达,并且其表达水平和胃癌的转移密切相关。MEG3通过内源性竞争机制,上调细胞内miR-181a的靶基因Bcl-2的表达水平,其过表达能够抑制HGC-27和MGC-803细胞增殖、迁移、浸润,并促进细胞凋亡。众所周知,Bcl-2是Bcl-2家族的一个抗凋亡基因,其定位在线粒体膜上,可通过改变线粒体巯基的氧化还原状态,控制其膜电位,调节线粒体膜对某些凋亡蛋白前体,如Bax、细胞色素C的通透性等发挥抗凋亡作用。MEG3以miR-181位点依赖的方式影响胃癌细胞中Bcl-2的表达,进而影响细胞表型,其作为抑癌基因[47],可以作为抗肿瘤药物的潜在治疗靶标。
随着分子生物学技术的不断发展、肿瘤生物信息学分析手段的进步以及转录组学数据的积累,更多的具有潜在ceRNA功能的lncRNA将被发现,将这些lncRNA与miRNA、mRNA构建一个分子网络,可以用于靶向或小分子抗癌药物的筛选。同时ceRNA在胃癌中扮演癌基因或抑癌基因的角色,因此通过干预ceRNA的表达水平发挥临床治疗作用在理论上是可行的。例如,HOTAIR能够与miR-331-3P竞争性结合进而上调HER2的表达。HER2是重要的癌基因,目前临床上已有靶向HER2的抗肿瘤药物赫赛汀。所以如果能够通过分子生物学技术下调HOTAIR的水平间接降低HER2的表达也可能发挥良好的治疗作用。ceRNA是胃癌发生发展进程中的标志性分子,将有望成为诊断标志物和预后标志物,辅助胃癌的早期诊断及预后判断。
4 展望长链非编码RNA(lncRNAs)作为非编码RNA的重要组成,广泛参与人体的多种生理功能,其表达水平的异常与多种人类肿瘤的发生发展密切相关,是潜在的诊断标志物和药物治疗靶标。近年来通过对内源性竞争机制的深入研究,发现lncRNAs通过与胃癌驱动基因竞争相同的miRNAs,降低miRNAs对靶基因的抑制效应,调节胃癌的进程。尽管较多的研究提示,lncRNAs可通过内源性竞争机制参与调控胃癌进程,但目前尚缺乏大样本量的验证数据以及深入的分子机制研究表明哪些lncRNAs有望成为胃癌诊断、预后的标志物或潜在的药物作用靶标,lncRNAs在胃癌进程中究竟是因是果还不十分明确,仍需要进一步的研究来确证。
| [1] | Cheng D L, Xiang Y Y, Ji L J, et al. Competing endogenous RNA interplay in cancer: mechanism, methodology, and perspectives[J]. Tumor Biol, 2015, 36 (2) : 479-88 doi:10.1007/s13277-015-3093-z. |
| [2] | Wan H Y, Guo L M, Liu T, et al. Regulation of the transcription factor NF-γB1 by microRNA-9 in human gastric adenocarcinoma[J]. Mol Cancer, 2010, 9 : 16 doi:10.1186/1476-4598-9-16. |
| [3] | Gao C, Pang M, Zhou Z, et al. Epidermal growth factor receptor-coamplified and overexpressed protein(VOPP1) is a putative oncogene in gastric cancer[J]. Clin Exp Med, 2015, 15 (4) : 469-75 doi:10.1007/s10238-014-0320-7. |
| [4] | Bertoli G, Cava C, Castiglioni I. MicroRNAs as biomarkers for diagnosis, prognosis and theranostics in prostate cancer[J]. Int J Mol Sci, 2016, 17 (3) : E421 doi:10.3390/ijms17030421. |
| [5] | Pileczki V, Cojocneanu-Petric R, Maralani M, et al. MicroRNAs?as regulators of apoptosis mechanisms in cancer[J]. Clujul Med, 2016, 89 (1) : 50-5 doi:10.15386/cjmed-2016.89.1. |
| [6] | Liu G H, Liu Y H, Yang Z, et al. MicroRNA-524-5p suppresses the growth and invasive abilities of gastric cancer cells[J]. Oncol Lett, 2016, 11 (3) : 1926-32 . |
| [7] | Qiu X, Zhang J, Shi W, et al. Circulating microRNA-26a in plasma and its potential diagnostic value in gastric cancer[J]. PLoS One, 2016, 11 (3) : e0151345 doi:10.1371/journal.pone.0151345. |
| [8] | Karreth FA1, Pandolfi P P. ceRNA cross-talk in cancer: when ce-bling rivalries go awry[J]. Cancer Discov, 2013, 3 (10) : 1113-21 doi:10.1158/2159-8290.CD-13-0202. |
| [9] | Ebert M S, Neilson J R, Sharp P A. MicroRNA sponges:competitive inhibitors of small RNAs in mammalian cells[J]. Nat Methods, 2007, 4 (9) : 721-6 doi:10.1038/nmeth1079. |
| [10] | Seitz H. Redefining microRNA targets[J]. Curr Biol, 2009, 19 (10) : 870-3 doi:10.1016/j.cub.2009.03.059. |
| [11] | Arvey A, Larsson E, Sander C, et al. Target mRNA abundance dilutes microRNA and siRNA activity[J]. Mol Syst Biol, 2010, 6 : 363 . |
| [12] | Ebert M S, Sharp P A. Emerging roles for natural microRNA sponges[J]. Curr Biol, 2010, 20 (19) : R858-61 doi:10.1016/j.cub.2010.08.052. |
| [13] | Salmena L, Poliseno L, Tay Y, et al. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language[J]. Cell, 2011, 146 (3) : 353-8 doi:10.1016/j.cell.2011.07.014. |
| [14] | Karreth F A, Tay Y, Perna D, et al. In vivo identification of tumor-suppressive PTEN ceRNAs in an oncogenic BRAF-induced mouse model of melanoma[J]. Cell, 2011, 147 (2) : 382-95 doi:10.1016/j.cell.2011.09.032. |
| [15] | Tay Y, Kats L, Salmena L, et al. Coding-independent regulation of the tumor suppressor PTEN by competing endogenous mRNAs[J]. Cell, 2011, 147 (2) : 344-57 doi:10.1016/j.cell.2011.09.029. |
| [16] | Sumazin P, Yang X, Chiu H S, et al. An extensive microRNA-mediated network of RNA-RNA interactions regulates established oncogenic pathways in glioblastoma[J]. Cell, 2011, 147 (2) : 370-81 doi:10.1016/j.cell.2011.09.041. |
| [17] | Zheng L, Li X, Gu Y, et al. The 3′UTR of the pseudogene CYP4Z2P promotes tumor angiogenesis in breast cancer by acting as a ceRNA for CYP4Z1[J]. Breast Cancer Res Treat,, 2015, 150 (1) : 105-18 doi:10.1007/s10549-015-3298-2. |
| [18] | Liu K, Guo L, Guo Y, et al. AEG-1 3′-untranslated region functions as a ceRNA in inducing epithelial-mesenchymal transition of human non-small cell lung cancer by regulating miR-30a activity[J]. Eur J Cell Biol, 2015, 94 (1) : 22-31 doi:10.1016/j.ejcb.2014.10.006. |
| [19] | 姜怀德, 李桂林. 长非编码RNA的功能研究[J]. 中国药理学通报, 2015, 31 (7) : 900-5 . Jiang H D, Li G L. On function of long noncoding RNA[J]. Chin Pharmacol Bull, 2015, 31 (7) : 900-5 . |
| [20] | Qiu M, Xu Y, Yang X, et al. CCAT2 is a lung adenocarcinoma-specific long non-coding RNA and promotes invasion of non-small cell lung cancer[J]. Tumour Biol, 2014, 35 (6) : 5375-80 doi:10.1007/s13277-014-1700-z. |
| [21] | Yang F, Bi J, Xue X, et al. Up-regulated long non-coding RNA H19 contributes to proliferation of gastric cancer cells[J]. FEBS J, 2012, 279 (17) : 3159-65 doi:10.1111/j.1742-4658.2012.08694.x. |
| [22] | Kumar M S, Armenteros-Monterroso E, East P, et al. HMGA2 functions as a competing endogenous RNA to promote lung cancer progression[J]. Nature, 2014, 505 (7482) : 212-7 . |
| [23] | Trimarchi T, Bilal E, Ntziachristos P, et al. Genome-wide mapping and characterization of Notch-regulated long noncoding RNAs in acute leukemia[J]. Cell, 2014, 158 (3) : 593-606 doi:10.1016/j.cell.2014.05.049. |
| [24] | Rossi M N, Antonangeli F. LncRNAs: new players in apoptosis control[J]. Int J Cell Biol, 2014, 2014 : 473857 . |
| [25] | Gu Y, Chen T, Li G, et al. LncRNAs:emerging biomarkers in gastric cancer[J]. Future Oncol, 2015, 11 (17) : 2427-41 doi:10.2217/fon.15.175. |
| [26] | Wang Y, Zhang D, Wu K, et al. Long noncoding RNA MRUL promotes ABCB1 expression in multidrug-resistant gastric cancer cell sublines[J]. Mol Cell Biol, 2014, 34 (17) : 3182-93 doi:10.1128/MCB.01580-13. |
| [27] | Xu C, Shao Y, Xia T, et al. lncRNA-AC130710 targeting by miR-129-5p is upregulated in gastric cancer and associates with poor prognosis[J]. Tumour Biol, 2014, 35 (10) : 9701-6 doi:10.1007/s13277-014-2274-5. |
| [28] | Qi P, Xu M D, Shen X H, et al. Reciprocal repression between TUSC7 and miR-23b in gastric cancer[J]. Int J Cancer, 2015, 137 (6) : 1269-78 doi:10.1002/ijc.29516. |
| [29] | Yoon J H, Abdelmohsen K, Srikantan S, et al. LincRNA-p21 suppresses target mRNA translation[J]. Mol Cell, 2012, 47 (4) : 648-55 doi:10.1016/j.molcel.2012.06.027. |
| [30] | Wang Y, Xu Z, Jiang J, et al. Endogenous miRNA sponge lincRNA-RoR regulates Oct4, Nanog, and Sox2 in human embryonic stem cell self-renewal[J]. Dev Cell, 2013, 25 (1) : 69-80 doi:10.1016/j.devcel.2013.03.002. |
| [31] | Cesana M, Cacchiarelli D, Legnini I, et al. A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J]. Cell, 2011, 147 (2) : 358-69 doi:10.1016/j.cell.2011.09.028. |
| [32] | Li T, Mo X, Fu L, et al. Molecular mechanisms of long noncoding RNAs on gastric cancer[J]. Oncotarget, 2016, 7 (8) : 8601-12 . |
| [33] | Fang X Y, Pan H F, Leng R X, et al. Long noncoding RNAs: novel insights into gastric cancer[J]. Cancer Lett, 2015, 356 (2 Pt B) : 357-66 . |
| [34] | Hu Y, Tian H, Xu J, et al. Roles of competing endogenous RNAs in gastric cancer[J]. Brief Funct Genomics, 2016, 15 (3) : 266-73 doi:10.1093/bfgp/elv036. |
| [35] | Guo L L, Song C H, Wang P, et al. Competing endogenous RNA networks and gastric cancer[J]. World J Gastroenterol, 2015, 21 (41) : 11680-7 doi:10.3748/wjg.v21.i41.11680. |
| [36] | Hu Y, Wang J, Qian J, et al. Long noncoding RNA GAPLINC regulates CD44-dependent cell invasiveness and associates with poor prognosis of gastric cancer[J]. Cancer Res, 2014, 74 (23) : 6890-902 doi:10.1158/0008-5472.CAN-14-0686. |
| [37] | Pan W, Liu L, Wei J, et al. A functional lncRNA HOTAIR genetic variant contributes to gastric cancer susceptibility[J]. Mol Carcinog,, 2016, 55 (1) : 90-6 doi:10.1002/mc.22261. |
| [38] | Cai B, Song X Q, Cai J P, et al. HOTAIR: a cancer-related long non-coding RNA[J]. Neoplasma, 2014, 61 (4) : 379-91 doi:10.4149/neo_2014_075. |
| [39] | Liu X H, Sun M, Nie F Q, et al. Lnc RNA HOTAIR functions as a competing endogenous RNA to regulate HER2 expression by sponging miR-331-3p in gastric cancer[J]. Mol Cancer, 2014, 13 : 92 doi:10.1186/1476-4598-13-92. |
| [40] | Lyu M H, Tang B, Zeng S, et al. Long noncoding RNA BC032469, a novel competing endogenous RNA, upregulates hTERT expression by sponging miR-1207-5p and promotes proliferation in gastric cancer[J]. Oncogene, 2016, 35 (27) : 3524-34 doi:10.1038/onc.2015.413. |
| [41] | Zhou X, Ye F, Yin C, et al. The Interaction Between MiR-141 and lncRNA-H19 in Regulating Cell Proliferation and Migration in Gastric Cancer[J]. Cell Physiol Biochem, 2015, 36 (4) : 1440-52 doi:10.1159/000430309. |
| [42] | 彭艳, 谢海棠, 孙红, 等. 人长链非编码RNA基因H19克隆、表达载体构建及对MCF-7细胞增殖的影响[J]. 中国药理学通报, 2015, 31 (4) : 555-60 . Peng Y, Xie H T, Sun H, et al. Constructing an expression vector for human lncRNA H19 and the effect of its overexpression on MCF-7 cell proliferation[J]. Chin Pharmacol Bull, 2015, 31 (4) : 555-60 . |
| [43] | Xia T, Liao Q, Jiang X M, et al. Long noncoding RNA associated-competing endogenous RNAs in gastric cancer[J]. Sci Rep, 2014, 4 : 6088 . |
| [44] | Guo J, Miao Y, Xiao B, et al. Differential expression of microRNA species in human gastric cancer versus non-tumorous tissues[J]. J Gastroenterol Hepatol,, 2009, 24 (4) : 652-7 doi:10.1111/jgh.2009.24.issue-4. |
| [45] | Papa A, Wan L, Bonora M, et al. Cancer-associated PTEN mutants act in dominant-negative manner to suppress PTEN protein function[J]. Cell, 2014, 157 (3) : 595-610 doi:10.1016/j.cell.2014.03.027. |
| [46] | Peng W, Si S, Zhang Q, et al. Long non-coding RNA MEG3 functions as a competing endogenous RNA to regulate gastric cancer progression[J]. J Exp Clin Cancer Res, 2015, 34 : 79 doi:10.1186/s13046-015-0197-7. |
| [47] | Zhou Y, Zhang X, Klibanski A. MEG3 noncoding RNA: a tumor suppressor[J]. J Mol Endocrinol,, 2012, 48 (3) : R45-53 doi:10.1530/JME-12-0008. |

