文章信息
- 张西轩, 李晔, 王亚航, 杜康龙, 张真, 阮海华
- ZHANG Xi-xuan, LI Ye, WANG Ya-hang, DU Kang-long, ZHANG Zhen, RUAN Hai-hua
- 蜡样芽孢杆菌ColR75E胶原酶的表达、纯化及酶学性质研究
- Expression, Purification and Enzymatic Characterization of ColR75E Collagenase of Bacillus cereus R75E
- 中国生物工程杂志, 2015, 35(10): 44-52
- China Biotechnology, 2015, 35(10): 44-52
- http://dx.doi.org/10.13523/j.cb.20151007
-
文章历史
- 收稿日期: 2015-07-02
- 修回日期: 2015-07-15
胶原酶(Collagenase)指的是在天然pH与温度条件下可以降解胶原纤维的一类蛋白酶[1, 2]。胶原酶来源广泛,多种微生物、动物的许多组织细胞都可产生胶原酶,根据来源可主要分为微生物胶原酶与动物胶原酶[3]。其中,微生物胶原酶以底物范围广、酶切位点多、生产成本低等优点应用更为广泛[3]。目前,商业化生产的微生物胶原酶主要是对Ⅰ型胶原蛋白具有特异降解能力的胶原酶,即Ⅰ型胶原酶[4],可作为富含胶原肉食品加工过程中的嫩化剂、在细胞培养时去除细胞外基质的细胞分散剂、以及在临床上治疗一些胶原沉积性疾病[2]。目前,微生物胶原酶中最广泛使用的商业化胶原酶是溶组织梭状芽孢杆菌(Clostridium histolyticum)分泌的胶原酶[5, 6]。
本实验室在前期研究中筛选到一株产胶原酶的蜡样芽孢杆菌Bacillus cereus R75E,并克隆到编码该胶原酶蛋白的编码基因colR75E(GenBank:KP987200)。该编码序列全长2 898bp,可编码由965个氨基酸残基构成的胶原酶ColR75E。序列分析表明,该基因与已经报道的溶组织梭状芽孢杆菌(Clostridium histolyticum)的胶原酶ColG编码基因具有69%的同源性,属于Ⅰ型胶原酶[6]。然而,迄今为止ColR75E胶原酶的酶学特性仍不清楚。基于此,本研究克隆了蜡样芽孢杆菌Bacillus cereusR75E的胶原酶基因colR75E,将其在大肠杆菌中表达和纯化[7],并进一步对其酶学性质进行研究,为微生物胶原酶在医疗、食品等工业领域的应用提供理论基础。
1 材料与方法 1.1 材 料 1.1.1 菌株和载体本实验室分离纯化的蜡样芽孢杆菌Bacillus cereus R75E保藏于中国微生物菌种保藏管理委员会普通微生物中心,保藏编号为CGMCC No.8614;感受态大肠杆菌[Trans10 和BL21(DE3)]均购自美国Promega公司;原核表达载体pET28a购自德国Novagen公司。
1.1.2 试剂与材料细菌基因组DNA提取试剂盒、琼脂糖凝胶DNA快速回收试剂盒购自天根生化科技(北京)有限公司;限制性内切酶BamHⅠ和XhoⅠ、质粒小提试剂盒、DNA marker DL5000购自日本TaKaRa公司;T4 DNA 连接酶购自美国Promega公司;KOD DNA聚合酶购自日本TOYOBO公司;His-Tag蛋白镍金属螯合层析柱购自美国Roche公司;牛跟腱来源的Ⅰ型胶原蛋白购自美国Sigma-Aldrich公司;其他生化试剂购自美国Amresco公司;引物合成以及DNA测序由Invitrogen Life Technology公司完成。
1.2 方 法 1.2.1 胶原酶ColR75E的序列分析及结构预测根据基因序列colR75E(GenBank:KP987200),利用ExPASy中Translate tool(http://web.expasy.org/translate/)将该DNA序列翻译成氨基酸序列,进一步使用ProtParam tool(http://web.expasy.org/protparam/)预测该蛋白的理化性质,利用NCBI的BLAST工具及Phyre在线分析平台(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)预测该蛋白的结构域及三维结构[8]。
1.2.2 构建胶原酶ColR75E的原核表达载体根据目的基因colR75E序列设计相应的扩增引物:上游引物colR75E-F为5′- CGCGGATCCATGAACAAGAAATCAAAGATCAATA-3′,下游引物colR75E-R为5′-CGCCTCGAGTTTTACTGACAATTCGTATGTTCC-3′(下划线标识的碱基序列依次分别为限制性内切酶BamHⅠ与XhoⅠ的识别位点)。以蜡样芽孢杆菌R75E的基因组DNA为模板进行PCR扩增,对扩增产物纯化后用限制性内切酶BamHⅠ与XhoⅠ双酶切过夜,随后用T4 DNA连接酶将目的基因连接至载体pET28a上,将连接产物转化至感受态大肠杆菌Trans10中,培养后挑取阳性重组克隆提质粒后经酶切验证,并送至Invitrogen Life Technology公司测序。
1.2.3 ColR75E的表达及纯化将测序验证的重组质粒pET28a/colR75E转化至大肠杆菌BL21(DE3)中,将该重组菌接种于5 ml LB液体培养基(含34μg/ml卡那霉素),37℃下培养12 h。随后,将上述培养物按1%的接种量接种至1 L LB液体培养基中,于37℃下振荡培养至OD600nm=0.6,并向培养物加入终浓度为0.5 mmol/L的IPTG,在20℃、150r/min的条件下诱导培养12 h。将培养液离心(12 000r/min,30min)后用20mmol/L PBS(含500 mmol/L NaCl,1 mmol/L PMSF,pH7.4)缓冲液重悬细胞沉淀,在冰水浴中超声破碎,12 000 g离心30 min后取上清,进行His标签亲和层析Ni-NTA纯化,并采用BradFord法测定纯化产物的蛋白质含量[9]。
1.2.4 ColR75E的电泳检测对1.2.3 节中所得的纯化产物进行SDS-PAGE及胶原酶谱检测。其中,胶原酶谱法为将浓度为0.1%(W/V)牛跟腱来源的Ⅰ型胶原蛋白加入SDS-PAGE凝胶中[10, 11],4℃、110V低温恒压电泳,电泳结束后将凝胶先置于洗脱液(50mmol/L Tris-HCl,pH7.5,2.5% Triton X-100,5mmol/L CaCl2)中振荡洗脱2次(60r/min),每次40 min;然后用漂洗液(除TritonX-100 外其余成分同洗脱液)漂洗2次(60 r/min),每次20 min;然后,将凝胶置于孵育液(50 mmol/L Tris-HCl,pH7.5,5 mmol/L CaCl2,0.02% Brij-35)中于37℃静置孵育15 min;最后,进行考马斯亮蓝染色(内含1%考马斯亮蓝R250、45%甲醇及10%乙酸的水溶液),以及甲醇-乙酸溶液(含10%甲醇以及10%乙酸的水溶液)脱色至条带清晰。
1.2.5 ColR75E的酶学性质分析ColR75E胶原酶活性测定采用目前通用的Mandl法[12, 13, 14],其酶活力单位(U)定义为:在37℃、pH 7.5以及0.36 mmol/L Ca2+存在的条件下每分钟分解Ⅰ型胶原蛋白(牛筋腱来源)释放1 μmol游离氨基酸所需的酶量为一个酶活力单位(1U)。其中,游离氨基酸的含量测定采用茚三酮法,以甘氨酸作为标准氨基酸,测定OD570nm处光吸收值,制备标准曲线。胶原酶的比活力计算为酶活力单位数除以通过BradFord法测得的等体积酶液中的蛋白质质量(单位:U/mg)。
(1)最适温度及其热稳定性:首先在不同反应温度(30℃、35℃、37℃、40℃、45℃、50℃、55℃、60℃、65℃和70℃)条件下测定ColR75E胶原酶活性以确定ColR75E胶原酶的最适反应温度。其次,将ColR75E胶原酶液在上述不同温度条件下预先处理1h,随后在最适反应温度下测定ColR75E的酶活力研究其热稳定性,以未预先处理的冷藏酶液的活力作为对照(即相对活力为100%)。
(2)最适pH及其酸碱稳定:首先,在不同反应pH值(pH3、pH 4、pH 5、pH 6、pH 7、pH 7.5、pH 8、pH 9、pH 10)及最适反应温度条件下测定ColR75E胶原酶的活性以确定ColR75E胶原酶的最适反应pH。其次,将ColR75E胶原酶溶于上述不同pH值的缓冲液中,于室温下预处理1 h,在最适温度及最适pH条件下测定ColR75E的酶活力研究其pH稳定性,以未预先处理的冷藏酶液的活力作为对照(即相对活力为100%)。其中所用不同pH的缓冲液为:pH3.0~6.0为乙酸钠-乙酸缓冲液(50 mmol/L),pH7.0~9.0为Tris-HCl缓冲液(50 mmol/L),pH10.0为Na2CO3-NaHCO3缓冲液(50 mmol/L)。
(3)不同金属离子或蛋白酶抑制剂对ColR75E酶活性的影响:在反应体系中加入不同的金属离子(K+、Na+、Zn2+、Ca2+、Fe2+、Mg2+、Mn2+、Pb2+、Cu2+,工作浓度为5 mmol/L)或者不同类型蛋白酶的抑制剂(EDTA、EGTA、PMSF、β-巯基乙醇、DTT、碘乙酸,工作浓度为1 mmol/L)。在最适反应条件下,测定上述反应体系中ColR75E的胶原酶活力,以未加金属离子或蛋白酶抑制剂的反应体系测得的活力为对照(即相对活力为100%)。
(4)酶动力学参数Km和Vmax的测定:在最适反应条件下,测定在含有不同浓度(0.5、1、2、4、6、8、10 mg/ml)Ⅰ型胶原蛋白底物的反应体系中,在最适反应条件下测定ColR75E的反应初速度(反应30 min测得的平均速度),经绘制Lineweaver-Bürk双倒数曲线,得出ColR75E对Ⅰ型胶原蛋白底物的Km和Vmax值。
1.2.6 ColR75E对鱼鳞中Ⅰ型胶原蛋白的降解作用为了分析ColR75E对Ⅰ型胶原蛋白的降解作用,采用胃蛋白酶提取法提取草鱼鱼鳞中的Ⅰ型胶原蛋白[15]。按照40∶1体积比将草鱼鱼鳞中提取的Ⅰ型胶原蛋白溶液(1 mg/ml,溶于50 mmol/L PBS,pH7.5)与ColR75E酶液(62.14 μg/ml)混合,于37℃的反应条件下在不同时间点取样,按照体积比为1∶5的比例将5×SDS上样缓冲液加入反应体系终止反应,于沸水浴加热10 min后进行浓度为12.5%的SDS-PAGE凝胶电泳以及考马斯亮蓝染色,检测上述反应产物的分子量变化。
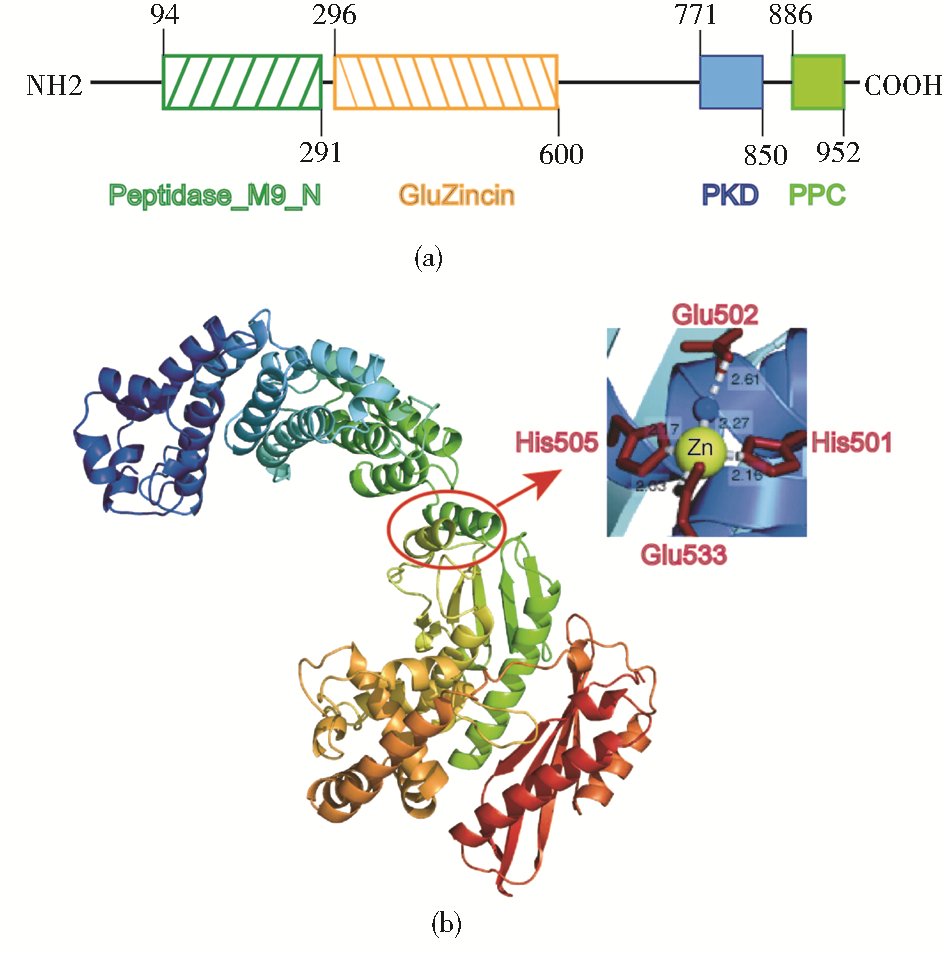
2 结 果 2.1 胶原酶ColR75E的理化性质以及结构预测colR75E基因序列(GenBank:KP987200)全长2 989 bp,可编码965个氨基酸残基。首先,由ProtParam在线分析平台预测结果显示:ColR75E胶原酶的理论分子量为110 076.4 Da,等电点pI为5.30,不稳定指数Ⅱ为29.28,脂溶指数为70.10,总平均疏水指数(GRAVY)为-0.692,表明该蛋白为稳定的亲水性蛋白。BLASTq分析结果表明ColR75E胶原酶含有如图 1a所示的4个保守的结构域:从94至291位氨基酸残基序列的M9肽酶家族N端结构域(Peptidase family M9 N-terminal);从296至600位氨基酸残基序列的谷氨酸-锌肽酶家族结构域(Peptidase Gluzincin family)、从771至850位氨基酸残基序列的多囊性肾病蛋白结构域(Polycystic kidney disease-like domain)以及从886至952位氨基酸残基序列的细菌前肽酶C末端结构域(bacterial pre-peptidase C-terminal)。上述4个结构域均是与降解或结合胶原蛋白相关的已知结构域[16]。Phyre在线分析平台以胶原酶Col G(序列同源性69%,2.8)[6]为模板模拟了ColR75E如图 1b所示的镊子状三维结构,同时由Phyre2的分析报告[17]可知ColR75E的活性中心位于中部Leu463-Leu546肽段上的两个α螺旋结构中。其中,第一个α螺旋结构中的His501、 His505以及通过氢键结合1个水分子的Glu502与相邻的α螺旋结构中Glu533四者作为配基共同结合一个Zn2+,由此构成了活性中心[6, 17]。
以蜡样芽孢杆菌R75E的基因组DNA为模板,用引物colR75E-F和colR75E-R以58℃为退火温度进行PCR扩增。扩增产物经1%琼脂糖凝胶电泳的结果如图 2a所示,在泳道中可观察到分子量约为3kb的亮带,表明colR75E基因(2 898 bp)扩增成功。将扩增的colR75E基因克隆至pET28a质粒。质粒的酶切鉴定结果如图 2b所示,pET28a/colR75E重组质粒酶切后产生约5 kb的空质粒条带和接近3 kb的目标基因条带。测序结果表明重组质粒的阅读框正确,表明重组质粒pET28a/colR75E构建成功。

|
| 图 2 colR75E基因PCR扩增产物琼脂糖凝胶(a)及重组质粒pET28a/colR75E BamHⅠ与XhoⅠ双酶切鉴定琼脂糖凝胶电泳(b) Fig. 2 Agarose gel electrophoresis of colR75E PCR products (a)and agarose gel electrophoresis of recombinant pET28a/colR75E plasmid cut with BamHⅠ and XhoⅠ(b) M:DNA marker;1:Products |
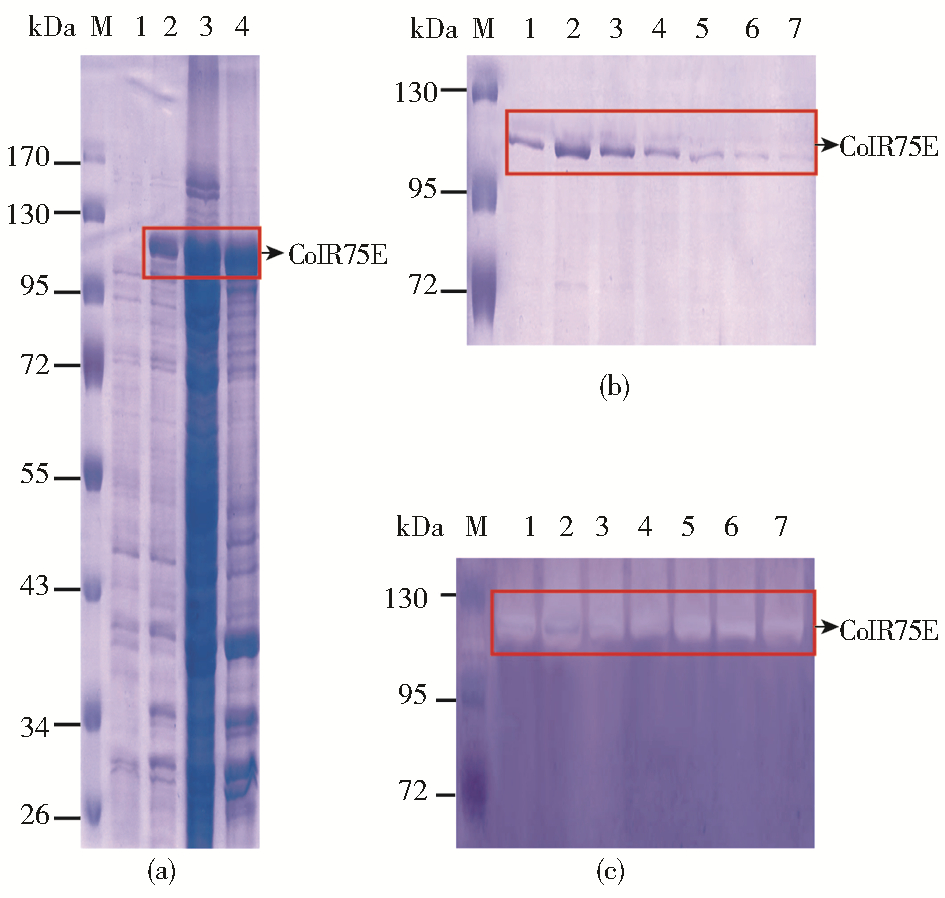
将重组质粒pET28a/colR75E转化至感受态大肠杆菌BL21(DE3)中,获得的重组菌可用于表达融合有His亲和标记物的ColR75E胶原酶蛋白。ColR75E胶原酶蛋白诱导表达的结果如图 3a所示,在经浓度为0.5 mmol/L的IPTG诱导后,与诱导前菌体相比(泳道1),表达了一个约114 kDa的蛋白(泳道2),这与预期的目标蛋白的分子量基本一致(ColR75E+His亲和标签)。将诱导后的重组菌体超声破碎后的沉淀(泳道3)以及上清液(泳道4)进行SDS-PAGE检测表明,诱导表达的ColR75E重组蛋白大量存在于上清液中,表明在大肠杆菌中表达的重组ColR75E胶原蛋白可溶。
|
| 图 3 大肠杆菌表达重组ColR75E胶原酶蛋白的可溶性检测(a)及重组ColR75E胶原酶蛋白的SDS-PAGE电泳(b)以及胶原酶谱检测结果(c)< Fig. 3 Solubility detection of recombinant ColR75E collagenase expressed in E.coli BL21 (DE3) (a) and analysis of SDS-PAGE (b) and collagen-zymography of purified recombinant ColR75E collagenase expressed in E.coli BL21 (DE3) with Ni-NTA column (c) (a) 1: The whole protein of E. coli BL21 (DE3) without induction; 2: The whole protein of E. coli BL21 (DE3) after IPTG induction; 3: The precipitated fraction after sonication of E. coli BL21 (DE3) after IPTG induction; 4: The supernatant fraction after sonication of E. coli BL21 (DE3) after IPTG induction (b)(c) 1~7 represents the number of elution tubes of recombinant ColR75E collagenase |
进一步利用Ni-NTA镍柱纯化大肠杆菌破碎上清液中的重组ColR75E胶原酶蛋白后,对所得重组蛋白进行SDS-PAGE电泳检测,结果如图 3B所示:凝胶上可观察到明显的纯化出来的ColR75E胶原酶蛋白条带,表明含有His标签的重组ColR75E胶原酶蛋白纯化成功。进一步对得到的重组蛋白进行胶原酶谱检测,结果如图 3C所示,可在含有Ⅰ型胶原蛋白的凝胶上观察到由于纯化的重组胶原酶的降解而产生的透明的负染条带(泳道1-7),表明经Ni-NTA镍柱纯化得到的蛋白确为重组ColR75E胶原蛋白酶。将所得重组蛋白集中在一起进行透析去除洗脱液中的咪唑等盐类,将所得的纯化产物经BradFord法[9]测定蛋白浓度为62.14 μg/ml。综上结果表明,利用大肠杆菌做为表达菌株获得有活性的重组胶原酶ColR75E是可行的。
2.4 重组ColR75E胶原蛋白酶的活性分析将纯化所得的重组ColR75E胶原蛋白酶作为待测溶液,测定其胶原酶活力。结果显示,在37℃、pH 7.5及存在0.36 mmol/L Ca2+的标准测活条件下ColR75E的比活力达到了3.62 U/mg。
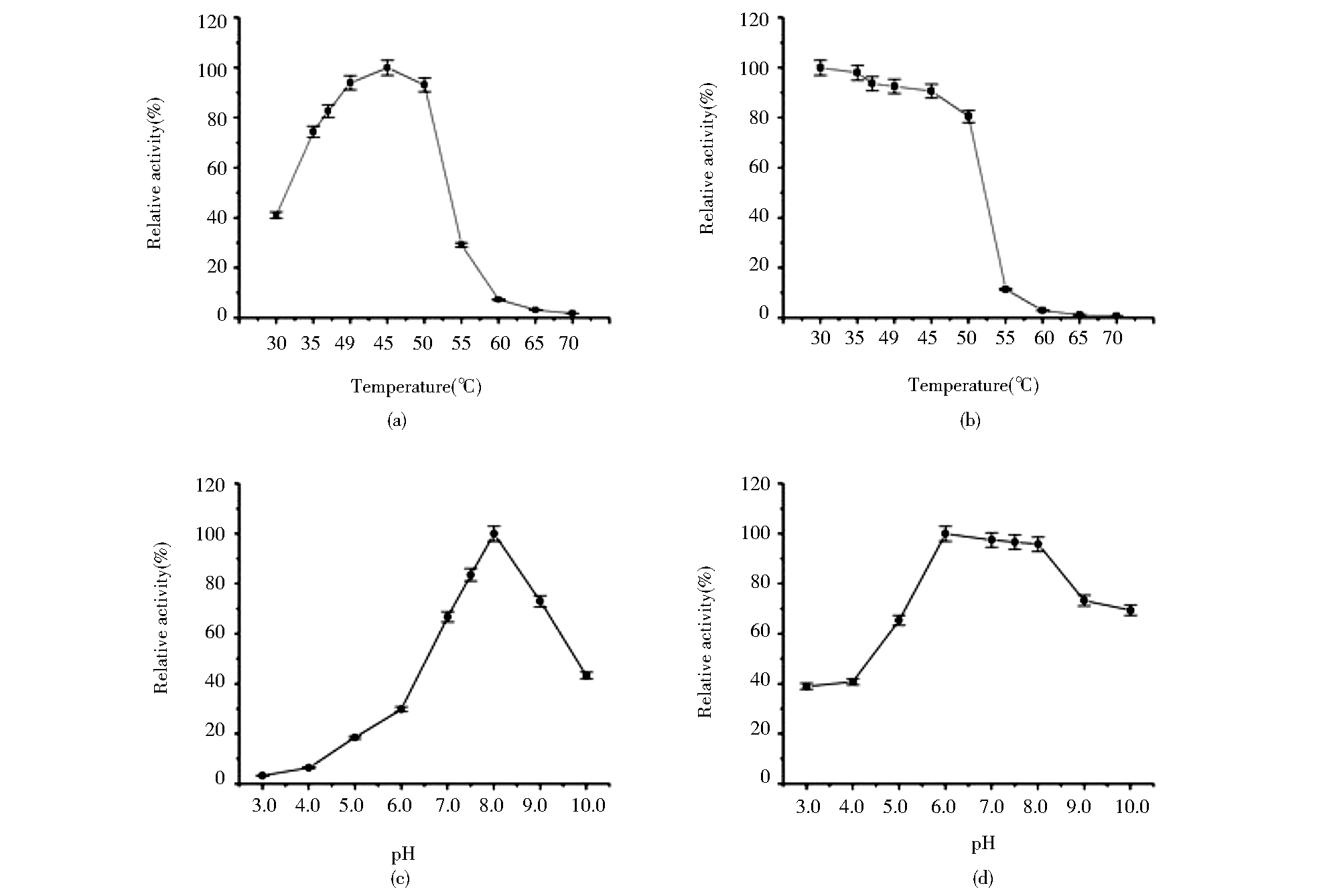
2.4.1 温度对ColR75E酶活性的影响在不同反应温度(30℃、35℃、37℃、40℃、45℃、50℃、55℃、60℃、65℃和70℃)下,ColR75E胶原蛋白酶的酶活力测定结果如图 4a所示,结果表明在30~45℃的温度范围内重组ColR75E胶原酶的酶活力随着反应温度上升而逐步提高,至45℃时达到最高值;在50℃时仍可以保持90%以上的酶活力。然而,当温度高于50℃时,酶活力迅速下降并最终丧失活性,表明45℃为重组ColR75E胶原酶的最佳反应温度。此外,将ColR75E在不同温度下处理1 h后测得的酶活力结果如图 4b所示,在30~45℃的温度范围内重组ColR75E胶原酶酶活力能够保持90%以上;在50℃时处理1 h的条件下重组ColR75E胶原酶的酶活力仍能够保持80%的活性左右,而当用高于50℃的温度(55℃,60℃,65℃,70℃)预处理后1h后,随着温度的升高,重组ColR75E胶原酶的酶活力显著降低,至65℃时完全失去活性。该结果表明,在低于50℃的温度范围内,重组ColR75E胶原酶可保持较高的稳定性。
|
| 图 4 不同反应温度对重组ColR75E胶原酶活力的影响 (a)及其热稳定性分析(b)和不同反应pH值对其活力影响(c)以及pH稳定性分析(d) Fig. 4 Effect of various temperature on the activity of recombinant ColR75E collagenase (a)and temperature stability analysis (b)and effect of various pH on the activity of recombinant ColR75E collagenase (c) and pH stability analysis of recombinant ColR75E collagenase(d) |
重组ColR75E胶原酶在不同pH反应体系中的酶活力如图 4c所示:在pH3.0~6.0的条件下,重组ColR75E胶原酶的酶活力较低,均低于最高值的30%;随着pH值的升高,重组ColR75E胶原酶的酶活力开始逐渐提高,直至pH8.0时其酶活力达到最高值;而当进一步提高反应溶液的pH值后,酶活力开始显著下降。例如pH9.0时活力下降27.2%,而至pH10.0时活力下降57.6%。上述结果表明,重组ColR75E胶原酶的最佳反应pH值为8.0。此外,为了测定重组ColR75E胶原酶对溶液酸碱度的耐受性,将重组ColR75E胶原酶用不同pH的溶液预处理1h后进行活力测定,结果如图 4d所示:在pH3.0~6.0的预处理条件下,随着pH的逐渐升高,重组ColR75E胶原酶的酶活力也逐渐提高;经pH6.0预处理后的酶具有最高的酶活力;而经pH7.0~8.0处理后酶活力也均保持在95%以上;然后,当pH继续升高至pH9.0~10.0后,重组ColR75E胶原酶的酶活力开始下降,但仍保持最高活力的70%左右。该结果表明,重组ColR75E胶原酶在pH6.0~8.0的条件下具备最好的稳定性,而与酸性溶液相比,该酶对于碱性溶液具有更高的耐受性。
2.4.3 不同金属离子以及蛋白酶抑制剂对重组ColR75E胶原酶酶活性的影响以不添加任何金属离子的反应体系测得的相对活力作为100%,当向反应体系内加入5 mmol/L不同类型的金属离子后测得的重组ColR75E胶原酶酶活力结果如表 1所示,Ca2+可显著促进该酶酶活性,将其酶活力提高了48.60%;Na+、K+、Mg2+对重组ColR75E胶原酶的酶活性影响不大,相对酶活力差异均小于5%;而Zn2+、Mn2+、Fe2+、Pb2+对重组ColR75E胶原酶酶活性有不同程度的抑制作用,其抑制能力Pb2+>Zn2+>Fe2+>Mn2+。Cu2+与Ag+则完全抑制重组ColR75E胶原酶的酶活性。而在所有蛋白酶抑制剂中,EDTA、EGTA可显著抑制重组ColR75E胶原酶的酶活性;与此不同,PMSF、β-巯基乙醇、DTT、碘乙酸等处理后,该酶活力仍可保持在85%以上,没有表现出敏感的抑制作用。总体来讲,重组ColR75E胶原酶的酶活性受金属螯合剂EDTA、EGTA的显著抑制,而对其他类型的蛋白酶抑制剂PMSF、β-巯基乙醇、DTT、碘乙酸不敏感,表明ColR75E胶原酶为金属蛋白酶。
| Reagent | Relative activity(%) | Reagent | Relative activity(%) |
| Na+ | 101.74 | Cu2+ | 0 |
| K+ | 100.00 | Ag+ | 0 |
| Zn2+ | 18.72 | EDTA | 11.15 |
| Ca2+ | 148.60 | EGTA | 23.82 |
| Fe2+ | 20.30 | PMSF | 95.50 |
| Mg2+ | 95.03 | βME | 86.94 |
| Mn2+ | 58.89 | DTT | 93.47 |
| Pb2+ | 2.59 | Iodoacetic acid | 89.70 |
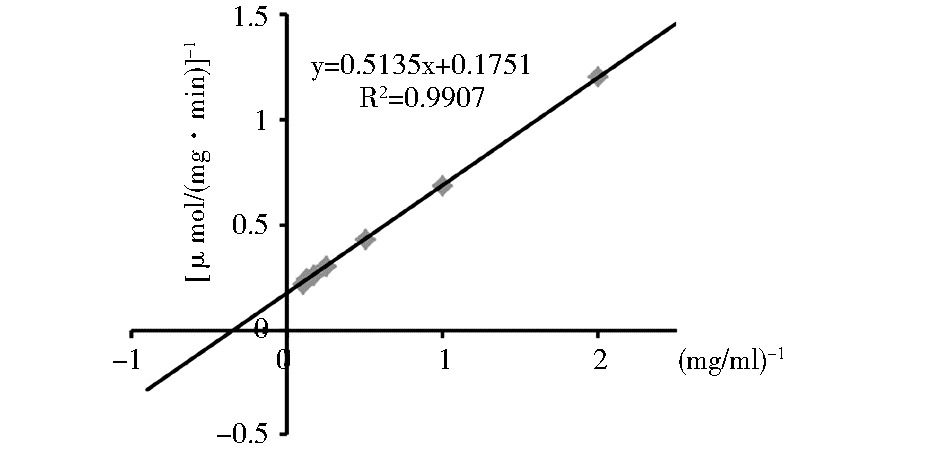
测定含有不同浓度(0.5、1.0、2.0、4.0、6.0、8.0、10.0 mg/ml)Ⅰ型胶原蛋白底物的反应体系中重组ColR75E胶原酶的反应初速度,随后绘制出如图 5所示的Lineweaver-Bürk双倒数曲线图,由该图可知:重组ColR75E胶原酶对于Ⅰ型胶原蛋白的最大反应速度Vmax值为5.71 μmol/(mg·min)、米氏常数Km值为25.55 μmol/L (即2.93 mg/ml)。
|
| 图 5 重组ColR75E胶原酶对于Ⅰ型胶原蛋白底物的Lineweaver-Bürk双倒数曲线图 Fig. 5 Lineweaver-Bürk plot of recombinant ColR75E collagenase to type Ⅰ collagen substrate |
重组ColR75E对草鱼鱼鳞中Ⅰ型胶原蛋白降解产物的SDS-PAGE电泳检测结果如图 6所示:图中泳道1为反应0 min的对照,在该泳道中可以清晰地观察到构成Ⅰ型胶原蛋白三股螺旋的肽链:两条重叠在一起的α1链(分子量为126 kDa)与一条α2链(分子量为116 kDa),此外还有一条高分子量的β链(α链的二聚体)[15];图中泳道2~8分别为重组ColR75E胶原酶与Ⅰ型胶原蛋白分别反应10 min、20 min、30 min、40 min、50 min、60min、120 min后的产物,该结果表明随着反应时间的增加,Ⅰ型胶原蛋白的α1链、α2链、β链逐渐减少,而分子量较小的水解肽比例逐渐增加,反应进行至120 min时Ⅰ型胶原蛋白中的α1链、α2链、β链几乎完全消失。该结果表明重组ColR75E胶原酶能够将天然构象的Ⅰ型胶原纤维完全降解为肽段。

|
| 图 6 重组ColR75E胶原酶对草鱼鱼鳞中Ⅰ型胶原蛋白水解产物的SDS-PAGE电泳检测 Fig. 6 Degradation of the native type I collagen extracted from scales of grass carp by recombinant ColR75E collagenase analyzed by SDS-PAGE |
本研究实现了蜡样芽孢杆菌colR75E胶原酶基因在大肠杆菌中的高效表达,而且重组colR75E胶原酶蛋白可溶性高。利用Ni-NTA亲和层析柱得到了高纯度蛋白,其比活力在标准条件下达到了3.62 U/mg,该活力显著高于Nagano等[13]于2000年报道的枯草芽孢杆菌FS-2所产的胶原酶(0.71 U/mg)的活力。最适反应条件分析表明,ColR75E胶原酶的最适反应温度为45℃。而该酶具有一定的温度耐受性,50℃处理1h后仍然保持80%以上的酶活。该结果与Lima等[18]及Kaminishi等[19]的研究结果相似,他们各自所发现的白色念珠菌(Candida albicans)所产的胶原酶最适反应温度也均为45℃,且在50℃的温度处理1 h后活力能够保持70%左右。表明ColR75E与报道的胶原酶的最适温度以及温度耐受性相似,且温度稳定性略高于上述两种胶原酶。目前,已经报道的常见的微生物胶原酶的最佳反应pH值一般都在 7.0以上,例如:Lima等[18]发现的胶原酶URM3622的最适反应pH为8.2,Suphatharaprateep等[20]发现的微生物胶原酶CNA1的最适反应pH为7.5,ColR75E也不例外,其最适反应pH为8.0,且在pH6.0~10.0的酸碱范围内都很稳定,可以保持超过70%以上的酶活性,是一种pH适用范围比较广泛的微生物胶原酶。
本研究将ColR75E的序列分析与结构预测与酶活力测定结果比较表明:(1)Ca2+可显著激活ColR75E胶原酶的活性,推测这是由于ColR75E胶原酶的PKD结构域,以及PPC结构域均依赖于Ca2+的激活,这与已经报道的微生物胶原酶的特点相符[6, 21],这两个结构域均与胶原蛋白底物的的结合密切相关,Ca2+通过激活这两个结构域增加与底物蛋白的结合,提高酶催化反应的效率;(2)Zn2+可明显抑制ColR75E胶原酶的活性,这是由于除了ColR75E本身自带的一个Zn2+之外,多余的Zn2+会严重影响ColR75E胶原酶活性中心的构象[6, 22],导致酶催化活性的下降;(3)EDTA与EGTA这两个金属螯合剂均可螯合Zn2+与Ca2+从而显著抑制ColR75E胶原酶的活性,进一步证实了ColR75E胶原酶是一种金属蛋白酶,这与结构预测的结果一致。综上所述,表明ColR75E胶原酶与胶原酶ColG一样,是一种依赖Ca2+与Zn2+的Ⅰ型胶原酶,属于金属蛋白酶一类[6, 23]。
目前,我国在微生物胶原酶的应用还处于刚起步阶段,由于高纯度胶原酶的提取工艺复杂,生产成本高,国内市场依赖大量高价进口胶原酶产品,限制了微生物胶原酶的应用。因此,重点研究ColR75E胶原酶的工业应用具有广泛的应用前景。
| [1] | Harrington D J. Bacterial collagenases and collagen-degrading enzymes and their potential role in human disease. Infect Immun, 1996, 64(6): 1885-1891. |
| [2] | 金敏, 李君文, 王忠彦. 微生物胶原酶研究进展. 氨基酸和生物资源, 2003, 25(1): 3-8. Jin M, Li J W, Wang Z Y. Researches on collagenase secreted by microbes. Amino Acids and Biotic Resources, 2002, 25(1): 3-8. |
| [3] | Mandl I. Bacterial collagenases and their clinical applications. Arzneimittelforschung, 1981, 32(10a): 1381-1384. |
| [4] | Van Wart H E, Steinbrink D R. Complementary substrate specificities of class I and class II collagenases from Clostridium histolyticum. Biochemistry, 1985, 24(23): 6520-6526. |
| [5] | Matsushita O, Jung C M, Katayama S, et al. Gene duplication and multiplicity of collagenases in Clostridium histolyticum. J Bacteriol, 1999, 181(3): 923-933. |
| [6] | Eckhard U, Schönauer E, Nüss D, et al. Structure of collagenase G reveals a chew-and-digest mechanism of bacterial collagenolysis. Nat Struct Mol Biol, 2011, 18(10): 1109-1114. |
| [7] | Lee P S, Lee K H. Escherichia coli——a model system that benefits from and contributes to the evolution of proteomics. Biotechnol Bioeng, 2003, 84(7): 801-814. |
| [8] | 刘阳, 杨雅麟, 张宇婷, 等. 维氏气单胞菌B565β-N-乙酰氨基葡萄糖苷酶的表达, 纯化及酶学性质. 中国生物工程杂志, 2015, 35(2): 38-44. Liu Y, Yang Y L, Zhang Y T, et al. Expression, purification and characterization of β-N-acetylglucosaminidase from Aeromonasveronii B565. China Biotechnology, 2015, 35(2): 38-44. |
| [9] | Bradford M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72(1): 248-254. |
| [10] | Gogly B, Groult N, Hornebeck W, et al. Collagen zymography as a sensitive and specific technique for the determination of subpicogram levels of interstitial collagenase. Anal Biochem, 1998, 255(2): 211-216. |
| [11] | Bildt M M, Bloemen M, Kuijpers-Jagtman A M, et al. Matrix metalloproteinases and tissue inhibitors of metalloproteinases in gingival crevicular fluid during orthodontic tooth movement. Eur J Orthodont, 2009, 31(5): 529-535. |
| [12] | Mandl I, MacLennan J D, Howes E L, et al. Isolation and characterization of proteinase and collagenase from C. histolyticum. J Clin Invest, 1953, 32(12): 1323-1329. |
| [13] | Nagano H, To K A. Purification of collagenase and specificity of its related enzyme from Bacillus subtilis FS-2. Biosci Biotech Bioch, 2000, 64(1): 181-183. |
| [14] | Kim T, Silva J L, Parakulsuksatid P, et al. Optimization of enzymatic treatments for deskinning of catfish nuggets. J Aquat Food Prod T, 2014, 23(4): 385-393. |
| [15] | Liu D, Liang L, Regenstein J M, et al. Extraction and characterisation of pepsin-solubilised collagen from fins, scales, skins, bones and swim bladders of bighead carp (Hypophthalmich thysnobilis). Food Chem, 2012, 133(4): 1441-1448. |
| [16] | Wilson J J, Matsushita O, Okabe A, et al. A bacterial collagen-binding domain with novel calcium-binding motif controls domain orientation. Embo J, 2003, 22(8): 1743-1752. |
| [17] | Kelley L A, Mezulis S, Yates C M, et al. The Phyre2 web portal for protein modeling, prediction and analysis. Nat Protoc, 2015, 10(6):845-858. |
| [18] | Lima C A, Rodrigues P, Porto T S, et al. Production of a collagenase from Candida albicans URM3622. Biochem Eng J, 2009, 43(3): 315-320. |
| [19] | Kaminishi H, Hagihara Y, Hayashi S, et al. Isolation and characteristics of collagenolytic enzyme produced by Candida albicans. Infect Immun, 1986, 53(2): 312-316. |
| [20] | Suphatharaprateep W, Cheirsilp B, Jongjareonrak A. Production and properties of two collagenases from bacteria and their application for collagen extraction. New Biotechnol, 2011, 28(6): 649-655. |
| [21] | Duarte A S, Correia A, Esteves A C. Bacterial collagenases—A review. Crit Rev Microbiol, 2014,(in print). |
| [22] | Bodiga V L, Eda S R, Chavali S, et al. In vitro biological evaluation of glyburide as potential inhibitor of collagenases. Int J Biol Macromol, 2014, 70: 187-192. |
| [23] | La Rocca G, Pucci-Minafra I, Marrazzo A, et al. Zymographic detection and clinical correlations of MMP-2 and MMP-9 in breast cancer sera. Brit J Cancer, 2004, 90(7): 1414-1421. |
 2015, Vol. 35
2015, Vol. 35