文章信息
- 刘阳, 杨雅麟, 张宇婷, 冉超, 周志刚
- LIU Yang, YANG Ya-lin, ZHANG Yu-ting, RAN Chao, ZHOU Zhi-gang
- 维氏气单胞菌B565β-N-乙酰氨基葡萄糖苷酶的表达、纯化及酶学性质
- Expression, Purification and Characterization of β-N-acetylglucosaminidase from Aeromonas veronii B565
- 中国生物工程杂志, 2015, 35(2): 38-44
- China Biotechnology, 2015, 35(2): 38-44
- http://dx.doi.org/10.13523/j.cb.20150206
-
文章历史
- 收稿日期:2014-10-23
2. 中国农业科学院饲料研究所 北京 100081
2. Feed Research Institute of Chinese Academy of Agriculture Sciences, Beijing 100081, China
N-乙酰氨基葡萄糖(N-Acetylglucosamine,GlcNAc)是一种以β-1,4糖苷键聚合的单糖,是仅次于纤维素的第二大丰富的天然多聚物几丁质的单体[1]。N-乙酰氨基葡萄糖在细胞表面结构起着重要的作用,是细菌细胞壁肽聚糖、真菌细胞壁几丁质和动物细胞的细胞外基质的关键组分[2]。N-乙酰氨基葡萄糖有广阔的应用前景,N-乙酰氨基葡萄糖及其衍生物几丁质和壳聚糖是自然界中至关重要的有机碳源和氮源;它们还可被用作生物质来生产生物燃料如生物乙醇;在临床上用来治疗关节损伤疾病,是潜在的治疗炎症性肠病药物;此外,N-乙酰氨基葡萄糖还可用于食品添加及化妆品等领域[1]。
酶法生产N-乙酰氨基葡萄糖由于反应条件温和、效率高、产物纯度高,具有环境友好和安全等优点,是现代N-乙酰氨基葡萄糖产业发展的方向。β-N-乙酰氨基葡萄糖苷酶能利用几丁质酶降解产物生产N-乙酰氨基葡萄糖,但目前已报道的β-N-乙酰氨基葡萄糖苷酶普遍存在活性低(多在每毫克蛋白几毫酶活单位到几十酶活单位之间)的缺点[3, 4, 5, 6],急需筛选性质优良的β-N-乙酰氨基葡萄糖苷酶。本实验室从曾爆发粘孢子虫病又恢复正常养殖的池塘底泥中筛选出一株具有降解粘孢子虫几丁质壳瓣能力的细菌(维氏气单胞菌Aeromonas veronii B565),已完成全基因组测序[7],其中编码1个β-N-乙酰氨基葡萄糖苷酶基因。本研究克隆了A. veronii B565的β-N-乙酰氨基葡萄糖苷酶基因nag565,在大肠杆菌中进行了表达纯化,进一步对其酶学性质进行了研究,该酶表现出非常高的活性,为深入研究酶的催化机理、分子改造及生产N-乙酰氨基葡萄糖储备了良好的材料,为实现β-N-乙酰氨基葡萄糖苷酶及N-乙酰氨基葡萄糖在水产、医药和食品等领域的应用奠定了基础。 1 材料与方法 1.1 材 料 1.1.1 菌株与质粒
维氏气单胞菌(A. veronii)B565为本实验室保存;感受态细胞E.coli BL21(DE3)购自北京全式金公司;载体pET28a(+)购自Invitrogen公司。 1.1.2 工具酶与试剂
基因组DNA提取试剂盒、质粒小提中量试剂盒、DNA纯化试剂盒均购自天根公司;各种限制性内切酶和DNA聚合酶购自TaKaRa公司;T4 DNA Ligase 购自NEB公司。 1.2 方 法 1.2.1 β-N-乙酰氨基葡萄糖苷酶基因全长序列的分析及结构预测
根据维氏气单胞菌B565全基因组序列(Genbank:CP002607)中的β-N-乙酰氨基葡萄糖苷酶nag565基因序列,利用ExPASy中Translate tool(http://web.expasy.org/translate)将DNA序列翻译成氨基酸序列。应用Signal P4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)对氨基酸序列进行信号肽分析。使用ProtParam tool(http://www.expasy.ch/cgi-bin/protparam)预测该蛋白的理化性质。使用BLASTp工具对氨基酸序列进行比对,查找相似性较高的氨基酸序列,Pfam网站(http://pfam.sanger.ac.uk/search)预测蛋白质的结构域。应用SWISS-MODEL(http://swissmodel.expasy.org/workspace/)预测该蛋白的三维结构。 1.2.2 原核表达载体nag565-pET28a(+)的构建
根据β-N-乙酰氨基葡萄糖苷酶nag565基因序列设计特异性引物:
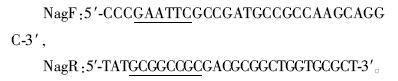
下划线分别为限制性酶切位点EcoR I和Not I。以提取的维氏气单胞菌B565基因组DNA为模板,进行PCR扩增,获得不含信号肽的目的基因。对PCR产物进行纯化后用限制性内切酶EcoR I和Not I进行双酶切,用T4 DNA连接酶将目的基因连接到用相同限制性内切酶处理过的载体pET28a(+)上。连接产物转化感受态BL21(DE3),挑取阳性重组克隆提质粒后经酶切验证,送至北京擎科新业公司测序。 1.2.3 NAG565的诱导及纯化
将经过酶切验证及测序正确的重组质粒nag565-pET28a(+)转化至E.coli BL21(DE3)感受态细胞,同时转化pET28a(+)空载体作为对照。将重组质粒接种于3ml液体LB培养基(含100 μg/ml卡那霉素),37℃振荡培养过夜,以1∶100的比例将菌液转接至液体LB培养基(含100 μg/ml卡那霉素)中,37℃振荡培养约3h(OD600=0.6),加入诱导剂IPTG,使其终浓度为1mmol/L,18℃、220r/min振荡培养12h。将培养液10 000r/min离心5min,收集细胞沉淀。用pH8.0、20mmol/L的Tris-HCl缓冲液重悬后超声破碎,离心后取上清,进行His标签融合蛋白亲和层析Ni-NTA纯化,SDS-PAGE检测。 1.2.4 NAG565酶活力测定
将pNP-GlcNAc溶于0.1mol/L磷酸二氢钠-柠檬酸缓冲液中,使其终浓度为1mmol/L,即为底物溶液。将10μl酶液、240μl 20mmol/L的磷酸氢二钠-柠檬酸缓冲液(pH7.0)和250μl 底物溶液在试管中混合并摇匀,37℃温育5min,然后加入2ml 0.5mol/L NaOH溶液终止反应,在405nm处测OD值,对照管在试管中依次加入10μl待测溶液,240μl缓冲液、2ml 0.5mol/L NaOH水溶液和250μl 底物溶液,在405nm处测OD值。以生成的对硝基酚(pNP)的量表示酶活力。每组平行测定 3 次,结果取平均值。在37℃条件下,每分钟分解pNP-GlcNAc释放1 μmol pNP所需的酶量定义为一个酶活力单位(1U)。 1.2.5 NAG565蛋白浓度的测定
采用Bradford[8]法测定NAG565蛋白质含量,以BSA(1mg/ml)作为标准蛋白。 1.2.6 NAG565酶学性质测定
(1)最适pH及pH稳定性:纯化后的 NAG565 酶液在不同 pH 3.0~11.0 条件下进行酶促反应,测定酶活性,以研究其最适 pH。将NAG565 纯酶液溶于不同 pH 的缓冲液中,37℃ 处理 1 h,在最适 pH 条件测定酶活性以研究酶的 pH 稳定性。酶活测定中分别采用如下缓冲液:pH3.0~4.0的0.1mol/L的乙酸钠缓冲液,pH5.0~7.0、0.1mol/L的磷酸氢二钠-柠檬酸缓冲液pH8.0~9.0的0.1mol/L的Tris-HCl缓冲液,pH10.0~11.0的0.1mol/L的甘氨酸-NaOH缓冲液。
(2)最适温度及温度稳定性:在最适 pH 缓冲液及不同温度(0℃、20℃、30℃、37℃、50℃、60℃和70℃)条件下测定酶活性以确定酶的最适反应温度。 在上述不同温度条件下将酶液处理 30 min,最适反应温度及最适 pH 环境中测定酶活性以研究其热稳定性,以未经过处理的酶液作为对照。
(3)不同金属离子或化学试剂对NAG565酶活的影响:在反应体系中加入不同的金属离子或化学试剂(K+、Na+、Ca2+、Li+、Co2+、Cr3+、Ni2+、Cu2+、Mg2+、Fe2+、Mn2+、Zn2+、Ag+、Hg2+、EDTA、SDS和β-巯基乙醇;终浓度均为5 mmol/L),以未加金属离子和化学试剂的反应体系作为对照。采用pH7.0、0.1mmol/L的磷酸氢二钠-柠檬酸缓冲液。
(4)不同蛋白酶对NAG565酶活力的影响:NAG565蛋白与不同蛋白酶(胰蛋白酶、胶原蛋白酶、糜蛋白酶、胃蛋白酶、蛋白酶K)按10∶1(质量比)在该蛋白酶溶液中处理30min后,按酶活测定方法检测处理酶的剩余酶活,以未加蛋白酶的反应体系作为对照(CK)。
(5)动力学参数Km及Vmax的测定:在最适反应条件下,测定NAG565对不同浓度(1、2、5、10、50、100、500、1000μmol/L) 的三种底物pNP-GlcNAc、pNP-(GlcNAc)2和pNP-(GlcNAc)3的反应初速度,经Lineweaver-Burk 绘图得出该酶对3种底物的Km和Vmax值。
(6)底物特异性:在最适条件下,底物采用4 -甲基伞形酮-N-乙酰基-α-D-氨基葡糖苷、4-甲基伞形酮-α-D-葡糖苷、4-甲基伞形酮-α-D-半乳糖、4-甲基伞形酮-α-D-木糖苷、4-甲基伞形酮-α-D-吡喃葡萄糖苷、乙二醇壳聚糖、羧甲基纤维素、壳聚糖粉末代替pNP-GlcNAc,测定纯化后NAG565的活性。 2 结果与讨论 2.1 nag565的序列分析及结构预测
该基因编码887个氨基酸,ProtParam预测结果显示:该蛋白分子理论分子量为98148.8Da,等电点PI为5.69,不稳定指数II为24.66,脂溶指数为74.77,总平均疏水指数(GRAVY)为-0.402,表示该蛋白为稳定的亲水性蛋白。Blast同源比对结果表明NAG565与气单胞菌属的β-N-乙酰氨基葡萄糖苷酶相似性高(85%到99%),与其它属相似性低,如与发光杆菌属、弧菌属、爱德华氏菌属的相似性为55%以下。应用Signal P4.1方法分析氨基酸序列,结果显示该蛋白含有一个信号肽,其切点在A22~A23之间,表明该蛋白为可分泌蛋白。如图 1,SWISS-MODEL分析表明NAG565含4个推测的保守的结构域:碳水化合物结合结构域 (CHB_HEX残基:37~198)、糖苷水解酶20家族结构域(Glyco_hydro_20b:残基215~330)、糖苷水解酶20家族催化结构域(Glyco_hydro_2:残基333~768)、β-氨基葡萄糖苷酶 C端结构域(CHB_HEX_C:残基816~887)。以粘质沙雷氏菌Serratia marcescens的β-N-乙酰氨基葡萄糖苷酶1C7T_A (序列同源性54.37%,1.9)为模板[9],预测模拟的NAG565催化域是一个a/b TIM-barrel,含有糖基水解酶20家族的保守氨基对(Asp533-Glu534),预测的活性为点R340、H446、V487、Q488、D533、E534、W615、W638、Y668、D670、F671、Y682、W684、W736、E738。
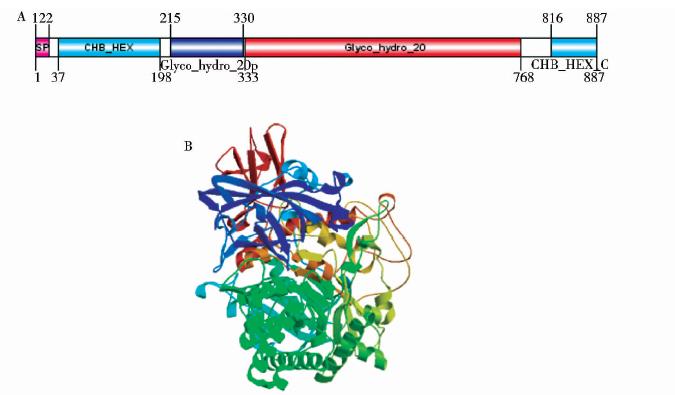
|
| 图 1 NAG565的模块结构(A)及以1C7T_A (54.37% sequence identity,1.9Å)为模板的NAG565三级结构预测(B) Fig. 1 Schematic representation of the modular structure of NAG565 and the predicted three-dimensional structure of NAG565 based on template 1C7T_A (54.37% sequence identity,1.9Å)NAG565 has one signal peptide (SP,residue:1-22)and four predicted conserved domains: Putative carbohydrate binding domain (CHB_HEX residue:37-198)、 Glycosyl hydrolase family 20(Glyco_hydro_20b residue:215-330)、Glycosyl hydrolase family 20,catalytic domain(Glyco_hydro_2 residue: 333-768)、Chitobiase/beta-hexosaminidase C-terminal domain(CHB_HEX_C residue:816-887) |
以维氏气单胞菌B565 基因组DNA为模板,用引物NagF和NagR进行扩增,经1.0%琼脂糖凝胶电泳分析,在2.6 kb左右出现单一的目的条带,与预期值相同,如图 2。经过EcoR I和Not I双酶切,连接至载体pET28a(+),经酶切鉴定及测序正确,说明nag565-pET28a(+)原核表达载体成功构建。
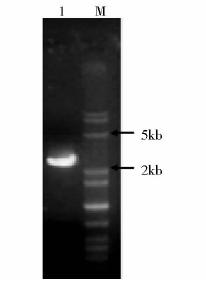
|
| 图 2 nag565 PCR产物琼脂糖凝胶电泳图Fig. 2 Agarose gel electrophoresis of nag565M: DNA marker; 1: PCR products of nag565 |
将重组质粒nag565-pET28a(+)转化BL21(DE3)感受态细胞,获得重组菌,经过活化后添加诱导剂IPTG在18℃诱导12h。离心细胞培养液后获得沉淀,经pH8.0、20mmol/L的Tris-HCl缓冲液重悬后超声破碎,离心后取上清,进行His标签Ni-NTA纯化,SDS-PAGE检测。纯化后的蛋白显示95 kDa的单一条带,与预期分子量相符(图 3)。
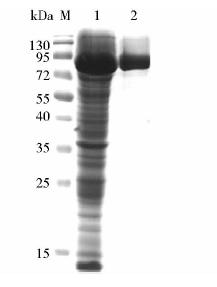
|
| 图 3 E.coli BL21(DE3)表达NAG565的SDS-PAGE电泳图Fig. 3 SDS-PAGE of NAG565 expressed in E. coli BL21(DE3)M: Protein marker; 1: Intracellular protein of E. coli BL21(DE3) containing recombinant plasmid pET-28a(+)-nag565 induced by IPTG; 2:Purified NAG565 |
2.4 纯化后NAG565酶活力测定
将经过Ni-NTA纯化后的NAG565蛋白液作为待测溶液,测定其酶活力及蛋白质含量。结果NAG565蛋白液的酶活力为225.7U/ml发酵液,比活力为7328U/mg。 2.5 NAG565的酶学性质 2.5.1 NAG565最适pH及pH稳定性
在不同pH3.0~11.0缓冲液体系中测定NAG565的酶活力,结果如图 4a所示。其最适pH为7.0,在pH8.0的缓冲液中,可以保持80%以上的相对酶活。将NAG565与不同pH3.0~11.0缓冲液混合37℃处理12h后在pH7.0测定酶活力,以研究其pH稳定性。由图 4b所示,NAG565在pH7.0的缓冲液中稳定性最高,稳定范围在pH5.0~9.0,能保持70%的相对酶活。

|
| 图 4 NAG565的最适pH(a)、pH稳定性(b)、最适温度(c)及热稳定性(d)Fig. 4 The optimal pH(a),pH stability (b),optimal temperature (c) and thermostability(d) of NAG565 |
测定NAG565在不同温度(0℃、20℃、30℃、37℃、50℃、60℃和70℃)下的酶活力,NAG565在37℃时酶活力最高(图 4c),在30℃时可以保持70%以上的相对酶活,当温度高于50℃时,酶活迅速下降。将NAG565在上述不同温度下处理30min后进行酶活力测定以研究其温度稳定性,结果如图 4d所示。在0~30℃,NAG565稳定性较好,在30~70℃之间,随着温度的升高,酶稳定性逐渐降低。 2.5.3 不同金属离子或化学试剂对NAG565酶活性的影响
不同金属离子或化学试剂对NAG565酶活性的影响不同,如表 1所示,Na+、Mn2+促进NAG565酶活性,而Ag+、Hg2+显著抑制NAG565酶活,Co2+、Cr3+、Cu2+等对NAG565酶活有不同程度的抑制作用。K+、Ca2+、Mg2+等则对NAG565蛋白的酶活的影响很小。NAG565蛋白对SDS和β-巯基乙醇具有一定的抵抗能力。
| Reagent | Relative activity(%) | Reagent | Relative activity(%) |
| Na+ | 108.52 | Mn2+ | 108.51 |
| K+ | 95.20 | Zn2+ | 82.65 |
| Ca2+ | 97.66 | Pb2+ | 83.83 |
| Li+ | 67.71 | Fe3+ | 86.00 |
| Co2+ | 43.40 | Ag+ | 18.33 |
| Cr3+ | 64.19 | Hg2+ | 0 |
| Ni+ | 86.91 | SDS | 64.71 |
| Cu2+ | 77.79 | β-Met | 74.01 |
分别用胰蛋白酶、胶原蛋白酶、糜蛋白酶、胃蛋白酶、蛋白酶K处理NAG565酶液30min后,NAG565蛋白的剩余相对酶活保持在68.9%~89.1%,如图 5所示,由此可以看出NAG565对多种蛋白酶都具有较强的抗性。
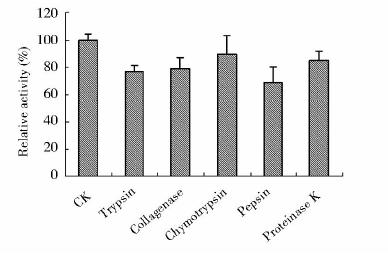
|
| 图 5 不同蛋白酶对NAG565酶活性的影响Fig. 5 Effect of proteases on NAG565 activity |
在37℃、pH7.0测定NAG565对不同浓度(1、2、5、10、50、100、500、1000μmol/L) 的底物的反应初速度,经Lineweaver-Burk法绘图得出NAG565对pNP-GlcNAc、pNP-(GlcNAc)2和pNP-(GlcNAc)3 3种底物的Km和Vmax值,结果如表 2所示,在3种底物中,pNP-(GlcNAc)3的Km值最小,Vmax值最大。
| Substrates | Vmax [μmol/(mg·min)] | Km (mg/ml) |
| pNP-GlcNAc | 270 | 2366.4 |
| pNP-(GlcNAc)2 | 980 | 501.2 |
| pNP-(GlcNAc)3 | 1050 | 276.0 |
NAG565蛋白对β-N-乙酰氨基葡萄糖苷键具有专一性,NAG565蛋白对其它底物如:4-甲基伞形酮-N-乙酰基-α-D-氨基葡糖苷、4-甲基伞形酮-α-D-葡糖苷、4-甲基伞形酮-α-D-半乳糖、4-甲基伞形酮-α-D-木糖苷、4-甲基伞形酮-α-D-吡喃葡萄糖苷、乙二醇壳聚糖、羧甲基纤维素、壳聚糖粉末均没有降解能力。
3 讨 论
目前报道的β-N-乙酰氨基葡萄糖苷酶主要来自细菌,这些酶的酶学基本性质呈以下特点:(1)最适pH多在5到8之间,个别菌来源的β-N-乙酰氨基葡萄糖苷酶能适应偏碱性的条件,如链霉菌属Streptomyces sp.的β-N-乙酰氨基葡萄糖苷酶的最适pH为10 [10],革兰氏阴性菌的β-N-乙酰氨基葡萄糖苷酶的最适pH多为中性或弱碱性(pH7~8);(2)最适温度多在37到50℃之间,少数菌的如热紫链霉菌Streptomyces thermoviolaceus和Streptomyces sp.最适温度偏高(60~70℃);(3)酶活多在每毫克蛋白几毫酶活单位到几十酶活单位之间,个别菌来源的β-N-乙酰氨基葡萄糖苷表现出较高的酶活,如革兰氏阴性菌粘质沙雷氏菌S.marcescens属β-N-乙酰氨基葡萄糖苷酶活为62750 U/mg[4, 5, 11, 12, 13, 14]。
本研究实现了β-N-乙酰氨基葡萄糖苷酶基因nag565在大肠杆菌系统中的高效表达,每ml发酵液的重组酶的酶活为225.7U,经纯化后达到电泳纯,比活为7328 U/mg,活性仅次于Tews等[15]1996年报道S. marcescens属β-N-乙酰氨基葡萄糖苷酶的活性,是目前报道的气单胞菌属中比活最高的β-N-乙酰氨基葡萄糖苷酶[6, 16, 17],其活性也远高于多数革兰氏阳性和阴性菌的β-N-乙酰氨基葡萄糖苷酶。该酶的最适pH为7.0,且在pH 5.0~9.0范围内都很稳定,可以保持70%以上的相对酶活性;最适温度为37℃,30℃以下处理对酶活性没有影响,均符合水产动物的养殖环境条件(养殖温度20~40℃,多数养殖鱼胃肠道pH值呈中性),具有较高的应用价值。
金属离子和化学试剂在酶的催化反应过程中起重要作用。 Fe3+、Mn2+、Ag+、Cu2+、Zn2+、Co2+、Hg2+、SDS、Urea、DTT等离子被报道对β-N-乙酰氨基葡萄糖苷酶酶活有较大程度的抑制[18, 19, 20, 21]。本研究发现 Mn2+对NAG565酶活性有弱的促进作用,Ag+、Hg2+显著抑制NAG565酶活,Co2+、Cr3+、Li+、Cu2+、SDS和β-巯基乙醇等对NAG565酶活有不同程度的抑制作用。在消化道中存在各种蛋白酶会对饲料用酶进行消化分解,从而影响饲用酶的使用效率,因此,具有蛋白酶抗性是其作为饲用酶的关键,本研究进一步分析了NAG565对多种蛋白酶的抗性,发现NAG565对多种蛋白酶都具有较强的抗性。基于以上性质,该酶符合水产养殖环境条件,也是一种潜在的水产饲用添加剂,可以帮助几丁质酶更有效地降解几丁质,提高几丁质的利用效率。
为推进其在水产中的应用,未来需进一步深入其水产养殖应用研究,为其在水产养殖中的应用奠定基础。由于酶的工业热处理中温度通常会超过45℃,因此未来可通过对该酶进行适当的分子改良来提高其热稳定性和活性,扩展其应用范围[22]。
| [1] | Chen J K, Shen C R, Liu C L. N-acetylglucosamine: production and applications. Mar Drugs, 2010,8(9):2493-2516. |
| [2] | Konopka J B. N-acetylglucosamine (GlcNAc) functions in cell signaling. Scientifica (Cairo), 2012,Article ID 489208. |
| [3] | Yang S, Song S, Yan Q, et al. Biochemical characterization of the first fungal glycoside hydrolyase family 3 beta-N-acetylglucosaminidase from Rhizomucor miehei. J Agric Food Chem, 2014,62(22):5181-5190. |
| [4] | Ueda M, Fujita Y, Kawaguchi T, et al. Cloning, nucleotide sequence and expression of the β-N-acetylglucosaminidase gene from Aeromonas sp. No. 10S-24. Journal of Bioscience and Bioengineering, 2000,89(2):164-169. |
| [5] | Sukprasirt P, Wititsuwannakul R. A chitinolytic endochitinase and beta-N-acetylglucosaminidase-based system from Hevea latex in generating N-acetylglucosamine from chitin. Phytochemistry, 2014,104:5-11. |
| [6] | Lan X, Zhang X, Kodaira R, et al. Gene cloning, expression, and characterization of a second beta-N-acetylglucosaminidase from the chitinolytic bacterium Aeromonas hydrophila strain SUWA-9. Biosci Biotechnol Biochem, 2008,72(2):492-498. |
| [7] | Li Y, Liu Y, Zhou Z, et al. Complete genome sequence of Aeromonas veronii strain B565. J Bacteriol, 2011,193(13):3389-3390. |
| [8] | Bradford M M.A rapid and sensitive method for the quantization of microgram quantities of protein utilizing the principle of protein-dye binding.Anal Biochem,1976,72:248-254. |
| [9] | Prag G, Papanikolau Y, Tavlas G, et al. Structures of chitobiase mutants complexed with the substrate Di-N-acetyl-d-glucosamine: the catalytic role of the conserved acidic pair, aspartate 539 and glutamate 540. J Mol Biol, 2000,300(3):611-617. |
| [10] | Prakash D, Nawani N, Kapadnis B. Cloning, expression and characterization of thermophilic and alkalophilic N-acetylglucosaminidase from Streptomyces sp. NK52 for the targeted production of N-acetylglucosamine. Proceedings of the National Academy of Sciences, India Section B: Biological Sciences, 2013, 83(3): 431-437. |
| [11] | Eckert C, Lecerf M, Dubost L, et al. Functional analysis of AtlA, the major N-acetylglucosaminidase of Enterococcus faecalis. J Bacteriol, 2006,188(24):8513-8519. |
| [12] | Yang S, Hua C, Yan Q, et al. Biochemical properties of a novel glycoside hydrolase family 1 β-glucosidase (PtBglu1) from Paecilomyces thermophila expressed in Pichia pastoris. Carbohydrate Polymers, 2013,92(1):784-791. |
| [13] | Kim S, Matsuo I, Ajisaka K, et al. Cloning and characterization of the nagA gene that encodes beta-n-acetylglucosaminidase from Aspergillus nidulans and its expression in Aspergillus oryzae. Biosci Biotechnol Biochem, 2002,66(10):2168-2175. |
| [14] | Ling Z, Suits M D, Bingham R J, et al. The X-ray crystal structure of an Arthrobacter protophormiae endo-β-N-acetylglucosaminidase reveals a (β/α)8 catalytic domain, two ancillary domains and active site residues key for transglycosylation activity. Journal of Molecular Biology, 2009,389(1):1-9. |
| [15] | Tews I, Vincentelli R, Vorgias C E. N-Acetylglucosaminidase (chitobiase) from Serratia marcescens: gene sequence, and protein production and purification in Escherichia coli. Gene, 1996,170(1):63-67. |
| [16] | Tsujibo H, Fujimoto K, Tanno H, et al. Gene sequence, purification and characterization of N-acetyl-beta-glucosaminidase from a marine bacterium, Alteromonas sp. strain O-7. Gene, 1994,146(1):111-115. |
| [17] | Lin J, Xiao X, Zeng X, et al. Expression, characterization and mutagenesis of the gene encoding β-N-acetylglucosaminidase from Aeromonas caviae CB101. Enzyme and Microbial Technology, 2006,38(6):765-771. |
| [18] | Tsujibo H, Hatano N, Mikami T, et al. A novel beta-N-acetylglucosaminidase from Streptomyces thermoviolaceus OPC-520: gene cloning, expression, and assignment to family 3 of the glycosyl hydrolases. Appl Environ Microbiol, 1998,64(8):2920-2924. |
| [19] | Park J K, Kim W J, Park Y I. Purification and characterization of an exo-type beta-N-acetylglucosaminidase from Pseudomonas fluorescens JK-0412. J Appl Microbiol, 2011,110(1):277-286. |
| [20] | Murakami S, Takaoka Y, Ashida H, et al. Identification and characterization of endo-beta-N-acetylglucosaminidase from methylotrophic yeast Ogataea minuta. Glycobiology, 2013,23(6):736-744. |
| [21] | O'Connell E, Murray P, Piggott C, et al. Purification and characterization of a N-acetylglucosaminidase produced by Talaromyces emersonii during growth on algal fucoidan. Journal of Applied Phycology, 2008, 20(5): 557-565. |
| [22] | Bassler B L, Yu C, Lee Y C, et al. Chitin utilization by marine bacteria. Degradation and catabolism of chitin oligosaccharides by Vibrio furnissii. J Biol Chem, 1991,266(36):24276-24286. |
 2015, Vol. 35
2015, Vol. 35




