癌症是当今世界重要的公共健康问题。根据世界卫生组织国际癌症研究机构的数据显示,2018年约有1 810万新病例和960万癌症相关死亡病例,影响到所有国家和地区的人口。尤其是乳腺癌在女性癌症新发病例占比最高(占比24.2%),而且死亡率非常高[1]。
病理组织学分析仍然是诊断乳腺癌最广泛使用的方法[2],而且大多数诊断仍由病理学家在显微镜下对组织学样本进行目视检查来完成,需要专业的病理学家的大量工作,专家之间的诊断一致性平均约为75%[3]。因此运用计算机来进行病理组织学图像的自动分类可以使乳腺癌诊断更快,而且更不容易出错。目前关于乳腺癌识别的研究主要分为2种方法:1)基于手工特征提取结合传统机器学习的方法。Belsare等[4]使用统计纹理特征训练K-NN(k近邻)和支持向量机(support vector machine,SVM)分类器,在一个40倍放大的私人乳腺组织学数据集上达到了70%~100%的准确率。Spanhol等[5]公开了乳腺癌病理图像数据集BreaKHis,研究了6种包括邻接阈值统计[6]在内的手工提取的纹理特征结合包含SVM在内的4种分类器共24组的分类性能,作为区分良性和恶性肿瘤的参考基线,达到了80%~85%的准确率;2)基于深度学习的分类方法,深度学习使得模型可以直接从输入的图片中提取特征,避免了人工提取特征的程序,节省了大量的人力物力。近年来,卷积神经网络(convolutional neural networks,CNN)作为深度学习的重要方法在图像识别领域取得了巨大的成功, 其在医疗图像分析方面也取得了快速的发展[7]。Araújo等[8]利用卷积神经网络将乳腺癌病理图像分成了癌和非癌2大类,最高达到了88.3% 的识别率。进一步划分为正常组织、良性病变、原位癌和浸润性癌4类时,最高达到了77.8%的总体准确率。何雪英等[9]使用卷积神经网络模型对BreaKHis数据集进行了良性和恶性分类,经过数据增强处理之后的识别率最高可达到91%。Spanhol等[10-11]在BreaKHis数据集上采用卷积神经网络和深度特征的方法将乳腺癌组织病理图像分为良性和恶性2类,最高分别达到了90%和86.3%的准确度。Bayramoglu等[12]提出了一种与放大系数无关的乳腺癌组织病理图像分类方法,该方法在BreaKHis数据集上,可同时分类病理图像的良性或恶性和分类放大倍数,其实验结果在良性或恶性分类上达到了84.3%的准确度。
为了进一步提高乳腺病理图像分类精度,本文提出了基于卷积神经网络的乳腺癌病理图像分类方法,同时针对高分辨病理图像提出了图像分块的思想,并在BreaKHis数据集上,验证了此方法的性能。
1 BreaKHis数据集BreaKHis数据集[5]是巴拉那联邦大学的Spanhol等论文中发布的公开数据集。该数据集采集自82个病人,其中良性24人,恶性58人。目前为止,该数据集一共包含7 909张图片,分为4个放大系数40、100、200、400。每个放大系数又可分为良性和恶性肿瘤2大类。具体分布情况如表 1所示。
| 表 1 按放大系数和类别的图像分布 Table 1 Image distribution by magnification factor and class |
乳腺良性和恶性肿瘤在显微镜下又可根据肿瘤细胞的不同分为不同类型,本文只研究不同放大系数下的良性和恶性二元分类问题。图 1展示了40放大系数下的部分病理图像,图 1(a)~(d)图像为4种良性肿瘤,图 1(e)~(h)为4种恶性肿瘤。所有图像存储格式为三通道RGB格式,固定尺寸为700×460像素。

|
Download:
|
| 图 1 乳腺癌组织病理学图像样本 Fig. 1 Sample breast cancer histopathology image | |
随着深度学习的快速发展,在图像识别领域,深度学习方法特别是采用CNN的方法已经优于传统的机器学习方法。CNN体系结构的主要由3种层构成:卷积层、池化层和全连接层。卷积层是用一组参数可学习的滤波器对输入图像进行卷积运算,每个滤波器经过运算产生特征映射。池化层对输入特征映射进行向下采样以降低输入的空间维度。全连接层一般用在模型末端,用来将前边提取到的特征综合起来进行分类等操作。通常,完整的CNN架构是通过各种方式叠加这些层来获得的。LeNet-5模型[13]运用CNN模型结构进行图像分类实例,它对输入尺寸为32×32的灰度图像进行2次卷积加池化操作,最后加上3层全连接层进行图像的10分类。
目前,各大研究机构相继发布了一些CNN模型结构,如LeNet、VGG、AlexNet和ResNet等,这些模型都曾在图像识别上取得过优异的成绩。在一些数据集上的识别上,基于这些成熟的模型来构建模型,往往比自己从头搭建模型效果更好,更方便快捷。如基于AlexNet模型进行了乳腺图像的特征提取[10],该模型相对简单,限制了其分类精度。故本文基于结构更加复杂的Inception V3[14]模型搭建模型,进行病理图像的分类。
如图 2所示,模型由特征提取过程和分类过程构成。因为InceptionV3模型针对图片大小为299×299搭建,所以采用图像缩放和Inception V3模型构成特征提取过程,其中InceptionV3模型不包含原模型最后2层全连接层。分类过程由2层新的全连接层神经网络构成,其中第1层全连接层神经网络具有512个节点,第2层具有2个节点,并在最后一层神经网络采用SoftMax函数作为激活函数进行分类。
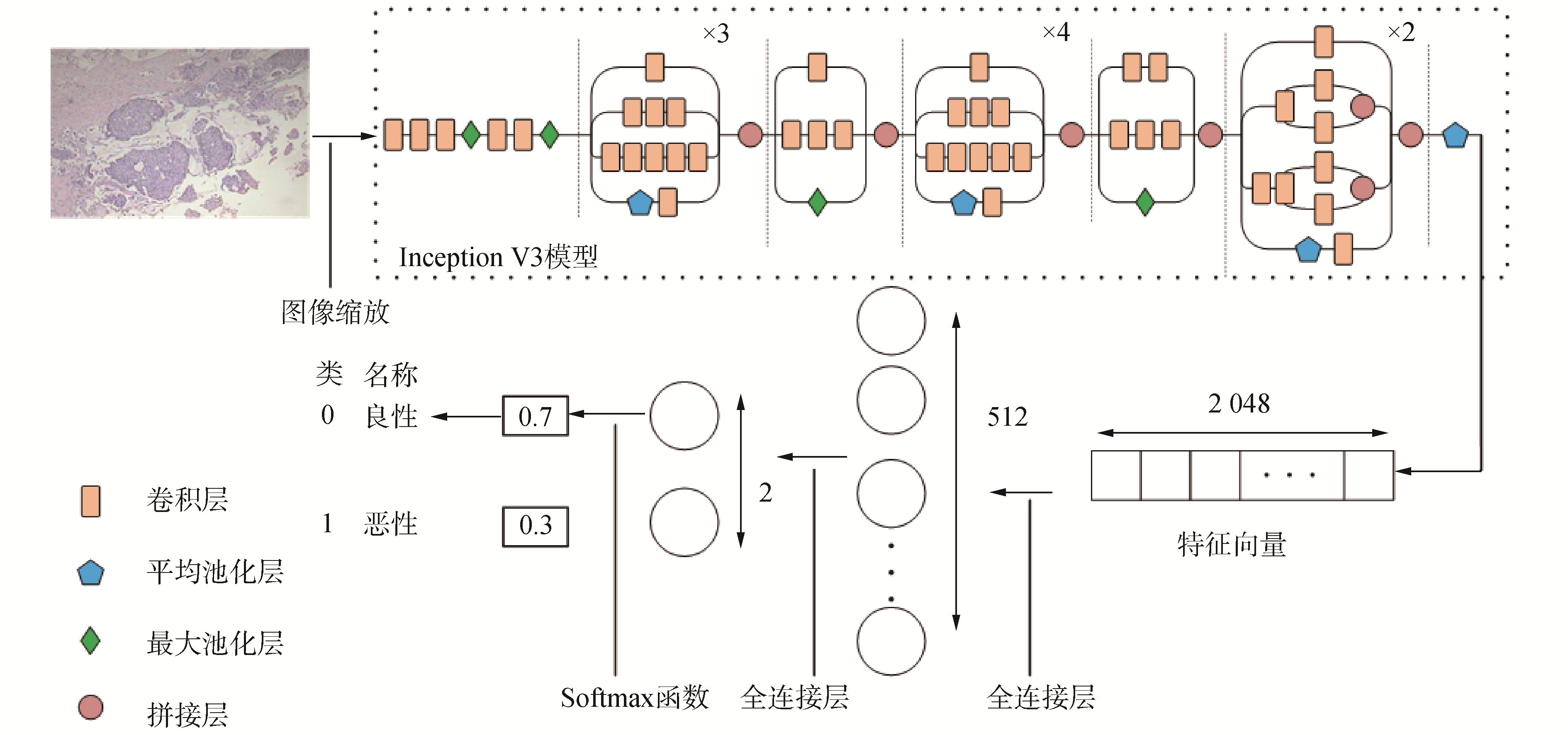
|
Download:
|
| 图 2 图像基于Inception V3模型分类过程 Fig. 2 Image classification based on inception V3 model | |
单张图像分类具体操作步骤为:1)将图片通过缩放使尺寸大小固定为299×299;2)将缩放后的图片送入InceptionV3模型进行计算,取InceptionV3模型最后一层池化层的输出参数作为图片的特征向量,其尺寸为1×2 048;3)将特征向量送入全连接层神经网络进行计算,第2层每个节点的数值经过SoftMax函数计算之后的输出结果即为输入图像属于某一类的概率;4)将图像分类到输出概率最大的类别。
由于数据量的限制,本文只训练特征提取之后2层全连接层的参数。对于特征提取阶段Inception V3模型的参数,采用了迁移学习[15]的方法获得。迁移学习的是先在1个大型数据集上训练CNN模型的参数,然后将训练好的参数作为在新的数据集训练相同CNN模型的初始化权值,迁移学习又可分为微调和固定权值的2种方式,微调方式在每轮训练之后对CNN模型的参数进行更新,而固定权值的方式是CNN模型参数在初始化之后便固定不变。
本文采用在ImageNet[16]数据集上训练的InceptionV3模型参数作为特征提取阶段模型的初始化权值。且微调方式导致模型参数的改变,使得每次训练都需要重新计算图像的特征向量,计算任务大、训练时间长,故本文采用固定权值的方法。由此本文在计算出图像特征向量之后将其存储为文本文档,这样可使本张图片在下轮训练过程中,可直接读取文本文档获得特征向量,避免重复特征提取过程的计算,节约计算时间。
在训练过程中,采用通过构建SoftMax函数的输出结果与图像标签之间的交叉熵作为损失函数,通过梯度下降法进行全连接层神经网络的参数优化,进而对损失函数进行最小化。单张图片交叉熵损失函数为:
| $ L\left( {x, y} \right) = - \sum\limits_k {{y_k}{\rm{log}}\;{p_k}\left( x \right)} $ | (1) |
式中:pk(x)是输入图像x被分类器分为第k类的概率;yk是指示性函数,当输入图像x的标签y是k类时yk=1,否则yk=0。
2.2 基于四叉树图像分割的数据增强在训练CNN模型时,若使用的数据集的样本数较少,该网络容易过度拟合。常用的方法为数据增强。数据增强是通过旋转、翻转、滑动窗口等操作提升数据量,本文采用更适合高分辨率图像的基于四叉树分割的数据增强方法[17]。四叉树由1个连续的结构组成,在每一层,将上一层的输入图像均等分成4个部分。即在每一级L(L≥1)处,将输入图像划分为(2L-1)2个大小相等且不重叠的块。这意味着在第1级输入图像保持为原始图像,在第2级输入图像被分成4个图像,在第3级输入图像被分成16个图像,依此类推,如图 3所示。本文对乳腺图像的第2级和第3级切分结果进行了研究,即每张图片分割为4块和16块。经过分割之后训练数据分别增加为原数据量的4倍和16倍,在分割之后,每块子图像都被认为与原始图像具有相同的类标签。
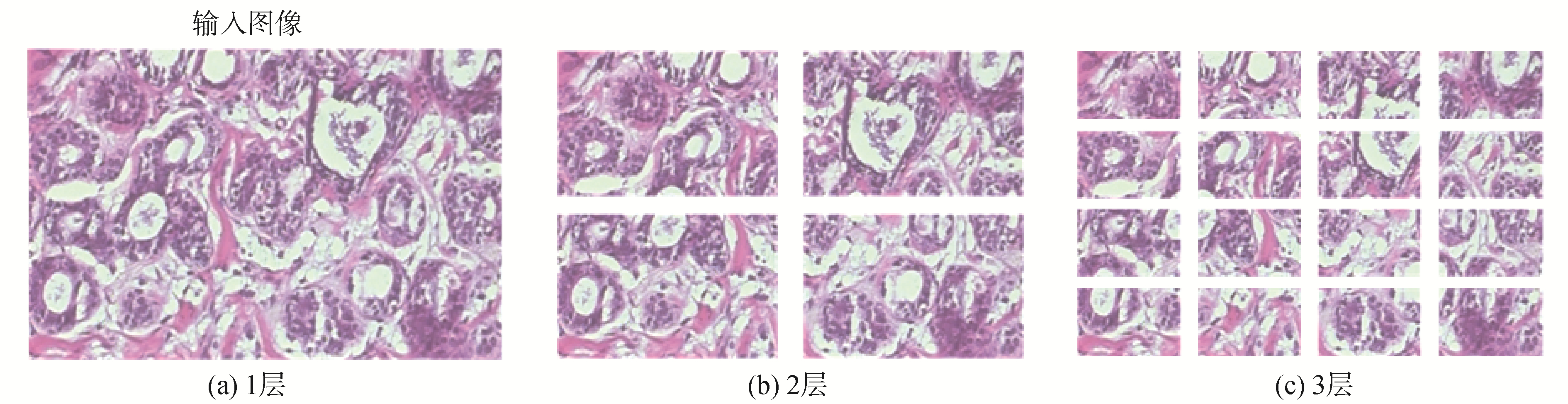
|
Download:
|
| 图 3 基于四叉树方法的图像分割方法 Fig. 3 Image cutting method based on quadtree | |
测试时由于采用了基于四叉树的图像分割方法,一张图片被分割成多个子图像块,每个图像块在经过模型计算可能产生不同的分类结果,故需要融合算法将所有图像块的分类结果整合起来。常用的算法有和规则、乘积规则、最大值规则、多数投票规则等。为了研究不同的融合算法对对乳腺图像分类结果的影响,本文选取了和规则,乘积规则和最大值规则[18]算法进行了实验验证,算法计算过程为:
和规则:
| $ \phi = \mathop {{\rm{argmax}}}\limits_{k = 1}^K \sum\limits_{i = 1}^N {{p_i}\left( k \right)} $ | (2) |
乘积规则:
| $ \phi = \mathop {{\rm{argmax}}}\limits_{k = 1}^K \mathop \Pi \limits_{\text{i} = 1}^N {p_i}\left( k \right) $ | (3) |
最大值规则:
| $ \phi = \mathop {{\rm{argmax}}}\limits_{k = 1}^K \mathop {{\rm{max}}}\limits_{i = 1}^N {p_i}\left( k \right) $ | (4) |
式中:pi(k)表示为1张图片的第i个子图像块被模型分为k类的概率值;K表示分类类别总数;N表示1张图片被切分块的数量。图 4展示1张病理图片的完整分类示例过程,在本例中将原始图像分割为4块,采用的融合函数为最大值规则。
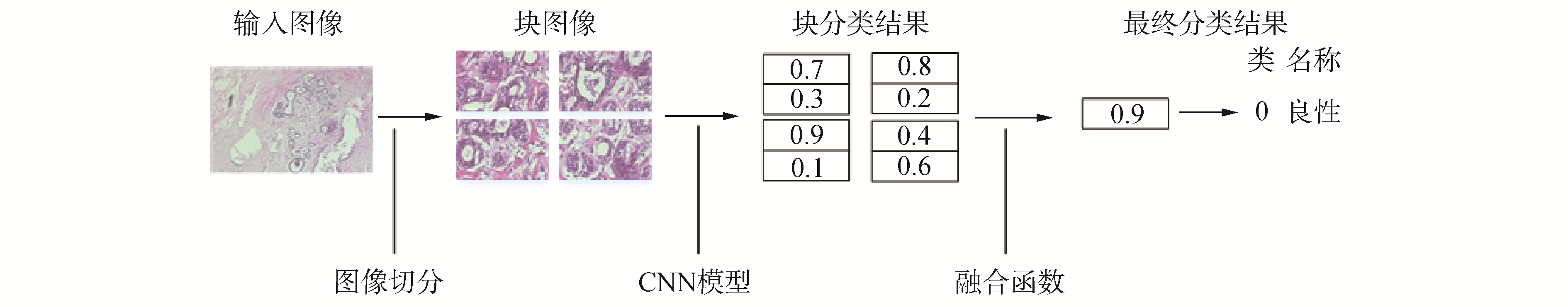
|
Download:
|
| 图 4 图像完整分类过程 Fig. 4 Image integrity classification process | |
本文中所有试验均在相同试验环境下完成,试验平台为1台CPU为Intel I7-8700,GPU为NVIDIA GTX1060 6 GB显存,内存为16 GB的计算机,试验环境为Windows 10操作系统,基于Python语言的TensorFlow架构进行编程。
3.1 实验细节与评价标准本文将实验数据集按照75%、15%、15%的比例切分为训练集、验证集、测试集。训练集用来训练模型,验证集用来调节模型的超参数。模型的超参数选定之后,合并使用训练集和验证集的数据进行重新训练模型,利用测试集来输出测试结果。本文实验结果为5次测试结果的平均取值,且5次实验独立进行,每次实验前重新随机划分训练集、验证集和测试集。
实验结果以乳腺病理图像在图像层面和病人层面的分类准确率作为评价标准,并在每个放大系数上独立评估。基于图片层面的分类准确率只是考虑分类正确的图像占全部图像的比例,图像层面分类准确率IA为:
| $ {I_A} = \frac{{{N_c}}}{{{N_{{\rm{im}}}}}} $ | (5) |
式中:Nim表示全部图片的数量;Nc是分类正确的图片数量。
基于病人层面的分类准确率则先对每个患者的多张病理图像计算分类准确率,再对所有患者的分类准确率取平均值。病人层面分类准确率PA为:
| $ {P_A} = \frac{{\sum\limits_{p = 1}^{{N_{\rm{p}}}} {\frac{{N_c^p}}{{N_{{\rm{im}}}^p}}} }}{{{N_p}}} $ | (6) |
式中:Np是患者的数量;Ncp是对应患者P分类正确的图片数量;Nimp是对应患者P图片的总数。
3.2 实验结果与对比 3.2.1 实验结果根据3.1节所定义的实验细节和评价标准,本节在BreaKHis数据集上进行本文提出的乳腺病理图像识别方法的性能验证实验,实验结果如表 2和表 3所示。表 2和表 3分别展示了测试集在图像层面和病人层面的识别准确度。从实验结果上看,将图片基于四叉树策略分割后,无论4切分还是16切分,准确度都明显高于原图,其中4切分提高了2.0%~4.4%的准确率,16切分提高了0.9%~4.9%的准确率。说明图片分割方法结合融合算法能有效提升乳腺病理图像识别准确度。
| 表 2 图像层面的准确率 Table 2 Accuracy at picture level |
| 表 3 病人层面的准确率 Table 3 Accuracy at patient level |
而从实验结果上并不能得出哪一个融合算法更适合本实验,说明采用不同的融合算法规则对实验结果的影响很小。通过对比不同放大系数之间的实验数据,发现100放大系数下的识别率最高,400放大系数下的识别率最低,40放大系数略高于200放大系数的识别率。
图 5展示了100放大系数下使用原图和16切分的病理图像在训练过程中的损失函数随着训练步数的变化曲线,为了便于观察,曲线经过平滑处理。可以看出,在原图的情况下,本该逐渐减小收敛的损失函数却在训练步数大约达到2 000步时达到最小值,随后开始增大。这说明模型在训练步数超过2 000时开始过拟合。而在16切分情况下,损失函数是逐渐下降并收敛的,说明图像切分扩大了训练数据量,避免了过拟合情况的发生,这也是图像切分能提高识别准确度的原因之一。
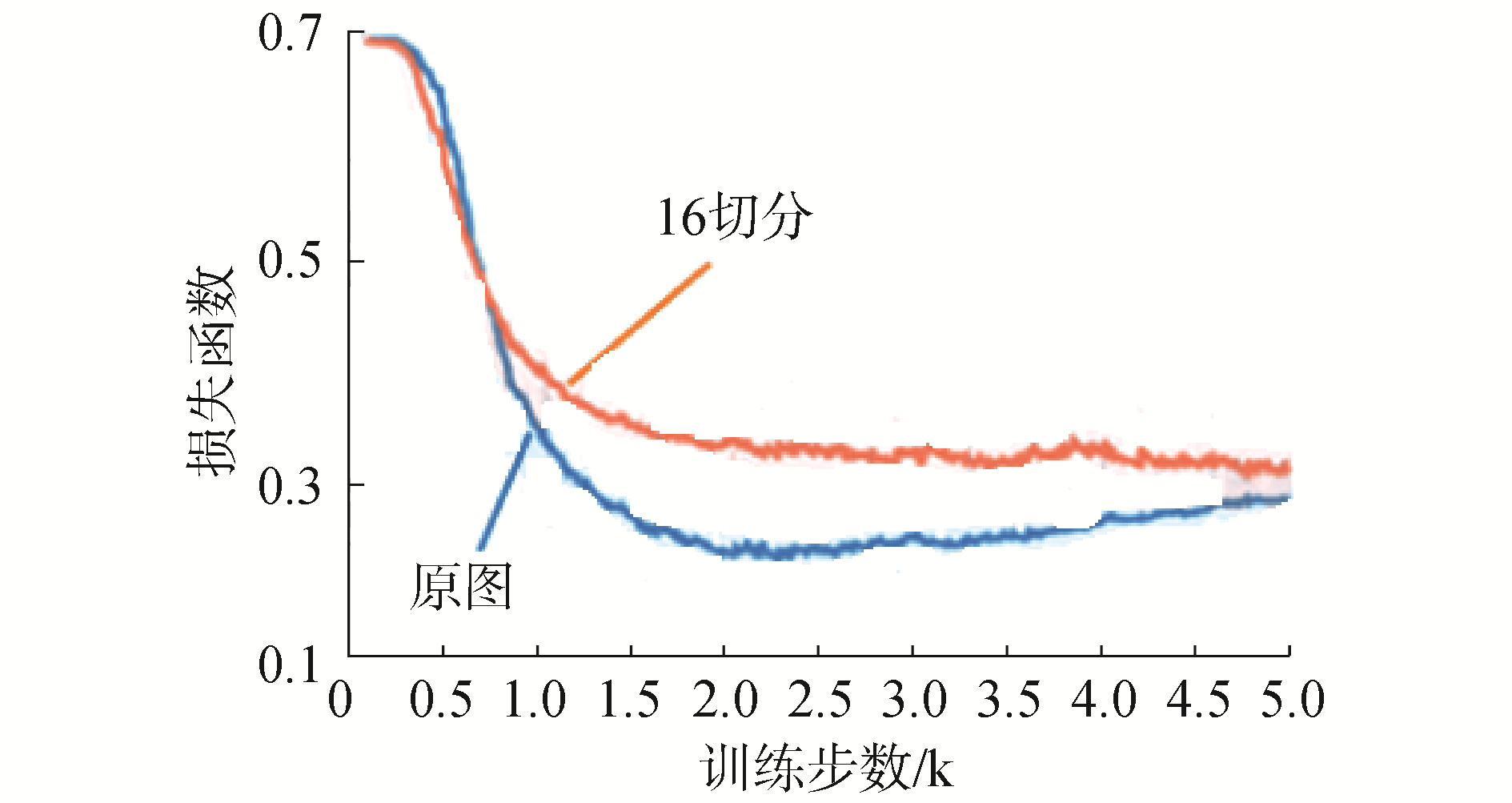
|
Download:
|
| 图 5 原图和16切分图像的损失函数对比 Fig. 5 Loss function comparison between original image and 16-sliced image | |
4切分的结果除了在100放大系数上都要略高于16切分的结果。这是由于在16切分的情况下,图像切分的相对较小,导致部分单张子图片所包含的信息不足以体现病理情况。
3.2.2 与其他实验对比为了更好的评估实验结果,我们选取了文献[5, 10-12]和本文中各个放大系数中最好的实验结果进行对比,如表 4所示。文献[12]中未展示图片层面识别准确度。从表中可以看到基于卷积神经网络和四叉树图片切分的方法,在各个放大系数上达到了最高的识别准确度。相对文献[5]中采用手工特征提取和传统的机器学习的方法,本文的方法提高了9.9%~12.2%的图像识别准确度;相对文献[9]采用卷积神经网络提取特征,旋转、翻转的方法增强数据,本文的方法提高了2.3%~5.1%的图像识别率;相对文献[10]中采用卷积神经网络提取特征,滑动窗口数据增强的方法,本文的方法提高了5%~11.5%的图像识别率;相对文献[11]中采用深度特征和特征融合的方法,本文的方法提高了8.2%~10.9%的图像识别率;相对文献[12]中采用多任务卷积神经网络的方法,本文方法提高了10.2%~12%的图像识别率。并且经过试验结果对比可以得出,基于卷积神经网路特征提取的方法,在分类性能上要优于手工特征提取的方法。
| 表 4 与其他方法的实验结果比较 Table 4 Comparisons with experimental results of other methods |
1) 图像分割的数据增强方法能够明显提高乳腺图像识别率;4切分的性能要高于16切分。
2) 不同融合算法对分块图像识别率的影响较小。通过与其他相关方法的对比,验证了利用深度学习方法提取特征的性能要优于基于视觉的手工特征。
未来将要优化图像的特征提取方法,如采用多模型提取特征结合特征融合的方法。通过优化图片的切分方法,数据增强方法,进一步提高分类的精度。在二分类的基础上,对于良性和恶性肿瘤进行进一步的分类,做到细分。
| [1] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: a cancer journal for clinicians, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 (  0) 0)
|
| [2] |
杨峰, 李金锋. 十年来乳腺肿瘤组织学分类的变化及临床意义[J]. 中华外科杂志, 2014, 52(1): 1-3. YANG Feng, LI Jinfeng. WHO classification of tumors of the breast[J]. Chinese journal of surgery, 2014, 52(1): 1-3. (  0) 0)
|
| [3] |
ELMORE J G, LONGTON G M, CARNEY P A, et al. Diagnostic concordance among pathologists interpreting breast biopsy specimens[J]. JAMA, 2015, 313(11): 1122-1132. DOI:10.1001/jama.2015.1405 (  0) 0)
|
| [4] |
BELSARE A D, MUSHRIF M M, PANGARKAR M A, et al. Classification of breast cancer histopathology images using texture feature analysis[C]//Proceedings of 2015 IEEE Region 10 Conference. Macao, China: IEEE, 2015.
(  0) 0)
|
| [5] |
SPANHOL F A, OLIVEIRA L S, PETITJEAN C, et al. A dataset for breast cancer histopathological image classification[J]. IEEE transactions on biomedical engineering, 2016, 63(7): 1455-1462. DOI:10.1109/TBME.2015.2496264 (  0) 0)
|
| [6] |
HAMILTON N A, PANTELIC R S, HANSON K, et al. Fast automated cell phenotype image classification[J]. BMC bioinformatics, 2007, 8(1): 110. DOI:10.1186/1471-2105-8-110 (  0) 0)
|
| [7] |
ZHANG Yanbo, YU Hengyong. Convolutional neural network based metal artifact reduction in X-ray computed tomography[J]. IEEE transactions on medical imaging, 2018, 37(6): 1370-1381. DOI:10.1109/TMI.2018.2823083 (  0) 0)
|
| [8] |
ARAÚJO T, ARESTA G, CASTRO E, et al. Classification of breast cancer histology images using Convolutional Neural Networks[J]. PLoS one, 2017, 12(6): e0177544. DOI:10.1371/journal.pone.0177544 (  0) 0)
|
| [9] |
何雪英, 韩忠义, 魏本征. 基于深度学习的乳腺癌病理图像自动分类[J]. 计算机工程与应用, 2018, 54(12): 121-125. HE Xueying, HAN Zhongyi, WEI Benzheng. Breast cancer histopathological image auto-classification using deep learning[J]. Computer engineering and applications, 2018, 54(12): 121-125. (  0) 0)
|
| [10] |
SPANHOL F A, OLIVEIRA L S, PETITJEAN C, et al. Breast cancer histopathological image classification using convolutional neural networks[C]//Proceedings of 2016 International Joint Conference on Neural Networks. Vancouver, BC, Canada: IEEE, 2016.
(  0) 0)
|
| [11] |
SPANHOL F A, OLIVEIRA L S, CAVALIN P R, et al. Deep features for breast cancer histopathological image classification[C]//Proceedings of 2017 IEEE International Conference on Systems, Man, and Cybernetics (SMC). Banff, AB, Canada: IEEE, 2017.
(  0) 0)
|
| [12] |
BAYRAMOGLU N, KANNALA J, HEIKKILÄ J. Deep learning for magnification independent breast cancer histopathology image classification[C]//Proceedings of the 23rd International Conference on Pattern Recognition. Cancun, Mexico: IEEE, 2017.
(  0) 0)
|
| [13] |
LECUN Y, BOTTOU L, BENGIO Y, et al. Gradient-based learning applied to document recognition[J]. Proceedings of the IEEE, 1998, 86(11): 2278-2324. DOI:10.1109/5.726791 (  0) 0)
|
| [14] |
SZEGEDY C, VANHOUCKE V, IOFFE S, et al. Rethinking the inception architecture for computer vision[C]//Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Las Vegas, NV, USA: IEEE, 2016: 2818-2826.
(  0) 0)
|
| [15] |
YOSINSKI J, CLUNE J, BENGIO Y, et al. How transferable are features in deep neural networks?[C]//Proceedings of the 27th International Conference on Neural Information Processing Systems. Montreal, Quebec, Canada: ACM, 2014.
(  0) 0)
|
| [16] |
DENG Jia, DONG Wei, SOCHER R, et al. ImageNet: a large-scale hierarchical image database[C]//Proceedings of 2009 IEEE Conference on Computer Vision and Pattern Recognition. Miami, Florida, USA: IEEE, 2009.
(  0) 0)
|
| [17] |
VAMVAKAS G, GATOS B, PERANTONIS S J. Handwritten character recognition through two-stage foreground sub-sampling[J]. Pattern recognition, 2010, 43(8): 2807-2816. DOI:10.1016/j.patcog.2010.02.018 (  0) 0)
|
| [18] |
KITTLER J, HATEF M, DUIN R P W, et al. On combining classifiers[J]. IEEE transactions on pattern analysis and machine intelligence, 1998, 20(3): 226-239. (  0) 0)
|
 2021, Vol. 42
2021, Vol. 42


