文章信息
- 李爽, 高志勇, 李伟红, 田祎, 刘白薇, 沈玲羽, 严寒秋, 贾蕾, 张代涛, 王全意.
- Li Shuang, Gao Zhiyong, Li Weihong, Tian Yi, Liu Baiwei, Shen Lingyu, Yan Hanqiu, Jia Lei, Zhang Daitao, Wang Quanyi
- 北京市2019年腹泻病例中札如病毒基因特征分析
- Gene characteristics analysis on Sapovirus from diarrhea cases in Beijing, 2019
- 中华流行病学杂志, 2023, 44(11): 1787-1794
- Chinese Journal of Epidemiology, 2023, 44(11): 1787-1794
- http://dx.doi.org/10.3760/cma.j.cn112338-20230505-00278
-
文章历史
收稿日期: 2023-05-05
1976年英国学者Madeley和Cosgrove[1]在儿童腹泻粪便样本中首次发现札如病毒,该病毒属于杯状病毒科,可以引起各年龄段人群发病,是非细菌性急性胃肠炎散发和暴发疫情的常见病原之一[2-6]。札如病毒为单股正链小RNA病毒,基因组全长约7.3~7.5 kb,根据完整的衣壳蛋白区(VP1)基因序列。札如病毒分5个基因组(GⅠ~GⅤ组),其中GⅠ、GⅡ、GⅣ和GⅤ组可感染人类。目前,至少确定有GⅠ.1~7、GⅡ.1~8、GⅣ.1和GⅤ.1~2共18种基因型[6-9]。本研究收集2019年北京市监测哨点医院肠道门诊的散发腹泻病例粪便样本,分析札如病毒基因分型特征,为札如病毒感染性腹泻防控工作提供依据。
对象与方法1. 研究对象:2019年1-12月在北京市监测哨点医院采集散发腹泻监测病例粪便样本。≥5岁和 <5岁散发腹泻病例分别来自于综合医院肠道门诊和儿童专科医院肠道门诊。病例定义为24 h内出现排便≥3次且有性状改变,和/或24 h内出现呕吐者,主诉症状为腹泻和/或呕吐等消化道症状的门诊病例,发病3 d内的初诊病例。
2. 样本采集和保存运输:采集液体水样便3~5 ml,置于无菌便盒中,成形便达到便盒1/2以上。采样后密封并做好记录,样本4 ℃~8 ℃转运到北京市CDC,立即检测,或短期保存(-20 ℃冰箱)和长期保存(-70 ℃冰箱)。整个过程避免反复冻融。所有样本的保存及运输有详细记录。
3. 样本处理和病毒核酸提取:固体粪便或液体粪便样本分别取0.1 g或0.1 ml于1 ml pH值7.4磷酸盐缓冲液中,经旋涡振荡器混匀,制成10%的便悬液。该悬液室温静置10 min后,以5 000 r/min离心半径3.5 cm,离心5 min,取上清液用于病毒RNA提取,或-20 ℃保存备检。取140 μl上清液按QIAamp ViralRNA Mini Kit[德国凯杰(QIAgen)生物公司]说明书进行病毒RNA提取,最终核酸洗脱体积为100 μl,用于札如病毒检测,或-70 ℃保存备检,试剂在有效期内使用。
4. 实时荧光RT-PCR方法检测札如病毒[10-11]:使用札如病毒核酸检测试剂盒(江苏硕世生物科技有限公司)和7500实时荧光定量PCR仪(美国应用生物系统公司)对样本进行札如病毒核酸检测。札如病毒阳性的样本使用3种RT-PCR(RT-PCR、半巢式RT-PCR和巢式RT-PCR)方法扩增札如病毒VP1区基因和聚合酶(RdRp)区基因,使用One Step RT-PCR Kit[德国凯杰(QIAgen)生物公司]进行一轮扩增,使用AmpliTaq Gold®360 MasterMix(美国生命技术公司)进行二轮扩增。见表 1。使用QIAxcel全自动毛细管电泳仪[德国凯杰(QIAgen)生物公司]对扩增产物进行电泳。试剂均有效期内使用,试验操作和结果判断按说明书进行。
5. 基因型鉴定和进化分析:将3种RT-PCR扩增产物均送生工生物工程(上海)股份有限公司进行测序。使用DNASTAR Lasergene SeqMan Pro软件对测序成功的序列进行拼接,使用BioEdit 7.0.9.0软件中clustalW Multiple alignment对序列进行编辑和比对,使用诺如病毒在线分型工具Norovirus Typing Tool Version 2.0(https://www.rivm.nl/mpf/typingtool/norovirus)进行初步的基因分型和鉴定。使用Mega 6.06软件的邻接法(Neighbor-Joining)构建VP1区和Rap区基因进化树,bootstrap值设置1 000次,札如病毒参考序列选自GenBank。
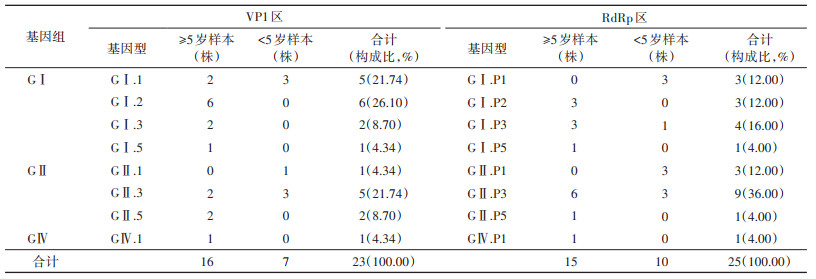
结果1. 札如病毒检出结果:共采集1 522件粪便样本,其中≥5岁病例样本983件,<5岁病例样本539件。用实时荧光RT-PCR对1 522件腹泻病例便样本进行札如病毒检测,总检出率为2.89%(44/1 522),其中≥5岁病例的检出率为2.64%(26/983),<5岁病例的检出率为3.34%(18/539)。
2. 札如病毒测序及鉴定:对札如病毒阳性的44件样本分别扩增VP1区基因和RdRp区基因,扩增阳性产物进行基因测序。VP1区基因共23株实验株测序成功,共检出8个基因型别,以GⅠ组为主(60.87%,14/23);≥5岁病例以GⅠ.2型为主要型别(37.50%,6/16),<5岁病例中GⅠ.1型和GⅡ.3型占比最高(均为42.86%,3/7);RdRp区基因共25株测序成功,共检出8个基因型别,其中GⅡ组占比较高(52.00%,13/25);≥5岁病例以GⅡ.P3型为主要型别(40.00%,6/15),<5岁病例中则以GⅠ.P1型、GⅡ.P1型和GⅡ.P3型为主要型别(均为30.00%,3/10)。见表 2。VP1区和RdRp区基因型合并分型结果显示,共有18株在2个区均获得结果,包含9个基因型别,其中5株GⅡ.P3~GⅡ.3型,GⅠ.P1~GⅠ.1型和GⅠ.P2~GⅠ.2型各3株,2株GⅠ.P3~GⅠ.3型,GⅠ.P5~GⅠ.5型、GⅡ.P1~ GⅡ.1型、GⅡ.P5~GⅡ.5型、GⅣ.P1~GⅣ.1型和GⅡ.P3~GⅠ.2型各1株。
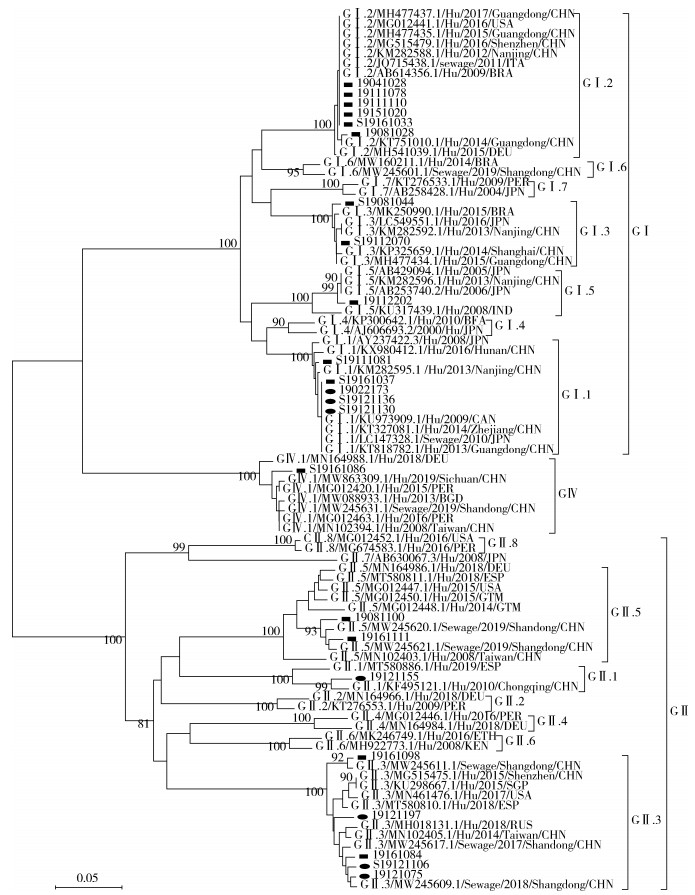
3. VP1区札如病毒基因组序列同源性和进化分析:VP1区测序成功的可分8个基因型,每个基因型内部相似性为95.5%~100.0%,其中5株GⅠ.1型中4株实验株S19161037、19022173、S19121136和S19121130相似性为100.0%,6株GⅠ.2型中5株实验株S19161033、19151020、19111110、19111078和19041028相似性为100.0%。23株8个基因型与对应的51株参考株相似性为92.2%~100.0%,其中GⅠ.1型实验株S19161037、19022173、S19121136和S19121130与4株参考株KU973909.1/Hu/2009/CAN、KT327081.1/Hu/2014/Zhejiang/CHN、LC147328.1/Sewage/2010/JPN和KT818782.1/Hu/2013/Guangdong/CHN相似性均为100.0%,进化最为接近。GⅠ.2型实验株S19161033、19151020、19111110、19111078和19041028与7株参考株AB614356.1/Hu/2009/BRA、JQ715438.1/sewage/2011/ITA、KM282588.1/Hu/2012/Nanjing/CHN、MG515479.1/Hu/2016/Shenzhen/CHN、MH477435.1/Hu/2015/Guangdong/CHN、MH 477437.1/Hu/2017/Guangdong/CHN和MG012441.1/Hu/2016/USA相似性均为100.0%,进化最为接近。GⅠ.3型实验株S19112070与参考株MH477434.1/Hu/2015/Guangdong/CHN相似性为100.0%,进化最为接近。GⅠ.5型实验株19112202与参考株KU317439.1/Hu/2008/IND等4株相似性为95.1%~99.1%,其中与KM282596.1/Hu/2013/Nanjing/CHN相似性为99.1%,进化最为接近。GⅡ.1型实验株19121155与参考株MT580886.1/Hu/2019/ESP和KF495121.1/Hu/2010/Chongqing/CHN相似性为92.2%~97.1%,该实验株与KF495121.1/Hu/2010/Chongqing/CHN相似性为97.1%,进化最为接近。GⅡ.3型实验株19161084和19161098与MT580810.1等7株参考株相似性为94.6%~99.1%,其中实验株19161098与MW245611.1/Sewage/Shangdong/CHN相似性为92.2%,进化最为接近。19121075等3株GⅡ.3型实验株与MT580810.1等9株参考株相似性为95.1%~99.1%,其中实验株19121075与参考株MW245609.1/Sewage/2018/Shangdong/CHN相似性为99.1%,进化最为接近。GⅡ.5型实验株19161111和19081100与MW245621.1等8株参考株相似性为91.4%~98.3%,其中实验株19161111与参考株MW245621.1/Sewage/2019/Shangdong/CHN相似性为98.3%,进化最为接近。GⅣ.1型实验株S19161086与MW863309.1等7株参考株相似性为95.9%~98.3%,该实验株与参考株MW088933.1/Hu/2013/BGD相似性为98.3%,进化最为接近。见图 1。
|
注: :≥5岁样本的(16株实验株)基因序列; :≥5岁样本的(16株实验株)基因序列; :<5岁样本的(7株实验株)基因序列;参考株序列信息包括基因型、GenBank序列号、宿主来源、时间和地点
图 1 2019年北京市腹泻病例中23株札如病毒实验株VP1区(245 bp)进化分析 :<5岁样本的(7株实验株)基因序列;参考株序列信息包括基因型、GenBank序列号、宿主来源、时间和地点
图 1 2019年北京市腹泻病例中23株札如病毒实验株VP1区(245 bp)进化分析
|
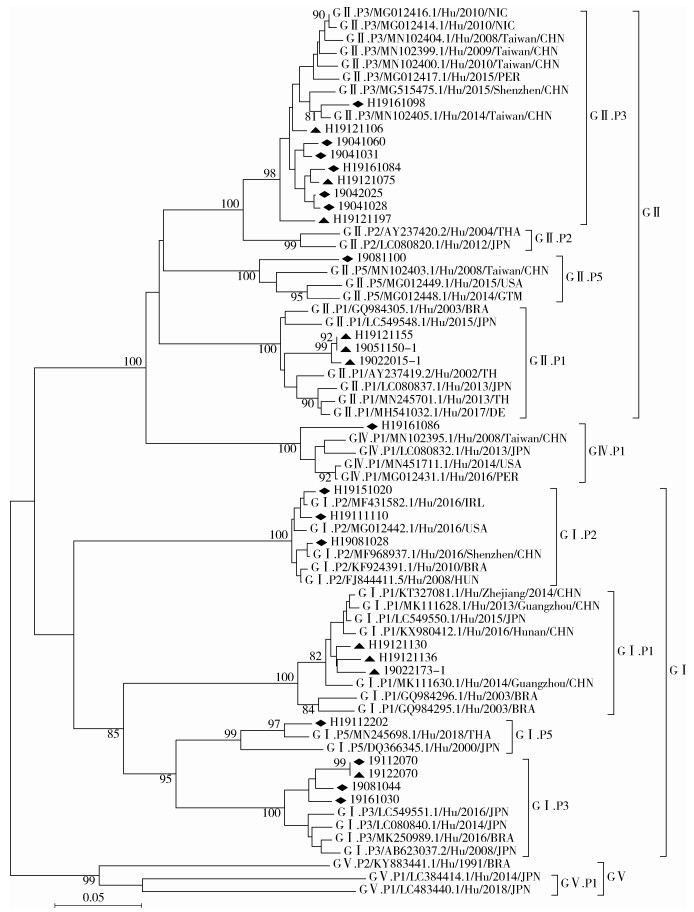
4. RdRp区札如病毒基因组序列同源性及进化分析:RdRp区测序成功的25株可分8个基因型,每个基因型内部相似性为94.0%~100.0%,其中4株GⅠ.P3型中2株实验株19112070和19122070相似性为100.0%,3株GⅡ.P1型中2株实验株H19121155和19051150-1相似性为100.0%。8个基因型与对应的39株参考株相似性为90.2%~99.1%,H19121130等3株GⅠ.P1型实验株与GQ984296.1等7株参考株相似性为90.7%~98.7%,其中实验株H19121130与参考株KT327081.1/Hu/Zhejiang/2014/CHN相似性为98.7%,进化最为接近。H19151020等3株GⅠ.P2实验株与MG012442.1等5株参考株相似性为97.0%~99.1%,其中H19151020实验株与参考株MF431582.1/Hu/2016/IRL相似性为99.1%,H19081028实验株与参考株MF968937.1/Hu/2016/Shenzhen/CHN相似性为99.1%,进化最为接近。19122070等4株GⅠ.P3型实验株与LC549551.1等4株参考株相似性为92.8%~95.7%,其中19112070与19122070实验株与参考株AB623037.2/Hu/2008/JPN相似性均为95.7%,进化最为接近。GⅠ.P5型实验株H19112202与参考株DQ366345.1和MN245698.1相似性为91.5%~96.2%,该实验株与参考株MN245698.1/Hu/2018/THA相似性为96.2%,进化最为接近。H19121155等3株GⅡ.P1型实验株与GQ984305.1等6株参考株相似性为92.8%~95.3%,其中H19121155、19051150-1实验株与参考株GQ984305.1/Hu/2003/BRA、AY237419.2/Hu/2002/TH相似性均为95.3%,进化最为接近。H19121197等9株GⅡ.P3型实验株与MG515475.1等8株参考株相似性为94.0%~97.8%,其中H19161098实验株与参考株MN102405.1/Hu/2014/Taiwan/CHN相似性为97.8%,进化最为接近。GⅡ.P5型实验株19081100与MG012449.1等3株参考株相似性为90.2%~91.9%,其中该实验室与参考株MG012449.1/Hu/2015/USA相似性为91.9%,进化较为接近。GⅣ.P1型实验株H19161086与MN102395.1等4株参考株相似性为92.4%~94.5%,其中该实验株与参考株MN102395.1/Hu/2008/Taiwan/CHN、MN451711.1/Hu/2014/USA相似性均为94.5%,进化最为接近。见图 2。
|
注: : ≥5岁样本的(15株实验株)基因序列; : ≥5岁样本的(15株实验株)基因序列; :<5岁样本的(10株实验株)基因序列;参考株序列信息包括基因型、GenBank序列号、宿主来源、时间和地点
图 2 2019年北京市腹泻病例中25株札如病毒实验株RdRp区(237 bp)进化分析 :<5岁样本的(10株实验株)基因序列;参考株序列信息包括基因型、GenBank序列号、宿主来源、时间和地点
图 2 2019年北京市腹泻病例中25株札如病毒实验株RdRp区(237 bp)进化分析
|
目前病毒性感染已成为腹泻的主要原因之一[12],而国内外研究表明,札如病毒是继诺如病毒后腹泻的重要病原体[2-6]。2019年北京市门诊腹泻病例中札如病毒总检出率为2.89%。通过对北京市札如病毒监测腹泻病例样本进行病原学分析发现,VP1区共有23株测序成功,其中≥5岁病例共16株测序成功,检出7个基因型,其中6株GⅠ.2型(37.50%),GⅠ.1型、GⅠ.3型、GⅡ.3型和GⅡ.5型各2株(均为12.50%)、GⅠ.5型和GⅣ.1型各1株(均为6.25%),基因型别多样化且以GⅠ.2型为主要型别。札如病毒在我国不同地区和不同年份的流行株均有不同,靳淼等[13]研究显示,2008-2009年北京市成年人急性胃肠炎病例的札如病毒主要型别为GⅣ.1型,其次为GⅠ.2型和GⅡ.3型,Wang等[14]研究2011-2013年上海市门诊成年人腹泻病例报道的主要流行株为GⅠ.2型。有研究发现,GⅠ.2型已成为我国多个地区札如病毒新的流行型别[14-17]。本研究发现,≥5岁病例以GⅠ.2型为主要基因型,与这些文献报道的结论一致,而国外文献报道的主要流行型别为GⅠ.1型[18-20]。说明我国各地区≥5岁病例的札如病毒基因型别呈多样性和复杂性,有必要开展连续和长期监测工作。
<5岁病例VP1区共7株测序成功,检出3个基因型,GⅠ.1型与GⅡ.3型各3株(42.86%)和GⅡ.1型1株(14.29%),主要型别为GⅠ.1型和GⅡ.3型。而李响等[15]的研究显示2014年吉林省札如病毒感染病例均为 <5岁儿童,流行株为GⅠ.2型,本研究结论与其结果并不相同,说明国内各地区 <5岁病例札如病毒的流株有所差异。相关文献研究认为GⅡ.3型易在儿童中检出且易引起暴发疫情[21-23],这也有可能是本研究中GⅡ.3型多在24~59月龄的儿童中检出的原因。提示今后应加强 <5岁儿童及暴发疫情中札如病毒感染情况的研究。
RdRp区共测序成功25株,其中≥5岁病例有15株,共检出6个基因型别,其中GⅡ.P3型为主要基因型别。目前,国内外较少研究札如病毒RdRp区的流行型别,无法获得其流行特征,提示应继续加强相关研究。<5岁病例RdRp区共测序成功10株,GⅠ.P1型、GⅡ.P1型和GⅡ.P3型各3株(30.00%),均为主要型别,1株GⅠ.P3型(10.00%)。泰国的 <5岁儿童多项研究发现,主要型别为GⅡ.P3型[22-24],与本研究结论不完全一致。
在≥5岁病例中,共11株在VP1和RdRp的2个区均获得分型,仅检出1株为GⅡ.P3与GⅠ.2型,考虑为混合感染或重组株。在 <5岁病例中,在VP1和RdRp的2个区均获得分型结果的7株中,未检出混合感染或重组株。说明2019年北京市札如病毒的多基因混合感染及重组株的现象较少。蒋翠莲等[25]对2株GⅠ.1型札如病毒进行了全基因测序,未发现重组现象。所以,今后应对札如病毒监测中出现的混合感染或重组株进行全基因组测序,以获得更准确的结论。
通过同源性及进化分析发现,测序成功的48株实验株中,有10株实验株(4株GⅠ.1型、5株GⅠ.2型和1株GⅠ.3型)与不同年份不同国家的人源参考株和污水源参考株(共12株)的相似性为100.0%,其他38株实验株与对应基因型的不同人源及污水源参考株(共78株)的相似性为90.2%~99.1%,与多项研究报道的观点一致,说明札如病毒在世界范围可通过环境、水源和人际传播。
本研究存在局限性。一是未下载到国内外食物来源的参考株,无法深入分析札如病毒的食源性传播。二是实验株与国内多株参考株高度同源,但无北京地区参考株。另外,同源性分析发现,与实验株高度同源的污水源参考株,来自我国其他地区且年份不同。北京地区尚未对水源、食源中札如病毒检出情况进行研究,提示今后应开展对环境、水源及食源性札如病毒的监测。
利益冲突 所有作者声明无利益冲突
作者贡献声明 李爽:研究设计、样品检测、论文撰写/修改、试验数据分析;高志勇:研究设计;李伟红、田祎、刘白薇、沈玲羽:数据收集、样品检测;严寒秋:研究设计、样品检测、试验数据分析、论文修改;贾蕾、张代涛、王全意:研究设计、经费支持
| [1] |
Madeley CR, Cosgrove BP. Caliciviruses in man[J]. Lancet, 1976, 1(7952): 199-200. DOI:10.1016/s0140-6736(76)91309-x |
| [2] |
Oka T, Wang QH, Katayama K, et al. Comprehensive review of human sapoviruses[J]. Clin Microbiol Rev, 2015, 28(1): 32-53. DOI:10.1128/CMR.00011-14 |
| [3] |
马小珍, 曹冉冉, 廖雪春, 等. 2011-2020年成都某哨点医院5岁以下儿童病毒性腹泻病原谱分析[J]. 国际病毒学杂志, 2022, 29(2): 113-118. DOI:10.3760/cma.j.issn.1673-4092.2022.02.006 Ma XZ, Cao RR, Liao XC, et al. Pathogen spectrum analysis of viral diarrhea in children under 5 years old in a sentinel hospital in Chengdu from 2011 to 2020[J]. Int J Virol, 2022, 29(2): 113-118. DOI:10.3760/cma.j.issn.1673-4092.2022.02.006 |
| [4] |
李爽, 严寒秋, 高志勇, 等. 北京市2019年肠道门诊腹泻病例札如病毒感染特征分析[J]. 国际病毒学杂志, 2021, 28(3): 222-225. DOI:10.3760/cma.j.issn.1673-4092.2021.03.011 Li S, Yan HQ, Gao ZY, et al. Infection characteristics of sapoviruses in diarrhea cases admitted to the intestinal clinics in 2019, Beijing[J]. Int J Virol, 2021, 28(3): 222-225. DOI:10.3760/cma.j.issn.1673-4092.2021.03.011 |
| [5] |
于悦, 郭新慧, 严寒秋, 等. 札如病毒急性胃肠炎暴发特征系统综述[J]. 中华流行病学杂志, 2019, 40(1): 93-98. DOI:10.3760/cma.j.issn.0254-6450.2019.01.019 Yu Y, Guo XH, Yan HQ, et al. Systematic review on the characteristics of acute gastroenteritis outbreaks caused by sapovirus[J]. Chin J Epidemiol, 2019, 40(1): 93-98. DOI:10.3760/cma.j.issn.0254-6450.2019.01.019 |
| [6] |
Iritani N, Yamamoto SP, Abe N, et al. Epidemics of GⅠ. 2 sapovirus in gastroenteritis outbreaks during 2012-2013 in Osaka City, Japan[J]. J Med Virol, 2016, 88(7): 1187-1193. DOI:10.1002/jmv.24451 |
| [7] |
Oka T, Mori K, Iritani N, et al. Human sapovirus classification based on complete capsid nucleotide sequences[J]. Arch Virol, 2012, 157(2): 349-352. DOI:10.1007/s00705-011-1161-2 |
| [8] |
Xue L, Cai WC, Gao JS, et al. Genome characteristics and molecular evolution of the human sapovirus variant GⅡ. 8[J]. Infect Genet Evol, 2019, 73: 362-367. DOI:10.1016/j.meegid.2019.05.017 |
| [9] |
Tsinda EK, Malasao R, Furuse Y, et al. Complete coding genome sequences of uncommon GII. 8 sapovirus strains identified in diarrhea samples collected from Peruvian children[J]. Genome Announc, 2017, 5(43): e01137-17. DOI:10.1128/genomeA.01137-17 |
| [10] |
Pang XL, Honma S, Nakata S, et al. Human caliciviruses in acute gastroenteritis of young children in the community[J]. J Infect Dis, 2000, 181(Suppl 2): S288-294. DOI:10.1086/315590 |
| [11] |
Jiang X, Huang PW, Zhong WM, et al. Design and evaluation of a primer pair that detects both Norwalk- and Sapporo-like caliciviruses by RT-PCR[J]. J Virol Methods, 1999, 83(1/2): 145-154. DOI:10.1016/S0166-0934(99)00114-7 |
| [12] |
苏通, 刘莹莹, 赵文娜, 等. 2015-2020年河北省其它感染性腹泻流行特征分析[J]. 国际病毒学杂志, 2021, 28(6): 494-497. DOI:10.3760/cma.j.issn.1673-4092.2021.06.013 Su T, Liu YY, Zhao WN, et al. Epidemiological characteristics of other types of infectious diarrhea in Hebei province, 2015-2020[J]. Int J Virol, 2021, 28(6): 494-497. DOI:10.3760/cma.j.issn.1673-4092.2021.06.013 |
| [13] |
靳淼, 李慧莹, 孔翔羽, 等. 北京2008-2009年成人急性胃肠炎病例中札如病毒的检测和分型[J]. 中华实验和临床病毒学杂志, 2015, 29(1): 62-64. DOI:10.3760/cma.j.issn.1003-9279.2015.01.021 Jin M, Li HY, Kong XY, et al. Detection and typing assay of sapovirus in adults with acute gastroenteritis from 2008 to 2009 in Beijing[J]. Chin J Exp Clin Virol, 2015, 29(1): 62-64. DOI:10.3760/cma.j.issn.1003-9279.2015.01.021 |
| [14] |
Wang G, Shen Z, Qian FX, et al. Genetic diversity of sapovirus in non-hospitalized adults with sporadic cases of acute gastroenteritis in Shanghai, China[J]. J Clin Virol, 2014, 59(4): 250-254. DOI:10.1016/j.jcv.2014.01.007 |
| [15] |
李响, 吴东林, 杨显达, 等. 2014年吉林省札如病毒的检测及基因分型[J]. 中国卫生工程学, 2015, 14(6): 508-510. DOI:10.19937/j.issn.1671-4199.2015.06.008 Li X, Wu DL, Yang XD, et al. Detection and genotype analysis of sapovirus in Jilin province in 2014[J]. Chin J Public Health Eng, 2015, 14(6): 508-510. DOI:10.19937/j.issn.1671-4199.2015.06.008 |
| [16] |
张海龙, 叶郁辉, 何雅青, 等. 2016年深圳市腹泻患者札如病毒感染特征分析[J]. 国际病毒学杂志, 2019, 26(1): 31-34. DOI:10.3760/cma.j.issn.1673-4092.2019.01.09 Zhang HL, Ye YH, He YQ, et al. The characteristics of sapovirus infection among patients with diarrhea in Shenzhen in 2016[J]. Int J Virol, 2019, 26(1): 31-34. DOI:10.3760/cma.j.issn.1673-4092.2019.01.09 |
| [17] |
石鑫, 许军, 舒畅, 等. 黑龙江省札如病毒感染流行病学及基因型分析[J]. 中国公共卫生管理, 2021, 37(1): 69-71. DOI:10.19568/j.cnki.23-1318.2021.01.0017 Shi X, Xu J, Shu C, et al. Genotype and epidemiology analysis of sapovirus in Heilongjiang Province[J]. Chin J Public Health Manage, 2021, 37(1): 69-71. DOI:10.19568/j.cnki.23-1318.2021.01.0017 |
| [18] |
庞蓓蓓, 周璇, Kobayashi N, 等. 2011年和2013年武汉市病毒性胃肠炎病原监测与分析[J]. 中国人兽共患病学报, 2016, 32(5): 457-461. DOI:10.3969/j.issn.1002-2694.2016.05.008 Pang BB, Zhou X, Kobayashi N, et al. Surveillance and analysis of the pathogens of viral gastroenteritis in 2011 and 2013, Wuhan, China[J]. Chin J Zoonoses, 2016, 32(5): 457-461. DOI:10.3969/j.issn.1002-2694.2016.05.008 |
| [19] |
Becker-Dreps S, González F, Bucardo F. Sapovirus: an emerging cause of childhood diarrhea[J]. Curr Opin Infect Dis, 2020, 33(5): 388-397. DOI:10.1097/QCO.0000000000000671 |
| [20] |
Varela MF, Rivadulla E, Lema A, et al. Human sapovirus among outpatients with acute gastroenteritis in Spain: a one-year study[J]. Viruses, 2019, 11(2): 144. DOI:10.3390/v11020144 |
| [21] |
龙冬玲, 庄辉元, 靳淼, 等. 一起由札如病毒引起的急性胃肠炎暴发疫情的分子流行病学研究[J]. 国际病毒学杂志, 2017, 24(3): 183-186. DOI:10.3760/cma.j.issn.1673-4092.2017.03.009 Long DL, Zhuang HY, Jin M, et al. Molecular epidemiology of an acute gastroenteritis outbreak caused by sapovirus[J]. Int J Virol, 2017, 24(3): 183-186. DOI:10.3760/cma.j.issn.1673-4092.2017.03.009 |
| [22] |
Khamrin P, Maneekarn N, Thongprachum A, et al. Emergence of new norovirus variants and genetic heterogeneity of noroviruses and sapoviruses in children admitted to hospital with diarrhea in Thailand[J]. J Med Virol, 2010, 82(2): 289-296. DOI:10.1002/jmv.21640 |
| [23] |
Chaimongkol N, Khamrin P, Malasao R, et al. Molecular characterization of norovirus variants and genetic diversity of noroviruses and sapoviruses in Thailand[J]. J Med Virol, 2014, 86(7): 1210-1218. DOI:10.1002/jmv.23781 |
| [24] |
Kittigul L, Pombubpa K, Taweekate Y, et al. Molecular characterization of rotaviruses, noroviruses, sapovirus, and adenoviruses in patients with acute gastroenteritis in Thailand[J]. J Med Virol, 2009, 81(2): 345-353. DOI:10.1002/jmv.21380 |
| [25] |
蒋翠莲, 曹雪, 艾静, 等. 两株GⅠ. 1型札如病毒全基因组测序及初步分析[J]. 国际病毒学杂志, 2022, 29(5): 408-411. DOI:10.3760/cma.j.issn.1673-4092.2022.05.015 Jiang CL, Cao X, Ai J, et al. Whole genome sequencing and preliminary analysis of two strains of GⅠ. 1 sapovirus[J]. Int J Virol, 2022, 29(5): 408-411. DOI:10.3760/cma.j.issn.1673-4092.2022.05.015 |
 2023, Vol. 44
2023, Vol. 44