文章信息
- 徐响, 蔡雨欣, 刘永红, 沈莹, 潘阳, 姚慧, 王小莉, 杨鹏.
- Xu Xiang, Cai Yuxin, Liu Yonghong, Shen Ying, Pan Yang, Yao Hui, Wang Xiaoli, Yang Peng
- 北京市新型冠状病毒Omicron BA.2和BF.7变异株感染者潜伏期特征分析
- Comparison of incubation periods of infections of Omicron variants BA.2 and BF.7 in Beijing
- 中华流行病学杂志, 2023, 44(9): 1397-1401
- Chinese Journal of Epidemiology, 2023, 44(9): 1397-1401
- http://dx.doi.org/10.3760/cma.j.cn112338-20230316-00153
-
文章历史
收稿日期: 2023-03-16
2. 北京市疾病预防控制中心传染病地方病控制所, 北京 100013;
3. 北京市疾病预防控制中心办公室, 北京 100013
2. Institute for Infectious Disease and Endemic Disease Control, Beijing Center for Disease Prevention and Control, Beijing 100013, China;
3. Central Office, Beijing Center for Disease Prevention and Control, Beijing 100013, China
新型冠状病毒(新冠病毒)Omicron变异株具有多分支谱系和显著的遗传多样化,变异类型多,其分支及子代分支包括BA.2、BA.5、BA.5.2、BF.7、BQ.1、XBB等。2022年下半年国内流行以Omicron BA.5.2和BF.7变异株为主。潜伏期指病原体侵入机体到机体最早出现临床症状的时间[1]。一般来说,潜伏期越短,病毒的传播速度越快。估算传染病的潜伏期可判定感染者的感染时间,进而明确传染源和传播途径,评估疫情传播风险。早期Omicron变异株潜伏期短于先前出现的毒株,但目前BA.2和BF.7变异株相关的潜伏期研究较少,且多数研究中感染者的暴露时间不明确。本研究收集北京市2022年由BA.2和BF.7变异株引起的2起疫情中暴露日期明确的病例信息,利用流行病学调查(流调)结果,估算2种变异株的潜伏期及其影响因素。
资料与方法1. 资料来源:来源于中国疾病预防控制信息系统的传染病报告信息管理系统,按报告日期收集2022年4月22日至5月8日、10月27日至11月20日北京市新冠病毒本土感染者传染病报告卡个案,根据现场流调报告选取传播链清晰且具有明确单次暴露史的感染者作为研究对象,对其进行病毒全基因组测序,其中包括64例BA.2变异株感染者和58例BF.7变异株感染者。现场流调的内容包括感染者发病时间、末次接触阳性感染者时间、是否接种疫苗、疫苗接种剂次、末次疫苗接种时间等信息。
2. 相关定义:①潜伏期:从感染疾病到出现临床症状的时间[2],选择流调资料准确、发病时间明确、暴露史明确的确诊感染者进行计算[3],潜伏期=发病日期-暴露日期。②单次暴露:传播链清晰且流调资料准确,有唯一暴露来源且仅暴露一次。③临床分型:感染者具体分类及判定标准参照当时发布的最新版《新型冠状病毒肺炎防控方案》。2022年4月疫情参照第八版[4],2022年10月疫情参照第九版[5],两版防控方案对于感染者的分类及判定标准一致。④病原学检测:参照《新型冠状病毒肺炎防控方案(第八版)》[4]和《新型冠状病毒肺炎防控方案(第九版)》[5]中《新冠病毒标本采集和检测技术指南》,对采集的病例咽拭子应用实时荧光定量RT-PCR方法进行新冠病毒核酸检测。
3. 统计学分析:应用Excel 2016软件建立数据库和绘图,SPSS 22.0软件进行统计学分析,R 4.1.1软件进行潜伏期的分布拟合。计数资料采用频数、构成比或率描述,计量资料采用M(Q1,Q3)描述。分类变量采用χ2检验和秩和检验进行分析。采用Weibull、Gamma和lognormal概率密度分布模型拟合相关数据。采用Akaike信息准则(AIC)进行定量分析,其中AIC值越小,说明模型拟合程度越高[6]。采用多因素方差分析探究潜伏期是否受感染者的年龄、性别、是否接种疫苗以及变异株类型的影响。双侧检验,检验水准α=0.05。
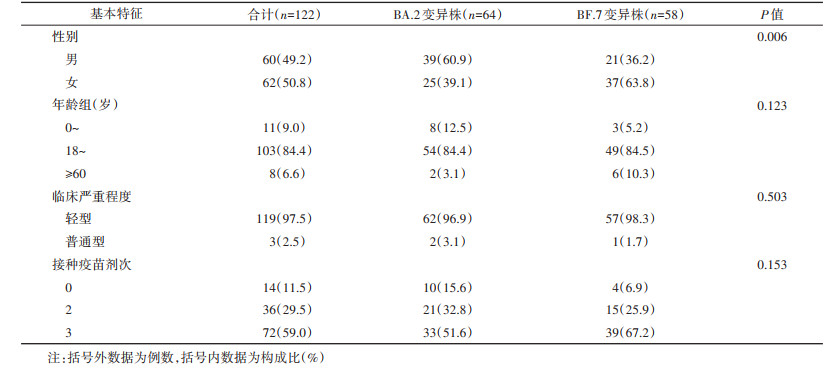
结果1. 基本特征:对122例Omicron BA.2和BF.7变异株感染者进行分析,其中BA.2变异株感染者64例,BF.7变异株感染者58例。2起疫情中,男性60例,女性62例;年龄范围2~69岁,中位年龄30.5岁,84.4%的感染者年龄在18~59岁;轻型119例(97.5%),普通型3例(2.5%);36例(29.5%)感染者接种2剂次疫苗但未完成加强针接种,72例(59.0%)感染者完成加强针接种,14例(11.5%)感染者未接种疫苗(BA.2变异株感染者10例,BF.7变异株感染者4例)。见表 1。
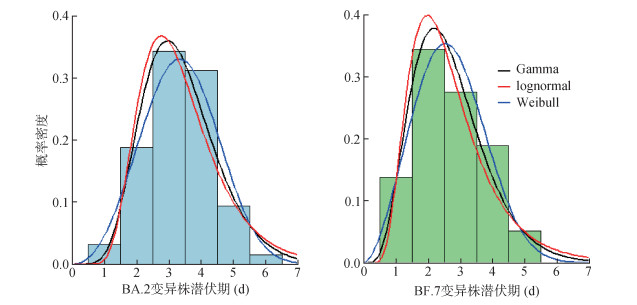
2. 潜伏期分布特征:利用122例感染者的已知暴露信息和发病信息,估算出不同Omicron变异株潜伏期。64例测序结果为BA.2变异株的感染者中,潜伏期最长7.00 d,最短1.00 d,潜伏期M(Q1,Q3)为3.00(3.00,4.00)d;58例测序结果为BF.7变异株的感染者中,潜伏期最长5.00 d,最短1.00 d,潜伏期M(Q1,Q3)为3.00(2.00,3.25)d。Omicron BF.7变异株较BA.2变异株潜伏期有所缩短。
3种分布模型中,潜伏期的估计以lognormal分布对应的AIC值最小(BA.2和BF.7变异株的lognormal分布AIC值分别为57.44和77.55)。基于3种分布模型的概率密度分布见图 1。对于BA.2变异株,80%的感染者潜伏期在4.30 d内,90%的感染者潜伏期在5.06 d内;对于BF.7变异株,80%的感染者潜伏期在3.57 d内,90%的感染者的潜伏期在4.36 d内。
|
| 图 1 北京市Omicron变异株潜伏期的概率密度分布 |
3.潜伏期影响因素分析:多因素方差分析结果显示,不同变异株的主效应差异有统计学意义(F=4.97,P=0.029),性别(F=0.37,P=0.547)、年龄(F=0.88,P=0.351)、是否接种疫苗(F=0.71,P=0.401)的主效应差异无统计学意义。
讨论本研究根据流调结果估算潜伏期,并探究影响潜伏期的因素,发现Omicron BF.7变异株的潜伏期较BA.2变异株有缩短迹象。一项Meta分析表明,Omicron变异株感染者的潜伏期较原始株和其他变异株均有所缩短[7]。在Omicron变异株流行初期,韩国(Delta和Omicron BA.1变异株的潜伏期分别为6.50和3.50 d)[8]、日本(原始株、Alpha和Omicron BA.1变异株的潜伏期分别为6.30、4.50和2.80 d)[9]、英国(Alpha、Delta、Omicron BA.1和Omicron BA.2变异株的潜伏期分别为4.19、3.87、3.67和3.48 d)[10]和中国香港地区(Delta和Omicron BA.1变异株的潜伏期分别为4.80和3.60 d)[11]的研究均表明,感染Omicron变异株比感染既往变异株的潜伏期更短。目前尚不清楚Omicron变异株不同分支间的潜伏期是否具有相似特征。本研究利用122例感染者已知的暴露信息和发病信息,得出BA.2和BF.7变异株潜伏期M(Q1,Q3)分别为3.00(3.00,4.00)d和3.00(2.00,3.25)d。其中,BA.2变异株的潜伏期与中国吉林省的研究结果一致[12]。
本研究发现潜伏期的最优分布为lognormal分布,这与Achangwa等[13]的研究结果一致,但也有部分学者发现Gamma分布[14]和Weibull分布[15]为潜伏期最优分布。这可能与研究目的和病例的选择有关。
本研究探究了多种因素对于潜伏期的影响,结果显示不同类型的Omicron变异株潜伏期存在一定差异,BF.7变异株感染者的潜伏期短于BA.2变异株。此外,本研究还考虑了性别、年龄对潜伏期的影响,结果均差异无统计学意义。这和Yang等[16]和Achangwa等[13]的研究结果一致。然而Su等[17]的研究表明,男性潜伏期比女性长3.2 d;Dai等[18]和Kong[19]的研究表明,老年病例可能存在更长的潜伏期。这些结果表明潜伏期的分布某种程度上可能受性别或年龄的影响,但仍需通过进一步的研究来证明。
本研究未发现接种疫苗对潜伏期长短的影响。值得注意的是,新冠病毒疫苗与其他疫苗一样,接种后会随着时间的延长出现抗体水平下降的现象,部分人群通过接种新冠病毒疫苗加强针,刺激机体产生一定滴度的抗体,从而增强疫苗的免疫效果。在本研究中,有51.6%的BA.2变异株感染者、84.5%的BF.7变异株感染者的末次新冠病毒疫苗接种距感染时已超过6个月,其机体内免疫性抗体滴度较接种时有所下降[20],疫苗保护效力也有不同程度的降低,可能是疫苗接种对潜伏期无明显影响的原因之一。
本研究存在局限性。研究中涉及感染者的发病时间主要来自患者主诉,可能存在回忆偏倚;不同剂次、品牌的疫苗保护效果不同,可能会随着时间的增加有不同程度地降低[21-22],因此,为验证疫苗和潜伏期的相关性,需要考虑混杂因素做进一步分析。
综上所述,与Omicron BA.2变异株相比,BF.7变异株的潜伏期更短,传播速度更快。通过研究变异株的流行病学特征,可以更好地了解病毒特性和传播动态,为公共卫生政策制定提供有力的依据。
利益冲突 所有作者声明无利益冲突
作者贡献声明 徐响:数据整理、统计分析、实施研究、论文撰写;蔡雨欣、刘永红:采集/分析解释数据、论文修改审阅;沈莹、潘阳:分析解释数据、论文修改审阅;姚慧:分析解释数据、统计分析;王小莉、杨鹏:研究指导、论文修改审阅
| [1] |
宋倩倩, 赵涵, 方立群, 等. 新型冠状病毒肺炎的早期传染病流行病学参数估计研究[J]. 中华流行病学杂志, 2020, 41(4): 461-465. DOI:10.3760/cma.j.cn112338-20200205-00069 Song QQ, Zhao H, Fang LQ, et al. Study on assessing early epidemiological parameters of COVID-19 epidemic in China[J]. Chin J Epidemiol, 2020, 41(4): 461-465. DOI:10.3760/cma.j.cn112338-20200205-00069 |
| [2] |
Wong KY, Zhou QN, Hu T. Semiparametric regression analysis of doubly-censored data with applications to incubation period estimation[J]. Lifetime Data Anal, 2023, 29(1): 87-114. DOI:10.1007/s10985-022-09567-3 |
| [3] |
吴双胜, 孙瑛, 窦相峰, 等. 北京市3起新型冠状病毒Omicron变异株聚集性疫情的感染来源和传播链分析[J]. 中华流行病学杂志, 2023, 44(3): 373-378. DOI:10.3760/cma.j.cn112338-20221112-00966 Wu SS, Su Y, Dou XF, et al. Analysis on infection sources and transmission chains of three outbreaks caused by 2019-nCoV Omicron variant in Beijing, China[J]. Chin J Epidemiol, 2023, 44(3): 373-378. DOI:10.3760/cma.j.cn112338-20221112-00966 |
| [4] |
国务院应对新型冠状病毒肺炎疫情联防联控机制综合组. 关于印发新型冠状病毒肺炎防控方案(第八版)的通知[EB/OL]. (2021-05-14)[2023-04-24]. http://www.gov.cn/xinwen/2021-05/14/content_5606469.htm.
|
| [5] |
国务院应对新型冠状病毒肺炎疫情联防联控机制综合组. 关于印发新型冠状病毒肺炎防控方案(第九版)的通知[EB/OL]. (2022-06-28)[2023-04-24]. http://www.gov.cn/xinwen/2022-06/28/content_5698168.htm.
|
| [6] |
Ejima K, Kim KS, Ludema C, et al. Estimation of the incubation period of COVID-19 using viral load data[J]. Epidemics, 2021, 35: 100454. DOI:10.1016/j.epidem.2021.100454 |
| [7] |
Wu Y, Kang LY, Guo ZR, et al. Incubation period of COVID-19 caused by unique SARS-CoV-2 strains: a systematic review and meta-analysis[J]. JAMA Netw Open, 2022, 5(8): e2228008. DOI:10.1001/jamanetworkopen.2022.28008 |
| [8] |
Liu YW, Zhao S, Ryu S, et al. Estimating the incubation period of SARS-CoV-2 Omicron BA. 1 variant in comparison with that during the Delta variant dominance in South Korea[J]. One Health, 2022, 15: 100425. DOI:10.1016/j.onehlt.2022.100425 |
| [9] |
Tanaka H, Ogata T, Shibata T, et al. Shorter incubation period among COVID-19 cases with the BA. 1 omicron variant[J]. Int J Environ Res Public Health, 2022, 19(10): 6330. DOI:10.3390/ijerph19106330 |
| [10] |
Ward T, Glaser A, Overton CE, et al. Replacement dynamics and the pathogenesis of the Alpha, Delta and Omicron variants of SARS-CoV-2[J]. Epidemiol Infect, 2023, 151: e32. DOI:10.1017/s0950268822001935 |
| [11] |
Du ZW, Liu CF, Wang L, et al. Shorter serial intervals and incubation periods in SARS-CoV-2 variants than the SARS-CoV-2 ancestral strain[J]. J Travel Med, 2022, 29(6): taac052. DOI:10.1093/jtm/taac052 |
| [12] |
Liu H, Wang S, Yang SQ, et al. Characteristics of the severe acute respiratory syndrome coronavirus 2 Omicron BA. 2 subvariant in Jilin, China from march to May 2022[J]. J Transl Int Med, 2023, 10(4): 349-358. DOI:10.2478/jtim-2022-0054 |
| [13] |
Achangwa C, Park H, Ryu S. Incubation period of wild type of SARS-CoV-2 infections by age, gender, and epidemic periods[J]. Front Public Health, 2022, 10: 905020. DOI:10.3389/fpubh.2022.905020 |
| [14] |
杜志成, 顾菁, 李菁华, 等. 基于区间删失数据估计方法的COVID-19潜伏期分布估计[J]. 中华流行病学杂志, 2020, 41(7): 1000-1003. DOI:10.3760/cma.j.cn112338-20200313-00331 Du ZC, Gu J, Li JH, et al. Estimating the distribution of COVID-19 incubation period by interval-censored data estimation method[J]. Chin J Epidemiol, 2020, 41(7): 1000-1003. DOI:10.3760/cma.j.cn112338-20200313-00331 |
| [15] |
黄勇, 郑志伟, 陈纯, 等. 广州市2起由新型冠状病毒奥密克戎变异株引起的本地疫情流行病学参数研究[J]. 中华流行病学杂志, 2022, 43(11): 1705-1710. DOI:10.3760/cma.j.cn112338-20220523-00450 Huang Y, Zheng ZW, Chen C, et al. Epidemiological characteristics of two local COVID-19 outbreaks caused by 2019-nCoV Omicron variant in Guangzhou, China[J]. Chin J Epidemiol, 2022, 43(11): 1705-1710. DOI:10.3760/cma.j.cn112338-20220523-00450 |
| [16] |
Yang L, Dai JY, Zhao J, et al. Estimation of incubation period and serial interval of COVID-19: analysis of 178 cases and 131 transmission chains in Hubei province, China[J]. Epidemiol Infect, 2020, 148: e117. DOI:10.1017/s0950268820001338 |
| [17] |
Su YJ, Kuo KC, Wang TW, et al. Gender-based differences in COVID-19[J]. New Microbes New Infect, 2021, 42: 100905. DOI:10.1016/j.nmni.2021.100905 |
| [18] |
Dai JY, Yang L, Zhao J. Probable longer incubation period for elderly COVID-19 cases: analysis of 180 contact tracing data in Hubei Province, China[J]. Risk Manag Healthc Policy, 2020, 13: 1111-1117. DOI:10.2147/rmhp.S257907 |
| [19] |
Kong TK. Longer incubation period of coronavirus disease 2019 (COVID-19) in older adults[J]. Aging Med (Milton), 2020, 3(2): 102-109. DOI:10.1002/agm2.12114 |
| [20] |
Zhang HY, Jia YY, Ji Y, et al. Inactivated vaccines against SARS-CoV-2: neutralizing antibody titers in vaccine recipients[J]. Front Microbiol, 2022, 13: 816778. DOI:10.3389/fmicb.2022.816778 |
| [21] |
Suah JL, Husin M, Tok PSK, et al. Waning COVID-19 vaccine effectiveness for BNT162b2 and CoronaVac in malaysia: an observational study[J]. Int J Infect Dis, 2022, 119: 69-76. DOI:10.1016/j.ijid.2022.03.028 |
| [22] |
Adams K, Rhoads JP, Surie D, et al. Vaccine effectiveness of primary series and booster doses against covid-19 associated hospital admissions in the United States: living test negative design study[J]. BMJ, 2022, 379: e072065. DOI:10.1136/bmj-2022-072065 |
 2023, Vol. 44
2023, Vol. 44