文章信息
- 骆晨雨, 张愉涵, 卢明, 陆斌, 蔡洁, 李娜, 周月阳, 骆家辉, 张丁丁, 杨欣壮, 陈宏达, 代敏.
- Luo Chenyu, Zhang Yuhan, Lu Ming, Lu Bin, Cai Jie, Li Na, Zhou Yueyang, Luo Jiahui, Zhang Dingding, Yang Xinzhuang, Chen Hongda, Dai Min
- 基于遗传和环境危险因素构建的风险评分在结直肠肿瘤分级筛查中的应用评价
- Research on applying genetic and environmental risk score in risk-adapted colorectal cancer screening
- 中华流行病学杂志, 2023, 44(6): 999-1005
- Chinese Journal of Epidemiology, 2023, 44(6): 999-1005
- http://dx.doi.org/10.3760/cma.j.cn112338-20221102-00935
-
文章历史
收稿日期: 2022-11-02
2. 国家癌症中心/国家肿瘤临床医学研究中心/中国医学科学院北京协和医学院肿瘤医院流行病学研究室, 北京 100021;
3. 中国医学科学院北京协和医学院北京协和医院基本外科, 北京 100730
2. Department of Cancer Epidemiology, National Cancer Center/National Clinical Research Center for Cancer/Cancer Hospital, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100021, China;
3. Department of General Surgery, Peking Union Medical College Hospital, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100730, China
结直肠癌是全球常见的恶性肿瘤之一。全球癌症统计报告显示,2020年全球结直肠癌新发病例约190万,死亡病例约90万[1]。我国的结直肠癌疾病负担较为沉重,2020年我国估计结直肠癌发病55.5万例,死亡28.6万例[1],是严重危害我国居民健康的公共卫生问题。流行病学研究证据表明,遗传因素与环境因素共同作用于结直肠癌的发生发展[2]。得益于全基因组关联研究(GWAS)的广泛开展,目前已鉴定出若干与结直肠癌相关的遗传易感位点[3]。环境风险因素(吸烟、肥胖等)与结直肠癌的发病风险增加同样密切相关[2, 4]。本研究利用中国人群新型结直肠肿瘤筛查方案比较的多中心随机对照试验(Target-C研究,中国临床试验平台http://www.chictr.org.cn,注册号:ChiCTR1800015506),探讨结直肠肿瘤发生风险与遗传及环境之间的关系,并进一步评价遗传联合环境风险评分分级筛查方案的应用效果,以期提高我国结直肠肿瘤的筛查早诊水平。
对象与方法1. 研究对象:数据来源于Target-C研究,此项目在中国6个地区(安徽省合肥市、湖南省长沙市、江苏省徐州市、云南省昆明市、浙江省温岭市、浙江省兰溪市)开展研究,详细介绍见文献[5-7]。本研究中,在基线筛查阶段共有2 296例参与者进行了结肠镜检查且血液样本DNA质量符合单核苷酸多态性(SNP)分型要求。在排除了缺少病理确诊结果(n=57)、至少1个SNP位点检测失败(n=79)的参与者后,最终纳入的研究对象为2 160例,包括19例结直肠癌、204例进展期腺瘤、471例非进展期腺瘤和1 466例健康对照。
2. 研究内容:
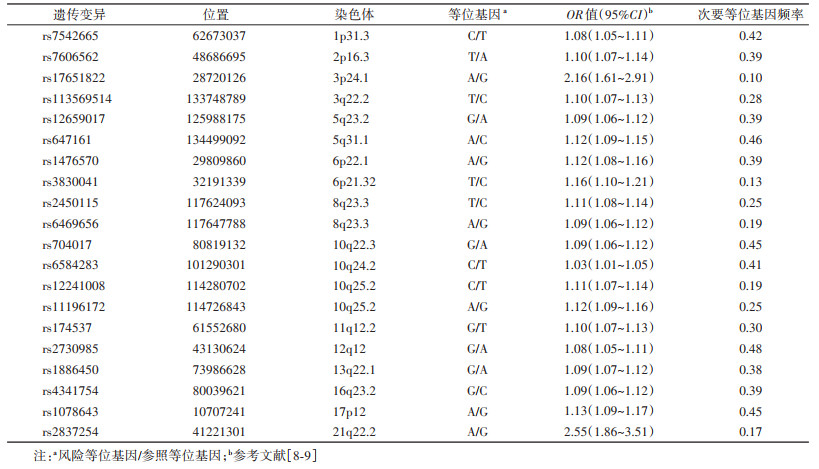
(1)遗传风险评分:在既往GWAS中[8-9],选取已知与东亚地区人群结直肠癌风险显著相关的SNP位点,利用美国Sequenom公司的MassARRAY®分子量阵列技术对相关位点进行实验室检测。遗传变异质控标准:①去除分型成功率 < 95%的遗传变异;②去除次要等位基因频率 < 0.1%的遗传变异;③去除人群中分布频率不满足Hardy-Weinberg遗传平衡定律(P < 0.001)的遗传变异,最终纳入20个遗传易感位点(表 1)。将各位点依据其效应OR值的自然对数(lnOR)加权求和后得到遗传风险评分,计算公式为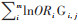
(2)环境风险评分:研究对象的基线调查均采用标准的结构化流行病学问卷,包括一般人口学资料、生活方式、既往病史、一级亲属结直肠癌家族史等信息。利用亚太结直肠筛查评分系统进行环境风险评估。该评分系统对年龄(50~岁=0分;55~岁=1分;65~74岁=2分)、性别(女=0分;男=1分)、一级亲属结直肠癌家族史(无=0分;有=1分)、吸烟状况(不吸烟=0分;吸烟/戒烟=1分)和BMI(< 23 kg/m2=0分;≥23 kg/m2=1分)赋分,相加得到最终的环境风险评分[11]。根据前期的研究结果,本研究将总分≥4分的人群定义为高风险组,< 4分的人群定义为低风险组。
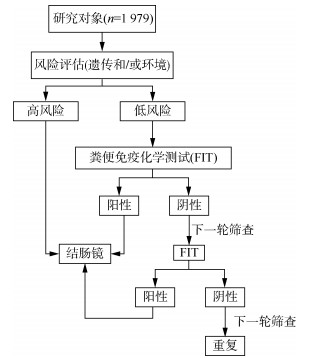
(3)结直肠肿瘤风险分级筛查方案:基于上述环境和遗传风险评分,本研究设计了3种个体化的分级筛查方案,分别为单独使用遗传风险评分、单独使用环境风险评分以及使用遗传和环境联合风险评分(任一评分高风险即为高风险人群)。所有研究对象首先接受遗传和/或环境风险评估,对于评估为高风险的研究对象,接受单次结肠镜筛查,并不再纳入下一轮次的筛查;对于评估为低风险的研究对象,接受定量粪便免疫化学检测(FIT)筛查。FIT结果阳性的研究对象将接受结肠镜检查,阴性者进入下一年的FIT筛查,如FIT阳性且完成了结肠镜检查,则将不被纳入下一轮次的筛查(图 1)。本研究使用的FIT产品为日本荣研公司(Eiken Chemical)生产的OC-Sensor,根据制造商推荐,将≥100 ngHb/ml作为阳性截断值。2 160例研究对象中有181例无定量FIT检测结果,分级筛查方案的效果评价研究最终纳入1 979例。本研究假定在理想情况下,研究对象对筛查方案全部依从。
|
| 图 1 基于遗传和环境风险评分的结直肠肿瘤分级筛查示意图 |
3. 筛查结局定义:最终的诊断结果根据结肠镜检查和病理报告来确定。本研究中,结直肠肿瘤包括进展期肿瘤和非进展期腺瘤。进展期肿瘤包括经病理组织学确诊的结直肠癌(《国际疾病分类》第十次修订版,代码C18~C20)、进展期腺瘤和进展期锯齿状息肉。其中,进展期腺瘤定义为至少具有以下特征之一:①有重度异型增生或高级别上皮内瘤变;②绒毛状或管状绒毛状组织学特征;③直径 > 10 mm的腺瘤[12]。进展期锯齿状息肉定义为传统锯齿状腺瘤或直径 > 10 mm的无蒂锯齿状腺瘤[13]。
4. 统计学分析:使用R 4.3.1软件进行数据分析。分类变量以例数(%)描述,并使用χ2检验进行比较。使用logistic回归分别分析遗传风险单独与结直肠肿瘤风险之间的关联,以及遗传联合环境风险与结直肠肿瘤风险之间的关联。为确定分级筛查方案的效果,计算各方案每轮次的累计检出率、阳性预测值,结肠镜负荷以及筛查出1例进展期肿瘤所需的结肠镜检查数,并使用χ2检验与所有研究对象接受结肠镜检查的方案进行率的比较。以P < 0.05为差异有统计学意义。
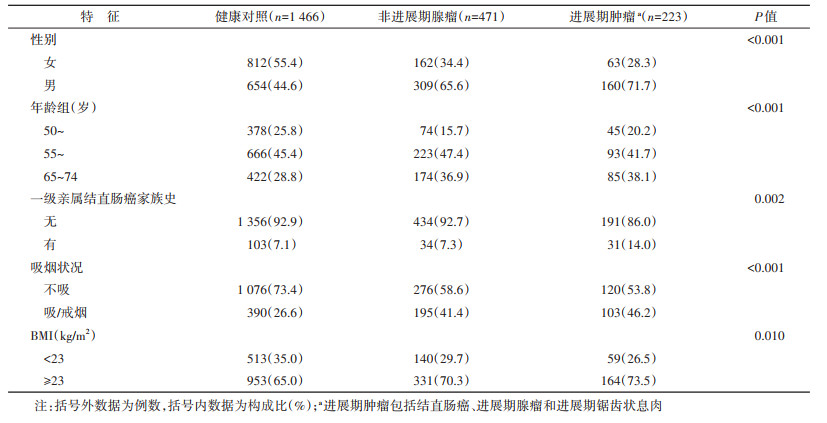
结果1. 一般情况:共纳入2 160例研究对象,包括1 466例健康对照,471例非进展期腺瘤和223例进展期肿瘤(204例进展期腺瘤和19例结直肠癌)。与健康对照相比,在结直肠非进展期腺瘤和进展期肿瘤患者中,男性、年龄65~74岁、有一级亲属结直肠癌家族史、吸/戒烟、BMI≥23 kg/m2者占比较高(表 2)。
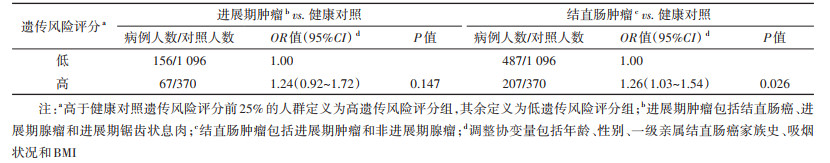
2. 遗传风险评分与结直肠肿瘤风险:在调整环境危险因素后,与低遗传风险评分组相比,高遗传风险评分组发生结直肠肿瘤的风险增加了26%(OR=1.26,95%CI:1.03~1.54,P=0.026)。未观察到高遗传风险评分与进展期肿瘤发病风险之间存在统计学关联(OR=1.24,95%CI:0.92~1.72,P=0.147)。见表 3。
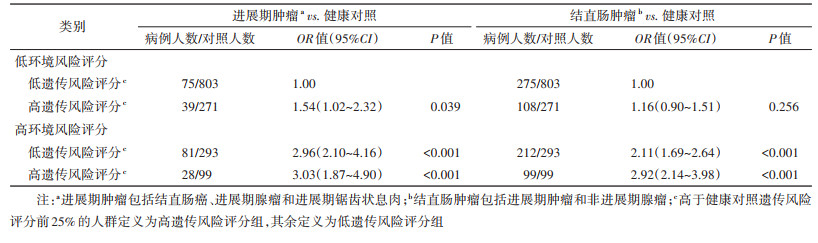
3. 遗传和环境风险联合评分与结直肠肿瘤风险:随着遗传和环境风险评分等级的增加,结直肠肿瘤的发病风险也随之增加。遗传和环境风险评分均最高的研究对象患所有结直肠肿瘤的风险是两评分均最低的研究对象的2.92倍(OR=2.92,95%CI:2.14~3.98,P < 0.001),患进展期肿瘤的风险是两评分均最低的研究对象的3.03倍(OR=3.03,95%CI:1.87~4.90,P < 0.001)。见表 4。
4. 筛查方案的效果评价:在不进行分级筛查,即研究对象全部接受结肠镜检查时,进展期肿瘤的检出率为10.46%,阳性预测值为10.46%,检出1例进展期肿瘤所需的结肠镜检查数为9.6[14]。分级筛查的结果显示,当进行到第3轮筛查时,基于遗传联合环境风险评分构建的分级筛查方案,其进展期肿瘤累计检出率与全部接受结肠镜检查方案差异无统计学意义(8.79% vs. 10.46%,P=0.075),阳性预测值更高(14.11% vs. 10.46%,P < 0.001),每检出1例进展期肿瘤所需的结肠镜检查数更低(7.1 vs. 9.6,P < 0.001)。当进行到第5轮筛查时,遗传联合环境风险评分方案的检出率为9.46%(P=0.288),结肠镜检查数减少了31.39%,阳性预测值为13.79%,检出1例进展期肿瘤所需的结肠镜检查数为7.3(表 5)。
基于中国人群新型结直肠癌筛查方案比较的多中心随机对照试验,本研究探讨了结直肠肿瘤发生风险与遗传和环境风险之间的关系,并提出了风险评估结合FIT筛查的结直肠肿瘤个体化分级筛查方案。结果表明,高遗传风险评分的个体患结直肠肿瘤的风险更高。环境因素可能对遗传风险与结直肠肿瘤的关系具有效应修饰作用。联合遗传风险评分和环境风险评分的分级筛查方案,在提高检测进展期肿瘤的阳性预测值和降低结肠镜检查负荷方面具有一定的转化价值。
本研究使用20个已知与东亚地区人群结直肠癌相关的易感位点来计算遗传风险评分,发现高遗传风险评分的个体发生结直肠肿瘤的风险是健康对照的1.26倍。尽管单个遗传易感位点对结直肠肿瘤遗传风险的影响微弱,但却具有累积效应。因此,研究者常通过计算遗传风险评分来整合遗传变异信息。一项基于英国生物银行的研究利用了37个欧洲地区人群结直肠癌易感位点构建遗传风险评分,结果显示评分居于前1%的人群患结直肠癌的风险是中位数人群的2.9倍[15]。我国一项病例对照研究利用13个遗传位点构建遗传风险评分,同样得到遗传风险评分越高,患结直肠癌风险越高的结果[16]。
本研究结果显示,随着遗传和环境风险评分的增加,结直肠肿瘤的发病风险也随之增加。遗传和环境风险评分均高组的研究对象患进展期肿瘤的风险高于遗传和环境风险评分均低组。分层分析的结果还提示,环境因素可能对遗传风险与结直肠肿瘤的关系具有效应修饰作用。国外一项探究环境和遗传因素对结直肠肿瘤发生风险影响的前瞻性队列研究显示,遗传因素与环境因素间存在加性交互作用[17]。Wang等[18]对11项人群研究的荟萃分析显示,与结直肠癌低遗传风险评分者相比,具有高遗传风险评分的人可能会从践行健康生活方式中获益更多。环境因素的影响可能因个体的遗传易感性而异,可能的机制是某些基因只有在被环境因素激活时才表达[19]。目前,基因-环境交互作用对结直肠肿瘤发病风险的影响仍然有待探索,需要更多的大样本研究提供证据支撑。
本研究结果显示,联合遗传和环境风险评分的分级筛查方案,其进展期肿瘤的检出率在第3轮即与全部接受结肠镜方案差异无统计学意义,并可提高阳性预测值和降低结肠镜检查负荷。该结果提示,使用问卷调查和基因检测有助于结直肠肿瘤风险分层和高危人群的识别,从而确定适宜接受筛查的个体。一些学者已经关注到遗传和环境风险评分在结直肠癌筛查中的应用。例如,Jeon等[20]的研究表明,基于19个环境因素与63个遗传易感位点构建的环境与遗传风险评分模型,可显著提高结直肠癌的风险分层效果,为制订不同人群的筛查适宜年龄提供参考。然而,在筛查人群中应用遗传风险评分的效果尚存在争议。有研究者在基于FIT的筛查人群中将遗传风险评分作为分层依据和FIT阳性后的二次诊断工具,结果显示遗传风险评分对人群风险分层的效果欠佳,阳性预测值的提高甚微[21]。
本研究存在局限性。首先,本研究只纳入了20个遗传易感位点,如果纳入更多位点,风险预测的准确性可能会进一步提高。然而,从人群筛查的转化价值来看,检测有限的位点具有成本低廉和方便快捷的优点。其次,本研究在比较筛查方案效果时,假定各轮次的研究对象对筛查方案全部依从。然而,在实际筛查过程中,各轮次的参与率和依从率会降低。
综上所述,本研究利用Target-C研究的样本开展回顾性分析,结果显示,与基于结肠镜的传统筛查方法相比,联合遗传和环境风险评分、FIT和结肠镜的分级筛查方案,有助于人群风险分层,实现更好的筛查效果。上述结果有助于制定有效的结直肠肿瘤个体化筛查方案,为优化我国结直肠肿瘤防控策略提供参考价值。
利益冲突 所有作者声明无利益冲突
作者贡献声明 骆晨雨:数据整理、统计学分析、论文撰写;张愉涵、卢明、陆斌、蔡洁、李娜、周月阳、骆家辉:数据整理、论文修改;张丁丁、杨欣壮:研究指导;陈宏达、代敏:研究指导、论文修改、经费支持
| [1] |
Sung H, Ferlay J, Siegel RL, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [2] |
Dekker E, Tanis PJ, Vleugels JLA, et al. Colorectal cancer[J]. Lancet, 2019, 394(10207): 1467-1480. DOI:10.1016/S0140-6736(19)32319-0 |
| [3] |
Huyghe JR, Bien SA, Harrison TA, et al. Discovery of common and rare genetic risk variants for colorectal cancer[J]. Nat Genet, 2019, 51(1): 76-87. DOI:10.1038/s41588-018-0286-6 |
| [4] |
Li N, Lu B, Luo CY, et al. Incidence, mortality, survival, risk factor and screening of colorectal cancer: a comparison among China, Europe, and northern America[J]. Cancer Lett, 2021, 522: 255-268. DOI:10.1016/j.canlet.2021.09.034 |
| [5] |
Chen HD, Lu M, Liu CC, et al. Comparative evaluation of participation and diagnostic yield of colonoscopy vs fecal immunochemical test vs risk-adapted screening in colorectal cancer screening: interim analysis of a multicenter randomized controlled trial (TARGET-C)[J]. Am J Gastroenterol, 2020, 115(8): 1264-1274. DOI:10.14309/ajg.0000000000000624 |
| [6] |
Chen HD, Li N, Ren JS, et al. Participation and yield of a population-based colorectal cancer screening programme in China[J]. Gut, 2019, 68(8): 1450-1457. DOI:10.1136/gutjnl-2018-317124 |
| [7] |
Chen HD, Li N, Shi JF, et al. Comparative evaluation of novel screening strategies for colorectal cancer screening in China (TARGET-C): a study protocol for a multicentre randomised controlled trial[J]. BMJ Open, 2019, 9(4): e025935. DOI:10.1136/bmjopen-2018-025935 |
| [8] |
Lu YC, Kweon SS, Tanikawa C, et al. Large-scale genome-wide association study of East Asians identifies loci associated with risk for colorectal cancer[J]. Gastroenterology, 2019, 156(5): 1455-1466. DOI:10.1053/j.gastro.2018.11.066 |
| [9] |
Wang JP, Carvajal-Carmona LG, Chu JH, et al. Germline variants and advanced colorectal adenomas: adenoma prevention with celecoxib trial genome-wide association study[J]. Clin Cancer Res, 2013, 19(23): 6430-6437. DOI:10.1158/1078-0432.CCR-13-0550 |
| [10] |
Archambault AN, Jeon J, Lin Y, et al. Risk stratification for early-onset colorectal cancer using a combination of genetic and environmental risk scores: an international multi-center study[J]. J Natl Cancer Inst, 2022, 114(4): 528-539. DOI:10.1093/JNCI/DJAC003 |
| [11] |
Sung JJY, Ng SC, Chan FKL, et al. An updated Asia Pacific Consensus Recommendations on colorectal cancer screening[J]. Gut, 2015, 64(1): 121-132. DOI:10.1136/gutjnl-2013-306503 |
| [12] |
Gupta S, Lieberman D, Anderson JC, et al. Recommendations for follow-up after colonoscopy and polypectomy: a consensus update by the US multi-society task force on colorectal cancer[J]. Gastroenterology, 2020, 158(4): 1131-1153. e5. DOI:10.1053/j.gastro.2019.10.026 |
| [13] |
Rex DK, Ahnen DJ, Baron JA, et al. Serrated lesions of the colorectum: review and recommendations from an expert panel[J]. Am J Gastroenterol, 2012, 107(9): 1315-1329. DOI:10.1038/ajg.2012.161 |
| [14] |
Chen HD, Shi JF, Lu M, et al. Comparison of colonoscopy, fecal immunochemical test, and risk-adapted approach in a colorectal cancer screening trial (TARGET-C)[J]. Clin Gastroenterol Hepatol, 2023, 21(3): 808-818. DOI:10.1016/j.cgh.2022.08.003 |
| [15] |
Frampton MJE, Law P, Litchfield K, et al. Implications of polygenic risk for personalised colorectal cancer screening[J]. Ann Oncol, 2016, 27(3): 429-434. DOI:10.1093/annonc/mdv540 |
| [16] |
汤德珍, 孟杨, 朱礼明, 等. 遗传和生活方式风险评分与结直肠癌发生风险的评估研究[J]. 医药论坛杂志, 2021, 42(19): 97-101. Tang DZ, Meng Y, Zhu LM, et al. Research of genetic and lifestyle risk score on risk assessment of colorectal cancer[J]. J Med Forum, 2021, 42(19): 97-101. |
| [17] |
Choi J, Jia GC, Wen WQ, et al. Healthy lifestyles, genetic modifiers, and colorectal cancer risk: a prospective cohort study in the UK Biobank[J]. Am J Clin Nutr, 2021, 113(4): 810-820. DOI:10.1093/ajcn/nqaa404 |
| [18] |
Wang XL, O'Connell K, Jeon J, et al. Combined effect of modifiable and non-modifiable risk factors for colorectal cancer risk in a pooled analysis of 11 population-based studies[J]. BMJ Open Gastroenterol, 2019, 6(1): e000339. DOI:10.1136/bmjgast-2019-000339 |
| [19] |
俞维萍. 环境暴露、基因多态性与结直肠癌关系的流行病学研究[D]. 杭州: 浙江大学, 2004. Yu WP. An epidemiological study on environmental exposure factors and genetic polymorphisms of colorectal cancer[D]. Hangzhou: Zhejiang University, 2004. |
| [20] |
Jeon J, Du MM, Schoen RE, et al. Determining risk of colorectal cancer and starting age of screening based on lifestyle, environmental, and genetic factors[J]. Gastroenterology, 2018, 154(8): 2152-2164. e19. DOI:10.1053/j.gastro.2018.02.021 |
| [21] |
Obón-Santacana M, Díez-Villanueva A, Alonso MH, et al. Polygenic risk score across distinct colorectal cancer screening outcomes: from premalignant polyps to colorectal cancer[J]. BMC Med, 2021, 19(1): 261. DOI:10.1186/s12916-021-02134-x |
 2023, Vol. 44
2023, Vol. 44