文章信息
- 王安然, 吴思竹, 刘盛宇, 修晓蕾, 周佳茵, 胡拯涌, 段一凡.
- Wang Anran, Wu Sizhu, Liu Shengyu, Xiu Xiaolei, Zhou Jiayin, Hu Zhengyong, Duan Yifan
- 面向FAIR数据共享的医学通用数据模型比较研究
- Comparative study of medical common data models for FAIR data sharing
- 中华流行病学杂志, 2023, 44(5): 828-836
- Chinese Journal of Epidemiology, 2023, 44(5): 828-836
- http://dx.doi.org/10.3760/cma.j.cn112338-20221025-00908
-
文章历史
收稿日期: 2022-10-25
FAIR原则是科学数据管理的重要指导方针,它倡导实现数据的可发现(findable)、可访问(accessible)、可互操作(interoperable)和可重用(reusable),四个目标层层递进,旨在促进基于优质数据资源的数据密集型科学研究,最大限度发挥数据价值[1-2]。在开放科学背景下,健康医疗大数据作为国家基础性战略资源,其FAIR化建设和开放共享利用对推动数据驱动的医学科技创新至关重要。尤其在流行病学研究领域,研究人员正积极探索如何基于健康医疗数据构建大规模人群队列,开展更快速、更广泛的真实世界研究,为解决人群健康和公共卫生重大问题提供决策依据。美国精准医学“All of Us”研究计划要求参与者授权共享个人电子健康记录(EHR)数据,并基于定期的EHR更新保障人群队列随访[3]。英国通过整合不同医疗卫生系统的多类数据,构建了覆盖超过5 400万人群的国家队列,用以探究疾病风险因素、流行趋势和临床特征[4]。我国同样高度重视健康医疗大数据的积累、共享和应用,以大型数据中心和区域信息平台为载体,构建数据汇聚体系和共享通道,推进数据生命周期管理和数据资源FAIR化建设。虽然我国实体数据汇聚总量增长迅速,但数据资源因采集标准不一、数据结构各异以及语义信息缺失或模糊等问题无法进行有效整合和分析利用,极大地限制了数据潜在价值的挖掘。FAIR原则也强调了构建数据语义模型和互操作的重要性,但我国现有实践更多是在元数据维度建立标准规范。因此,如何强化健康医疗大数据内容层面的语义表达准确性和一致性,提高数据标准化整合和共享利用水平,成为亟需解决的关键问题。
通用数据模型(CDM)定义了统一的数据表示框架并支持引入丰富的术语体系,通过数据结构转换和语义映射处理,实现数据字段、内容和语义多层面的标准化组织。近年来,CDM已发展成为跨区域、跨机构健康医疗数据互联互通、集成整合、协同分析利用的有效工具,在保障多中心队列建设、流行病学分析、公共卫生决策等研究的数据一致性方面发挥重要作用,促进了大规模、低成本的观察性研究开展和临床研究证据的快速生成[5-7]。在新型冠状病毒感染(COVID-19)流行期间,多项基于CDM的跨国、多中心队列研究也为疾病监测、感染预防、临床特征分析等提供了更多有效信息[8-10]。目前发展较为成熟的医学CDM均为国外开发,包括美国国家生物医学计算中心资助开发的i2b2(Informatics for Integrating Biology & the Bedside)CDM[11]、美国观察性健康数据科学和信息学合作组织(Observational Health Data Sciences and Informatics,OHDSI)维护的OMOP(Observational Medical Outcomes Partnership)CDM[12]、美国患者导向医疗效果研究所研发的PCORnet(National Patient-Centered Clinical Research Network)CDM[13]。本研究期望通过探究国外典型医学CDM的先进技术理念与实践模式,为实现我国健康医疗大数据的语义标准化和多层次互联互通,提高数据资源建设质量和共享利用率提供参考。
一、CDMCDM规范了数据结构化存储格式和标准化语义描述方法。它通常根据学科中受关注的主题领域设置不同域表(如诊断、检查、用药、手术等),并对域表的字段结构、字段类型及表间关系等内容进行统一约束,使不同来源数据呈现为相同的存储格式。另一方面,CDM使用概念进行数据实体的标准描述,通过引入术语标准建立原始数据内容(字段属性、字段值)与标准术语概念、编码的一一对应关系,即语义映射,实现数据内容的标准化语义表达。映射后的标准概念、编码和术语词表信息都将存储于CDM域表。为避免数据映射过程中的信息丢失同时方便数据质控与溯源,CDM一般还支持映射前的原始数据和原始语义信息的存储。不同域表通过主外键相链接,构成了标准化关系数据模型,支撑整体数据结构、语义内容的关联。
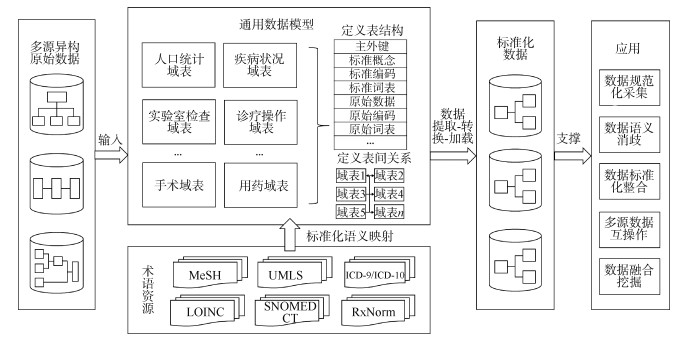
应用统一的术语进行数据内容描述是CDM数据标准化的基础。CDM既支持基于单一术语标准的语义映射,也支持多源术语标准的整合应用,如使用SNOMED CT术语进行原始数据中患者诊断信息的映射、使用LOINC术语进行检验数据映射、使用RxNorm术语进行药物数据映射等。但CDM只提供了数据组织框架,数据结构重组和语义映射过程还需依托开发的提取-转换-加载(ETL)工具或应用程序实现。最终形成的标准化数据集合在多源数据互操作、多方协同研究和多模式数据融合挖掘等方面发挥重要价值。基于CDM的数据标准化模式见图 1。
|
| 图 1 基于通用数据模型的数据标准化模式 |
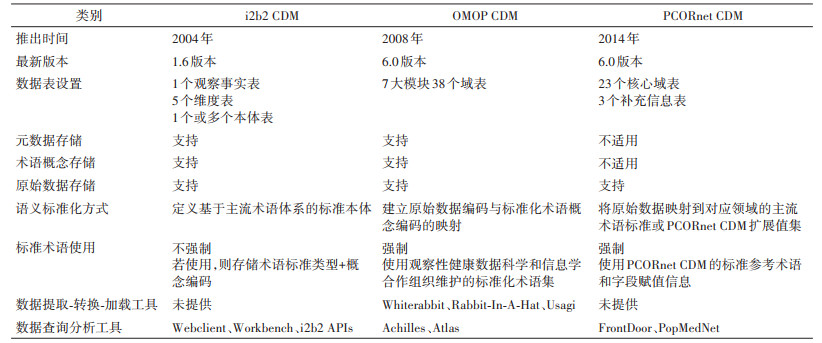
i2b2是一个开源的临床数据仓储和分析平台,该项目开发了i2b2 CDM和一组模块化软件以支持大规模数据的标准化集成存储、管理和查询。OHDSI开放科学社区基于OMOP CDM开发了一系列开源软件,致力于促进数据处理和分析流程的标准化。PCORnet是一个以患者为中心的国家级临床研究网络,基于开发的PCORnet CDM进行大规模医疗保健数据的标准化与分析研究。本研究面向数据存储结构、术语映射模式和辅助工具研发三个方面,比较分析i2b2 CDM、OMOP CDM和PCORnet CDM的设计特点,并探究不同模型的优势与局限。见表 1,2。
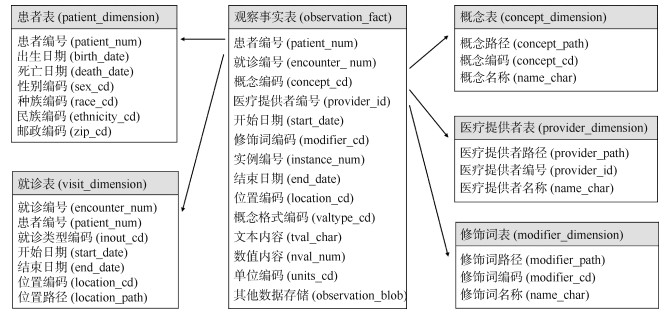
1. 数据存储结构: i2b2 CDM的核心结构由1个观察事实表和5个维度表(概念表、患者表、医疗提供者表、就诊表和修饰词表)组成。患者的所有观察结果(如诊断、用药、实验室检查等)都以实体-属性-值的结构存储于观察事实表中。维度表存储了进一步表征医疗事实的相关描述信息,如用药剂量、主要/次要诊断等信息可存储在modifier_dimension中,并通过modifier_cd字段与观察事实表中的记录关联。i2b2 CDM支持集成、共享、标准化和分析来自医疗保健和临床研究的多类型数据,涵盖EHR、用药、检查、临床文本、医学影像、基因组学、临床试验等。i2b2 CDM数据表的关系结构与核心字段见图 2。
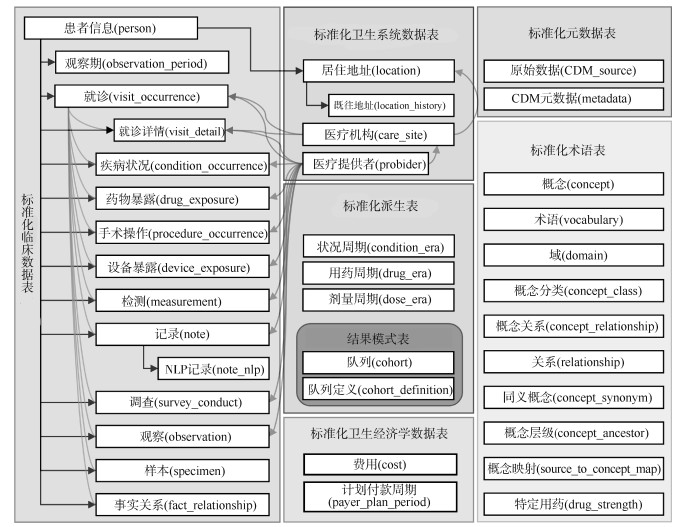
OMOP CDM采用“以患者为中心”的模型架构,最新的6.0版本包括10个术语表、2个元数据表、15个临床数据表、4个卫生系统数据表、2个卫生经济学数据表,3个派生表和2个结果模式表。OMOP CDM最初的创建目的是进行药物和医疗器械上市后的安全性监测,因此域表设计侧重于观察结果、药物暴露、医疗设备暴露、医疗保险索赔等领域,在后续的版本更新中又增加了临床文本记录、生物样本、队列以及患者用药和疾病状况的扩展表。在OMOP CDM中,绝大多数临床事件表都通过person_id字段与患者信息表关联,不同事件域表也通过唯一标识符[event]_id相关联,允许按“患者”纵向查看所有医疗事件。OMOP CDM数据表的主要关系结构见图 3。
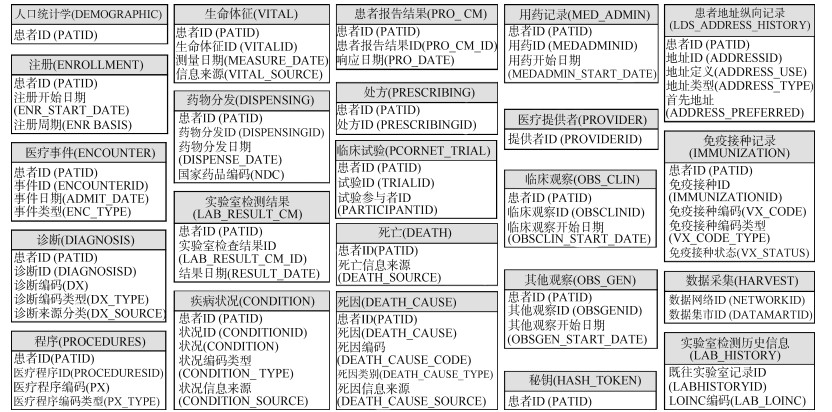
PCORnet CDM同样采用“以患者为中心”的模型架构,最新6.0版本的数据模型包含23个核心域表和3个补充信息表。研发PCORnet CDM的核心目的是创建覆盖全国范围医疗保健数据的标准化集合,从而提高国家进行大规模、多站点临床研究的能力,尤其是疗效比较研究。因此,PCORnet CDM在药物处方、实验室检验、免疫接种、临床试验、患者报告结局等领域有更大的适应性,但尚未对医疗设备、临床文本记录等领域进行设置。与OMOP CDM类似,PCORnet CDM将不同数据对应存储于不同的临床域表(如诊断、实验室检查等),大部分域表可通过PATID字段关联。PCORnet CDM的数据表与核心字段见图 4。
2. 术语映射模式: i2b2 CDM以概念编码的形式定义存储的医疗事实,编码的层次结构、描述性术语和其他相关信息共同构成了i2b2本体,即i2b2元数据。concept_dimension表用于存储术语概念,其中concept_path字段记录了概念层次结构。i2b2 CDM通过本体驱动的方法进行数据存储,支持预定义一个通用本体或多个领域本体,研究人员可通过修改本体实现数据的更新与访问查询,而无需修改数据库结构[17]。i2b2本体是实现数据质量控制和集成整合的主要机制,通常依赖构建衍生于LOINC、SNOMED CT、ICD等术语体系的标准本体实现与其他数据源的互操作。
OMOP CDM提供concept、concept_relationship、concept_ancestor等多个表单存储术语、概念信息。与i2b2 CDM相比,OMOP CDM的术语层更为复杂,支持多级层次结构、多种概念关系和概念同义词存储。区别于其他数据模型,OHDSI维护着一套独有的“标准化术语集”(https://athena.ohdsi.org/),并明确要求使用该术语集进行数据标准化转换和语义映射。OHDSI的术语专家负责将内部术语以及从第三方标准组织采集的术语概念、概念关系组织为规范格式,并重新划分领域。目前OHDSI已整合来自100余个术语体系的840多万个概念,其中药物、疾病状况、手术操作、观察、设备、检验等领域包含的概念最多。
PCORnet CDM并未提供术语层维护,主要基于定义的“PCORnet CDM实施规范”,直接将与术语词表映射后的数据组织成既定数据结构,通过强制映射模式实现数据标准化和互操作[18]。PCORnet CDM支持20余种国际主流术语体系,此外还通过定义值集来约束模型各字段属性。在数据ETL过程中,PCORnet CDM要求将映射后的标准术语类型、概念编码和原始数据信息填充到对应的模型字段,便于数据分析查询等操作。
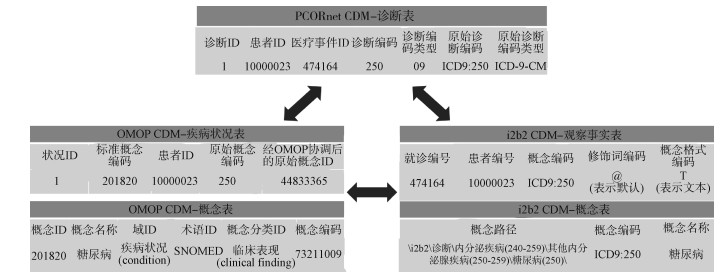
图 5简要展示了不同CDM关于“糖尿病”这一诊断结果的存储模式。
|
| 图 5 三种通用数据模型(CDM)的数据存储示例 |
3. CDM辅助工具研发: 在数据标准化处理方面,OHDSI开发了一组数据ETL处理的流程设计工具(Whiterabbit、Rabbit-In-A-Hat、Usagi)和标准术语查询工具Athena,这些工具在一定程度上简化了原始数据与目标模型在结构、语义层面的映射逻辑构建。针对数据查询分析利用,i2b2平台开发了一系列API接口、网络客户端(Webclient)和工作台客户端(Workbench)等,实现了基于web服务的模块管理和底层数据协调通信,支持可视化的本体(概念)浏览和拖拽形式的查询构建,协助研究人员全面了解数据内容结构、查找特定类型的样本数据、进行队列定义和数据统计分析等[19]。OHDSI开发了用于映射质量评估的可视化数据表征工具Achilles、队列定义和数据查询分析工具Atlas等开源软件,支持患者级的观察性数据分析、预测建模[20]。PCORnet研究网络的数据查询由协调中心统筹管理,中心通过基于PopMedNet开源平台的分布式数据网络查询门户向各网络合作伙伴发送患者数据查询请求,并接收返回的结果,实现跨机构的数据查询响应和联合分析研究[21]。
4. 优势与局限: 不同CDM的设计理念存在明显差异,主要体现在三个方面。
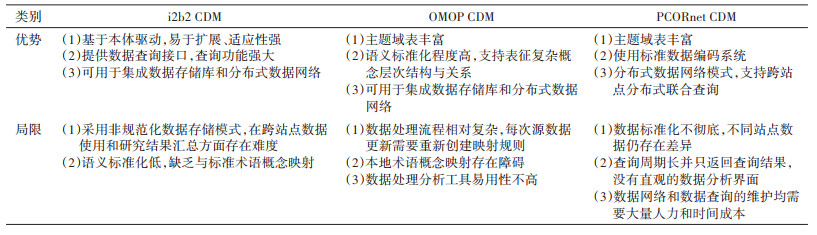
(1)在数据存储结构设计方面,i2b2 CDM未规定特定类型数据的存储格式,因此模型的扩展性和适应性更强。PCORnet CDM和OMOP CDM都设置了更为丰富的域表、细粒度的数据元素,在实现跨数据集的一致性方面表现更好。
(2)针对数据转换和语义映射模式,i2b2 CDM支持从各种源系统中直接摄取数据,且通过修改本体实现数据更新和查询访问,其相对灵活、非规范化的数据转换模式在跨不同数据源使用并以统一形式汇总研究结果方面存在难度。PCORnet CDM在数据ETL过程中尽可能保留了原始数据的语义信息,存在标准化不彻底的问题。OMOP CDM的术语覆盖度和语义标准化程度最高,但语义协调的过程也更复杂,每次数据更新都需要重新创建映射规则。此外,针对未使用国际通用术语标准的医疗保健数据,OMOP CDM存在一定的本地术语概念映射障碍[22-23]。
(3)多源数据的标准化处理是为了促进大规模、高质量数据的联合分析利用,i2b2平台开发了基于医学本体的数据查询工具,支持使用预定义的术语实现高效的跨库数据检索与访问。PCORnet研究网络同样支持数据的查询,但其底层数据采用分布式存储,数据处理和维护依托各站点完成,数据查询需基于协调中心分发,不支持研究人员进行直观的数据浏览访问。
三、医学CDM的发展应用CDM的标准化数据组织模式为跨区域、跨机构健康医疗大数据的横向汇聚和个人医疗数据的纵向整合提供了解决方案,极大地推动了数据资源的规范化收集、整合、共享和分析利用。国外针对医学CDM的研究已日趋成熟,在医疗数据协作网络构建、大规模潜在临床队列设计与识别、多中心观察性研究开展等方面进行了有效实践。美国的临床试验数据网络(Accrual to Clinical Trials Network)和可扩展的学习型健康医疗系统合作架构(Scalable Collaborative Infrastructure for a Learning Health System)均使用i2b2 CDM作为底层存储库设计模式,致力于为开展高效、安全的多中心临床试验和转化研究提供标准化数据[24-25]。COVID-19临床特征国际联盟(Consortium for Clinical Characterization of COVID-19 by EHR)也通过将数据映射到i2b2 CDM实现了跨国籍的患者数据标准化集成,为开展疾病流行病学和临床特征分析提供了有效信息来源[26]。OMOP CDM也已被许多跨国、跨机构研究项目采纳,建立了由研究人员和观察性健康医疗数据库组成的国际研究网络。Hripcsak等[6]基于OHDSI网络的2.5亿患者数据探究了糖尿病、抑郁症、高血压三种疾病的治疗途径特征,揭示了不同疾病治疗模式的地理差异。美国精准医学“All of Us”研究计划也在基于OMOP CDM进行人群队列中的患者EHR数据标准化[27]。PCORnet CDM目前主要应用于美国的临床研究网络和健康计划研究网络,已整合超过8 000万美国民众的EHR和健康计划数据,支持面向全国范围的患者数据分布式查询,在实用性临床试验、流行病学研究和罕见病研究等领域表现突出[21]。i2b2 CDM、OMOP CDM和PCORnet CDM的国际应用情况见表 3。
不同CDM由于其设计原理的独特性,为跨模型的数据转换与访问应用带来了新的挑战。国外学者在不同CDM的数据互操作领域也开展了丰富的研究,包括建立多模型间的数据互操作标准[32]或数据协调架构[33]、进行多模型间的数据转换实践[17, 34]、研发跨模型的数据查询调用算法、工具[35-36]等。CDM间互操作性的实现,促进了更大规模健康医疗数据的标准化集成整合、互联互通和关联分析,有助于数据利用率和数据潜在价值的提升。
国内关于CDM的研究仍处于起步阶段。近年来,已有研究人员关注到i2b2 CDM和PCORnet CDM在数据规范化收集、标准化处理、高效检索和开放共享等方面的作用,但缺少相关技术的实践探索。当前国内的研究应用多围绕OMOP CDM展开,研究人员致力于探索基于OMOP CDM的数据语义化建设和关联融合途径,提高健康医疗数据的整合利用率,涉及方法理论研究、临床队列标准和医学数据平台建设研究以及真实世界数据标准化转换研究等方面。北京大学公共卫生学院和北京大学健康医疗大数据国家研究院建立的中国队列共享平台正在开展基于OMOP CDM的队列数据标准模型研发,以促进多队列资源的协调整合服务[37],其研发团队还基于OMOP CDM和国内外专业领域标准制定了呼吸系统疾病专病队列标准框架[38]。此外,岳和欣等[39]基于不同数据模型对适用于我国临床队列的通用数据模块进行了归纳总结。也有研究人员基于OMOP CDM实现了大规模临床患者数据的结构化转换与标准语义映射,为实现跨区域、跨医院的临床数据互联互通和共享利用奠定了良好基础[40-41]。
四、机遇与挑战1. 深入开展FAIR数据语义化研究。数据标准化、语义化是健康医疗大数据开放共享、分析利用面临的主要难点。当前我国健康医疗数据资源的FAIR化建设仍停留在元数据、分类编码等字段属性的表层描述层面,数据语义表达的规范性严重不足,导致大量数据并不能被有效地发现、理解和使用。现阶段我国大力发展精准医学并着重建设大型自然人群、专病队列,但队列研究仍面临着纳入人群不够宽泛、长期随访监测困难,且不同队列间壁垒严重无法实现资源整合等挑战[42]。CDM支持数据元数据、实体内容(包括字段、值域)、术语概念等多层次的标准化处理,有助于增强数据语义理解的一致性、提高健康医疗数据的利用率。CDM不仅为不同队列研究的数据整合提供有效方法,并且推动了基于健康医疗数据资源的大型队列构建和随访的新模式。我国应深入开展基于CDM的健康医疗大数据FAIR化建设实践,打破数据标准化壁垒,推进跨区域、跨机构的数据资源互联互通和开放共享利用。
2. 实现多源术语标准的整合应用。FAIR原则指出,使用可被计算机识别的术语、词表、本体等通用编码语言描述数据,确保能够以相同方式表示不同数据资源的数据内容和关联规则是实现数据互操作的基础[2]。CDM为多种术语编码系统、本体词表资源的协调整合应用提供了标准框架,避免了单一词表映射不完全问题,在提升数据语义映射的完整度和一致性方面有着良好表现。国际相关机构十分重视医学术语体系建设,积累了UMLS、MeSH、SNOMED CT、LOINC等经典术语标准,让医学CDM的研究和应用更有代表性。近年来,我国也建设形成了“中文一体化医学语言系统”“中文医学主题词表”“临床检验项目分类与代码”等医学术语标准,但仍存在来源词汇少、覆盖范围局限、更新维护滞后等问题[43]。未来,我国还需持续加强中文医学术语标准的规划建设和国际医学术语标准的本地化实施,并进一步实现多源术语体系的集成应用。
3. 推动医学CDM的本地适配性实施。国外典型医学CDM的架构设计多围绕医疗卫生、临床实践领域中关注度高的方向进行结构化域表划分,在数据ETL标准化处理、跨库数据访问查询、多源数据整合分析等方面研发的辅助工具也多基于英文数据和英文术语标准,因此并不能完全适配国内的应用场景。另一方面,由于不同CDM在领域类型、数据格式、字段赋值、术语映射等方面存在明显的异质性,国外研究机构已着手构建CDM混合解决方案,不再局限于单一模型的使用,从而促进更大规模的健康医疗数据整合、满足更全面的临床实践和科研转化需求。我国在进行CDM的引入、扩展和实施时,可综合考量多种数据模型在数据存储结构、语义映射方式以及联合查询模式等方面的设计优势与局限,面向我国多样化的医学研究需求,构建具有高度适配性的数据模型和配套工具。
总体而言,国外已开展了广泛的医学CDM研究,在理论方法研究、术语标准建设、基础设施支撑,以及多模型互操作领域均取得了显著成果,实现了规模化、系统化的多源异构健康医疗数据语义互操作、标准化整合和关联融合分析,尤其支撑了大规模、低成本人群队列等观察性研究的开展以及公共卫生问题的高质量临床决策证据获取。融合多维信息的健康医疗大数据作为重要生产要素蕴藏了巨大价值,其开放共享、深度挖掘和广泛应用对推动数据驱动的医学科技创新至关重要。CDM的技术理念与实践模式为解决我国健康医疗数据资源建设质量不佳、语义化程度低、无法实现打通共享和重复利用等实际问题提供了宝贵的借鉴思路,值得深入探索。
利益冲突 所有作者声明无利益冲突
作者贡献声明 王安然:研究设计和实施、论文撰写;吴思竹:研究指导、论文修改、经费支持;刘盛宇、修晓蕾:研究设计和实施;周佳茵、胡拯涌、段一凡:论文修改
| [1] |
Wilkinson MD, Dumontier M, Aalbersberg IJ, et al. The FAIR guiding principles for scientific data management and stewardship[J]. Sci Data, 2016, 3: 160018. DOI:10.1038/sdata.2016.18 |
| [2] |
宋佳, 温亮明, 李洋. 科学数据共享FAIR原则: 背景、内容及实践[J]. 情报资料工作, 2021, 42(1): 57-68. Song J, Wen LM, Li Y. Scientific data sharing FAIR data principles: background, content and practice[J]. Inf Doc Serv, 2021, 42(1): 57-68. DOI:10.12154/j.qbzlgz.2021.01.007 |
| [3] |
祁子凡, 张凤旭, 张玲. 美国精准医学计划"All of Us"百万自然人群队列设计方案的经验和启示[J]. 中国循证医学杂志, 2021, 21(8): 980-985. Qi ZF, Zhang FX, Zhang L. Experience and enlightenment from the design of the "All of Us" research program in the US precision medicine program[J]. Chin J Evid-Based Med, 2021, 21(8): 980-985. DOI:10.7507/1672-2531.202104038 |
| [4] |
Wood A, Denholm R, Hollings S, et al. Linked electronic health records for research on a nationwide cohort of more than 54 million people in England: data resource[J]. BMJ, 2021, 373: n826. DOI:10.1136/bmj.n826 |
| [5] |
Himes BE, Dai Y, Kohane IS, et al. Prediction of chronic obstructive pulmonary disease (COPD) in asthma patients using electronic medical records[J]. J Am Med Inform Assoc, 2009, 16(3): 371-379. DOI:10.1197/jamia.M2846 |
| [6] |
Hripcsak G, Ryan PB, Duke JD, et al. Characterizing treatment pathways at scale using the OHDSI network[J]. Proc Natl Acad Sci USA, 2016, 113(27): 7329-7336. DOI:10.1073/pnas.1510502113 |
| [7] |
Marquis-Gravel G, Roe MT, Robertson HR, et al. Rationale and design of the aspirin dosing-a patient-centric trial assessing benefits and long-term effectiveness (ADAPTABLE) trial[J]. JAMA Cardiol, 2020, 5(5): 598-607. DOI:10.1001/jamacardio.2020.0116 |
| [8] |
Burn E, You SC, Sena AG, et al. Deep phenotyping of 34 128 adult patients hospitalised with COVID-19 in an international network study[J]. Nat Commun, 2020, 11(1): 5009. DOI:10.1038/s41467-020-18849-z |
| [9] |
Bourgeois FT, Gutiérrez-Sacristán A, Keller MS, et al. International analysis of electronic health records of children and youth hospitalized with COVID-19 infection in 6 countries[J]. JAMA Netw Open, 2021, 4(6): e2112596. DOI:10.1001/jamanetworkopen.2021.12596 |
| [10] |
Zhang HG, Dagliati A, Abad ZSH, et al. International electronic health record-derived post-acute sequelae profiles of COVID-19 patients[J]. NPJ Digit Med, 2022, 5(1): 81. DOI:10.1038/s41746-022-00623-8 |
| [11] |
i2b2 Community Wiki[EB/OL]. [2022-09-20]. https://community.i2b2.org/wiki/.
|
| [12] |
OMOP common data model[EB/OL]. [2022-09-20]. https://ohdsi.github.io/CommonDataModel/.
|
| [13] |
PCORnet common data model[EB/OL]. [2022-09-20]. https://pcornet.org/data/.
|
| [14] |
Weber GM, Klann J, Mendis M, et al. i2b2 common data model documentation[EB/OL]. (2021-01-31)[2022-09-20]. https://community.i2b2.org/wiki/display/BUN/2.+Quick+Start+Guide.
|
| [15] |
Blacketer C. Chapter 4 The common data model, The Book of OHDSI[EB/OL]. (2021-01-11)[2022-09-20]. https://ohdsi.github.io/TheBookOfOhdsi/CommonDataModel.
|
| [16] |
Common data model (CDM) specification, version 6.0[EB/OL]. (2020-10-22)[2022-09-20]. https://pcornet.org/wp-content/uploads/2022/01/PCORnet-Common-Data-Model-v60-2020_10_221.pdf.
|
| [17] |
Klann JG, Abend A, Raghavan VA, et al. Data interchange using i2b2[J]. J Am Med Inform Assoc, 2016, 23(5): 909-915. DOI:10.1093/jamia/ocv188 |
| [18] |
Rosenbloom ST, Carroll RJ, Warner JL, et al. Representing knowledge consistently across health systems[J]. Yearb Med Inform, 2017, 26(1): 139-147. DOI:10.15265/IY-2017-018 |
| [19] |
Wagholikar KB, Mendis M, Dessai P, et al. Automating installation of the integrating biology and the bedside (i2b2) platform[J]. Biomed Inform Insights, 2018, 10. DOI:10.1177/1178222618777749 |
| [20] |
Hripcsak G, Duke JD, Shah NH, et al. Observational health data sciences and informatics (OHDSI): opportunities for observational researchers[J]. Stud Health Technol Inform, 2015, 216: 574-578. DOI:10.3233/978-1-61499-564-7-574 |
| [21] |
Forrest CB, McTigue KM, Hernandez AF, et al. PCORnet® 2020: current state, accomplishments, and future directions[J]. J Clin Epidemiol, 2021, 129: 60-67. DOI:10.1016/j.jclinepi.2020.09.036 |
| [22] |
Yoon D, Ahn EK, Park MY, et al. Conversion and data quality assessment of electronic health record data at a Korean tertiary teaching hospital to a common data model for distributed network research[J]. Healthc Inform Res, 2016, 22(1): 54-58. DOI:10.4258/hir.2016.22.1.54 |
| [23] |
Lamer A, Depas N, Doutreligne M, et al. Transforming French electronic health records into the observational medical outcome Partnership's common data model: a feasibility study[J]. Appl Clin Inform, 2020, 11(1): 13-22. DOI:10.1055/s-0039-3402754 |
| [24] |
Visweswaran S, Becich MJ, D'Itri VS, et al. Accrual to clinical trials (ACT): a clinical and translational science award consortium network[J]. JAMIA Open, 2018, 1(2): 147-152. DOI:10.1093/jamiaopen/ooy033 |
| [25] |
Mandl KD, Kohane IS, McFadden D, et al. Scalable collaborative infrastructure for a learning healthcare system (SCILHS): architecture[J]. J Am Med Inform Assoc, 2014, 21(4): 615-620. DOI:10.1136/amiajnl-2014-002727 |
| [26] |
Brat GA, Weber GM, Gehlenborg N, et al. International electronic health record-derived COVID-19 clinical course profiles: the 4CE consortium[J]. NPJ Digit Med, 2020, 3: 109. DOI:10.1038/s41746-020-00308-0 |
| [27] |
Klann JG, Joss MAH, Embree K, et al. Data model harmonization for the all of us research program: transforming i2b2 data into the OMOP common data model[J]. PLoS One, 2019, 14(2): e0212463. DOI:10.1371/journal.pone.0212463 |
| [28] |
Herrett E, Gallagher AM, Bhaskaran K, et al. Data resource profile: clinical practice research datalink (CPRD)[J]. Int J Epidemiol, 2015, 44(3): 827-836. DOI:10.1093/ije/dyv098 |
| [29] |
You SC, Lee S, Cho SY, et al. Conversion of national health insurance service-national sample cohort (NHIS-NSC) database into observational medical outcomes partnership-common data model (OMOP-CDM)[J]. Stud Health Technol Inform, 2017, 245: 467-470. DOI:10.3233/978-1-61499-830-3-467 |
| [30] |
Canterberry M, Kaul AF, Goel S, et al. The patient-centered outcomes research network antibiotics and childhood growth study: implementing patient data linkage[J]. Popul Health Manag, 2020, 23(6): 438-444. DOI:10.1089/pop.2019.0089 |
| [31] |
Tison GH, Chamberlain AM, Pletcher MJ, et al. Identifying heart failure using EMR-based algorithms[J]. Int J Med Inform, 2018, 120: 1-7. DOI:10.1016/j.ijmedinf.2018.09.016 |
| [32] |
Belenkaya R, Mirhaji P, Khayter M, et al. Establishing interoperability standards between OMOP CDM v4, v5, and PCORnet CDM v1[C/OL]. [2022-09-20]. https://www.ohdsi.org/web/wiki/lib/exe/fetch.php?media=resources:ohdsi_poster_abstract_omop_to_pcornet.pdf.
|
| [33] |
U. S. Food & Drug Administration (FDA), National Institutes of Health (NIH), Office of the National Coordinator for Health Information Technology (ONC). Common data model harmonization (CDMH) and open standards for evidence generation: final report[EB/OL]. (2020-08-14)[2022-09-26]. https://aspe.hhs.gov/sites/default/files/private/pdf/259016/CDMH-Final-Report-14August2020.pdf.
|
| [34] |
Yu Y, Zong NS, Wen A, et al. Developing an ETL tool for converting the PCORnet CDM into the OMOP CDM to facilitate the COVID-19 data integration[J]. J Biomed Inform, 2022, 127: 104002. DOI:10.1016/j.jbi.2022.104002 |
| [35] |
Klann JG, Phillips LC, Herrick C, et al. Web services for data warehouses: OMOP and PCORnet on i2b2[J]. J Am Med Inform Assoc, 2018, 25(10): 1331-1338. DOI:10.1093/jamia/ocy093 |
| [36] |
Majeed RW, Fischer P, Günther A. Accessing OMOP common data model repositories with the i2b2 Webclient-algorithm for automatic query translation[J]. Stud Health Technol Inform, 2021, 278: 251-259. DOI:10.3233/SHTI210077 |
| [37] |
中国队列共享平台[EB/OL]. [2022-09-26]. http://chinacohort.bjmu.edu.cn.
|
| [38] |
孙一鑫, 裴正存, 詹思延. 呼吸系统疾病专病队列研究的标准制定与数据共享[J]. 中华流行病学杂志, 2018, 39(2): 233-239. Sun YX, Pei ZC, Zhan SY. Data harmonization and sharing in study cohorts of respiratory diseases[J]. Chin J Epidemiol, 2018, 39(2): 233-239. DOI:10.3760/cma.j.issn.0254-6450.2018.02.019 |
| [39] |
岳和欣, 湛永乐, 边峰, 等. 临床队列研究的数据标准与共享[J]. 中华流行病学杂志, 2021, 42(7): 1299-1305. Yue HX, Zhan YL, Bian F, et al. Data standard and data sharing in clinical cohort studies[J]. Chin J Epidemiol, 2021, 42(7): 1299-1305. DOI:10.3760/cma.j.cn112338-20200610-00831 |
| [40] |
张昕, 缪姝妹, 戴作雷, 等. 临床数据向通用数据模型转换研究及应用实践[J]. 中国数字医学, 2018, 13(10): 64-67. Zhang X, Miao SM, Dai ZL, et al. Research and application of conversion from clinical data to OMOP common data model[J]. China Dig Med, 2018, 13(10): 64-67. DOI:10.3969/j.issn.1673-7571.2018.10.022 |
| [41] |
洪娜, 刘飞, 张梦阳, 等. OHDSI通用数据模型在肿瘤大数据中的应用探索[J]. 中国数字医学, 2021, 16(11): 24-28. Hong N, Liu F, Zhang MY, et al. Exploration and practices on converting cancer big data to OHDSI common data model[J]. China Dig Med, 2021, 16(11): 24-28. DOI:10.3969/j.issn.1673-7571.2021.11.006 |
| [42] |
王笑峰, 金力. 大型人群队列研究[J]. 中国科学: 生命科学, 2016, 46(4): 406-412. Wang XF, Jin L. Large population-based cohort studies[J]. Sci Sin: Vit, 2016, 46(4): 406-412. DOI:10.1360/N052016-00104 |
| [43] |
谢雪娇, 张黎黎, 奈存剑, 等. 国外医学术语标准开发方法及对我国的启示[J]. 中华医学图书情报杂志, 2019, 28(11): 16-21. Xie XJ, Zhang LL, Nai CJ, et al. Development methods of foreign medical terminology standards and its enlightenments to our country[J]. Chin J Med Lib Inf Sci, 2019, 28(11): 16-21. DOI:10.3969/j.issn.1671-3982 |
 2023, Vol. 44
2023, Vol. 44