文章信息
- 钱璐, 梁胜楠, 崔方元, 程利红, 王江莘, 姜宁宁, 张香媛, 姜祥坤.
- Qian Lu, Liang Shengnan, Cui Fangyuan, Cheng Lihong, Wang Jiangshen, Jiang Ningning, Zhang Xiangyuan, Jiang Xiangkun
- 山东省聊城市2020-2021年小肠结肠炎耶尔森菌食品分离株分子特征分析
- Molecular characterization of foodborne Yersinia enterocolitica strains in Liaocheng City, Shandong Province, from 2020 to 2021
- 中华流行病学杂志, 2023, 44(2): 302-309
- Chinese Journal of Epidemiology, 2023, 44(2): 302-309
- http://dx.doi.org/10.3760/cma.j.cn112338-20220720-00645
-
文章历史
收稿日期: 2022-07-20
小肠结肠炎耶尔森菌(Yersinia enterocolitica,Y.e)是国内外广泛关注的食源性致病菌和人畜共患病原菌之一[1]。在冰箱及储存的生肉及其制品中常被发现[2-3],人类可通过食入污染的水源和食物被感染,引起腹泻等肠道疾病,严重的可引起反应性关节炎、败血病等[4]。目前对Y.e的研究多集中在传统方法主要包括生物分型、血清凝集和PCR毒力基因检测等[1-5],全基因组测序技术能够在基因水平上全面分析细菌的分子生物学特征,不仅能够通过多位点序列分型(MLST)、核心基因组多位点序列分型(cgMLST)和基于全基因组测序的单核苷酸多态性(wg-SNPs)分型等技术进行病原细菌分子分型,还能根据全基因组测序数据分析毒力基因和耐药基因,研究其致病性[6-7]。本研究于2020-2021年对山东省聊城市市售生肉及肉制品中Y.e污染状况进行监测,并对分离的Y.e进行全基因组测序,测序数据进行MLST、cgMLST和wg-SNPs分型以及毒力基因和耐药基因分析,同时进行药敏试验、PFGE和毒力基因PCR检测。旨在准确获得生肉与肉制品中Y.e的基因组特征,了解其遗传进化关系、耐药性和毒力性,为今后Y.e的感染防控和开展分子流行病学监测提供科学依据。
材料与方法1. 材料:
(1)样品来源:2020-2021年,从聊城市农贸市场及超市等监测点共采集165份生肉与肉制品,其中鸡肉30份、猪肉54份、牛肉46份、羊肉2份、培根制品30份。4 h内送至实验室开展检测工作。
(2)主要试剂与仪器:改良磷酸盐缓冲液(中国北京陆桥技术股份有限公司)、CIN-1培养基(中国北京陆桥技术股份有限公司)、改良Y培养基(中国北京陆桥技术股份有限公司)、限制性内切酶NotⅠ、XbaⅠ(日本TaKaRa公司)、小肠结肠炎耶尔森菌种属及毒力基因多重实时荧光PCR检测试剂盒(中国北京卓诚惠生生物科技股份有限公司)、基质液(法国生物梅里埃公司)、QIAamp DNA Mini Kit试剂盒(德国Qiagen公司)、96孔药敏板(美国赛默飞世尔科技公司)、建库试剂盒(中国深圳华大智造科技股份有限公司)、测序试剂盒(中国深圳华大智造科技股份有限公司)。
全自动微生物质谱分析仪(法国生物梅里埃公司)、全自动药敏试验菌液接种及判读系统(美国赛默飞世尔科技公司)、NanoDrop ONE超微量紫外-可见光分光光度计(美国赛默飞世尔科技公司)、Invitrogen Qubit 4荧光计(美国赛默飞世尔科技公司)、全基因组测序仪(中国深圳华大智造科技股份有限公司)、脉冲场凝胶电泳仪(美国BIO-RAD公司)。标准菌株:ATCC23715、ATCC25922、ATCC9812、ATCC8739。
2. 研究方法:
(1)菌株分离培养、鉴定及毒力基因PCR检测:以无菌操作取生肉及其制品25 g放入含有225 ml改良磷酸盐缓冲液增菌液的无菌均质袋中,以均质器均质1 min后于26 ℃增菌48 h。将增菌液0.5 ml与碱处理液4.5 ml充分混合15 s后分别接种至CIN-1培养基和改良Y培养基,26 ℃培养48 h,典型菌落在CIN-1培养基上为中心深红色,周围具有无色透明圈,在改良Y培养基上为无色透明、不黏稠的菌落。挑取可疑菌落接种至改良克氏双糖培养基,将斜面和底部皆变黄且不产气的培养物进一步接种至尿素酶,26 ℃培养24h,尿素酶反应阳性者再进一步接种至半固体培养基分别置于26 ℃和36 ℃培养24h,将26 ℃有动力而36 ℃无动力的可疑菌落挑至BHI培养基[8]进行培养,然后采用Vitek 2 compact 30进行生化鉴定,并结合质谱进行鉴定[9]。采用荧光定量PCR方法,对Y.e的ail、ystA、ystB、yadA、virF基因进行检测,参照试剂盒及仪器使用说明书要求进行操作[2]。
(2)药敏试验:鉴定为Y.e的菌落,药敏试验用微量肉汤稀释法进行,使用国家致病菌识别网肠道菌群专用革兰阴性需氧菌药敏检测板CHNENF,检测按照说明书完成,并做ATCC25922的药敏质控[4, 10-11]。试验结果用全自动药敏判读系统判读,分析其最低抑菌浓度值,根据2021年美国临床实验室标准化委员会的相应标准判读[12],分为敏感(S)、中介(I)和耐药(R)。共检测14种抗生素,分别为氯霉素(CH,4~32 μg/ml)、复方新诺明(SXT,0.5/9.5~8/152 μg/ml)、多粘菌素E(CT,0.25~8 μg/ml)、厄他培南(ETP,0.25~8 μg/ml)、美罗培南(MEM,0.12~2 μg/ml)、头孢噻肟(CTX,0.25~16 μg/ml)、头孢他啶(CAZ,0.25~16 μg/ml)、头孢他啶/阿维巴坦(CZA,0.25/4~8/4 μg/ml)、四环素(TET,1~16 μg/ml)、替加环素(TIG,0.25~8 μg/ml)、环丙沙星(CIP,0.015~0.12 μg/ml)、阿米卡星(AMI,4~64 μg/ml)、氨苄西林(AMP,2~32 μg/ml)、氨苄西林/舒巴坦(AMS,2/1~32/16 μg/ml)。
(3)PFGE分型:鉴定为Y.e的菌落,实验参照《国家致病菌识别网技术规范-小肠结肠炎耶尔森菌》进行。主要实验参数:细菌悬液浊度4.0~4.5;限制性内切酶NotⅠ(10 U/μl)37 ℃酶切2 h;电泳参数2~20 s,18.5 h;利用H9812作为分子量标准。指纹图谱上传至BioNumerics软件进行分析,构建聚类树状图,位置优化度和容许度为1.5%,带型相似度85.0%以上判定为一个带型簇[8]。
(4)全基因组测序:将鉴定为Y.e的菌落纯培养,用QIAamp DNA Mini Kit提取菌株基因组DNA,用超微量分光光度计测定基因组DNA纯度,用Invitrogen Qubit 4测定基因组DNA浓度,基因组DNA浓度需 > 4.59 ng/μl。使用华大基因二代测序仪完成全基因组测序,建库试剂盒与测序试剂盒均使用华大基因生产试剂盒。
(5)数据分析:测序数据利用微生物基因组注释系统进行全功能分析,包括基因组组装、耐药基因预测、毒力基因预测,同时使用k SNP 3.0软件对20株Y.e食品分离株的全基因组与美国生物技术信息中心上获得的不同年份、地区和来源的14株具有代表性菌株基因组(表 1)构建wg-SNPs进化树。MLST参考Y.e的7个管家基因(adk、argA、aroA、glnA、thrA、tmk、trpE),利用MLST 2.0(
1. Y.e来源标本的分布:从聊城市农贸市场及超市共监测165份肉与肉制品,其中鸡肉30份、猪肉54份、牛肉46份、羊肉2份、培根制品30份。经分离、培养和鉴定,共检出Y.e菌株21株,检出率为12.73%。其中在鸡肉中检出7份,检出率为4.24%;在牛肉中检出6份,检出率为3.64%;在猪肉中检出8份,检出率为4.85%。
2. 基因组组装报告:使用微生物基因组注释系统对质控合格的测序数据进行基因组组装,并利用测序数据对组装长度进行延伸,其中菌株Y.e 010两次测序失败,因此共得到20株Y.e基因组组装报告。20个Y.e基因组的支架(scaffold)N50均 > 20 kb,重叠群(contigs)N50均 > 20 kb。支架M(Q1,Q3)为34(22,106)个,重叠群M(Q1,Q3)为40(26,115)个,支架N50 M(Q1,Q3)为407 226(102 676,690 784)bp,重叠群N50 M(Q1,Q3)为382 714(85 816,576 513)bp,GC含量M(Q1,Q3)为47.22%(46.94%,47.52%)。20个菌株基因组分别最佳匹配到Y.e GCA-001304755、Y.e GCA-000597945和Y.e GCA-001219385等不同的最近缘序列。聊城市21株食源性Y.e的分离来源、分离时间及其基因组组装报告见表 2。
3. Y.e ST分型:MLST分型结果显示,本研究中20株Y.e可分为16个ST型。其中2株为ST360型,来自鸡肉;2株为ST335型,来自鸡肉和牛肉;2株为ST503型,来自猪肉;2株ST496型,来自牛肉和猪肉;其余ST型见表 2,无主要型别。
4. Y.e毒力基因谱分析:全基因组测序显示,21株Y.e中毒力基因检测结果见表 3。侵袭性相关基因inv携带率为100%(20/20);外毒素合成相关基因yplA、YaxAB携带率分别为20%(4/20)、80%(16/20);ystB基因携带率为90%(18/20);黏附性相关基因Myf、yapC携带率为65%(13/20)、20%(4/20);与分泌系统相关基因yst1携带率为35%(7/20)。荧光定量PCR结果显示21株Y.e呈现2种毒力基因特征,均为非致病性菌株型,11株Y.e毒力基因特征为ail-、ystA-、ystB-、yadA-、virF-;10株Y.e毒力基因特征为ail-、ystA-、ystB+、yadA-、virF-。
5. Y.e药敏试验结果与耐药基因检测:本研究对21株Y.e进行了药敏试验。见表 4。有10株菌对AMP耐药,耐药率为47.62%,有7株菌对CIP耐药,耐药率为33.33%,有2株菌对TET耐药,耐药率为9.52%,菌株Y.e03、Y.e07、Y.e09、Y.e015出现多重耐药现象。耐药基因检测结果显示17株菌携带blaA和vat(F)耐药基因;20株菌均携带y56 beta-lactamase耐药基因;10株菌携带氟喹诺酮类耐药基因qnrD1;2株菌携带氟喹诺酮类耐药基因qnrS1;2株菌携带四环素类耐药基因tet(A);1株菌携带二氨基嘧啶类耐药基因dfrA14。
6. PFGE分型和cgMLST分型:经PFGE分子分型后21株Y.e带型相似度为68.0%~100.0%,按相似度85.0%为标准分为14个带型簇[8],较为分散,没有优势带型簇,各带型簇涵盖的菌株数量在1~3之间。见图 1。相似度96.2%的Y.e012和Y.e011耐药基因携带情况一致,但是耐药性表现不一致,毒力基因携带情况不一致,ST分型不一致;相似度96.8%的Y.e004和Y.e014耐药基因携带情况一致,耐药性表现一致,ST分型一致,但是毒力基因携带情况不一致。相似度100.0%的Y.e018和Y.e020耐药基因和毒力基因携带情况均不一致,ST分型一致。ST分型一致的两株菌(ST503和ST335)PFGE指纹图谱相似度差异较大。

|
| 图 1 基于21株小肠结肠炎耶尔森菌(Y.e)PFGE指纹图谱构建的树状图 |
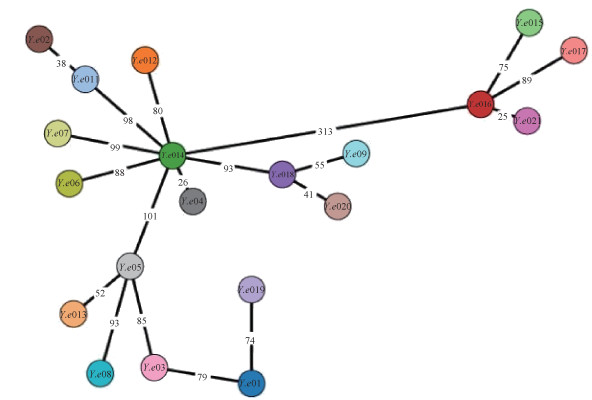
cgMLST聚类分析显示20株Y.e之间存在等位基因差异,数量在25~313个之间,可分为3个主进化分支。2株ST360型(Y.e04和Y.e014)菌株之间等位基因差异数量为26个,2株ST335型(Y.e02和Y.e011)菌株之间等位基因差异数量为38个,2株ST503型(Y.e016和Y.e021)菌株有25个等位基因差异,2株ST496型(Y.e018和Y.e020)菌株之间等位基因差异数量为41个。PFGE相似度96.2%的Y.e012和Y.e011两株菌位于同一主分支的2个的亚分支上,与PFGE聚类结果有差异。见图 2。
|
| 图 2 基于20株小肠结肠炎耶尔森菌(Y.e)核心基因组多位点序列分型最小生成树 |
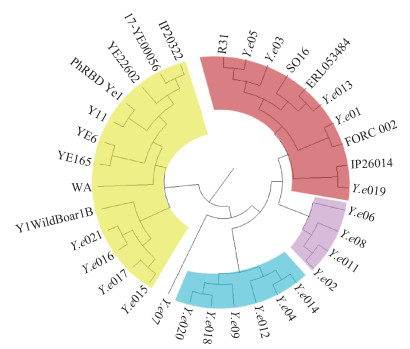
7. 基于wg-SNPs的遗传多样性分析:Y.e wg-SNPs系统进化树结果见图 3,34株菌株可分为2个主进化分支,2020-2021年聊城市分离的20株Y.e食品分离株则分别位于这两个主进化分支上,其中Y.e015、Y.e016、Y.e017和Y.e021位于主分支1,Y.e07位于主分支2的亚分支2a,Y.e04、Y.e09、Y.e012、Y.e014、Y.e018和Y.e020位于主分支2的亚分支2b,Y.e01、Y.e02、Y.e03、Y.e05、Y.e06、Y.e08、Y.e011、Y.e013和Y.e019位于主分支2的亚分支2c。
|
| 注:黄色部分为分支1;蓝色部分为分支2a;紫色部分为分支2b;红色部分为分支2c 图 3 34株小肠结肠炎耶尔森菌(Y.e)基于全基因组测序的单核苷酸多态性系统进化树 |
目前,Y.e病在我国主要以低水平散发为主。由于其在低温环境下易于生长,所以是“冰箱病”的主要致病菌之一[15]。本研究通过对2020-2021年聊城市市售生肉及其制品中Y.e进行监测,结果显示该致病菌在生肉与肉制品中分离率较高,达12.73%,高于傅祎欣等[2]报道的Y.e检出率,低于王丽丽等[8]的报道。提示居民肉制品应煮熟后食用,并避免交叉污染。
Y.e的致病性主要与毒力基因和耐药基因等特征有关,近年来,随着全基因组测序技术的发展和生物信息学分析工具的成熟,大大提升了细菌全基因组研究的水平,但Y.e全基因组研究在国内外仍是短板。目前,国内外学者常通过5个毒力基因(ail、ystA、ystB、yadA和virF)PCR实验来确定Y.e有无致病性[5]。致病性菌株通常携带ail、ystA、yadA和virF基因,非致病性菌株不携带上述基因[16]。本研究PCR结果显示,21株Y.e分离株均为非致病型菌株,这一结果与其他地区报道一致[2, 5, 8]。本研究同时通过全基因组测序技术监测Y.e其他毒力基因,如YaxAB、Myf等,结果显示,YaxAB和Myf基因在Y.e中携带率较高。Wagner等[17]证实了YaxAB编码的蛋白质有助于增强人类和小鼠细胞的细胞毒活性,致使YaxAB在Y.e的致病性中起作用;Bräuning等[18]认为YaxAB在哺乳动物Y.e感染中的确切作用仍然难以捉摸,但值得注意的是,YaxAB被发现对免疫细胞具细胞毒性,并影响小鼠感染模型中脾脏炎症的病变;Pakharukova等[19]的研究显示,在进化过程中Myf基因专门作为碳水化合物结合黏附素,促进Y.e到肠细胞,引起致病性。因此,该类菌仍存在一定潜在危害性,有必要对该类菌进行持续性监测。笔者认为进行5种毒力基因PCR检测的同时,通过全基因组测序技术增加Yax和Myf等毒力基因检测,能在一定程度上更加准确地判定Y.e的潜在致病力。
Y.e对AMP和一代头孢类抗生素天然耐药,测序结果显示可能与blaA和y56 beta-lactamase耐药基因携带有关。21株Y.e耐药试验报告显示,Y.e对氟喹诺酮类(CIP)耐药率较高,与国内研究情况略有不同[2, 8-20],也有研究显示,Y.e分离株对氟喹诺酮类抗生素敏感[2, 8, 19],本研究出现耐氟喹诺酮类(CIP)Y.e 6株,耐药基因检测结果显示可能与qnrD1和qnrS1等耐药基因的获得有关,为质粒介导的氟喹诺酮类耐药菌株,qnr基因编码的蛋白对DNA解旋酶产生保护作用,从而起到对氟喹诺酮类药物的抑制作用[21]。菌株Y.e016携带耐药基因qnrD1,但药敏结果显示对氟喹诺酮类(CIP)敏感,可能是氟喹诺酮类耐药决定区存在耐药基因表达突变的情况。本研究出现2株Y.e分离株对四环素耐药,可能与tet(A)耐药基因的获得有关。部分菌株携带blaA和y56 beta-lactamase耐药基因,但药敏结果显示对AMP敏感,可能是耐药基因未表达的原因。本研究出现对氟喹诺酮类、四环素类的耐药株,质粒的传播可以让耐药性在细菌间水平传播,增加不同种细菌的耐药性,提示临床上应谨慎使用氟喹诺酮类、四环素类抗生素。
现代分子分型方法以PFGE和MLST较常见,但都只利用了细菌有限的遗传信息,没有充分利用细菌整体的遗传信息去分析[7],本研究进一步在全基因组的水平基于序列多态性进行分型。cgMLST是以基因为单元进行比对和分型,其分型结果相较于MLST和PFGE,更具有可靠性[22],本研究结果也符合这一观点。如ST335的两株Y.e(菌株ID:Y.e02和Y.e011),cgMLST结果显示菌株之间等位基因差异数量为38个,采用cgMLST大大提高了常规MLST的分辨力。wg-SNPs系统进化树分析显示,主分支1中,4株中国山东省聊城市食品分离株(Y.e015、Y.e016、Y.e017和Y.e021)与意大利分离株Y1 WildBoar1B较近,而Y1 WildBoar1B则是从野生有蹄类动物尸体中分离出来的,并确定为高毒力株(生物型1B)[23];亚分支2c中,中国山东省聊城市食品分离株Y.e013与新西兰动物尸体分离株ERL053484、芬兰临床分离株SO16较近,以上分析提示Y.e食品分离株在聊城市有引起感染的风险,值得防控部门关注。
综上所述,相关部门应加强生肉与肉制品中Y.e的分子生物学监测,开展基因组测序及分子分型检测工作,不断完善食源性致病菌大数据,对食品安全风险评估和疾病暴发预警具有重要意义。
利益冲突 所有作者声明无利益冲突
作者贡献声明 钱璐、梁胜楠、崔方元、程利红:实验操作、论文撰写;王江莘、姜宁宁、张香媛:数据整理、统计学分析;姜祥坤:研究指导、论文修改、经费支持
| [1] |
Filik K, Szermer-Olearnik B, Wernecki M, et al. The podovirus ϕ80-18 targets the pathogenic American biotype 1B strains of Yersinia enterocolitica[J]. Front Microbiol, 2020, 11: 1356. DOI:10.3389/fmicb.2020.01356 |
| [2] |
傅祎欣, 洪锦春, 叶海梅, 等. 2019-2020年福建省生肉中小肠结肠炎耶尔森菌污染状况及耐药分析[J]. 中国预防医学杂志, 2021, 22(3): 193-196. Fu YX, Hong JC, Ye HM, et al. Prevalence and antibiotic resistance profile of Yersinia enterocolitica in row meat in Fujian province from 2019 to 2020[J]. Chin Prev Med, 2021, 22(3): 193-196. DOI:10.16506/j.1009-6639.2021.03.007 |
| [3] |
Klinzing DC, Matias RR, Skowronski E, et al. Shotgun genome sequence of a Yersinia enterocolitica isolate from the Philippines[J]. J Bacteriol, 2012, 194(2): 542-543. DOI:10.1128/JB.06451-11 |
| [4] |
罗嘉远, 陈洪友, 陈敏, 等. 食品中2株非常见耶尔森菌分离鉴定及耐药性分析[J]. 中国食品卫生杂志, 2020, 32(1): 20-24. Luo JY, Chen HY, Chen M, et al. Isolation and drug resistance of 2 stains of Yersinia from food[J]. Chin J Food Hyg, 2020, 32(1): 20-24. DOI:10.13590/j.cjfh.2020.01.004 |
| [5] |
张彦春, 杨波, 王苗, 等. 北京市顺义区零售生猪肉中小肠结肠炎耶尔森菌的检测与病原特征分析[J]. 中国人兽共患病学报, 2021, 37(10): 945-949. Zhang YC, Yang B, Wang M, et al. Distribution and characteristics of Yersinia enterocolitica isolated from retail raw pork meat in Shunyi, Beijing[J]. Chin J Zoonoses, 2021, 37(10): 945-949. DOI:10.3969/j.issn.1002-2694.2021.00.123 |
| [6] |
段然. 我国5岁以下儿童小肠结肠炎耶尔森菌病流行特征与致病性耶尔森菌比较基因组学初步研究[D]. 北京: 中国疾病预防控制中心, 2017: 1-77. Duan R. Epidemic characteristics and comparative genomics of pathogenic Yersinia enterocolitica in children under 5 years old in China[D]. Beijing: Chinese Center for Disease Control and Prevention, 2017: 1-77. |
| [7] |
周海健, 阚飙. 细菌基因组分型方法的应用研究进展[J]. 疾病监测, 2016, 31(8): 668-675. Zhou HJ, Kan B. Progress in application research of bacterium genome-based subtyping[J]. Dis Surveill, 2016, 31(8): 668-675. DOI:10.3784/j.issn.1003-9961.2016.08.013 |
| [8] |
王丽丽, 张寻, 段然, 等. 北京市市售生禽肉及社区居民冰箱耶尔森菌污染状况及分子特征研究[J]. 中国人兽共患病学报, 2020, 36(9): 752-757. Wang LL, Zhang X, Duan R, et al. Contamination status and molecular characteristics of Yersinia bacteria in raw meat and community refrigerators in Beijing[J]. Chin J Zoonoses, 2020, 36(9): 752-757. DOI:10.3969/j.issn.1002-2694.2020.00.108 |
| [9] |
张彦春, 张爽, 杨杰, 等. 自腹泻病例和家用冰箱分离的小肠结肠炎耶尔森菌病原特征分析[J]. 中国食品卫生杂志, 2018, 30(6): 582-585. Zhang YC, Zhang S, Yang J, et al. Analysis of pathogenic characteristics of Yersinia enterocolitica isolated from household refrigerators and diarrhea patients[J]. Chin J Food Hyg, 2018, 30(6): 582-585. DOI:10.13590/j.cjfh.2018.06.006 |
| [10] |
Råsbäck T, Rosendal T, Stampe M, et al. Prevalence of human pathogenic Yersinia enterocolitica in Swedish pig farms[J]. Acta Vet Scand, 2018, 60(1): 39. DOI:10.1186/s13028-018-0393-5 |
| [11] |
Liang JR, Kou ZQ, Qin S, et al. Novel Yersinia enterocolitica prophages and a comparative analysis of genomic diversity[J]. Front Microbiol, 2019, 10: 1184. DOI:10.3389/fmicb.2019.01184 |
| [12] |
Clinical and Laboratory Standards Institute (CLSI). CLSI M100 Performance standards for antimicrobial susceptibility testing[S]. Clinical and Laboratory Standards Institute, 2021.
|
| [13] |
Peng ZX, Zou MY, Li MH, et al. Prevalence, antimicrobial resistance and phylogenetic characterization of Yersinia enterocolitica in retail poultry meat and swine feces in parts of China[J]. Food Control, 2018, 93: 121-128. DOI:10.1016/j.foodcont.2018.05.048 |
| [14] |
Larsen MV, Cosentino S, Rasmussen S, et al. Multilocus sequence typing of total-genome-sequenced bacteria[J]. J Clin Microbiol, 2012, 50(4): 1355-1361. DOI:10.1128/JCM.06094-11 |
| [15] |
Chlebicz A, Śliżewska K. Campylobacteriosis, Salmonellosis, Yersiniosis, and Listeriosis as zoonotic foodborne diseases: a review[J]. Int J Environ Res Public Health, 2018, 15(5): 863. DOI:10.3390/ijerph15050863 |
| [16] |
Thoerner P, Bin Kingombe CI, Bögli-Stuber K, et al. PCR detection of virulence genes in Yersinia enterocolitica and Yersinia pseudotuberculosis and investigation of virulence gene distribution[J]. Appl Environ Microbiol, 2003, 69(3): 1810-1816. DOI:10.1128/AEM.69.3.1810-1816.2003 |
| [17] |
Wagner NJ, Lin CP, Borst LB, et al. YaxAB, a Yersinia enterocolitica pore-forming toxin regulated by RovA[J]. Infect Immun, 2013, 81(11): 4208-4219. DOI:10.1128/IAI.00781-13 |
| [18] |
Bräuning B, Bertosin E, Praetorius F, et al. Structure and mechanism of the two-component α-helical pore-forming toxin YaxAB[J]. Nat Commun, 2018, 9(1): 1806. DOI:10.1038/s41467-018-04139-2 |
| [19] |
Pakharukova N, Roy S, Tuittila M, et al. Structural basis for Myf and Psa fimbriae-mediated tropism of pathogenic strains of Yersinia for host tissues[J]. Mol Microbiol, 2016, 102(4): 593-610. DOI:10.1111/mmi.13481 |
| [20] |
王闻卿, 崔琪奇, 王筱, 等. 上海市浦东新区食源性小肠结肠炎耶尔森菌耐药及分子流行病学特征[J]. 中华流行病学杂志, 2019, 40(3): 354-359. Wang WQ, Cui QQ, Wang X, et al. Antimicrobial resistance and molecular epidemiology of foodborne Yersinia enterocolitica in Pudong New District, Shanghai[J]. Chin J Epidemiol, 2019, 40(3): 354-359. DOI:10.3760/cma.j.issn.0254-6450.2019.03.019 |
| [21] |
刘雪松, 张艳, 薛沾枚, 等. 一株喹诺酮耐药大肠杆菌耐药基因检测[J]. 现代畜牧科技, 2022(1): 35-37. Liu XS, Zhang Y, Xue ZM, et al. Detection of drug resistance gene in a quinolone resistant Escherichia col[J]. Mod Anim Husbandry Sci Technol, 2022(1): 35-37. DOI:10.19369/j.cnki.2095-9737.2022.01.013 |
| [22] |
畅晓晖. 基于全基因组测序的致病菌分型溯源技术研究进展[J]. 中国口岸科学技术, 2021, 3(增刊1): 41-47. Chang XH. Research on pathogenic traceability of pathogenic bacteria based on whole-genome sequencing[J]. China Port Sci Technol, 2021, 3(Suppl 1): 41-47. |
| [23] |
Macori G, Romano A, Adriano D, et al. Draft genome sequences of four Yersinia enterocolitica strains, isolated from wild ungulate carcasses[J]. Genome Announc, 2017, 5(15): e00192-17. DOI:10.1128/genomeA.00192-17 |
 2023, Vol. 44
2023, Vol. 44






