文章信息
- 刘佳, 耿杰, 刘家琪, 薛秀娟, 闫江舟, 袁源, 张向兵, 刘春华, 张国龙.
- Liu Jia, Geng Jie, Liu Jiaqi, Xue Xiujuan, Yan Jiangzhou, Yuan Yuan, Zhang Xiangbing, Liu Chunhua, Zhang Guolong
- 河南省部分地区HIV/AIDS肠道微生物群落结构的相关因素分析
- Analysis of factors associated with the structure of the gut microbial community in HIV/AIDS patients in some areas of Henan province
- 中华流行病学杂志, 2022, 43(4): 566-571
- Chinese Journal of Epidemiology, 2022, 43(4): 566-571
- http://dx.doi.org/10.3760/cma.j.cn112338-20211025-00816
-
文章历史
收稿日期: 2021-10-25
了解肠道微生物和宿主的相互作用可能为疾病的发病机制提供新的见解[1-2]。HIV的感染使宿主的肠道微生物易位和失调,从而影响宿主的免疫平衡和恢复,尤其是持续的免疫激活和炎症会加快疾病的进展[3-5]。近年有研究发现,HIV感染引起的宿主肠道微生物的改变由于年龄、性别、性取向、感染时间、抗病毒治疗、CD4+T淋巴细胞(CD4)计数和病毒载量等因素变得复杂[6-9],同时由于受试者特征、队列规模、地理区域、采样方法以及测序深度等的差异也有可能导致结果之间的不一致。因此,对于HIV感染者/AIDS病例(HIV/AIDS)肠道微生物变化的相关因素尚存在很多争议。肠道微生物可能受到生活环境和饮食等因素的影响,因而不同国家/地区人群之间的差别是显著的[10-12]。本研究分析河南省部分地区HIV/AIDS肠道微生物群落结构的相关因素,为提高HIV/AIDS生存质量相关干预研究提供参考。
对象与方法1. 研究对象:根据我国艾滋病综合防治信息系统查询河南省HIV/AIDS信息(数据截至2019年1月1日)。纳入标准:①年龄范围18~60岁的HIV/AIDS;②BMI为18.5~30.0 kg/m2;③最近1个月内未使用抗生素或者益生菌;④无消化道症状;⑤无机会性感染。对于HIV/AIDS抗病毒治疗≥1年者,则要求最近1次CD4 > 500个/µl,病毒载量低于检测限。对于抗病毒治疗 < 1年者则不限制CD4和病毒载量。研究对象均签署知情同意书。本研究通过河南省CDC伦理委员会审批(批准文号:2019-KY- 005-02)。
2. 研究现场:2019年3月1日至5月1日在河南省许昌市、新乡市、永城市和确山县采用方便抽样方法预计选取HIV/AIDS 150例,其中,接受抗病毒治疗100例,未接受抗病毒治疗50例。通过现场调查的方式核实研究对象的社会人口学特征、感染途径、确诊时间和起始治疗时间等信息。
3. 样本采集和检测:使用EDTA-K2抗凝真空采血管采集8~10 ml全血标本,立即混匀,分出部分全血用于CD4检测,剩余全血离心(2 000 g×10 min)后,分离出血浆,用于HIV病毒载量、HCV抗体和HBsAg的检测。由患者自己使用灭菌拭子采集粪便样本(样本体积为黄豆大小),放入采样盒-70 ℃保存。所有样本在检测前均-70 ℃保存。
使用FACSCalibur/FACSCount流式细胞仪和配套试剂(BD,美国)进行CD4绝对计数。使用的COBASAmpliPrep/COBAS TaqMan HIV-1 test(Taqman)(美国罗氏制药有限公司)和HIV-1核酸定量检测试剂盒(中国东北制药集团)进行HIV病毒载量检测,最低检测限分别为20拷贝/ml和28拷贝/ml。使用胶体金法(中国广州万孚生物技术有限公司)检测HCV抗体和HBsAg。
4. 粪便的核酸提取和16S RNA基因测序:使用E.Z.N.ATM Mag-Bind Soil DNA Kit试剂盒(OMEGA,中国广州)提取粪便的基因组DNA,完全按照试剂盒操作。使用Qubit® 2.0荧光检测仪(life,美国)对DNA的质量和浓度进行检测。粪便样本微生物16S V3~V4高变区域采用通用引物16S-341F:CCTAYGGGRBGCASCAG和16S-806R:GGACTA CNNGGGTATCTAAT扩增,扩增产物经纯化后,采用Qubit® 2.0荧光检测仪对纯化后的扩增产物DNA进行精确定量。根据美国Illumina公司提供的标准建库流程对定量后的扩增产物构建双端(PE,paired-end)测序文库,最后采用NovaSeq 6000高通量测序系统(美国Illumina公司)进行2×250 bp双端(PE)测序。
5. 测序结果的处理和统计分析方法:高通量测序下机数据经过质量评估和过滤后利用QIIME2 v2019.7软件进行分析。使用fastq-join方法进行序列拼接,使用deblur方法进行去冗余并生成扩增序列变体(amplicon sequence variant,ASV),并基于greengenes参考数据库对每个样品的ASV进行分类学注释以及门、纲、目、科和属水平的统计。
为确定群落结构与多个因素(人口学信息/临床状态)的相关性,选取丰度最高的30个属作为肠道微生物群落结构分析的衡量指标。使用R v4.0.3软件中vegan包进行冗余分析(redundancy analysis,RDA),同时使用EnvFit函数(permu = 999)确定每个协变量的效应大小(R2)和P值,R2表示相关因素与物种分布的决定系数,该值越小则该因素对物种分布的影响越小;P表示相关因素与物种分布相关性的显著性检验值。以P < 0.05为差异有统计学意义。
结果1. 基本情况:实际调查164例HIV/AIDS,其中采集粪便样本152例,剔除不合格及信息缺失的样本30例,最终纳入122例。BMI为(23.62±2.78)kg/m2,年龄(47±13)岁,男性占66.39%(81/122),已婚占59.84%(73/122),小学和初中文化程度分别占31.15%(38/122)和40.16%(49/122)。异性性传播占51.64%(63/122)。CD4 < 250个/µl的占15.57%(19/122),病毒载量低于检测限占55.74%(68/122)。HCV抗体阳性占17.21%(21/122),HBsAg阳性占2.46%(3/122),未接受抗病毒治疗者占29.51%(36/122)。见表 1。
2. 肠道微生物群落结构特征分析:122例样本中,聚类分析共得到13 367个ASV,注释到30个门水平物种和336个属水平物种。前5位优势菌门是Firmicutes、Bacteroidetes、Proteobacteria、Fusobacteria和Actinobacteria,前5位优势菌属及其平均相对丰度占比是Prevotella(17.42%)、Roseburia(7.96%)、Megamonas(7.87%)、Bacteroides(7.71%)和Faecalibacterium(7.57%)。在86例接受抗病毒治疗的病例样本中,聚类分析共得到10 679个ASV,注释到29个门水平物种和312个属水平物种。前5位优势菌门是Firmicutes、Bacteroidetes、Proteobacteria、Fusobacteria和Actinobacteria,前5位优势菌属及其平均相对丰度占比是Prevotella(17.05%)、Megamonas(9.19%)、Bacteroides(8.48%)、Roseburia(7.21%)和Faecalibacterium(5.57%)。在36例未接受抗病毒治疗的病例样本中,聚类分析共得到4 242个ASV,注释到20个门水平物种和212个属水平物种。前5位优势菌门是Firmicutes、Bacteroidetes、Proteobacteria、Fusobacteria和Actinobacteria,前5位优势菌属及其平均相对丰度占比是Prevotella(18.29%)、Faecalibacterium(12.35%)、Roseburia(9.77%)、Bacteroides(5.87%)和Megamonas(4.73%)。以上菌属仅列明确属分类水平的物种。
3. 肠道微生物群落结构的相关因素:本研究使用RDA和Envfit对肠道微生物群落结构和各因素的相关性进行分析。Envfit分析结果显示,抗病毒治疗(P < 0.001)是群落结构最显著的相关因素。其他显著因素有:确诊时间(P=0.009)、病毒载量(P=0.022)和HCV抗体(P=0.018)(表 2)。
RDA显示抗病毒治疗与其他3个显著因素在群落结构的构建过程中呈负相关性。RDA同时还显示了特定的属与这些因素的相关性。抗病毒治疗与高丰度的Megamonas和Bacteroides呈正相关,与Prevotella、Roseburia和Faecalibacterium呈负相关,而确诊时间、病毒载量和HCV抗体3个因素则与高丰度的Prevotella、Roseburia和Faecalibacterium呈正相关,与Megamonas和Bacteroides呈负相关(图 1)。
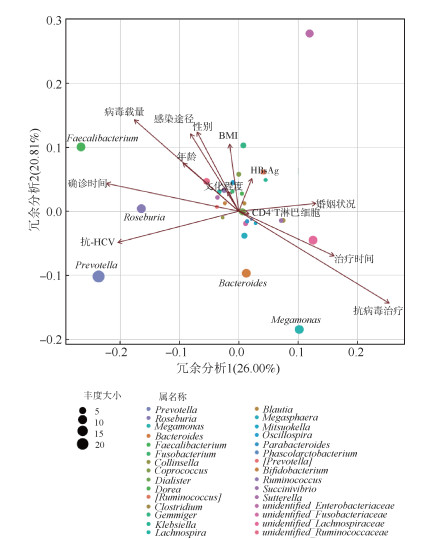
|
| 注:箭头代表各因素,射线越长表示该因素的相关性越显著。各因素之间的夹角为锐角时表示两个因素之间呈正相关关系,夹角为钝角时呈负相关关系 图 1 河南省部分地区HIV/AIDS肠道微生物群落结构与相关因素的冗余分析 |
4. 未抗病毒治疗HIV感染者中肠道微生物群落结构的相关因素:上述分析显示抗病毒治疗是与肠道微生物结构最相关的因素。为了探讨其他因素,本研究分析其中的36例未抗病毒治疗HIV感染者病例。见表 1。
Envfit分析结果显示,确诊时间(P=0.003)是与未抗病毒治疗HIV感染者肠道微生物群落结构显著相关的因素(表 2)。RDA显示确诊时间与高丰度的Roseburia、Faecalibacterium、Megamonas和Prevotella呈正相关,与Fusobacterium和Bacteroides呈负相关(图 2)。

|
| 注:箭头代表各因素,射线越长表示该因素的相关性越显著;各因素之间的夹角为锐角时表示两个因素之间呈正相关关系,夹角为钝角时呈负相关关系 图 2 河南省部分地区未抗病毒治疗HIV感染者肠道微生物群落结构与相关因素冗余分析 |
本研究对河南省HIV/AIDS肠道微生物群落结构的特征进行了描述,并重点探讨了肠道微生物群落结构和各因素之间的相关性,同时也对高丰度的属与这些因素的相关性进行了分析。
既往大多数研究都提供了抗病毒治疗改变肠道微生物群的证据[9, 13-14],不过在具体的影响以及细节上尚未达成共识[15]。本研究发现,在总人群中抗病毒治疗是与群落结构最显著的相关因素,因此除了对总人群进行分析,也对其中的抗病毒治疗和未抗病毒治疗的人群都进行了分析和比较。在总人群中,前5位优势菌属为:Prevotella、Roseburia、Megamonas、Bacteroides和Faecalibacterium,抗病毒治疗人群中的是Prevotella、Megamonas、Bacteroides、Roseburia和Faecalibacterium,未抗病毒治疗人群中的是Prevotella、Faecalibacterium、Roseburia、Bacteroides和Megamonas。3个人群前5位优势菌属的种类一样,但是丰度排序不同,主要体现在Faecalibacterium和Megamonas,因此推测这种差别可能与抗病毒治疗有较强的相关性。随后的RDA也印证了这一推测,抗病毒治疗与Megamonas呈正相关,与Faecalibacterium呈负相关,且这种相关性较之其他菌属更强。更进一步的证据表现在确诊时间与Megamonas的正负相关性的不同上,在未抗病毒治疗人群中,确诊时间与Megamonas呈正相关,而在总人群中,确诊时间与Megamonas呈负相关,其他主要高丰度菌属的正负相关性在两个人群中都一致,这说明抗病毒治疗可能逆转其他因素对于Megamonas的影响。因此,抗病毒治疗不仅是影响肠道微生物群落结构的重要因素,影响着多个主要菌属的丰度,而且可能对Megamonas有着更为重要的影响。
HIV感染能够改变宿主的肠道菌群,但是有一些研究发现:未抗病毒治疗的HIV感染者没有明显改变直肠微生物群[16],并且在中国人群的研究也发现HIV感染者和健康对照之间的α-多样性并没有显著性差异的情况[17]。这种情况被研究者解释为肠道菌群的改变可能是与疾病进展和感染持续时间有关。确有研究发现在HIV感染的早期,肠道细菌的丰富度和组成未发生HIV特异性变化,而在慢性HIV感染者中,最终可以发现肠道细菌组中的HIV特异性特征[18]。这些研究提示了感染时间与HIV感染者肠道微生物之间存在关联。本研究发现确诊时间是未抗病毒治疗人群中与群落结构显著相关的因素之一,而且在总人群中,确诊时间也是除抗病毒治疗之外与群落结构显著相关的因素。RDA显示,在未抗病毒治疗人群中,确诊时间与高丰度的Roseburia、Faecalibacterium、Megamonas和Prevotella呈正相关,与Bacteroides呈负相关。在总人群中,确诊时间与高丰度的Prevotella、Roseburia和Faecalibacterium呈正相关,与Megamonas和Bacteroides呈负相关。这反映了确诊时间对于诸多高丰度菌属的影响是广泛的,在HIV/AIDS肠道微生物群落构建过程中起着重要的作用。因此,在设计肠道微生物研究时,应将病例的确诊时间作为重要的考虑因素。
近来很多研究发现MSM状态或性偏好是HIV/AIDS肠道微生物改变的影响因素[7-8, 19]。更有Meta分析认为MSM状态对于肠道微生物的影响更甚于HIV感染[9, 20]。我国一项研究中也发现HIV感染者中的肠道微生物的改变与HIV的传播途径有关[21]。本研究中未发现感染途径与HIV/AIDS肠道微生物群落结构的相关性。这为HIV/AIDS的性偏好对肠道微生物影响的研究提供了不一样的证据。
本研究存在局限性。尚不能准确知晓病例真正的感染时间,因此本研究用确诊时间代表感染时长进行了讨论,这可能使结果存在一定的误差。本研究使用丰度最高的30个属作为肠道微生物群落结构分析的衡量指标,与一些研究中使用的β多样性略有差别。本研究仅对各因素的相关性进行了分析,要证明这些因素的因果关系还需要更合理的人群设计以及更多临床数据才能说明。
综上所述,抗病毒治疗和确诊时间是河南省部分地区HIV/AIDS肠道微生物群落结构显著相关的因素,并且对多个高丰度菌属有着重要影响。
利益冲突 所有作者声明无利益冲突
作者贡献声明 刘佳:实施研究、撰写文章;耿杰、刘家琪、薛秀娟、闫江舟、袁源、张向兵:实施研究;刘春华、张国龙:技术指导和支持
| [1] |
Kamada N, Seo SU, Chen GY, et al. Role of the gut microbiota in immunity and inflammatory disease[J]. Nat Rev Immunol, 2013, 13(5): 321-335. DOI:10.1038/nri3430 |
| [2] |
Pickard JM, Zeng MY, Caruso R, et al. Gut microbiota: Role in pathogen colonization, immune responses, and inflammatory disease[J]. Immunol Rev, 2017, 279(1): 70-89. DOI:10.1111/imr.12567 |
| [3] |
Zevin AS, McKinnon L, Burgener A, et al. Microbial translocation and microbiome dysbiosis in HIV-associated immune activation[J]. Curr Opin HIV AIDS, 2016, 11(2): 182-190. DOI:10.1097/COH.0000000000000234 |
| [4] |
Dillon SM, Frank DN, Wilson CC. The gut microbiome and HIV-1 pathogenesis: a two-way street[J]. AIDS, 2016, 30(18): 2737-2751. DOI:10.1097/QAD.0000000000001289 |
| [5] |
Bandera A, de Benedetto I, Bozzi G, et al. Altered gut microbiome composition in HIV infection: causes, effects and potential intervention[J]. Curr Opin HIV AIDS, 2018, 13(1): 73-80. DOI:10.1097/COH.0000000000000429 |
| [6] |
Lozupone CA, Li M, Campbell TB, et al. Alterations in the gut microbiota associated with HIV-1 infection[J]. Cell Host Microbe, 2013, 14(3): 329-339. DOI:10.1016/j.chom.2013.08.006 |
| [7] |
Noguera-Julian M, Rocafort M, Guillén Y, et al. Gut microbiota linked to sexual preference and HIV infection[J]. EBioMedicine, 2016, 5: 135-146. DOI:10.1016/j.ebiom.2016.01.032 |
| [8] |
Armstrong AJS, Shaffer M, Nusbacher NM, et al. An exploration of Prevotella-rich microbiomes in HIV and men who have sex with men[J]. Microbiome, 2018, 6(1): 198. DOI:10.1186/s40168-018-0580-7 |
| [9] |
Tuddenham SA, Koay WLA, Zhao N, et al. The impact of human immunodeficiency virus infection on gut microbiota α-diversity: An individual-level meta-analysis[J]. Clin Infect Dis, 2020, 70(4): 615-627. DOI:10.1093/cid/ciz258 |
| [10] |
Vangay P, Johnson AJ, Ward TL, et al. US immigration westernizes the human gut microbiome[J]. Cell, 2018, 175(4): 962-972. DOI:10.1016/j.cell.2018.10.029 |
| [11] |
Kaplan RC, Wang Z, Usyk M, et al. Gut microbiome composition in the Hispanic Community Health Study/Study of Latinos is shaped by geographic relocation, environmental factors, and obesity[J]. Genome Biol, 2019, 20(1): 219. DOI:10.1186/s13059-019-1831-z |
| [12] |
Yatsunenko T, Rey FE, Manary MJ, et al. Human gut microbiome viewed across age and geography[J]. Nature, 2012, 486(7402): 222-227. DOI:10.1038/nature11053 |
| [13] |
Ji YJ, Zhang FD, Zhang RF, et al. Changes in intestinal microbiota in HIV-1-infected subjects following cART initiation: influence of CD4+T cell count[J]. Emerg Microbes Infect, 2018, 7(1): 1-4. DOI:10.1038/s41426-018-0117-y |
| [14] |
Ancona G, Merlini E, Tincati C, et al. Long-term suppressive cART is not sufficient to restore intestinal permeability and gut microbiota compositional changes[J]. Front Immunol, 2021, 12: 639291. DOI:10.3389/fimmu.2021.639291 |
| [15] |
Pinto-Cardoso S, Klatt NR, Reyes-Terán G. Impact of antiretroviral drugs on the microbiome: unknown answers to important questions[J]. Curr Opin HIV AIDS, 2018, 13(1): 53-60. DOI:10.1097/COH.0000000000000428 |
| [16] |
Nowak RG, Bentzen SM, Ravel J, et al. Rectal microbiota among HIV-uninfected, untreated HIV, and treated HIV-infected in Nigeria[J]. AIDS, 2017, 31(6): 857-862. DOI:10.1097/QAD.0000000000001409 |
| [17] |
Ling ZX, Jin CZ, Xie TS, et al. Alterations in the fecal microbiota of patients with HIV-1 infection: an observational study in a Chinese population[J]. Sci Rep, 2016, 6: 30673. DOI:10.1038/srep30673 |
| [18] |
Rocafort M, Noguera-Julian M, Rivera J, et al. Evolution of the gut microbiome following acute HIV-1 infection[J]. Microbiome, 2019, 7(1): 73. DOI:10.1186/s40168-019-0687-5 |
| [19] |
Kelley CF, Kraft CS, de Man TJ, et al. The rectal mucosa and condomless receptive anal intercourse in HIV-negative MSM: implications for HIV transmission and prevention[J]. Mucosal Immunol, 2017, 10(4): 996-1007. DOI:10.1038/mi.2016.97 |
| [20] |
Zhou J, Zhang Y, Cui P, et al. Gut microbiome changes associated with HIV infection and sexual orientation[J]. Front Cell Infect Microbiol, 2020, 10: 434. DOI:10.3389/fcimb.2020.00434 |
| [21] |
Xu HM, Ou ZT, Zhou YJ, et al. Intestinal mucosal microbiota composition of patients with acquired immune deficiency syndrome in Guangzhou, China[J]. Exp Ther Med, 2021, 21(4): 391. DOI:10.3892/etm.2021.9822 |
 2022, Vol. 43
2022, Vol. 43




