文章信息
- 张鑫, 张文竹, 李文革, 赵红庆, 武艳华, 李虎, 刘正洁, 吴媛, 卢金星.
- Zhang Xin, Zhang Wenzhu, Li Wenge, Zhao Hongqing, Wu Yanhua, Li Hu, Liu Zhengjie, Wu Yuan, Lu Jinxing
- 我国艰难梭菌核糖体分型库的标准化及应用
- Standardization and application on ribotyping library of Clostridioides difficile in China
- 中华流行病学杂志, 2019, 40(12): 1624-1628
- Chinese Journal of Epidemiology, 2019, 40(12): 1624-1628
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2019.12.022
-
文章历史
收稿日期: 2019-05-06
2. 中国疾病预防控制中心传染病预防控制所, 北京 102206;
3. 河北北方学院医学检验学院预防医学教研室, 张家口 075000;
4. 新疆维吾尔自治区阿克苏地区疾病预防控制中心 843000
2. State Key Laboratory of Infectious Disease Prevention and Control, National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China;
3. Department of Prevention College of Laboratory Medicine, Hebei North University, Zhangjiakou 075000, China;
4. Aksu Regional Center for Disease Control and Prevention of Xinjiang Uygur Autonomous Region, Aksu 843000, China
艰难梭菌(Clostridioides difficile)是导致住院患者发生抗生素性腹泻(antibiotic-associated diarrhea)的一种常见病原菌[1]。2000年以来,艰难梭菌感染(Clostridioides difficile infection,CDI)在全球范围内持续上升,发病率增高,疾病严重程度不断升级,而且有数据表明这种趋势仍在继续[2-3]。目前应用于艰难梭菌分子分型方法主要有多位点序列分析(MLST)、PFGE、限制性核酸内切酶分析、毒素基因分型(toxin typing)、核糖体分型(ribotyping)、随机扩增多态性DNA分析、扩增片段长度多态性分析、多位点可变数目串联重复序列分析(MLVA)、表层蛋白A基因序列分型(Slpa typing)等[4]。每种分型方法各有其特点和适用范围,核糖体分型方法为欧洲地区艰难梭菌分型的首选方法,其对于提供我国CDI流行病学数据、提供数字化分型信息、构建艰难梭菌监测网络和评价各地区流行菌株也有着重要意义。本实验室此前初步建立了基于QIAxcel毛细管电泳仪方法的艰难梭菌核糖体分型库[5],但其操作流程过于简单,分型效果也未评价。因此,为提高各级网络实验室数据间可比性、实现该方法的应用推广,制定核糖体分型实验室标准操作流程、应用该流程标准化已建立的分型库、纳入已收集的374株艰难梭菌分离株补充该库型别、并对应用该库分型得到的结果进行一致性评价十分必要。
材料与方法1.菌株来源:54株标准株包括来自美国国家菌种保藏中心(ATCC)标准菌株30株和欧洲CDC的标准菌株24株,374株实验株来自2010-2018年我国7个省份(北京、上海、山东、陕西、浙江、广东和云南)人和动物分离株。
2.实验室操作流程标准化及核糖体数据库的评价:在本实验室原有的操作流程基础上,参照国外PCR方法[6]和不同参数下的分型效果,依据核糖体分型库的分析要求,优化实验方案,制定相应参考范围,编写实验室核糖体分型标准操作流程和QIAxcel仪器结果处理参考标准。遵循上述标准流程和标准,由3位不同实验人员分别于2017、2018和2019年3个不同年份,按照同一实验方案独立完成54株标准菌株分型,包括以下步骤:
(1)传代培养:于脑心浸液培养基上,37 ℃厌氧培养24 h;
(2)细菌DNA提取:挑取细菌至Chelex-100溶液中煮沸12 min,离心保留上清液;
(3)核糖体分型PCR扩增程序:95 ℃ 10 min;94 ℃ 1 min,58 ℃ 1 min,72 ℃ 2 min,25个循环;72 ℃延伸7 min;
(4)PCR产物纯化使用德国QIAxcel公司MinElute PCR purification Kit;凝胶电泳使用德国QIAxcel公司QIAxcel毛细管电泳仪;数据处理只保留两端Maker间清晰的电泳条带;
(5)聚类分析:导入毛细管电泳结果于BioNumerics 7.6(基于QIAxcel模块)软件,分析类型选择平均连锁聚类法(Unweighted Pair Group Method Arithmetic Clustering)。
3.核糖体分型库数据补充:应用本实验室核糖体分型标准操作流程,对374株艰难梭菌分离株采用QIAxcel毛细管电泳仪进行核糖体分型实验,按照标准处理电泳结果后使用BioNumerics 7.6(基于QIAxcel模块)软件识别,导入Excel表格填充菌株背景资料,选择平均连锁聚类法进行聚类分析,人工矫正结果,统一原则命名。
4.数据库分型一致性检验:采用简单随机抽样的方法,从2017-2019年本实验室已有核糖体分型结果的分离株中随机抽取194株生长状况良好的菌株作为样本株,按照本实验室核糖体分型标准流程、数据处理方案进行分型。应用SPSS 19.0软件进行统计学分析,对本次分型结果与原分型结果的一致性采用Kappa检验,并进行假设检验,P<0.05为差异有统计学意义。一致性强弱程度分为6个区段:当K<0.00,一致性强度极差;0.00~0.20,微弱;0.21~0.40,弱;0.41~0.60,中度;0.61~0.80,高度;0.81~1.00,极强[7]。
结果1.标准流程及评价:根据本实验室已有的QIAXcel毛细管电泳核糖体分型方法,此次研究明确了核糖体分型实验室操作流程,包括菌株传代培养条件,细菌基因组DNA提取量,PCR反应相应参数、PCR产物纯化试剂盒种类,毛细管电泳操作前DNA参考浓度、毛细管电泳结果处理标准与建议,核糖体分型数据库使用说明和注意事项等步骤。
由3位不同实验者在2017、2018和2019年按照标准实验室操作流程,分别对标准菌株进行核糖体分型实验部分结果,在严格遵守实验操作流程的条件下,两种型别经不同人员7次传代培养后核糖体分型电泳条带均表现为高度清晰,条带位置与数量稳定,分型效果良好。见图 1。
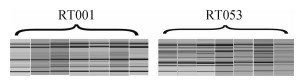
|
| 图 1 RT001与RT053型各7次重复电泳条带 |
此次应用的54株标准菌株中共有40个RT型别,包括:RT001、RT002、RT003、RT005、RT009、RT010、RT012、RT014、RT015、RT017、RT020、RT023、RT024、RT027、RT029、RT031、RT038、RT046、RT053、RT054、RT056、RT057、RT060、RT070、RT075、RT076、RT077、RT078、RT081、RT085、RT087、RT095、RT106、RT118、RT126、RT131、RT140、RT207、RT220和RT251。在BioNumerics 7.6软件聚类结果中HL2与ATCC1874、HL12与ATCC1382、HL20与ATCC1808、HL27与ATCC1803、HL53与ATCC1804虽未能100%匹配,但相似性极高,仍可以根据条带数目和分布确认分型关系,其余44个标准株相似度均为100%,可直接快速地从聚类结果中得到分型结果,见图 2。
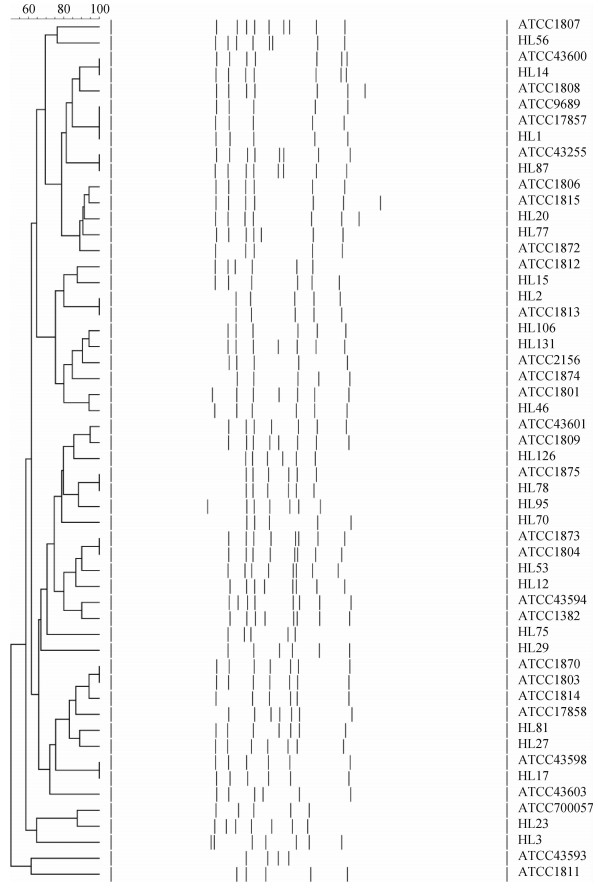
|
| 图 2 54株标准菌株核糖体分型聚类分析 |
2.核糖体分型数据库概况:2017-2019年本实验室共对374株艰难梭菌分离株进行核糖体实验,已有核糖体型别占74.87%(280株),新定义核糖体型别占25.13%(94株),其中RT012占12.03%(45株),RT001和RT126占9.63%(36株),RT017占9.36%(35株),RT046占8.29%(31株),RT014占4.81%(18株),RT009占3.74%(14株),RT002占2.67%(10株),RT078和RT220占2.41%(9株),RT140和RT207占1.87(7株),RT085占1.07(4株),RT038、RT070和RT077占0.53%(2株),RT015、RT020、RT023、RT024、RT027、RT031、RT056和RT131占0.27(1株),以及本次实验中未出现的型别RT003、RT005、RT029、RT053、RT054、RT057、RT060、RT075、RT076、RT081、RT087、RT095、RT106、RT118和RT251,见图 3。
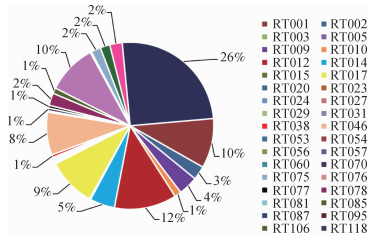
|
| 注:未显示百分比的RT型别对应实验菌株数为0 图 3 实验菌株RT分型结果 |
本次实验菌株来源省份主要以北京(148株)、山东(89株)和云南(72株)为主,上海、广东、河南、四川、浙江等地较少,动物宿主标本主要来自山东(51株),少量来自云南。
3.核糖体分型一致性分析:抽取源自2010-2018年中国13个城市194株实验室分离株,其中RT001型33株,RT009型13株,RT012型42株,RT017型32株,RT046型21株,RT078型8株,RT085型5株,RT126型34株,RT207型4株和RT220型2株。分别由两名实验人员独立进行核糖体分型分析。两次分型结果假设检验χ2值为1 342.313,P<0.001,可认为两次结果具有一致性,Kappa值为0.891,P<0.001,根据参考标准,该方法一致性为极强。
讨论在欧洲地区、美国及澳大利亚,艰难梭菌感染引起的大范围暴发流行已造成了沉重的疾病负担,这些国家或地区CDC和医院已将其列为常规检测监测项目[8-11],我国近10年也涌现了大量关于艰难梭菌的研究报道[12-13]。但艰难梭菌分子分型方案和方法的不同,使得不同实验室间有关艰难梭菌分子分型报道可能存在偏差。目前,在进化种群关系研究、艰难梭菌流行监测、暴发溯源方面,MLST、PFGE和PCR-ribotyping有很好的应用。其中MLST作为国际上常用的分型方法,虽然具有较好的重复性,操作简便,易于实验室间数据共享。但其ST1型对应4种核糖体型别RT027、RT016、RT036、RT176;ST11型对应10种核糖体型别:RT078、RT033、RT045、RT066、RT126、RT127、RT193、RT237、RT280、RT281等情况而表现出对于识别流行菌株存在不足[14-15]。北美地区则将PFGE分型作为“金标准”,但其操作繁琐、DNA易降解、分析较复杂且数据不便交流。分辨能力较好的MLVA在分析主要流行株和溯源研究中也存在不足,而以WGS为基础的cgMST作为新兴的方法应用于各级网络实验室流行病学监测检测还需要时间的证明和检验。因此,核糖体分型方法在识别主要流行株(RT027和RT078)和相关研究上有极大的优势。本研究采用了分辨力和分型能力较好,操作相对简单、快速,适用于艰难梭菌感染暴发溯源的QIAxcel毛细管电泳仪核糖体分型方法[16-18]。
数据库标准化和一致性检验结果表明:使用54株引自国外的标准菌株在遵循标准核糖体分型实验流程前提下,毛细管电泳结果具有清晰、稳定的条带和良好的重复性,在使用BioNumerics 7.6软件进行聚类分析时,易得到直观准确的分型结果。两次分型结果一致性较好。艰难梭菌核糖体分型库的补充结果表明,不同地区、不同宿主来源的艰难梭菌分离株核糖体型别存在差异[19-20],可以为我国开展艰难梭菌感染流行病学监测提供可靠信息。然而需要注意的是,核糖体分型方法受细菌生长状况、细菌变异速度以及实验室环境等因素的影响,在使用BioNumerics 7.6软件分析时仍需人工矫正数据和检查结果。
综上所述,本研究标准化后的核糖体分型数据库在实际分型操作中简单快捷,仪器要求较低,适合我国大部分地区分型工作的使用,结果一致性好,方便实验室间数据快速比对,具有较强的实用性。标准化后的实验室操作流程和核糖体分型数据库可以架构在我国已有的国家致病菌识别网上,为艰难梭菌感染从市到省到国家的三级监测检测提供解决方案。并且在建立我国核糖体分型网络数据库,构建全国艰难梭菌监测网络,制定CDI防控策略,应用于临床诊疗,提供预防指导,感染暴发时溯源和开展疾病预防控制工作等方面有着重要意义。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Burnham CAD, Carroll KC. Diagnosis of Clostridium difficile infection:an ongoing conundrum for clinicians and for clinical laboratories[J]. Clin Microbiol Rev, 2013, 26(3): 604-630. DOI:10.1128/CMR.00016-13 |
| [2] |
Burke KE, Lamont JT. Clostridium difficile infection:A worldwide disease[J]. Gut Liver, 2014, 8(1): 1-6. DOI:10.5009/gnl.2014.8.1.1 |
| [3] |
Knight DR, Briony E, Chang BJ, et al. Diversity and evolution in the genome of Clostridium difficile[J]. Clin Microbiol Rev, 2015, 28(3): 721-741. DOI:10.1128/CMR/00127-14 |
| [4] |
Rodriguez C, van Broeck J, Taminiau B, et al. Clostridium difficile infection:Early history, diagnosis and molecular strain typing methods[J]. Microb Pathog, 2016, 97: 59-78. DOI:10.1016/j.micpath.2016.05.018 |
| [5] |
刘笑舒.我国艰难梭菌毒素基因库及核糖体分型标准库的建立及应用[D].北京: 中国疾病预防控制中心, 2017. Liu XS. Establish and application of toxin genes profile and standard ribotyping library of Clostridium difficile in China[D]. Beijing: Chinese Center for Disease Control and Prevention, 2017. |
| [6] |
O'Neill GL, Ogunsola FT, Brazier JS, et al. Modification of a PCR ribotyping method for application as a routine typing scheme for Clostridium difficile[J]. Anaerobe, 1996, 2(4): 205-209. DOI:10.1006/anae.1996.0028 |
| [7] |
夏邦世, 吴金华. Kappa一致性检验在检验医学研究中的应用[J]. 中华检验医学杂志, 2006, 29(1): 83-84. Xia BS, Wu JH. Application of Kappa consistency test in laboratory medicine research[J]. Chin J Lab Med, 2006, 29(1): 83-84. DOI:10.3760/j:issn:1009-9158.2006.01.030 |
| [8] |
Miller M, Gravel D, Mulvey M, et al. Health care-associated Clostridium difficile infection in Canada:patient age and infecting strain type are highly predictive of severe outcome and mortality[J]. Clin Infect Dis, 2010, 50(2): 194-201. DOI:10.1086/649213 |
| [9] |
Davies KA, Ashwin H, Longshaw CM, et al. Diversity of Clostridium difficile PCR ribotypes in Europe:results from the European, multicentre, prospective, biannual, point-prevalence study of Clostridium difficile infection in hospitalised patients with diarrhoea (EUCLID), 2012 and 2013[J]. Euro Surveill, 2016, 21(29). DOI:10.2807/1560-7917.ES.2016.21.29.30294 |
| [10] |
Bouwknegt M, van Dorp S, Kuijper E. Burden of Clostridium difficile infection in the United States[J]. N Engl J Med, 2015, 372(24): 2368-2370. DOI:10.1056/NEJMc1505190 |
| [11] |
Slimings C, Armstrong P, Beckingham WD, et al. Increasing incidence of Clostridium difficile infection, Australia, 2011-2012[J]. Med J Aust, 2014, 200(5): 272-276. DOI:10.5694/mja13.11153 |
| [12] |
徐英春, 张曼. 中国成人艰难梭菌感染诊断和治疗专家共识[J]. 协和医学杂志, 2017, 8(2): 131-138. Xu YC, Zhang M. Expert consensus on diagnosis and treatment of Clostridium difficile infection in Chinese adults[J]. Med J Peking Union Med Coll Hosp, 2017, 8(2): 131-138. |
| [13] |
杨雪妹, 吴允孚. 艰难梭菌感染的流行病学和诊治进展[J]. 中国感染与化疗杂志, 2013, 13(4): 312-316. Yang XM, Wu YF. Infections caused by Clostridium difficile:changing epidemiology and management update[J]. Chin J Infect Chemother, 2013, 13(4): 312-316. DOI:10.3969/j.issn.1009-7708.2013.04.017 |
| [14] |
Janezic S, Rupnik M. Genomic diversity of Clostridium difficile strains[J]. Res Microbiol, 2015, 166(4): 353-360. DOI:10.1016/j.resmic.2015.02.002 |
| [15] |
Suo JJ, Yan ZQ, Wu Y, et al. Clostridium difficile RT078/ST11:A threat to community population and pigs identified in elder hospitalized patients in Beijing, China[J]. Infect Control Hosp Epidemiol, 2017, 38(11): 1383-1385. DOI:10.1017/ice.2017.206 |
| [16] |
Stubbs SLJ, Brazier JS, O'Neill GL, et al. PCR targeted to the 16S-23S rRNA gene intergenic spacer region of Clostridium difficile and construction of a library consisting of 116 different PCR ribotypes[J]. J Clin Microbiol, 1999, 37(2): 461-463. |
| [17] |
Huber CA, Foster NF, Riley TV, et al. Challenges for standardization of Clostridium difficile typing methods[J]. J Clin Microbiol, 2013, 51(9): 2810-2814. DOI:10.1128/JCM.00143-13 |
| [18] |
Fenner L, Widmer AF, Goy G, et al. Rapid and reliable diagnostic algorithm for detection of Clostridium difficile[J]. J Clin Microbiol, 2008, 46(1): 328-330. DOI:10.1128/JCM.01503-07 |
| [19] |
Liu XS, Li WG, Zhang WZ, et al. Molecular characterization of Clostridium difficile isolates in China from 2010 to 2015[J]. Front Microbiol, 2018, 9: 845. DOI:10.3389/fmicb.2018.00845 |
| [20] |
Liao F, Li WG, Gu WP, et al. A retrospective study of community-acquired Clostridium difficile infection in southwest China[J]. Sci Rep, 2018, 8: 3992. DOI:10.1038/s41598-018-21762-7 |
 2019, Vol. 40
2019, Vol. 40


