文章信息
- 王晓林, 贾磊, 李韩平, 刘永健, 韩婧婉, 李天一, 李敬云, 李林.
- Wang Xiaolin, Jia Lei, Li Hanping, Liu Yongjian, Han Jingwan, Li Tianyi, Li Jingyun, Li Lin.
- 1996-2014年中国HIV-1毒株CRF01_AE亚型传播簇和传播网络研究
- Transmission cluster and network of HIV-1 CRF01_AE strain in China, 1996-2014
- 中华流行病学杂志, 2019, 40(1): 84-88
- Chinese Journal of Epidemiology, 2019, 40(1): 84-88
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2019.01.017
-
文章历史
收稿日期: 2018-07-23
我国自1985年报道了第一例HIV感染者以来,感染人数不断增加,HIV主要传播途径不断变化,HIV-1流行亚型逐渐多样化[1]。目前,CRF01_AE是我国流行最广的HIV-1亚型[2-3]。通过对流行簇的研究发现其主要经性传播途径扩散传播[4-8]。
HIV的传播和流行受人群行为特征及多种社会因素的影响,揭示这些特征和规律对指导防控具有重要意义。分子进化分析通过研究HIV传播过程中各因素的作用,能够发现特定时期和地域的HIV传播规律。传播簇是分子进化中存在传播关系的序列集群。通过对传播簇的分析可从分子层面推断某些传播事件中有关联的感染者间可能存在的传播关系,如Chalmet等[9]通过传播簇研究,发现北美地区的MSM人群推动了当地HIV的快速传播扩散。传播网络分析方法在HIV分子进化研究中的应用,使发现及定位高风险传播者成为可能[10]。本研究结合传播簇和传播网络研究方法,对我国HIV-1毒株CRF01_AE亚型的传播和流行规律进行了分析。
材料和方法1.病毒基因序列的收集和处理:下载Los Alamos数据库(http://www.hiv.lanl.gov)中采样时间截至2014年12月31日的所有中国CRF01_AE亚型pol区基因序列(HXB2位置:2 053~3 052)。分析我国HIV流行状况,自本实验室数据库中进一步补充CRF01_AE流行省份的病毒基因序列。汇总序列背景信息,如传播途径、采样时间和采样省份等。同时具有多条序列的感染者只保留首次采样获得的序列。利用Los Alamos数据库的在线质量控制(Quality Control)去除质量较差序列,利用NCBI病毒基因型分析工具(https://www.ncbi.nlm.nih.gov/projects/genotyping/formpage.cgi)、在线REGA重组分析工具、Comet 2.2软件进一步确认病毒亚型并删除重组序列。加入HIV-1 A1亚型序列为参照序列,利用HIV align在线软件比对序列,利用BioEdit 7.2软件进行手工校正,最后删除所有序列主要耐药突变位点后进行系统进化分析。
2.系统进化树构建:利用jModelTest 2.1.7软件选择碱基替代模型,利用PhyML 3.0软件最大似然法(Maximum Likelihood,ML)构建系统进化树[11],采用SH(Shimodaira-Hasegawa)法进行可靠性检验,利用MEGA 6.0软件计算簇内平均基因距离(Tamura-Nei 93碱基替代模型)[12],利用Cluster Picker 1.2.5软件挑选传播簇[13](SH≥95且簇内平均基因距离≤0.015)。
3.传播网络结构分析:选择基因距离≤0.015的序列,利用Cytoscape 3.6.0软件分析序列间关系[14],绘制传播网络结构图,分析传播关系。以传播网络中的单个序列为节点,与其具有传播关系的节点数量为“度”。度是传播网络分析最主要的参数,指的是网络中一个点连接其他点的个数,表示一个人可能传播其他人的人数。度的值越高,传播的风险越大:低传播风险者的度≤3;高传播风险者的度≥4。
4.统计学分析:应用SPSS 18.0软件进行统计学分析,采用χ2检验或Fisher精确概率法比较不同人群的构成差异。采用趋势χ2检验比较各时段不同序列分布趋势差异。以P<0.05为差异有统计学意义。
结果1.序列基本情况:共获得2 094条质量较高、背景信息齐全的CRF01_AE亚型毒株序列。其中1 323条来自于Los Alamos数据库,771条由本实验室序列数据库补充。序列采样时间覆盖1996-2014年,地区分布包含我国19个省份(吉林省、辽宁省、北京市、河北省、天津市、山东省、河南省、安徽省、江苏省、上海市、浙江省、福建省、陕西省、四川省、云南省、贵州省、广西壮族自治区、广东省、湖南省)。感染途径包括异性性传播(775例,37.01%),注射吸毒传播(83例,3.96%)、男男性传播(1 236例,59.03%)。
2.传播簇分析:采用GTR+I+G碱基替代模型,构建基于所有CRF01_AE亚型pol区基因序列的系统进化树(图 1)。根据传播簇判断标准获得82个传播簇,包含255条序列(12.18%)。传播簇序列采样时间为2002-2014年,地域包含16个省份。以包含≥2个省份来源序列的传播簇为跨省传播簇,以所有序列来自同一省份的传播簇为省内传播簇进一步分析传播簇特点。所有传播簇中,省内传播簇数量和包含序列数(61个,173条)明显多于跨省传播簇(21个,82条),提示地域对于CRF01_ AE亚型的传播具有一定的影响。在21个跨省传播簇中,1个分布于地理上相邻的2个省、15个分布于地理上不相邻的2个省,5个分布于3~6个省份。
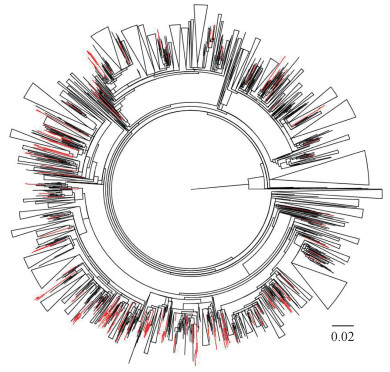
|
| 注:CRF01_AE亚型传播簇内序列以红色标记,簇外序列以黑色标记 图 1 1996-2014年我国HIV-1毒株CRF01_AE亚型的最大似然法构建系统进化树 |
在82个传播簇中,男男性传播比例最高(66.27%,169/255)。男男性传播人群的成簇比例随时间升高,由1996-2008年的2.41%(2/83),上升为2009-2010年的12.77%(60/470)、2011-2012年的18.25%(69/378)、2013-2014年的23.61%(72/305),在4个时间段具有明显的上升趋势(χ2=27.800,df=1,P=0.000)(图 2)。异性性行为传播在4个时段差异无统计学意义,趋于稳定。跨省传播簇内异性性传播占13.41%(11/82),男男性传播占86.59%(71/82);省内传播簇内异性性传播占39.88%(69/173),男男性传播占56.65%(98/173)注射吸毒占3.47%(6/173);跨省传播簇中男男性传播的比例(86.59%,71/82)明显高于省内传播簇(56.65%,98/173),差异有统计学意义(χ2=22.792,P=0.000)。省内传播簇的序列在各时段成簇比例均高于跨省传播簇,但在4个时段差异无统计学意义。跨省传播簇在4个时段成簇序列比例由1996-2008年的0.67%(2/297),上升为2009-2010年的2.89%(25/865)、2011-2012年的5.43%(25/460)、2013-2014年的6.36%(30/472),具有明显的上升趋势(χ2=20.276,df=1,P=0.000)(图 2)。
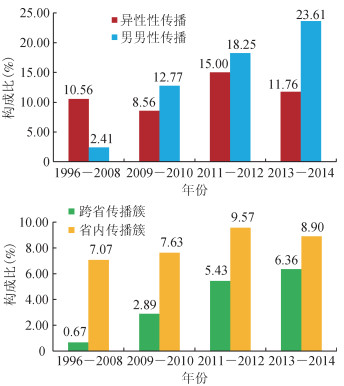
|
| 图 2 1996-2014年我国HIV-1毒株CRF01_AE亚型不同时段内成簇序列构成比 |
部分传播簇含有2种及以上的传播途径,表明不同高危人群之间存在潜在的传播关系[15]。在82个传播簇中,共有15个传播簇含有2种及以上传播途径,其中8个为省内传播簇(13.11%,8/61);7个为跨省传播簇(33.33%,7/21),差异有统计学意义(χ2=4.273,P=0.039)。
3.传播网络分析:对构成82个传播簇的255条序列进行了传播网络分析,确定了构成传播网络的247个点(node)、345条边(edge)和75个具有传播关系的网络。其中,度≥4的高传播风险者比例为34.51%(88/255),跨省传播簇高传播风险者的比例为51.22%(42/82),低传播风险者的比例为48.78%(40/82);省内传播簇高传播风险者的比例为26.59%(46/173),低传播风险者的比例为73.41%(127/173),差异有统计学意义(χ2=14.932,P=0.000),提示跨省传播簇有更大的传播风险。

|
| 注:彩色圆点为不同度(绿色表示度≤3;黄色表示度≥4);彩色连接线为不同传播簇(红色为跨省传播簇,灰色为省内传播簇) 图 3 1996-2014年我国HIV-1毒株CRF01_AE亚型82个传播簇的成簇序列的传播网络 |
高传播风险者中,男男性传播占86.36%(76/88),异性性行为传播占13.64%(12/88)。跨省传播簇男男性传播占95.24%(40/42),异性性行为传播占4.76%(2/42);省内传播簇男男性传播占78.26%(36/46),异性性行为传播占21.74%(10/46)。低传播风险者中,跨省传播簇男男性传播占77.50%(31/40),异性性行为传播占22.50%(9/40);省内传播簇男男性传播占48.82%(62/127),异性性行为传播占46.46%(59/127),注射吸毒传播占4.72%(6/127)。
讨论CRF01_AE亚型自20世纪90年代初传入我国以来快速传播流行,成为我国主要流行亚型之一。
HIV-1 pol区蛋白酶和反转录酶基因序列的替换率最低,且由于HIV-1耐药检测和监测的需要而易于大量获得,最适合用于HIV大数据分子进化分析[16]。传播簇是通过分子进化方法确认的可能存在传播关系的序列集群,分支可信度和基因距离是定义传播簇的关键指标。以SH≥95标准获得的成簇结果,假阳性率和成簇真实性与标准的bootstrap方法最为接近[17-19]。HIV-1不同亚型的型内平均基因距离存在差异,CRF01_AE与B亚型的型内平均基因距离最相似,而B亚型pol区基因距离≤0.015被证实存在确定的传播关系[20-22]。本研究参考B亚型相关的研究结果,采用的传播簇的成簇标准是SH≥95且簇内平均基因距离≤0.015,可认为是有效的成簇标准。用TN93进化模型,可以纠正替换偏倚和减少简并碱基对结果的影响[23-24],减少漏判和误判。
虽然地域对于CRF01_AE的传播具有影响,如省内传播簇的数量和包含的序列明显多于跨省传播簇,但跨省传播簇的传播地域更广,20个簇分布于≥2个地理不相邻的省份,最多覆盖6个省份。因此,重点分析跨省传播人群的传播风险构成对有效防控具有重要的指导意义。男男性传播人群在传播簇中的成簇比例随年份增加显示出明显的增长趋势,在CRF01_AE亚型的传播中扮演了越来越重要的角色。男男性传播人群在跨省传播簇的比例显著高于省内传播,以及跨省传播簇成簇序列比例上升趋势明显的结果表明,男男性传播可能造成了跨省传播簇的快速增长。此外,2种及以上传播途径的簇在跨省传播簇中的比例显著高于省内传播簇,揭示了跨省传播簇中的男男性传播人群可能具有更加复杂的传播网络关系,传播风险更高。有研究表明2012年上海市13.9%的男男性传播人群在过去半年有异性性伴,48.6%存在多次偶然性伴的性行为[25]。Esbj?rnsson等[15]的研究表明,多种传播途径混杂的传播簇中存在从男男性传播人群向其他人群扩散传播的分子特征。Dennis等[26]的研究指出2种及以上传播途径的簇中包含同异性发生性行为的男男性传播感染者,意味着其传播风险和危害大于单一传播途径的传播簇。
自2014年在美国波士顿举办的“反转录病毒和机会性感染大会”提出HIV传播网络研究方法以来[27],社会和分子传播网络研究在艾滋病领域得到迅速应用,基于传播网络的精准干预理念得到广泛认可。传播簇无法评估感染者对传播的贡献,而传播网络可以以直观的形式展示网络的结构特征、网络节点及网络成员之间的关系。Oster等[28]认为对度≥4的人群进行精准干预对预防HIV的传播将更加有效,因此,一般将度≥4的网络人群作为高风险群体。为了进一步阐明跨省传播簇中传播风险,本研究比较了跨省和省内传播簇中人群的传播风险,结果表明传播网络中男男性传播是主要的传播风险,是跨省传播的主要传播途径。
综上所述,通过传播簇和传播网络的分析表明,我国HIV-1毒株CRF01_AE亚型的跨省传播相对省内传播增长趋势显著。男男性传播成簇人群增长迅速,是跨省传播高传播风险的主要人群。密切关注跨省传播簇内风险目标,对跨省传播簇内男男性传播人群进行重点教育、实施精准干预、加强实时监测和管理,或许能有效阻断HIV-1毒株CRF01_AE亚型的传播,控制新发感染。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
陶艳琳, 赵雪涛, 汤宇帆, 等. 2000-2012年中国HIV基因亚型时空分布的系统综述[J]. 中华疾病控制杂志, 2013, 17(12): 1073-1082. Tao YL, Zhao XT, Tang YF, et al. A systematic review of temporal and geographical distributions of HIV genotypes in China during 2000-2012[J]. Chin J Dis Control Prev, 2013, 17(12): 1073-1082. |
| [2] |
朱可心, 庄勋, 周元, 等. 人类免疫缺陷病毒-1 CRF01_AE毒株在我国的分子流行特征分析[J]. 中华传染病杂志, 2015, 33(7): 396-402. Zhu KX, Zhuang X, Zhou Y, et al. Molecular epidemiological characterization of human immunodeficiency virus-1 CRF01_AE strains in China[J]. Chin J Infect Dis, 2015, 33(7): 396-402. DOI:10.3760/cma.j.issn.1000-6680.2015.07.005 |
| [3] |
苏迎盈, 刘慧鑫, 吴静, 等. 中国HIV-1基因亚型分布及流行趋势[J]. 中华流行病学杂志, 2014, 35(10): 1164-1168. Su YY, Liu HX, Wu J, et al. Distribution of HIV-1 genotypes in China:a systematic review[J]. Chin J Epidemiol, 2014, 35(10): 1164-1168. DOI:10.3760/cma.j.issn.0254-6450.2014.10.020 |
| [4] |
He X, Xing H, Ruan YH, et al. A comprehensive mapping of HIV-1 genotypes in various risk groups and regions across China based on a nationwide molecular epidemiologic survey[J]. PLoS One, 2012, 7(10): e47289. DOI:10.1371/journal.pone.0047289 |
| [5] |
Li L, Chen LL, Liang SJ, et al. Subtype CRF01_AE dominate the sexually transmitted human immunodeficiency virus type 1 epidemic in Guangxi, China[J]. J Med Virol, 2013, 85(3): 388-395. DOI:10.1002/jmv.23360 |
| [6] |
Wang XL, He X, Zhong P, et al. Phylodynamics of major CRF01_AE epidemic clusters circulating in mainland of China[J]. Sci Rep, 2017, 7(1): 6330. DOI:10.1038/s41598-017-06573-6 |
| [7] |
Li XG, Liu HZ, Liu L, et al. Tracing the epidemic history of HIV-1 CRF01_AE clusters using near-complete genome sequences[J]. Sci Rep, 2017, 7(1): 4024. DOI:10.1038/S41598-017-03820-8 |
| [8] |
Li XS, Gao R, Zhu KX, et al. Genetic transmission networks reveal the transmission patterns of HIV-1 CRF01_AE in China[J]. Sex Transm Infect, 2018, 94(2): 111-116. DOI:10.1136/sextrans-2016-053085 |
| [9] |
Chalmet K, Staelens D, Blot S, et al. Epidemiological study of phylogenetic transmission clusters in a local HIV-1 epidemic reveals distinct differences between subtype B and non-B infections[J]. BMC Infect Dis, 2010, 10: 262. DOI:10.1186/1471-2334-10-262 |
| [10] |
Little SJ, Kosakovsky Pond SL, Anderson CM, et al. Using HIV networks to inform real time prevention interventions[J]. PLoS One, 2014, 9(6): e98443. DOI:10.1371/journal.pone.0098443 |
| [11] |
Guindon S, Dufayard JF, Lefort V, et al. New algorithms and methods to estimate maximum-likelihood phylogenies:assessing the performance of PhyML 3.0[J]. Syst Biol, 2010, 59(3): 307-321. DOI:10.1093/sysbio/syq010 |
| [12] |
Tamura K, Stecher G, Peterson D, et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [13] |
Ragonnet-Cronin M, Hodcroft E, Hué S, et al. Automated analysis of phylogenetic clusters[J]. BMC Bioinformatics, 2013, 14(1): 317. DOI:10.1186/1471-2105-14-317 |
| [14] |
Elia J, Glessner JT, Wang K, et al. Genome-wide copy number variation study associates metabotropic glutamate receptor gene networks with attention deficit hyperactivity disorder[J]. Nat Genet, 2012, 44(1): 78-84. DOI:10.1038/ng.1013 |
| [15] |
Esbjörnsson J, Mild M, Audelin A, et al. HIV-1 transmission between MSM and heterosexuals, and increasing proportions of circulating recombinant forms in the Nordic Countries[J]. Virus Evol, 2016, 2(1): vew010. DOI:10.1093/ve/vew010 |
| [16] |
Hué S, Clewley JP, Cane PA, et al. HIV-1 pol gene variation is sufficient for reconstruction of transmissions in the era of antiretroviral therapy[J]. AIDS, 2004, 18(5): 719-728. DOI:10.1097/00002030-200403260-00002 |
| [17] |
Grabowski MK, Herbeck JT, Poon AFY. Genetic cluster analysis for HIV prevention[J]. Curr HIV/AIDS Rep, 2018, 15(2): 182-189. DOI:10.1007/s11904-018-0384-1 |
| [18] |
Hué S, Brown AE, Ragonnet-Cronin M, et al. Phylogenetic analyses reveal HIV-1 infections between men misclassified as heterosexual transmissions[J]. AIDS, 2014, 28(13): 1967-1975. DOI:10.1097/Qad.0000000000000383 |
| [19] |
Anisimova M, Gil M, Dufayard JF, et al. Survey of branch support methods demonstrates accuracy, power, and robustness of fast likelihood-based approximation schemes[J]. Syst Biol, 2011, 60(5): 685-699. DOI:10.1093/sysbio/syr041 |
| [20] |
Patiño-Galindo JA, González-Candelas F. The substitution rate of HIV-1 subtypes:a genomic approach[J]. Virus Evol, 2017, 3(2): vex029. DOI:10.1093/ve/vex029 |
| [21] |
Zhao J, Chen L, Chaillon A, et al. The dynamics of the HIV epidemic among men who have sex with men (MSM) from 2005 to 2012 in Shenzhen, China[J]. Sci Rep, 2016, 6: 28703. DOI:10.1038/Srep28703 |
| [22] |
Smith DM, May SJ, Tweeten S, et al. A public health model for the molecular surveillance of HIV transmission in San Diego, California[J]. AIDS, 2009, 23(2): 225-232. DOI:10.1097/QAD.0b013e32831d2a81 |
| [23] |
Poon AFY. Impacts and shortcomings of genetic clustering methods for infectious disease outbreaks[J]. Virus Evol, 2016, 2(2): vew031. DOI:10.1093/ve/vew031 |
| [24] |
Hassan AS, Pybus OG, Sanders EJ, et al. Defining HIV-1 transmission clusters based on sequence data[J]. AIDS, 2017, 31(9): 1211-1222. DOI:10.1097/QAD.0000000000001470 |
| [25] |
张永, 石国政, 殷方兰, 等. 上海MSM人群拥有性伴情况及其性行为特征[J]. 中国艾滋病性病, 2012, 18(11): 720-723, 727. Zhang Y, Shi GZ, Yin FL, et al. Study on MSM's sex partners and sexual behaviors characteristics in Shanghai[J]. Chin J AIDS STD, 2012, 18(11): 720-723, 727. DOI:10.13419/j.cnki.aids.2012.11.011 |
| [26] |
Dennis AM, Hué S, Hurt CB, et al. Phylogenetic insights into regional HIV transmission[J]. AIDS, 2012, 26(14): 1813-1822. DOI:10.1097/QAD.0b013e3283573244 |
| [27] |
Buchbinder SP, Liu AY. CROI 2014:New tools to track the epidemic and prevent HIV infections[J]. Top Antivir Med, 2014, 22(2): 579-593. |
| [28] |
Oster AM, Wertheim JO, Hernandez AL, et al. Using molecular HIV surveillance data to understand transmission between subpopulations in the united states[J]. J Acquir Immune Defic Syndr, 2015, 70(4): 444-451. DOI:10.1097/QAI.0000000000000809 |
 2019, Vol. 40
2019, Vol. 40


