文章信息
- 刘文宣, 张晓琳, 高霞, 杨磊, 李保欣, 刘殿武.
- Liu Wenxuan, Zhang Xiaolin, Gao Xia, Yang Lei, Li Baoxin, Liu Dianwu.
- HLA-DQ基因多态性与HBV感染不同临床结局的关联研究
- Association between HLA-DQ gene polymorphisms and different outcomes of hepatitis B virus infection
- 中华流行病学杂志, 2016, 37(3): 384-388
- Chinese Journal of Epidemiology, 2016, 37(3): 384-388
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2016.03.019
-
文章历史
- 收稿日期: 2015-11-03
2. 050031 石家庄, 河北医科大学第一附属医院传染病科
2. Department of Infectious Diseases, The First Hospital of Hebei Medical University, Shijiazhuang 050031, China
HBV感染后机体会可出现不同的慢性化转归,多数为慢性乙型肝炎(乙肝),20%发展为肝硬化,5%进展为原发性肝细胞癌等[1]。HBV感染的结局除了受病毒本身的影响外,宿主遗传因素也起着重要的作用,而广泛存在于基因中的单核苷酸多态性(SNP)可导致机体对各种慢性损伤的反应具有明显的个体差异,影响不同的疾病转归[2]。人白细胞抗原(HLA)基因多态性影响不同个体对HBV的易感性及HBV感染的不同转归[3, 4]及机体对乙肝疫苗的应答[5]。日本的一项全基因组关联研究(GWAS)显示[6],
1. 研究对象:来自石家庄市第五医院、河北医科大学第一医院、第二医院和第四医院。入院时间为2011年1月至2012年1月,乙肝相关疾病就诊总数为752例,经严格的排除标准排除181例,按标准纳入HBV自然清除者175例、慢性HBV感染者(包括慢性乙肝 112例和肝硬化130例)242例、HBV相关性肝癌患者154例及健康体检者254例,共纳入研究对象825例。
纳入标准:中国北方汉族;性别和年龄不限;符合病例的诊断标准;签署知情同意书。排除标准:合并其他类型病毒如HCV、HEV或HIV感染;乙醇、自身免疫、药物、寄生虫及其他微生物等原因所致肝损害;急性乙肝、非HBV相关肝癌病例;不能参加或不愿在知情同意书上签字者。
健康对照者入选标准:未接种过HBV疫苗;抗-HBs、抗-HBc、抗-HBe、HBeAg及HBsAg均为阴性;血常规及生化指标均在正常参考范围内;无内分泌、心血管、肾脏疾病及其他肝脏疾病。
HBV自然清除者的入选标准:未接种过乙肝疫苗;肝功能各项指标在正常值范围内;血清抗-HBs及抗-HBe阳性;HBsAg、HBeAg及 HBV DNA均为阴性,同时符合以上四项条件者,定义为HBV自然清除。
病例诊断符合2000年《病毒性肝炎防治方案》[8]中的诊断标准。
2. 调查方法:采用自行设计的调查问卷,内容包括患者基本情况(性别、年龄、吸烟、饮酒、有无乙肝疫苗接种史等)、发病情况(病程、病情、有无其他疾病、有无家族史等)、检查结果(乙肝五项、HBV-DNA定量、ALT和AST水平等)。同时,收集外周血,用基质辅助激光解吸电离飞行时间质谱(MALDI- TOF MS)实验平台进行SNP分型。SNP分析模型:共显性模型、显性模型、隐性模型。假设某一突变位点的野生碱基为W,突变碱基为V。在共显性模型中定义该位点可能出现的3种基因型WW、WV、VV分别为0、1、2,基因型分析时的比较方式为WV/VV与WW比较;在显性模型中定义该位点可能出现的3种基因型WW、WV、VV分别为0、1、1,基因型分析时的比较方式为WV+VV与WW比较;在隐性模型中定义该位点可能出现的3种基因型WW、WV、VV分别为0、0、1,基因型分析时的比较方式为VV与WW+WV比较。根据研究目的,将研究对象分成不同亚组,分别研究2个SNP位点与HBV易感性(HBV感染组和健康组)、HBV自然清除(HBV感染组和HBV自然清除组)、肝硬化进展(肝硬化组和慢性乙肝组)、原发性肝细胞癌进展(原发性肝细胞癌组和肝硬化+慢性乙肝组)的关联。
3. 质量控制:①设置阴性对照:按样品数的1%加入阴性对照(水代替样品DNA),以监控实验中的扩增产物污染及交叉污染的情况;②室内质控标准品对照:按照样品数的1%加入室内质控标准品对照(“炎黄一号”细胞株DNA作为模板,DNA样品的每个SNP位点的确切数据可以在炎黄数据库中查询),以检测每个SNP位点的引物设计可靠度;③样本重复实验复核:随机选取样品数的5%进行重复实验复核,以验证重复性及稳定性。每一位点的合格样品数据符合度均达98%以上,提示实验具有一定的重复性和稳定性。本研究调查人员均接受统计培训,以保证流行病学调查资料的质量。所有调查表进行编码,复核无误后,采用EpiData软件,由双人录入计算机,统计学分析前,再次核查数据库。
4. 统计学分析:符合正态分布的计量资料用x±s描述,非正态分布资料用M和四分位数间距(Q)描述。分析SNP位点与HBV易感性及其感染结局的关联,先进行单因素分析,再用非条件logistic回归分析校正混杂因素,并计算OR值及其95%CI,以P<0.05为差异有统计学意义,检验水准均为双侧,采用SPSS 16.0软件进行数据处理和分析。
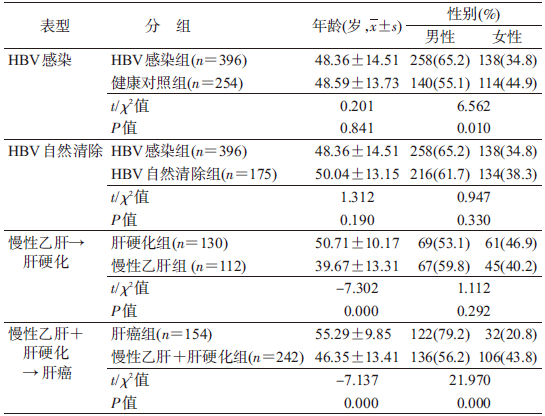
结 果1. 一般特征:HBV感染组男性比例高于健康对照组(65.2% vs. 55.1%,P=0.010);肝癌组男性比例高于慢性乙肝+肝硬化组(79.2% vs. 56.2%,P<0.001);HBV 自然清除组与HBV感染组、肝硬化组与慢性乙肝组比较,男女比例差异无统计学意义(P>0.05)。肝硬化组年龄高于慢性乙肝组[(50.71±10.17)岁 vs. (39.67±13.31)岁,P<0.001];肝癌组年龄高于慢性乙肝+肝硬化组[(55.29±9.85)岁 vs. (46.35±13.41)岁,P<0.001];健康对照组平均年龄为(48.59±13.73)岁,HBV感染组平均年龄为(48.36±14.51)岁,两组年龄差异无统计学意义(P>0.05);HBV 自然清除组平均年龄为(50.04±13.15)岁,与HBV感染组比较,两组年龄差异无统计学意义(P>0.05),见表 1。
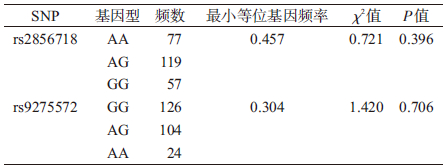
2. Hardy-Weinberg遗传平衡(H-W平衡)检验:基因分型完成后,对健康者(共254例研究对象)的2个位点基因型频率进行H-W平衡检验。结果显示,2个位点均符合H-W平衡(P>0.05),提示选取的研究对象具有群体代表性(表 2)。
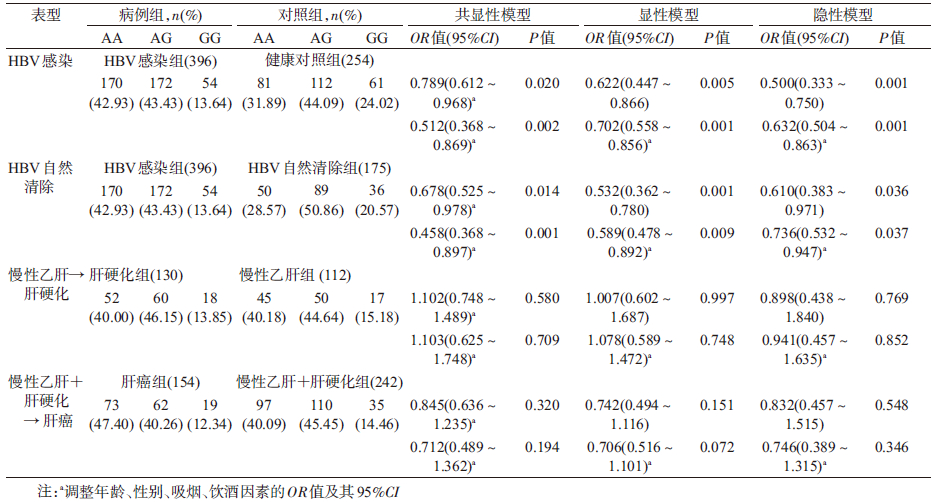
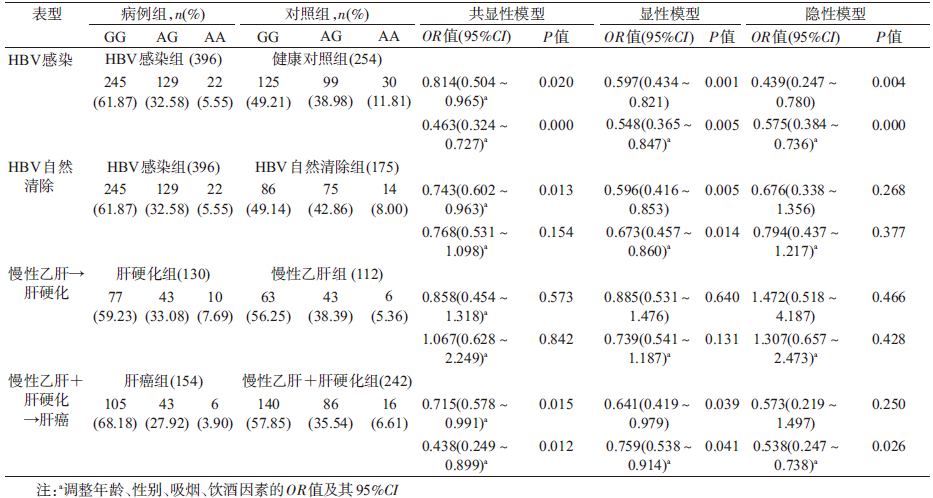
3. rs2856718与HBV感染的关系:在共显性模型下,携带rs2856718AG 和rs2856718GG基因型的个体感染HBV的风险降低,OR值分别为0.789(95%CI:0.612~0.968,P=0.020)和0.512(95%CI:0.368~0.869,P<0.001);在显性模型(GG+AG vs. AA)和隐性模型(GG vs. AA+AG)下,OR值分别为0.702(95%CI:0.558~0.856,P<0.001)和0.632(95%CI:0.504~0.863,P<0.001)。rs2856718与HBV自然清除相关,在共显性模型下,rs2856718与HBV自然清除有关联,携带rs2856718AG 和rs2856718GG基因型的个体HBV自然清除能力强(AG vs. AA,OR=0.678,95%CI:0.525~0.978,P=0.014; GG vs. AA,OR=0.458,95%CI:0.368~0.897,P=0.001);在显性模型下,rs2856718GG+AG基因型携带者HBV自然清除能力更强,发展为HBV慢性感染的可能性为rs2856718AA基因型携带者的0.589倍(95%CI:0.478~0.892,P<0.001);在隐性模型下,rs2856718GG基因型携带者HBV自然清除能力强,发展为HBV慢性感染的可能性为rs2856718AA+AG基因型携带者的0.736倍(95%CI:0.532~0.947,P=0.037)(表 3)。
4. rs9275572与HBV感染的关系:rs9275572与HBV易感性有关联。在共显性模型下,携带rs9275572AG或rs9275572AA基因型的个体感染HBV的风险降低,OR值分别为0.814(95%CI:0.504~0.965,P=0.020)和0.463(95%CI:0.324~0.727,P<0.001);在显性模型(AA+AG vs. GG)下和隐性模型(AA vs. GG+AG)下,OR值分别为0.548(95%CI:0.365~0.847,P<0.001)和0.575(95%CI:0.384~0.736,P<0.001)。rs9275572与HBV的自然清除相关,在共显性模型下,携带rs9275572AG基因型的个体HBV自然清除率更高,发展为HBV慢性感染的可能性为rs9275572GG基因型携带者的0.743倍(95%CI:0.602~0.963,P=0.013);在显性模型下,携带rs9275572AA+AG基因型的个体HBV自然清除率更高,发展为HBV慢性感染的可能性为rs9275572GG基因型携带者的为0.673(95%CI:0.457~0.860,P=0.014),见表 4。
机体在感染HBV后,HLA分子将抗原肽递呈给CD4+T淋巴细胞(CD4),病毒特异性的CD4支持细胞毒性T淋巴细胞能直接杀伤HBV感染的肝细胞[9, 10]。本研究显示,在中国北方汉族人群中,HLA-DQ的两个SNP位点(rs9275572和rs2856718)与HBV易感性、HBV 自然清除及HBV感染后的相关疾病进展有关,该研究结果与Hu等[11]在中国汉族人群的研究一致,其研究结果显示,HLA-DQ的SNP位点rs2856718与HBV相关肝癌及HBV自然清除有关。
Kumar等[7]报道了一项GWAS研究结果,SNP位点rs9275572与HCV感染有关,AA为HCV感染的危险碱基,且rs9275572与慢性丙型肝炎到原发性肝细胞癌的进展有关。而本研究显示,在中国北方汉族人群中,AA碱基为HBV感染的保护性因素,在HBV自然清除、慢性乙肝和肝硬化到原发性肝细胞癌的进展中起到保护作用,与Kumar等在日本人群中HCV的研究中的结果恰恰相反,这可能与HBV和HCV的致病机制不同及研究人群不同有关。关于HLA与HBV和HCV感染的关系,也有类似的报道,在日本人群中,HLA DR9是HCV感染的保护性因素[12],但在中国人群中却是HBV感染的危险因素[13]。SNP位点rs2856718与HBV易感性的报道来自一项日本的GWAS研究[6],该研究表明rs2856718A在健康人中的等位基因频率为0.47,在HBV患者中为0.59,为HBV感染的危险碱基;本研究中rs2856718A在健康人中的等位基因频率为0.54,在HBV患者中为0.64,同样为HBV感染的危险碱基,而rs2856718G为HBV感染的保护性碱基,与日本人群的研究一致。另外,本研究发现rs2856718G为HBV自然清除的保护因素,rs2856718G等位基因携带者HBV自然清除率能力高于rs2856718A等位基因携带者,且在原发性肝细胞癌进展中也发现了微弱保护作用,与另一项来自中国人群的rs2856718与HBV自然清除及在原发性肝细胞癌进展中的研究结果基本一致[13]。
本研究显示,HLA-DQ rs9275572和 rs2856718两个SNP位点与HBV的易感性、HBV 自然清除及HBV感染后的相关疾病进展有关,提示HLA-DQ可能在HBV感染结局中可能起重要作用。HLA复合体与免疫应答和免疫调节等有关,其多态性决定了HLA系统对致病因子的反应能力,这可能是宿主HLA基因多态性与HBV感染的免疫病理反应和转归密切相关的机制。
利益冲突 无| [1] Ott JJ,Stenens GA,Groeger J,et al. Global epidemiology of hepatitis B virus infection:new estimates of age-specific HBsAg seroprevalence and endemicity[J]. Vaccine,2012,30(12):2212-2219. DOI:10.1016/j.vaccine.2011.12.116. |
| [2] Zeng Z. Human genes involved in hepatitis B virus infection[J]. World J Gastroenterol, 2014,20(24):7696-7706. DOI:10.3748/wjg.v20.i24.7696. |
| [3] Tao JJ,Su KK,Yu CB,et al. Fine mapping analysis of HLA-DP/DQ gene clusters on chromosome 6 reveals multiple susceptibility loci for HBV infection[J]. Amino Acids,2015,47(12):2623-2634. DOI:10.1007/s00726-015-2054-6. |
| [4] Kamatani Y,Wattanapokayakit S,Ochi H,et al. A genome-wide association study identifies variants in the HLA-DP locus associated with chronic hepatitis B in Asians[J]. Nat Genet,2009,41(5):591-595. DOI:10.1038/ng.348. |
| [5] Li ZK,Nie JJ,Li J,et al. The effect of HLA on immunological response to hepatitis B vaccine in healthy people:a meta-analysis[J]. Vaccine,2013,31(40):4355-4361. DOI:10.1016/j.vaccine. 2013.06.108. |
| [6] Mbarek H,Ochi H,Urabe Y,et al. A genome-wide association study of chronic hepatitis B identified novel risk locus in a Japanese population[J]. Hum Mol Genet,2011,20(19):3884-3892. DOI:10.1093/hmg/ddr301. |
| [7] Kumar V,Kato N,Urabe Y,et al. Genome-wide association study identifies a susceptibility locus for HCV-induced hepatocellular carcinoma[J]. Nat Genet,2011,43(5):455-458. DOI:10.1038/ng.809. |
| [8] 中华医学会传染病与寄生虫病学分会、肝病学分会联合修订. 病毒性肝炎防治方案[J]. 中华传染病杂志,2001,19(1):56-62. Joint revision of Society of Infectious Diseases and Parasitic Diseases,Chinese Society of Hepatology,Chinese Medical Association. Guideline:prevention and treatment of viral hepatitis[J]. Chin J Infect Dis,2001,19(1):56-62. |
| [9] Zhang M,Zhou J,Zhao T,et al. Dissection of a circulating and intrahepatic CD4+ Foxp3+ T-cell subpopulation in chronic hepatitis B virus (HBV) infection:a highly informative strategy for distinguishing chronic HBV infection states[J]. J Infect Dis,2012,205(7):1111-1120. DOI:10.1093/infdis/jis011. |
| [10] 苏明宽,曾勇彬,陈静,等. HLA-DP、DQ基因多态性与福建汉族人群乙型肝炎病毒感染及不同结局的关联研究[J]. 中华医学遗传学杂志,2014,31(6):765-769. DOI:10.3760/cma.j.issn. 1003-9406.2014.06.019. Su MK,Zeng YB,Chen J,et al. Studies on the association of single nucleotide polymorphisms of HLA-DP and DQ genes with the outcome of chronic hepatitis B virus infection[J]. Chin J Med Genet, 2014,31(6):765-769. DOI:10.3760/cma.j.issn.1003-9406.2014.06.019. |
| [11] Hu LM,Zhai XJ,Liu JB,et al. Genetic variants in human leukocyte antigen/DP-DQ influence both hepatitis B virus clearance and hepatocellular carcinoma development[J]. Hepatology,2012,55(5):1426-1431. DOI:10.1002/hep.24799. |
| [12] Miki D,Ochi H,Takahashi A,et al. HLA-DQB1*03 confers susceptibility to chronic hepatitis C in Japanese:a genome-wide association study[J]. PLoS One,2013,8(12):e84226. DOI:10.1371/journal.pone.0084226. |
| [13] An P,Winkler C,Guan L,et al. A common HLA-DPA1 variant is a major determinant of hepatitis B virus clearance in Han Chinese[J]. J Infect Dis,2011,203(7):943-947. DOI:10.1093/infdis/jiq154. |
 2016, Vol. 37
2016, Vol. 37