微生物污染是药品生产过程控制及终产品质量评估的重要指标,也是影响消费者用药安全的关键因素[1]。实现药品生产工艺和生产环境中污染微生物“种”水平的准确鉴定是加强药品生产过程控制,提升产品质量,降低用药风险的有效手段,也是药品微生物污染溯源性分析的必要要求[2]。研究表明,葡萄球菌是药品生产和检验环境的主要污染微生物,占到全部污染微生物的50%以上[2-5]。其中,常见污染葡萄球菌主要为表皮葡萄球菌(Staphylococcus epidermidis)、溶血葡萄球菌(Staphylococcus haemolyticus)、沃氏葡萄球菌(Staphylococcus warneri)等血浆凝固酶阴性葡萄球菌(Coaglucose Negative Staphylococcus,CoNS)。近年的研究发现,除金黄色葡萄球菌等典型的凝固酶阳性葡萄球菌(Coaglucose Positive Staphylococcus,CoPS)外,凝固酶阴性葡萄球菌,如表皮葡萄球菌、溶血葡萄球菌等,也是人类疾病的重要病原体,与院内感染的发生密切相关[6-7]。因此,在药品微生物检验及日常环境监控中实现多种常见葡萄球菌种水平的准确鉴定,对药品生产过程的质量控制以及消费者的用药安全均具有重要意义。
随着分子生物学的发展,利用细菌特定基因核酸序列的比对分析可以实现对大多数细菌的鉴定,与传统生化鉴定技术相比更加快速准确。美国药典40版(USP 40)、欧洲药典9.0版(EP 9.0)、日本药局方17版(JP 17)和《中华人民共和国药典》2015年版均收载了基于核酸序列分析用于药品检验的方法和指导原则[8]。特别是基于16S rRNA核酸序列的比对方法已被广泛应用于细菌的鉴定。但是研究表明16S rRNA核酸序列在葡萄球菌属内高度保守,对亲缘关系较近的种区分能力较差[9]。此外,文献中报道tufA基因(编码延伸因子Tu)[10]、femA基因(编码氨酰转移酶)[11]、rpoB基因(编码RNA聚合酶β亚基)[12]和gap基因(编码甘油醛-3-磷酸脱氢酶)[13]等看家基因序列可以应用于葡萄球菌种水平的鉴定。因此本研究拟通过对不同靶基因序列的多样性分析及葡萄球菌“种”水平鉴定能力的比较评价,从中筛选出性能较优的靶基因序列或序列组合,从而提高对葡萄球菌种水平分子鉴定的准确性。
1 材料 1.1 主要仪器VITEK 2 Compact全自动微生物生化鉴定系统(梅里埃公司);ABI 9700型PCR扩增仪(ABI公司);琼脂糖凝胶电泳仪及成像系统(Bio-Rad公司);SANYOMIR-254型培养箱(SANYO公司);NuAire LABGARD型生物安全柜(NUAIRE公司)。
1.2 试验菌株收集整理60株菌种信息明确的葡萄球菌,涵盖18个常见葡萄球菌种,其中葡萄球菌标准菌株购自美国菌种保藏中心(ATCC)和中国工业微生物菌种保藏管理中心(CICC),其余葡萄球菌为本实验室菌种库保藏药品及生产环境分离株,实验室已严格按照《中华人民共和国药典》2015年版“微生物鉴定指导原则9204”要求,对葡萄球菌环境分离株进行了全面菌种鉴定分析,获得了可靠的菌种鉴定结果。
2 实验方法 2.1 16S rRNA分子鉴定挑取单克隆菌落,接种于胰酪大豆胨液体培养基(TSB,MERCK公司)中,37 ℃过夜培养。采用细菌基因组DNA小量纯化试剂盒(TAKARA,宝生物工程有限公司),提取60株葡萄球菌的基因组DNA。利用引物27F(5′-AGAGTTTGATCMTGGCTCAG-3′)和907R(5′-CCGTCAATTCMTTTRAGTTT-3′)扩增葡萄球菌的16S rRNA序列[14]。琼脂糖凝胶电泳检测后,PCR阳性扩增产物进行双向核酸测序(委托上海生物工程有限公司完成)。利用软件Lasergene 7.0校正和拼接核酸序列;然后进行16S rRNA序列的Blast比对分析,依据比对结果进行60株葡萄球菌的菌种判定[15]。
2.2 VITEK 2 Compact生化鉴定挑取菌株单克隆菌落,划线接种于胰酪大豆胨琼脂培养基平板(TSA,广东环凯微生物科技有限公司)。37 ℃过夜培养后,利用0.45%的无菌盐水(广东环凯微生物科技有限公司),制备浓度合适的菌悬液(比浊度0.8~1.0)。选取GP鉴定卡,按照VITEK 2 Compact系统操作说明进行试验操作,对60株葡萄球菌进行生化鉴定,可信度 > 90%为好的鉴定结果。
2.3 靶基因的PCR扩增和核酸测序采用PCR扩增60株葡萄球菌的靶基因tufA、femA、rpoB和gap序列,扩增引物信息如表 1所示。琼脂糖凝胶电泳检测后,对阳性扩增产物进行双向核酸测序(委托上海生物工程有限公司完成)。利用软件Lasergene 7校正和拼接靶基因序列。
|
|
表 1 靶基因PCR扩增引物信息 Table 1 Primers for PCR amplification of target genes |
利用软件Mega 7.0比对分析60株葡萄球菌的16S rRNA、tufA、femA、rpoB和gap的核酸扩增序列。去除两端准确度较低的核酸序列后,在具有单核苷酸多态性的区域截取每个靶基因的标准分析序列(约500 bp)。利用软件DNASP V 5.10对每个靶基因的标准分析序列及16S rRNA分别与tufA、femA、rpoB和gap的串联序列进行多样性分析(等位基因数,单核苷酸多态性等)。然后利用软件Mega 7.0分别对单个靶基因的标准分析序列及16S rRNA分别与tufA、femA、rpoB和gap的串联比对序列,采用邻接法(Neighbor-Joining method)进行60株葡萄球菌的聚类关系分析,依据聚类分析结果,评价不同靶基因及其串联序列对葡萄球菌属不同种的分子鉴定能力。
3 实验结果 3.1 16S rRNA序列比对和VITEK生化鉴定结果16S rRNA序列比对和VITEK生化鉴定结果如表 2所示。结果表明,基于16S rRNA序列比对不能够区分山羊葡萄球菌(S. caprae)和头状葡萄球菌(S. capitis),两者16S rRNA序列同源性为100%。3株巴氏葡萄球菌(S. pasteuri)通过VITEK方法被判定为沃氏葡萄球菌(S. warneri)(可信度 > 95%)。除山羊葡萄球菌和巴氏葡萄球菌外,其余葡萄球菌的16S rRNA序列比对和VITEK鉴定结果一致。
|
|
表 2 60株葡萄球菌的16S rRNA序列比对和VITEK 2生化鉴定结果 Table 2 Identification information of 60 Staphylococcus isolates by 16S rRNA sequence alignment and VITEK 2 compact system |
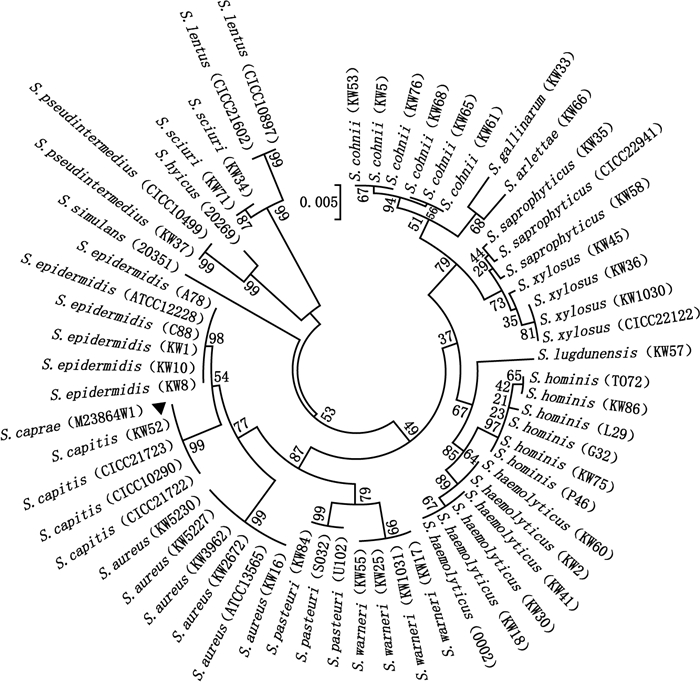
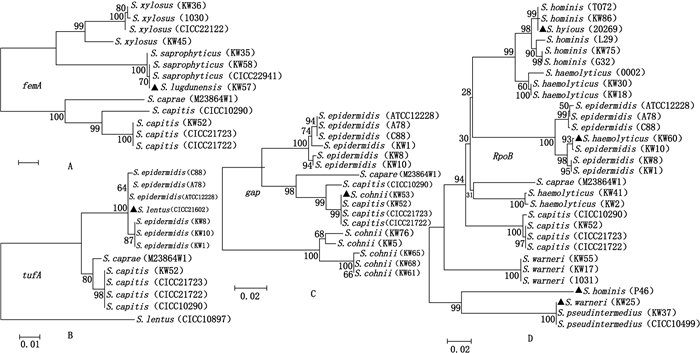
图 1显示了60株葡萄球菌的16S rRNA序列聚类关系,结果表明16S rRNA序列聚类分析能够准确鉴定葡萄球菌属的16个常见种,但不能有效区分山羊葡萄球菌(S. caprae)与头状葡萄球菌(S. capitis)。靶基因femA、tufA、gap和rpoB的核酸序列能够准确区分山羊葡萄球菌和头状葡萄球菌,但对其他一些葡萄球菌易造成种水平的错误判定(图 2)。其中femA序列不能准确区分路邓葡萄球菌(S. lugdunensis)和腐生葡萄球菌(S. saprophyticus)(图 2-A),tufA序列不能准确鉴定缓慢葡萄球菌(S. lentus)和表皮葡萄球菌(S. epidermidis)(图 2-B),gap序列不能准确区分头状葡萄球菌(S. capitis)和科氏葡萄球菌(S. cohnii)(图 2-C),而rpoB序列则对多种葡萄球菌不能有效区分,如猪葡萄球菌(S. hyicus)、溶血葡萄球菌(S. haemolyticus)、人葡萄球菌(S. hominis)和沃氏葡萄球菌(S. warneri)等(图 2-D)。综上所述,基于单个靶基因序列的聚类分析易造成部分葡萄球菌种水平的错误鉴定。
|
▲表示不能正确区分鉴定的菌株(▲demonstrated the falsely identified strain) 图 1 基于16S rRNA序列的60株葡萄球菌聚类关系 Figure 1 Neighbor-joining tree showing the phylogenetic relationships of 60 Staphylococcus isolates based on 16S rRNA sequence |
|
▲表示不能正确区分鉴定的菌株(▲ demonstrated the falsely identified strain) 图 2 基于靶基因femA(A)、tufA(B)、gap(C)和rpoB(D)的葡萄球菌聚类关系 Figure 2 Neighbor-Joining tree showing the phylogenetic relationships of some Staphylococcus isolates based on femA(A), tufA(B), gap(C)and rpoB(D)sequence, respectively |
由于靶基因rpoB对葡萄球菌的分子鉴定能力较差,因此不对其核酸序列进行多样性分析。16S rRNA、femA、tufA和gap在60株葡萄球菌中的核酸序列多样性分析结果如表 3所示。其中16S rRNA共有25个基因型,可变单核苷酸位点占分析序列全长的9.57%(67/687),与其他靶基因相比,16S rRNA序列在不同葡萄球菌之间的变异程度较低。而femA在葡萄球菌之间的变异程度最高,具有40个基因型,特别是可变单核苷酸位点占到分析序列全长的60.2%。
|
|
表 3 不同靶基因及相关串联序列多样性分析及葡萄球菌种鉴定结果 Table 3 Nucleotide sequence variation of different target genes and identification results ofStaphylococcus isolates based on different nucleotide sequences |
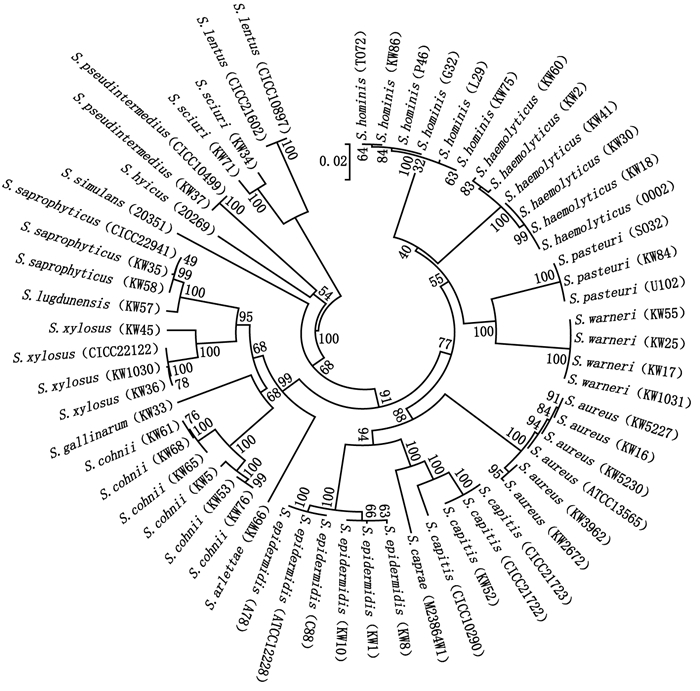
上述结果表明,尽管femA基因在葡萄球菌不同菌株中的变异程度最强,但仍然不能够区分路邓葡萄球菌和腐生葡萄球菌。因此为提高对葡萄球菌种水平的准确鉴定能力,本研究进一步评价了靶基因的串联序列对葡萄球菌种水平的鉴定能力。分别将16S rRNA序列与靶基因femA、tufA和gap的序列进行串联,序列多样性分析表明, 串联序列比单个靶基因序列具有更好的多样性。16S rRNA与不同靶基因的串联序列对葡萄球菌不同种的鉴定结果如表 3所示。与单个靶基因序列相比,16S rRNA与tufA和gap的串联序列对葡萄球菌的鉴定能力没有明显改善,仍然不能有效区分缓慢葡萄球菌与表皮葡萄球菌(tufA),科氏葡萄球菌与头状葡萄球菌(gap)。而16S rRNA与femA的串联序列,相比单个的16S rRNA序列或femA基因序列,能够更好地区分鉴定山羊葡萄球菌与头状葡萄球菌,路邓葡萄球菌与腐生葡萄球菌,克服了单一靶基因种水平鉴定能力的不足。60株葡萄球菌的16S rRNA与femA串联序列聚类关系(图 3),表明基于16S rRNA与femA的串联序列能够实现对18种葡萄球菌的准确鉴定。
|
图 3 基于16S rRNA和femA串联序列的60株葡萄球菌聚类关系 Figure 3 Neighbor-Joining tree showing the phylogenetic relationships of 60 Staphylococcus isolates based on compilation of 16S rRNA with femA sequence |
葡萄球菌属在自然界中分布广泛,其中多个葡萄球菌种都是重要的致病菌,也是药品生产和检验环境中的典型污染物。实现葡萄球菌属不同种葡萄球菌的快速准确鉴定,对药品中污染微生物的溯源控制以及院内感染的防控都具有重要意义。目前市场上存在着多种基于生化鉴定的全自动微生物鉴定系统,如VITEK 2和BD Phoenix等。Delmas等学者利用VITEK GP鉴定卡,对VITEK数据库中已有葡萄球菌种的鉴定准确率为93.2%[16]。而本研究结果表明, VITEK系统不能准确鉴定巴氏葡萄球菌。相比传统生化鉴定试验,微生物全自动鉴定系统尽管能够快速实现大部分细菌的种属鉴定,但细菌表型特征的不稳定性及参比数据库菌种数量、来源的局限性,严重影响着细菌最终鉴定结果的准确性[17-18]。基于靶基因序列检测及分析的方法,特别是基于16S rRNA序列比对的方法,被视为目前细菌鉴定与分类的“金标准”[19], 但研究发现,由于16S rRNA序列保守性较高,不能区分部分亲缘关系较近的葡萄球菌菌种, 本研究结果与文献报道[9]一致,山羊葡萄球菌和头状葡萄球菌的16S rRNA序列相似性为100%,基于16S rRNA序列无法区分山羊葡萄球菌和头状葡萄球菌。以上分析表明,采用全自动微生物鉴定系统或16S rRNA序列比对分析方法都会造成部分葡萄球菌种的错误鉴定。
除16S rRNA序列外,文献也报道了分别基于tufA[10]、femA[11]、rpoB[12]和gap[13]等看家基因序列建立的葡萄球菌鉴定方法。但目前对这些看家基因的种水平鉴定能力缺乏比较评价,极大地限制了这些看家靶基因的实际应用。本研究结果表明,靶基因tufA、femA、rpoB和gap在葡萄球菌种间和种内都表现出了比16S rRNA序列更强的序列多样性,但对部分葡萄球菌仍然不能准确鉴定。这可能是由于看家靶基因在一些葡萄球菌种内的变异超过了种间变异,导致单个靶基因序列的聚类分析不能全面反映菌株之间真实的亲缘进化关系。而多基因序列的串联分析通过增加分析靶基因的数目,可以提高对菌株的分辩能力,能够更好地反映菌株之间真实的亲缘关系。如基于多靶基因序列分析的多位点序列分型方法(MLST),已经广泛应用于多种致病菌的分型溯源[20]。本研究比较评价了不同靶基因串联序列对葡萄球菌的种水平鉴定能力,结果表明, 基于2个靶基因16S rRNA与femA的串联序列即可实现18种葡萄球菌的准确鉴定,克服了使用单一靶基因序列种水平鉴定不准确的问题,能够显著提高对葡萄球菌种水平的准确鉴定。
4.2 小结本研究比较评价了16S rRNA、tufA、femA、rpoB和gap序列对18种葡萄球菌的鉴定能力,结果表明,基于单个靶基因的序列分析会造成部分葡萄球菌种水平的错误鉴定。而通过16S rRNA与femA 2个靶基因的串联序列分析就能够准确鉴定常见的18种葡萄球菌,提高了葡萄球菌种水平鉴定的准确性。基于核酸序列比对分析的细菌鉴定方法,需要配套相应的参比靶基因核酸序列数据库,因此未来应加强对标准核酸序列数据库的建设,推进基于核酸序列比对分析方法在药品污染微生物鉴定和临床病原微生物诊断方面的应用。
| [1] |
LNGE R, DIETRICH D. Environmental risk assessment of pharmaceutical drug substances-conceptual considerations[J]. Toxicol Lett, 2002, 131(1-2): 97. DOI:10.1016/S0378-4274(02)00071-1 |
| [2] |
范一灵, 蒋波, 房蕊, 等. 药品无菌检查中微生物污染的鉴定和污染溯源分析[J]. 药物分析杂志, 2011, 31(6): 1067. FAN YL, JIANG B, FANG R, et al. Identification and characterization of bacterial contaminations isolated from drug sterility test[J]. Chin J Pharm Anal, 2011, 31(6): 1067. |
| [3] |
郑小玲, 王征南, 王知坚, 等. 药品微生物检测实验室环境菌库的建立[J]. 中国现代应用药学, 2015, 32(7): 847. ZHENG XL, WANG ZN, WANG ZJ, et al. Establishment of the environmentally microbial library in the drug sterility testing laboratory[J]. Chin J Mod Appl Pharm, 2015, 32(7): 847. |
| [4] |
户美玲, 陈佩, 严东珍, 等. 建立洁净区微生物数据库与无菌药品GMP生产过程控制的探讨[J]. 微生物学免疫学进展, 2013, 41(3): 33. HU ML, CHEN P, YAN DZ, et al. Study on establishing a microorganism database of clean areas and GMP manufacturing process-control for sterile medicinal products[J]. Prog Microbiol Immunol, 2013, 41(3): 33. |
| [5] |
范一灵, 冯震, 钟玮, 等. 无菌药品生产企业核心区微生物污染调查与分析[J]. 中国药事, 2014, 28(6): 586. FAN YL, FENG Z, ZHONG W, et al. Investigation and analysis of microbiological contamination in core area of sterile drug manufacturing enterprises[J]. Chin Pharm Aff, 2014, 28(6): 586. |
| [6] |
BRUMFITT W, HAMILTON-MILLER JMT. Coagulase-negative Staphylococci[J]. Int J Dermatol, 2014, 3(2): 161. |
| [7] |
PETTI CA, SIMMON KE, MIRO JM, et al. Genotypic diversity of coagulase-negative Staphylococci causing endocarditis:a global perspective[J]. J Clin Microbiol, 2008, 46(5): 1780. |
| [8] |
洪小栩, 冯震, 许华玉, 等. 分子生物学技术用于药品质量控制国家标准核酸数据库的建设规划研究[J]. 中国医药工业杂志, 2016, 47(5): 651. HONG XX, FENG Z, XU HY, et al. Program of national standard nucleotide sequence database used in quality control of phamaceutical products by molecular biology technology[J]. Chin J Pharm, 2016, 47(5): 651. |
| [9] |
SHAH MM, ⅡHARA H, NODA M, et al. dnaJ gene sequence-based assay for species identification and phylogenetic grouping in the genus Staphylococcus[J]. Int J Syst Evol Microbiol, 2007, 57(Pt 1): 25. |
| [10] |
HWANG SM, KIM MS, PARK KU, et al. Tuf gene sequence analysis has greater discriminatory power than 16S rRNA sequence analysis in identification of clinical isolates of coagulase-negative Staphylococci[J]. J Clin Microbiol, 2011, 49(12): 4142. |
| [11] |
VANNUFFEL P, HEUSTERSPREUTE M, BOUYER M, et al. Molecular characterization of femA from Staphylococcus hominis and Staphylococcus saprophyticus, and femA-based discrimination of staphylococcal species[J]. Res Microbiol, 1999, 150(2): 129. DOI:10.1016/S0923-2508(99)80030-8 |
| [12] |
DRANCOURT M, RAOULT D. rpoB gene sequence-based identification of Staphylococcus species[J]. J Clin Microbiol, 2002, 40(4): 1333. DOI:10.1128/JCM.40.4.1333-1338.2002 |
| [13] |
YUGUEROS J, TEMPRANO A, SNCHEZ M, et al. Identification of Staphylococcus spp.by PCR-restriction fragment length polymorphism of gap gene[J]. J Clin Microbiol, 2001, 39(10): 3693. |
| [14] |
BECKER K, HARMSEN D, MELLMANN A, et al. Development and evaluation of a quality-controlled ribosomal sequence database for 16S ribosomal DNA-based identification of Staphylococcus species[J]. J Clin Microbiol, 2004, 42(11): 4988. DOI:10.1128/JCM.42.11.4988-4995.2004 |
| [15] |
KIM M, OH HS, PARK S C, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. Int J Syst Evol Microbiol, 2014, 64(Pt 2): 346. |
| [16] |
DELMAS J, CHACORNAC J P, ROBIN F, et al. Evaluation of the Vitek 2 system with a variety of Staphylococcus species[J]. J Clin Microbiol, 2008, 46(1): 311. DOI:10.1128/JCM.01610-07 |
| [17] |
HEIKENS E, FLEER A, PAAUW A, et al. Comparison of genotypic and phenotypic methods for species-level identification of clinical isolates of coagulase-negative Staphylococci[J]. J Clin Microbiol, 2005, 43(5): 2286. |
| [18] |
KYOUNG P, DO-HOON L, MOON-WOO S, et al. Comparison of the MicroScan, VITEK 2, and Crystal GP with 16S rRNA sequencing and MicroSeq 500 v2[J]. BMC Microbiol, 2008, 8(1): 233. |
| [19] |
朱诗应, 戚中田. 16S rDNA扩增及测序在细菌鉴定与分类中的应用[J]. 微生物与感染, 2013, 8(2): 104. ZHU SY, QI ZT. Application of bacterial 16S rDNA amplication and sequencing in the classification and identification of bacteria[J]. J Microb Infect, 2013, 8(2): 104. |
| [20] |
AANENSEN DM, SPRATT BG. The multilocus sequence typing network:mlst.net.[J]. Nucleic Acids Res, 2005, 33(Web Server issue): W728. |
 2018, Vol. 38
2018, Vol. 38 
