2. 中央民族大学, 北京 100081;
3. 四川省食品药品检验检测院, 成都 611731;
4. 太原市食品药品检验所, 太原 030000;
5. 松原市食品药品检验所, 松原 138000
2. Minzu University of China, Beijing 100081, China;
3. Sichuan Institute for Food and Drug Control, Sichuan 611731, China;
4. Taiyuan Institute for Food and Drug Control, Taiyuan 030000, China;
5. Songyuan Institute for Food and Drug Control, Songyuan 138000, China
肉苁蓉Cistanches Herba,又名大芸、甜大芸、苁蓉、甜苁蓉、地精等,为我国传统名贵中药材,具有补肾阳,益精血,润肠通便的功效,主治阳痿、不孕、腰膝酸软、筋骨无力、肠燥便秘等[1]。其应用始载于《神农本草经》,被列为上品药材[2]。肉苁蓉在历代增力中药处方中出现率最高,同时在抗衰老类方剂中的出现率仅次于人参,居于第2位,故其具有“沙漠人参”的美誉[3-4]。
DNA条形码(DNA barcoding)是选用标准短的DNA片段对物种进行快速、准确的自动化的鉴定和识别[5],由Paul Hebert首先提倡将条形码技术引进生物的鉴定中[6]。中药鉴定是研究中药品种、质量,制定中药标准,寻找和扩大药源的前提和基础,DNA分子标记可以弥补和克服传统鉴定方法的一些缺陷和难题[7]。DNA条形码技术是近年来生物分类和鉴定的研究热点,在物种鉴定方面显示了广阔的应用前景[7-10]。
高分辨率熔解(high resolution melting,HRM)曲线技术是美国Utah大学Wittwer实验室在2003年首次提出的,基于新型饱和荧光染料LC Green的发明而进行基因突变检测的新技术[11-12]。该技术无需使用序列特异性探针,不受突变碱基位点和种类的局限,具有高灵敏度、特异性,简单方便,成本低,高通量,闭管操作等特点[13],是目前广泛应用于突变扫描和基因型分析和甲基化分析等的热点技术[14-16]。王志科等[17]采用HRM技术,应用ITS2序列对人参和西洋参药材进行快速鉴别;胡凯等[18]利用psbA-trnH序列设计引物,通过HRM曲线能够区分不同的金银花和山银花药材品种,并通过Bar-HRM联用技术,能在金银花类药材混合物中检测出低至5%的山银花样品;陈康等[19]将HRM技术应用于鹿茸真伪鉴别,建立了正品鹿茸药材熔解曲线模型;胡峻等[20]将HRM技术应用于木通药材的鉴别,并建立了正品木通药材的熔解曲线模型;蒋超等[21]将限制性内切酶引入熔解曲线分析中,建立了一种新的SNP分型方法,并分别以金银花/山银花、白术/苍术叶绿体序列SNP位点为例,检测了位于或不位于内切酶识别序列区的SNP位点分型能力。
本实验收集肉苁蓉和管花肉苁蓉的栽培品和野生品,以及产自乌兹别克斯坦的与肉苁蓉性状比较相近的野生种,采用DNA条形码技术,选取ITS和ITS2两对引物进行鉴别,并建立3种肉苁蓉(肉苁蓉、管花肉苁蓉及采自乌兹别克斯坦的肉苁蓉相近种)的HRM鉴定方法。
1 材料和方法 1.1 材料AB135-S分析天平(Mettler公司),Milli-Q Biocel纯水仪(Millipore公司),MM400球磨仪(Retsch公司),HH-4数显恒温水浴锅(友联仪器公司),ABI Veriti PCR仪(ThernoFisher公司),EPS-301电泳仪(Amersham公司),GelDoc XR+全自动凝胶成像系统(BIORAD公司),MyGo Pro荧光定量PCR仪(北方仪涛公司)。
DP305植物基因组DNA提取试剂盒(天根生化科技有限公司);2×Taq Master Mix缓冲液(货号GK8006,GENEray公司);Type-it HRM PCR Kit(400)(货号206544,Qiagen公司);GelRed(货号41003,Biotium公司);琼脂糖(Biowest公司);Tris-base(Sigma公司);冰醋酸(分析纯,国药集团化学试剂有限公司);EDTANa2·2H2O(国药集团化学试剂有限公司)。引物ITS4:5′-TCCTCCGCTTATTGATAT GC-3′,ITS5:5′-GGAAGTAAAAGTCGTAACAAGG-3′(ITS);引物ITS2F:5′-ATGCGATACTTGGTGTGAAT-3′,ITS3R:5′-GACGCTTCTCCAGACTACAAT-3′(上海捷瑞生物工程有限公司)。
从管花肉苁蓉主产区和田地区采集管花肉苁蓉Cistanche tubulosa样品19批,其中包括野生品6批和栽培品13批;从肉苁蓉主产区内蒙古自治区西部地区采集肉苁蓉Cistanche deserticola样品30批,其中包括野生品15批和栽培品15批;从乌兹别克斯坦境内采集肉苁蓉属植物19批,均为野生品。采集样品均由郑健研究员鉴定。《中国植物志》中肉苁蓉属共有6个种,分别为蒙古肉苁蓉Cistanche mongolica、肉苁蓉Cistanche deserticola、盐生肉苁蓉Cistanche salsa、管花肉苁蓉Cistanche tubulosa、兰州肉苁蓉Cistanche lanzho-uensis、沙苁蓉Cistanche sinensis。
1.2 植物总DNA的提取取样品20~30 mg,用MM400球磨仪粉碎,利用天根植物基因组提取试剂盒进行DNA提取。
1.3 聚合酶链式反应及测序PCR反应体系20 µL:2×Taq Master Mix缓冲液10 µL,上下游引物各0.4 µL,DNA模板1 µL,灭菌的双蒸水8.2 µL。PCR反应条件:95 ℃预变性4 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,循环35次;72 ℃延伸7 min。琼脂糖凝胶电泳检测,凝胶成像。将电泳结果显示为单一明亮条带的扩增产物送英潍捷基(上海)贸易有限公司进行测序。
1.4 数据处理测序峰图使用CodonCode Aligner 3.7.1软件(CodonCode Co.,USA)校对拼接,去除引物区段。将所有序列利用MEGA 5.0软件(molecular evolutionary genetics analysis)分析比对。采用NJ树(Neighbor-Joining tree)法建立系统发育树,通过bootstrap(1 000次重复)对各分支进行支持率检验。
1.5 引物设计根据ITS2引物(ITS2F:5′-ATG CGATACTTGGTGTGAAT-3′,ITS3R:5′-GACGCTTCT CCAGACTACAAT-3′)扩增得到的序列,利用Primer Premier 5软件设计引物。
1.6 HRM实验HRM PCR反应体系10 µL:2×HRM PCR Master Mix 5 µL,上下游引物各0.7 µL,DNA模板0.5 µL,灭菌的双蒸水3.1 µL。HRM PCR反应条件:95 ℃预变性5 min;95 ℃变性10 s,55 ℃退火30 s,72℃延伸1 s,循环40次;HRM熔解升温条件:65~95 ℃,0.1 ℃·s-1升温。
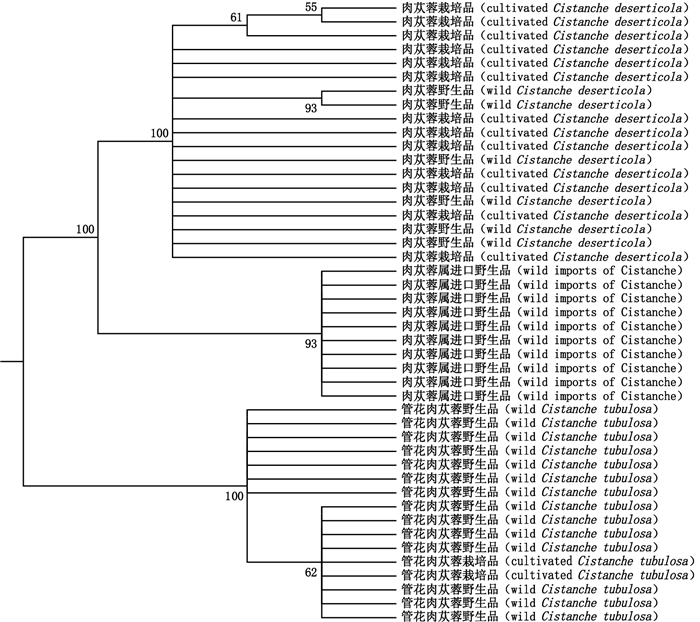
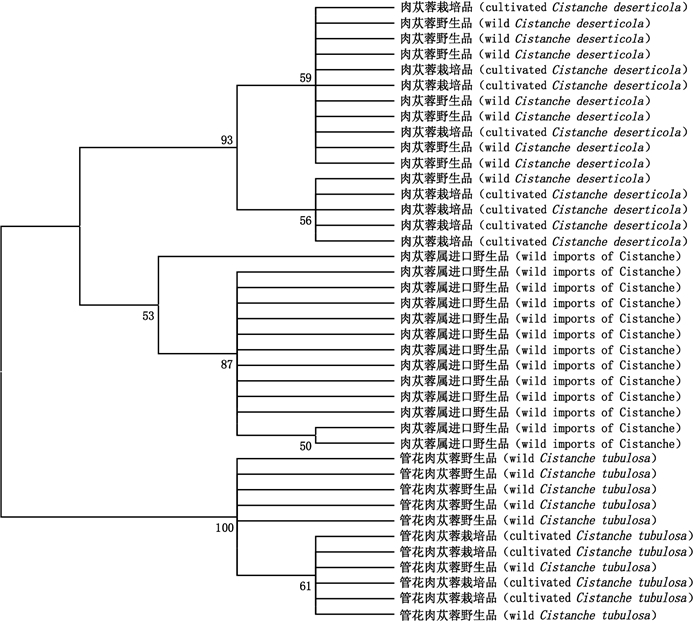
2 结果 2.1 NJ树聚类分析分别基于ITS和ITS2序列,利用MEGA 6.06软件建立肉苁蓉样品的NJ树图(图 1、2),结果表明不同种样品的ITS和ITS2序列都能分别聚到一支,且支持率达到50%以上。肉苁蓉和管花肉苁蓉的栽培品与野生品从ITS和ITS2序列上看,一致性高,没有明显差异,图 1和图 2中的肉苁蓉属进口野生品为采自乌兹别克斯坦境内的野生品,根据其性状初步鉴定为疑似肉苁蓉品种,但经DNA条形码鉴定,差异较大,怀疑为其他相近种。图 2中Cistanche deserticola和Cistanche tubulosa 2条序列分别为从NCBI上下载的肉苁蓉和管花肉苁蓉的ITS2序列,序列Cistanche deserticola与采集的肉苁蓉样品聚为一支,序列Cistanche tubulosa与采集的管花肉苁蓉样品聚为一支,也验证了采集样品的准确性。
|
Bootstap 1 000次重复,支上数值仅显示自展支持率≥50%[The bootstrap scores(1 000 replicates)are shown(≥50%)for each branch] 图 1 基于ITS序列建立的NJ树 Figure 1 Phylogenetic tree of the ITS sequences with NJ method |
|
Bootstap 1 000次重复,支上数值仅显示自展支持率≥50%[The bootstrap scores(1 000 replicates)are shown(≥50%)for each branch] 图 2 基于ITS2序列建立的NJ树 Figure 2 Phylogenetic tree of the ITS2 sequences with NJ method |
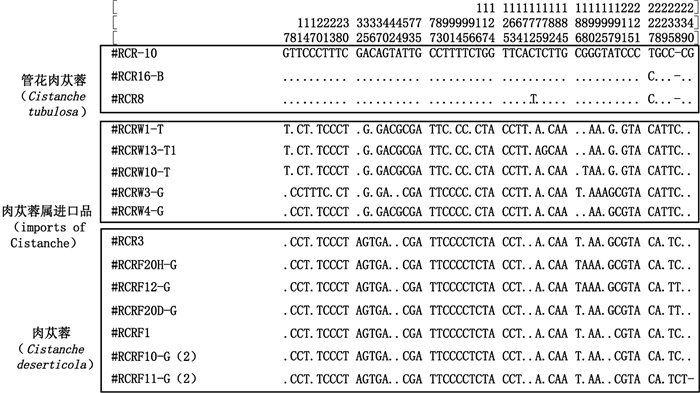
基于ITS2序列利用MEGA 6.06软件分析肉苁蓉样品的单倍型,结果如图 3所示,分别得到管花肉苁蓉3个单倍型,肉苁蓉属进口品5个单倍型,肉苁蓉7个单倍型。根据3种肉苁蓉样品的不同单倍型信息,利用Primer Premier 5软件设计HRM实验引物。
|
图 3 基于ITS2序列的单倍型分析 Figure 3 The haplotype analysis based on ITS2 sequences |
将3种肉苁蓉的不同单倍型的序列分别输入到Primer Premier 5软件中,综合考虑所得引物序列的评分和适用性,共筛选出8对引物,由捷瑞公司合成引物。引物详细信息如下:
Ysr-1(Ysr-1A:5′-TATGCAACAATGTCTACCGT-3′;
Ysr-S:5′-TATGCAACAATGTCTACCGT-3′)
Ysr-2(Ysr-2A:5′-CTGGTCGTGACGTGAGTC-3′;
Ysr-S:5′-TATGCAACAATGTCTACCGT-3′)
Ghr-1(Ghr-13A:5′-ATCGCACGATTGGGTATG-3′;
Ghr-1S:5′-CCCTCCTCTGTCCTTCTGG-3′)
Ghr-2(Ghr-2A:5′-AGATCGCACGATTGGGTA-3′;
Ghr-23S:5′-TCTGGGACAATGCTTAGGT-3′)
Ghr-3(Ghr-13A:5′-ATCGCACGATTGGGTATG-3′;
Ghr-23S:5′-TCTGGGACAATGCTTAGGT-3′)
Hmr-1(Hmr-1A:5′-ACGCCATACGTCACAACA-3′;
Hmr-S:5′-TTTAGGGTGATACTTAGGTGG-3′)
Hmr-2(Hmr-2A:5′-ATACGTCACAACAGCACGA-3′;
Hmr-S:5′-TTTAGGGTGATACTTAGGTGG-3′)
Hmr-3(Hmr-3A:5′-CGTCACAACAGCACGAGA-3′;
Hmr-S:5′-TTTAGGGTGATACTTAGGTGG-3′)
2.4 引物筛选对以上8对引物分别进行HRM预实验,根据预实验结果,对引物进行筛选,最终确定的引物序列如下:
Ysr-2(Ysr-2A:5′-CTGGTCGTGACGTGAGTC-3′;
Ysr-S:5′-TATGCAACAATGTCTACCGT-3′)
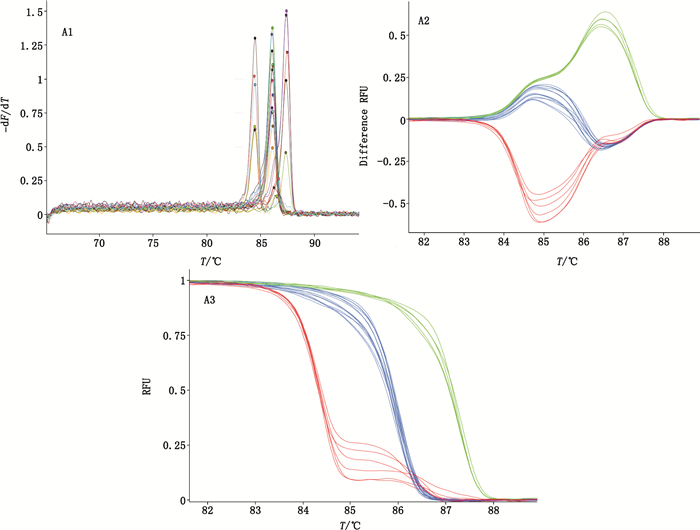
2.5 HRM实验结果利用最终确定的引物进行HRM实验,得到的结果如图 4-A1、A2、A3所示,A1、A2、A3分别为HRM实验的熔解解峰图、差异化曲线和归一化曲线。图 4-A1中存在3组熔解度曲线,表明3种肉苁蓉的熔解温度(melting tempreture,Tm)存在差异,且可区分;图 4-A2、A3中,红色曲线为管花肉苁蓉,蓝色为肉苁蓉,绿色为肉苁蓉属进口品,3组曲线的形态和位置存在差异,且聚类明显,可有效区分不同种。
|
A1.HRM峰图(peaks of HRM)A2.HRM的差异化曲线(difference of HRM)A3.HRM的归一化曲线(normalized of HRM) 图 4 HRM实验结果 Figure 4 The results of HRM test |
本研究利用通用引物ITS和ITS2对肉苁蓉、管花肉苁蓉及采自乌兹别克斯坦的肉苁蓉相近种这3种肉苁蓉进行了DNA条形码的研究,结果显示,ITS和ITS2两段序列均能将3种肉苁蓉有效区分。相同种肉苁蓉的野生品和栽培品在DNA条形码序列上不存在明显差异,因此判定采自乌兹别克斯坦与肉苁蓉性状相近的野生品为肉苁蓉相近种,而非肉苁蓉,排除了野生品与栽培品DNA条形码存在差异而导致其NJ树聚类分析中不能有效聚类的可能性。在DNA条形码结果的基础上,针对3种肉苁蓉样品的ITS2序列设计引物,并通过HRM实验对所设计引物进行筛选,通过判断不同引物扩增的不同样品间Tm值的差异程度,以及熔解曲线形态和位置的变化,最终确定Ysr-2(Ysr-2A:5′-CTGGTCGTGACGTGAGTC-3′;Ysr-S:5′-TATGCAACAATGTCTACCGT-3′)为最佳引物。利用引物Ysr-2对3种肉苁蓉的ITS2区域进行巢式实时扩增,然后进行熔解程序。熔解曲线分析结果显示,3种肉苁蓉样品可以通过HRM实验彼此区分,不同种样品的Tm值不同,且熔解曲线形态和位置存在明显差异,可以区分不同种,结果与DNA条形码一致。
HRM实验对实时荧光PCR仪的灵敏度要求较高,因为HRM实验的要求是能比较出不同序列间SNP(单核苷酸多态性)的差异,较高的灵敏度能保证仪器能区别差异较小序列间的Tm变化。HRM实验中选用的荧光染料为饱和染料,因为不饱和染料在DNA熔解过程中染料分子会跑到未熔解的区域,导致熔解曲线的温度变化很小或不均一,而使用饱和染料时,熔解区域的染料分子不能跑到未熔解部分,生成能准确反映DNA序列的熔解图像,且选用的饱和染料对PCR的抑制要低,才能保证实验的正常进行。另外为保证HRM实验的正常扩增,模板的浓度不能太低;优化合适的退火温度以保证扩增的特异性和高效率;合适的引物浓度为0.1~0.3 μmol·L-1,低引物浓度可以有效避免引物二聚体和非特异性扩增。
近年来,中药鉴定技术发展迅速,为药材的基原物种鉴定奠定了良好的基础。DNA条形码技术作为物种鉴定的新手段,在传统形态分类学对物种准确鉴定的基础上,通过对样品标准序列进行测序,建立DNA条形码数据库,可以实现物种鉴定的标准化。
Ririe及Wittwer等的研究[15-16]表明,DNA片段的GC含量(在DNA的4种碱基中,鸟嘌呤和胞嘧啶所占的比率)不同,其解链温度将会有区别,导致熔解曲线出现差异;片段GC含量越高,Tm值越大,熔解曲线越靠后。HRM曲线技术融合了PCR高灵敏性、DNA杂交的高特异性和光谱技术的高精确定量等优点,直接探测PCR过程中荧光信号的变化,以获得特定区段扩增产物定量的结果。
通过建立肉苁蓉的HRM检测方法,可以快速、高效地完成肉苁蓉品种的鉴定工作,且结果明显,同时也可以为其他中药民族药品种的HRM技术鉴定提供可参考的依据。
| [1] |
李振华, 龙平, 邹德志, 等. 基于ITS序列位点特异性PCR的肉苁蓉与其混伪品的鉴别研究[J]. 中国中药杂志, 2014, 39(19): 3684. LI ZH, LONG P, ZOU DZ, et al. Study on identification of Cistanches Herba and its adulterants by PCR amplification of specific alleles based on ITS sequences[J]. China J Chin Mater Med, 2014, 39(19): 3684. |
| [2] |
屠鹏飞, 何燕萍, 楼之岑. 肉苁蓉的本草考证[J]. 中国中药杂志, 1994, 19(1): 3. TU PF, HE YP, LOU ZC. Herbalogical studies on Roucongrong(Herba Cistanches)[J]. China J Chin Mater Med, 1994, 19(1): 3. |
| [3] |
郑荣, 张立国, 倪力军. 一种新的肉苁蓉甙类化合物的分离方法[J]. 天然产物研究与开发, 2001, 13(4): 55. ZHENG R, ZHANG LG, NI LJ. A new separation method of phenylpropanoid glycosides for Cistanche deserticola[J]. Nat Prod Res Dev, 2001, 13(4): 55. |
| [4] |
中国植物志编委会. 中国植物志.第69卷.第2分册[M]. 北京: 科学出版社, 1989, 83. Editorial Committee of Flora of China. Flora of China.69(2)[M]. Beijing: Science Press, 1989, 83. |
| [5] |
郭慧, 王谦博, 贾力维, 等. 中药材DNA条形码技术研究进展[J]. 中国药师, 2016, 19(3): 566. GUO H, WANG QB, JIA LW, et al. Study progress in DNA barcode of traditional Chinese medicine[J]. China Pharm, 2016, 19(3): 566. |
| [6] |
HEBERT PD, CYWINSKA A, BALL SL. Biological identifications through DNA barcodes[J]. Proc Biol Sci, 2003, 270(1512): 313. DOI:10.1098/rspb.2002.2218 |
| [7] |
陈士林, 庞晓慧, 姚辉, 等. 中药DNA条形码鉴定体系及研究方向[J]. 世界科学技术-中医药现代化, 2011, 13(5): 747. CHEN SL, PANG XH, YAO H, et al. Identification system and perspective of DNA barcoding traditional Chinese materia medical[J]. World Sci Technol Mod Tradit Chin Med Mater Med, 2011, 13(5): 747. |
| [8] |
陈士林, 姚辉, 宋经元, 等. 基于DNA barcoding(条形码)技术的中药材鉴定[J]. 世界科学技术-中医药现代化, 2007, 9(3): 7. CHEN SL, YAO H, SONG JY, et al. Use of DNA barcoding to identify Chinese medicinal materials[J]. Worl Sci Technol Mod Tradit Chin Med Mater Med, 2007, 9(3): 7. |
| [9] |
LI DZ, LIU JQ, CHEN ZD, et al. Plant DNA barcoding in China[J]. J Syste Evol, 2011, 49(3): 165. DOI:10.1111/jse.2011.49.issue-3 |
| [10] |
LI M, CAO H, BUT PPH, et al. Identification of herbal medicinal materials using DNA barcodes[J]. J Syst Evol, 2011, 49(3): 271. DOI:10.1111/jse.2011.49.issue-3 |
| [11] |
陈康, 蒋超, 袁媛, 等. 高分辨率熔解曲线技术及其在中药DNA分子鉴定中的应用[J]. 药学学报, 2015, 50(12): 1581. CHEN K, JIANG C, YUAN Y, et al. High resolution melting and its application to identity study of traditional Chinese medicine[J]. Acta Pharm Sin, 2015, 50(12): 1581. |
| [12] |
WITTWER CT, REED GH, GUNDRY CN, et al. High resolution genotyping by amplicon melting analysis using LCGreen[J]. Clin Chem, 2003, 49(6 Pt 1): 853. |
| [13] |
GUNDRY CN, VANDERSTEEN JG, REED GH, et al. Amplicon melting analysis with labeled primers:a closed-tube method for differentiating homozygotes and heterozygotes[J]. Clin Chem, 2003, 49(3): 396. DOI:10.1373/49.3.396 |
| [14] |
王英红, 孔庆鑫, 沈林海, 等. 用高分辨率熔解曲线做PCR条件优化[J]. 中国卫生检验杂志, 2012, 22(11): 2539. WANG YH, KONG QX, SHEN LH, et al. High resolution melting analysis in optimization of PCR condition[J]. Chin J Health Lab Technol, 2012, 22(11): 2539. |
| [15] |
李旦, 王加启, 卜登攀, 等. 高分辨率熔解曲线及其应用[J]. 生物技术通报, 2009(7): 48. LI D, WANG JQ, BU DP, et al. Study of high resolution melting and application[J]. Biotechnol Bull, 2009(7): 48. |
| [16] |
李东至. 高分辨率熔解曲线分析:一种新的用于遗传病基因突变筛查的技术[J]. 中国产前诊断杂志(电子版), 2011, 3(2): 1. LI DZ. High resolution melting analysis:a new technology to screen the genetic mutations[J]. Chin J Prenat Diagn(Electron Ver), 2011, 3(2): 1. |
| [17] |
王志科, 鲁放, 熊超, 等. Bar-HRM技术在人参和西洋参药材鉴定中的应用研究[J]. 世界科学技术-中医药现代化, 2016, 18(2): 191. WANG ZK, LU F, XIONG C, et al. Application of Bar-HRM technology in identification of medicinal material of Panax ginseng and Panax quinquefolium[J]. World Sci Technol Mod Tradit Chin Med Mater Med, 2016, 18(2): 191. |
| [18] |
胡凯, 王微, 游玉明. 基于DNA条形码和HRM技术对金银花及山银花药材的快速鉴别[J]. 时珍国医国药, 2015, 26(12): 2923. HU K, WANG W, YOU YM. The rapid identification of Lonicera japonica and Lonicera hypoglauca based on DNA barcodes and HRM technology[J]. Lishizhen Med Mater Med Res, 2015, 26(12): 2923. |
| [19] |
陈康, 蒋超, 袁媛, 等. 基于熔解曲线分析技术的鹿茸药材分子鉴别[J]. 中国中药杂志, 2015, 40(4): 619. CHEN K, JIANG C, YUAN Y, et al. Molecular identification of hairy antler by analysis of high resolution melting[J]. China J Chin Mater Med, 2015, 40(4): 619. |
| [20] |
胡峻, 詹志来, 袁媛, 等. 基于熔解曲线分析技术的木通类药材的分子鉴别[J]. 中国中药杂志, 2015, 40(12): 2304. HU J, ZHAN ZL, YUAN Y, et al. HRM identification of Chinese medicinal materials Mutong[J]. China J Chin Mater Med, 2015, 40(12): 2304. |
| [21] |
蒋超, 黄璐琦, 袁媛, 等. 酶切-熔解曲线分析:一种新的SNP分型方法及其在中药材鉴定中的应用[J]. 药学学报, 2014, 49(4): 558. JIANG C, HUANG LQ, YUAN Y, et al. Restriction endonuclease digest-melting curve analysis:a new SNP genotyping and its application to traditional Chinese medicine authentication[J]. Acta Pharm Sin, 2014, 49(4): 558. |
 2018, Vol. 38
2018, Vol. 38 
