2. 北京天坛生物制品有限公司, 北京 100176
2. Beijing Tiantan Biological Products Co., Ltd., Beijing 100176, China
Sabin株脊髓灰质炎减毒活疫苗(oral poliovirus vaccine,OPV)在我国消灭脊灰运动中发挥了巨大的作用。在疫苗生产过程中,尤其是病毒的增殖过程中,部分病毒发生突变,可导致其神经毒力升高,因此在疫苗的质控中,采用猴体神经毒力试验检测疫苗原液,以控制其神经毒力。但由于猴子来源困难,价格昂贵,技术操作也有一定难度,人们期望建立其他方法来代替,或者减少猴子的使用数量。通过对疫苗株及神经毒力回复株的序列比较,人们注意到基因组5′UTR非编码区域特定位点的改变,例如在Ⅲ型中472-U突变为C,Ⅰ型中525-T变为C,Ⅱ型中481-C变为G等与毒力的恢复密切相关,少量病毒发生突变即可影响其毒力。例如,Ⅲ型中472-C突变超过1%的疫苗批次即无法通过猴体神经毒力试验[1]。
基于以上研究,Chumakov等[2-3]于1980年代建立了MAPREC(mutant analysis by PCR and restriction enzyme cleavage)方法,对疫苗原液中涵盖突变位点的片段进行扩增并标记同位素,然后以特定内切酶进行切割,则涵盖突位点的片段可被切割成小片段,以电泳方法分离,对突变进行定量检测。经过国际协作研究,WHO将其列入OPV疫苗规程,作为猴体神经毒力试验前的预筛手段,规定Ⅲ型原液中472-C比例不得高于1% [4]。
鉴于突变靶位含量较低,传统的MAPREC操作中需要同位素标记来进行检测,这虽然增加了方法的敏感性和准确性,但也限制了其广泛应用。本研究在WHO的MAPREC标准操作规程的基础上[5],使用荧光标记代替同位素标记,建立了荧光MAPREC法来分析Ⅲ型Sabin株脊髓灰质病毒突变含量。现报道如下。
1 材料与方法 1.1 标准品和样品MAPREC国际标准品均购自英国国家生物制品检定所(NIBSC)。100% DNA(编号94/790)标准品为100% 472-C的合成单链DNA片段,用以评价实验的酶切效率,单次实验酶切效率不应低于90%。International Standard DNA(IS DNA,编号95/542)标准品为含有0.9% 472-C的合成单链DNA片段,用以模拟接近合格上限的样品。Low Mutant Virus Reference(LMVR,编号96/572)为含有0.7% 472-C的Ⅲ型Sabin株病毒,用以模拟合格的样品。High Mutant Virus Reference(HMVR,编号96/578)为含有1.1% 472-C的Ⅲ型Sabin株病毒,用以模拟不合格的样品。实验用的疫苗生产毒种,单次收获液和疫苗原液均来自北京天坛生物制品有限公司。
1.2 主要试剂与仪器病毒RNA提取试剂盒QIAamp Viral RNA Mini Kit货号52904为QIAGEN公司产品。反转录试剂盒ThermoScript RT-PCR System货号11146-032为Invitrogen公司产品。PCR酶AmpliTaq Gold 360 Master Mix货号4398881为ABI公司产品。DNA内切酶MboⅠ货号R0147L为NEB公司产品。PCR仪DNAEngine PTC-200为伯乐公司产品。电泳槽Hoefer SE 600,激光扫描仪Typhon 9500均为GE公司产品。PCR引物及荧光引物均为Invitrogen公司合成并使用HPLC方式进行纯化,见表 1。
|
|
表 1 PCR引物及荧光引物 Table 1 PCR primers and fluorescence labelled primers |
将不同荧光标记的引物使用TE缓冲液系列稀释为不同浓度,之后与1/3体积的30%甘油混合,取5 μL上样于10%聚丙烯酰胺P凝胶,TBE缓冲液进行电泳,上样量为12.5 ng至1 pg。500 V电泳30 min后,使用激光扫描仪Typhon 9500进行扫描,获取图像。使用Quantity One软件分析条带的荧光信号强度,并对信号强度与引物浓度进行线性回归后计算R2。选取不同稀释度间线性关系良好、灵敏度高的标记物进行进一步实验。
1.4 RNA提取与反转录将国际标准品LMVR与HMVR按照试剂盒说明书进行RNA提取后,按照反转录试剂盒ThermoScript RT-PCR System说明进行cDNA合成,反应条件,25 ℃反应10 min后,50 ℃反应40 min。反转录所得cDNA可以立即使用或在-20 ℃条件下保存1个月。
1.5 cDNA使用浓度的优化分别吸取360 Master Mix 875 μL,pS-3 175 μL(30 μg·mL-1),pA-3 175 μL(3 μg·mL-1),去离子水350 μL混合后作为PCR Mix。PCR反应体系的配制:取cDNA 6.25 μL与56.25 μL PCR Mix混合,之后取12.5 μL至50 μL PCR Mix中进行5倍稀释,共做7个稀释度。按照以下条件进行PCR反应:94 ℃ 10 min后,进行40个循环扩增,94 ℃30 s,55 ℃15 s,68 ℃ 1 min。以生成单链DNA扩增产物。取10 μL扩增产物,加入1 μL 360 Master Mix和1 μL 6 μg·mL-1的pA-3/CY5标记引物,混匀离心后72 ℃反应10 min,进行PCR产物的荧光标记。取6 μL标记产物加入2 μL 30%甘油,按照“1.2”项所述条件进行电泳和扫描分析。选取无剩余引物条带,并且进行125倍稀释后仍有PCR产物条带的cDNA浓度作为适宜浓度。
1.6 突变率的检测100% DNA与IS DNA标准品均使用100 μL去离子水复溶。吸取两DNA标准品与稀释至适宜浓度的LMVR与HMVR cDNA各5 μL,分别与45 μL的PCR Mix混合,按照“1.4”项条件进行PCR反应和荧光标记。吸取6 μL标记后的产物,加入1 μL 2 U·μL-1 DNA内切酶MboⅠ,37 ℃孵育2 h。将进行及未进行酶切的荧光标记产物进行电泳和扫描分析,按照图 1所示分析突变比例。样品的突变率检测同LMVR与HMVR标准品。
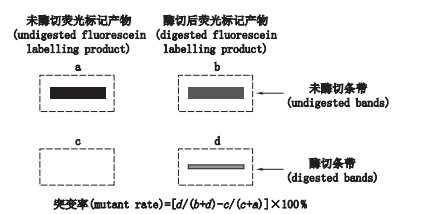
|
a、b、c、d指条带的荧光信号强度值(a、b、c、d indicate the fluorescence density of each band) 图 1 MAPREC实验中突变率的计算 Figure 1 The calculation of mutant contents in MAPREC assay |
将表 1中使用不同荧光标记的引物系列稀释后直接进行电泳分析。结果如表 2所示:不同荧光标记的引物检测限差异很大,其中pA-3/CY5最为灵敏,检测限达到5 pg,而pA-3/Rhodamine Green最不灵敏,检测限为500 pg。引物浓度对荧光强度进行线性回归后计算R2值,除pA-3/FAM外,R2值均大于0.98,线性良好。因此,选取最为灵敏的pA-3/CY5引物作为实验中使用的荧光标记引物。
|
|
表 2 不同荧光标记引物的检测限 Table 2 Detection limit of different fluorescence labelled primers |
对cDNA进行5倍系列稀释作为模板进行扩增,电泳结果如图 2所示,50至53稀释时扩增产物明显,且均无明显剩余引物,对后续试验干扰较小,本研究选择稀释51倍浓度进行后续研究。
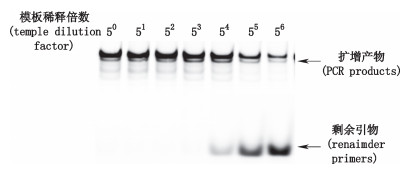
|
图 2 cDNA使用浓度的优化 Figure 2 Optimization of cDNA concentration |
采用建立方法,对4个国际标准品进行检测。如图 3所示,将扩增产物进行酶切后,均能够清楚分离酶切和未酶切条带,突变率计算结果见表 3。4个国际标准品的472-C突变检测值与其标示值基本一致,说明方法的准确性良好。并且,除LMVR标准品不同实验间RSD为10.0%外,其余标准品的RSD均小于10%。说明方法的精密度良好。100% DNA control标准品检测突变率为95.59%,主要是因为酶切效率难以达到100%,符合WHO SOP中该标准品酶切效率不应低于90%的要求[5]。

|
-.未经酶切处理(undigested)+.经过酶切处理(digested) 图 3 国际标准品及样品突变率检测电泳结果 Figure 3 Electrophoresis results for mutant content analysis of WHO international standards and samples |
|
|
表 3 荧光MAPREC法测定国际标准品突变率22次结果汇总 Table 3 Summary of results of 22 independent analysis experiments of WHO international standards with fluorescent MAPREC assay |
对Ⅲ型Sabin株脊髓灰质炎减毒活疫苗生产用的工作种子、疫苗原液和单次收获液进行检测,结果见表 4。所有样品均满足WHO关于突变率小于1%的要求。其中疫苗生产的工作种子突变率最低,为0.64%,经过传代的单次收获液和疫苗原液突变率略有升高,但均低于1%。并且不同的单次收获液和疫苗原液中突变率在0.76%~0.90%之间,较为稳定。
|
|
表 4 荧光MAPREC法测定疫苗样品突变率结果汇总 Table 4 Summary of results of mutant content analysis of vaccine samples with fluorescent MAPREC assay |
传统的MAPREC法采用同位素标记增加检测的灵敏度,以准确检测含量甚微的突变成分。而使用EtBr染色的方法仅能够检测到10%以上的突变[6]。SYBR Green-I染色的方法检测DNA具有高灵敏度低背景的特点[7-8],使用该染色方法能够检测到低至0.3%~0.4%的突变[9]。有学者建立了荧光定量PCR对突变进行检测的方法,以检测粪便和污水样品中病毒毒力回复的情况[10-11],相比传统的MAPREC方法快速简便,但灵敏度仍然较低。飞行时间质谱[12]和基因芯片[13-14]对突变率进行检测具有较高的灵敏度,但繁重的操作量和高昂的实验成本限制了其广泛应用。
本研究采用高灵敏度的荧光标记物代替同位素标记,建立了荧光MAPREC方法。通过对多个不同的荧光标记物进行筛选,发现CY5标记的荧光引物最为灵敏,检测限到低至5 pg。并且,引物量从12.5 ng到5 pg时与荧光信号强度间线性关系良好,R2值大于0.99。之后,以CY5标记引物代替同位素标记引物,成功建立了检测Ⅲ型Sabin株472 -C突变含量的荧光MAPREC方法,并以国际标准品对方法进行了验证。结果显示,4个国际标准品的检测值均与其标示值基本一致,并且,不同实验间RSD小于15%,说明所建立的方法较为稳定。采用建立的方法,对多个批次的OPV正常生产的工作种子,疫苗原液和单次收获液进行了检测,结果显示,所有样品的突变率均满足WHO的要求,与这些批次的猴体神经毒力试验结果一致。
除OPV疫苗外,WHO对Sabin株灭活疫苗也增加了MAPREC方法的要求[15]。但MAPREC方法本身操作较为复杂,加之采用同位素标记,因此,虽然该方法列入WHO规程,但仅有极少数国家能够开展此项检测。本研究建立的方法中采用荧光标记法,可以代替同位素法进行病毒突变比例的检测,从而消除了该方法推广的最大障碍,对该疫苗的质控有重要的意义。
| [1] |
EVANS DM, DUNN G, MINOR PD, et al. Increased neurovirulence associated with a single nucleotide change in a noncoding region of the Sabin type 3 poliovaccine genome[J]. Nature, 1985, 314(6011): 548. DOI:10.1038/314548a0 |
| [2] |
CHUMAKOV K, POWERS L, NOONAN KE, et al. Correlation between amount of virus with altered nucleotide sequence and the monkey test for acceptability of oral poliovirus vaccine[J]. Natl Acad Sci USA, 1991, 88(1): 199. DOI:10.1073/pnas.88.1.199 |
| [3] |
CHUMAKOV K, NORWOOD LP, PARKER M, et al. RNA sequence variants in live poliovirus vaccine and their relation to neurovirulence[J]. J Virol, 1992, 66(2): 966. |
| [4] |
WHO Expert Committee on Biological Standardization, sixty-third report. WHO technical report series; No. 980, Annex 2, Recommendations to Assure the Quality, Safety and Efficacy of Poliomyelitis Vaccines (Oral, Live, Attenuated)[S]. [2017-01-26]. http://www.who.int/biologicals/WHO_TRS_980_WEB.pdf
|
| [5] |
WHO SOP for Mutant Analysis by PCR and Restriction Enzyme Cleavage (MAPREC) for Oral Poliovirus (Sabin) Vaccine Types 1, 2 or 3. Version 5, 2012[S]. [2017-01-26] http://www.who.int/biologicals/vaccines/MAPREC_SOP_Final_09112012.pdf http://www.who.int/biologicals/vaccines/MAPREC_SOP_Final_09112012.pdf?ua=1
|
| [6] |
MARTINEZ CV, OLD MO, KWOCK DK, et al. Shedding of sabin poliovirus Type 3 containing the nucleotide 472 uracil-to-cytosine point mutation after administration of oral poliovirus vaccine[J]. J Infect Dis, 2004, 190(2): 409. DOI:10.1086/jid.2004.190.issue-2 |
| [7] |
MANSFIELD ES, WORLEY JM, MCKENZIE SE, et al. Nucleic acid detection using non-radioactive labelling methods[J]. Mol Cell Probes, 1995, 9(3): 145. DOI:10.1006/mcpr.1995.0023 |
| [8] |
WHITE HW, KUSUKAWA N. Agarose-based system for separation of short tandem repeat loci[J]. Biotechniques, 1997, 22(5): 976. |
| [9] |
HORIE H, TANO Y, DOI Y, et al. Estimation of the neurovirulence of poliovirus by non-radioisotope molecular analysis to quantify genomic changes[J]. Biologicals, 1998, 26(4): 289. DOI:10.1006/biol.1998.0159 |
| [10] |
GNANASHANMUGAM D, FALKOVITZHALPERN MS, DODGE A, et al. Shedding and reversion of oral polio vaccine type 3 in Mexican vaccinees:comparison of mutant analysis by PCR and enzyme cleavage to a real-time PCR assay[J]. J Clin Microbiol, 2007, 45(8): 2419. DOI:10.1128/JCM.02268-06 |
| [11] |
TROY SB, FERREYRAREYES L, HUANG C, et al. Use of a novel real-time PCR assay to detect oral polio vaccine shedding and reversion in stool and sewage samples after a Mexican national immunization day[J]. J Clin Microbiol, 2011, 49(5): 1777. DOI:10.1128/JCM.02524-10 |
| [12] |
AMEXIS G, OETH P, ABEL K, et al. Quantitative mutant analysis of viral quasispecies by chip-based matrix-assisted laser desorption/ionization time-of-flight mass spectrometry[J]. Natl Acad Sci USA, 2001, 98(21): 12097. DOI:10.1073/pnas.211423298 |
| [13] |
PROUDNIKOV D, KIRILLOV E, CHUMAKOV K, et al. Analysis of mutations in oral poliovirus vaccine by hybridization with generic oligonucleotide microchips[J]. Biologicals, 2000, 28(2): 57. DOI:10.1006/biol.1999.0241 |
| [14] |
LAASSRI M, DRAGUNSKY E, ENTERLINE J, et al. Genomic analysis of vaccine-derived poliovirus strains in stool specimens by combination of full-length PCR and oligonucleotide microarray hybridization[J]. J Clin Microbiol, 2005, 43(6): 2886. DOI:10.1128/JCM.43.6.2886-2894.2005 |
| [15] |
WHO Expert Committee on Biological Standardization, sixty-Fifth Report. WHO Technical Report Series; No. 993, Annex 3, Recommendations to Assure the Quality, Safety and Efficacy of Poliomyelitis Vaccines (Inactivated). [S]. [2017-01-26]. http://www.who.int/biologicals/expert_committee/WHO_TRS_993_web_FINAL.pdf?ua=1
|
 2018, Vol. 38
2018, Vol. 38 
