文章信息
- 王茹, 罗永坚, 邱道寿
- WANG Ru, LUO Yongjian, QIU Daoshou
- 二倍体紫苏线粒体基因组特征及其系统发育分析
- Mitochondrial genomic characterization and phylogenetic analysis of Perilla citriodora (Lamiaceae)
- 亚热带农业研究, 2024, 20(2): 83-92
- Subtropical Agriculture Research, 2024, 20(2): 83-92.
- DOI: 10.13321/j.cnki.subtrop.agric.res.2024.02.002
-
文章历史
- 收稿日期: 2024-04-07
2. 湖北民族学院林业与园艺学院, 湖北 恩施 445000
2. School of Forestry and Horticulture, Hubei University for Nationalities, Enshi, Hubei 445000, China
线粒体在真核细胞中通过有氧呼吸产生能量,不但参与调节细胞分化、凋亡、生长和分裂等关键代谢过程[1],而且与胁迫耐受性、植物生长活力以及细胞质雄性不育性等重要性状密切相关[2],是研究真核生物进化、物种鉴定、遗传多样性和育种的重要工具[3]。线粒体基因组相对较小、高度保守,并且密集地包含基因,同时还包含高度可变的非编码区域[4]。目前,对完整的植物线粒体基因组的研究远落后于完整的质体基因组。尽管在NCBI数据库中有近13 000个完整的质体基因组,但只有673个完整的植物线粒体基因组[5]。不同的植物线粒体基因组由于其结构和内容、核苷酸替代速率以及重复序列存在显著差异[6-7], 从而导致线粒体基因组存在环状、分支状和网状等复杂的结构类型,基因组长度也从海榄雌(Avicennia marina)的22 kb到落叶松的11.7 Mb不等,故对植物线粒体基因的组装具有挑战性[8]。因此,大多数植物系统学研究集中在核基因组和质体基因组上,对植物线粒体基因组的完整组装仍然是进化生物学中的一个瓶颈。随着高通量测序技术的发展和下一代系统基因组学的兴起,已开发了许多适用于线粒体基因组测序和组装的软件程序,如GetOrganelle[9]、Mitofiner[10]、GSAT[11]和PMAT[12]等,使线粒体基因组测序和组装更加精确和高效[13],也为深入了解物种的遗传特征及系统发育提供了良好的技术支撑。
二倍体紫苏(Perilla citriodora)是唇形科紫苏属(Perilla)1年生草本植物,具有独特香气,广泛分布于中国和东南亚地区[14]。紫苏是传统的药食两用植物,现已分离出萜类、黄酮类、生物碱、类固醇、喹啉和酚类等400多种化合物,具有抗过敏、抗抑郁、抗氧化和抗菌等药理功能[15-16]。紫苏种子含油量高达45%~55%,且富含不饱和脂肪酸[17];紫苏种子的α-亚麻酸含量最高可达55%~65%[18]。因此,紫苏具有重要的经济和药用价值[19-20]。目前,尚未见有关二倍体紫苏线粒体基因组的报道。本研究通过下载二倍体紫苏的二代和三代基因组测序数据,进一步加以组装和注释,并分析了其基因组的特征、重复序列、RNA编辑、密码子偏好以及系统进化等,以期为了解紫苏的遗传学信息提供参考。
1 材料与方法 1.1 数据获取从NCBI数据库中下载二代Illumina和三代PacBio二倍体紫苏基因组原始测序数据PRJNA431004(https://www.ncbi.nlm.nih.gov/bioproject/PRJNA431004)。
1.2 基因组组装和注释 1.2.1 组装使用默认设置的SMRT(V2.3.0)分析软件过滤测序接头和低质量原始长读长序列(raw long-read sequence),得到干净的长读长序列(clean long-read sequence, CLR)。使用Minimap 2(V2.1)比较软件对CLR与植物线粒体核心基因的参比基因序列数据进行比较。选取片段长度大于50 bp的序列作为候选序列,并以比对基因较多、比对质量较高的候选序列作为种子序列。使用NCBI叶绿体基因组数据对包含叶绿体基因组的序列进行过滤,移除了匹配百分比>90%的序列[21]。采用三代组装软件Canu[22]对获得的三代数据进行校正。然后用Bowtie 2(V2.3.5.1)将第2代数据与校正后的序列进行比对,再用Unicycler(V0.4.8)软件将短读测序数据和校正后的CLR以默认参数拼接在一起,最终得到平均深度为181.87×的环状紫苏线粒体基因组。
1.2.2 注释使用Blast对比紫苏,且与已发布的植物线粒体编码蛋白质和rRNA序列比对,然后根据相近物种进行手动调整。采用默认设置(http://lowelab.ucsc.edu/tRNAscan-SE/),使用tRNAscan-SE注释tRNA[23]。使用Open Reading Frame Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)注释ORFs。使用在线软件Draw Organelle Genome Maps[24],将线粒体基因组的环状地图可视化。
1.3 重复序列分析使用在线软件MISA(https://webblast.ipk-gatersleben.de/misa/)[25]对紫苏线粒体基因组进行了简单序列重复(simple sequence repeat, SSR)分析。使用在线工具Tandem Repeats Finder V4.09(http://tandem.bu.edu/trf/trf.submit.options.html)[26]检测长度>6 bp且重复单元匹配度超过95%的串联重复序列,参数设置为:2 7 7 80 10 50 2000 -f -d -m。使用Blastn(V2.10.1)检测分散重复序列,参数设置为:序列长度为7,E值为1e-5,去除冗余,去除串联重复。使用Circos(V0.69-5)(http://circos.ca/software/download/)[27]可视化以上重复序列。
1.4 叶绿体基因组同源序列分析从NCBI数据库下载紫苏(https://ngdc.cncb.ac.cn/cgir/genome?term=KT220684)的叶绿体基因组序列。利用NCBI上可用的BLAST软件识别线粒体和叶绿体基因组之间的同源片段,筛选标准设置为:匹配率≥70%、E值≤1e-5且长度≥30 bp,使用Circos(V0.69-5)[27]将筛选到的序列片段可视化。
1.5 密码子偏好性和选择压力分析使用生信云在线云平台(http://112.86.217.82:9919/#/)分析二倍体紫苏线粒体基因组的密码子组成和每个基因的相对同义密码子使用率(relative synonymous codon usage, RSCU)。将匍匐筋骨草(Ajuga reptans)、薰衣草(Lavandula angustifolia)、凉粉草(Platostoma chinense)、广藿香(Pogostemon heyneanus)、夏枯草(Prunella vulgaris)、三对节(Rotheca serrata)、半支莲(Scutellaria barbata)、犁头草(Scutellaria franchetiana)和蔓荆(Vitex trifolia)等9个唇形科物种的GenBank文件导入云工具Ka/Ks(异义替换/同义替换),获取32个保守基因的Ka/Ks比值。利用R编程语言对该32个蛋白质编码基因的Ka/Ks进行单因素方差分析。
1.6 系统发育分析从NCBI下载的线粒体基因组来自于50个物种,包括35个双子叶植物(含有9个唇形科物种)、13个单子叶植物、1个裸子植物和1个苔藓植物;并以苔藓植物粗裂地钱(Marchantia paleacea)作为外类群。利用PhyloSuite(V1.2.1)[28]识别并提取每个物种的23个保守基因(nad1、nad2、nad3、nad4、nad4L、nad5、nad6、nad7、nad9、ccmB、ccmC、cob、cox1、cox2、cox3、atp1、atp4、atp6、atp8、atp9、rps3、rps4、rps12)的基因序列。使用MAFFT(V7.450)[29]对保守基因序列进行比对,并将比对序列连接起来用于构建系统发育树。使用ModelFind在默认参数下构建最佳模型,并在RaxML(V8.2.4)[30]中进行具有1 000次Bootstrap重复的最大似然分析(ML)。使用MrBayes(V3.2.6)[31]进行贝叶斯推理(BI)分析,采用马尔科夫链蒙特卡罗方法进行200 000代的迭代,并每100代对树进行1次采样,舍弃前20%的样本树,剩余的进化树用来生成一个共识树。
2 结果与分析 2.1 紫苏线粒体基因组特征通过Denovo组装得到长度为301 156 bp的完整环状二倍体紫苏线粒体基因组(图 1),组装结果已上传至CNGB数据库。紫苏线粒体基因组的核苷酸组成为:A占27.3%,T占27.5%,G占22.5%,C占22.7%,GC含量为45.2%。从表 1可见,二倍体紫苏线粒体包含59个基因,其中蛋白质编码基因(PCGs)37个、tRNA基因20个和rRNA基因2个。
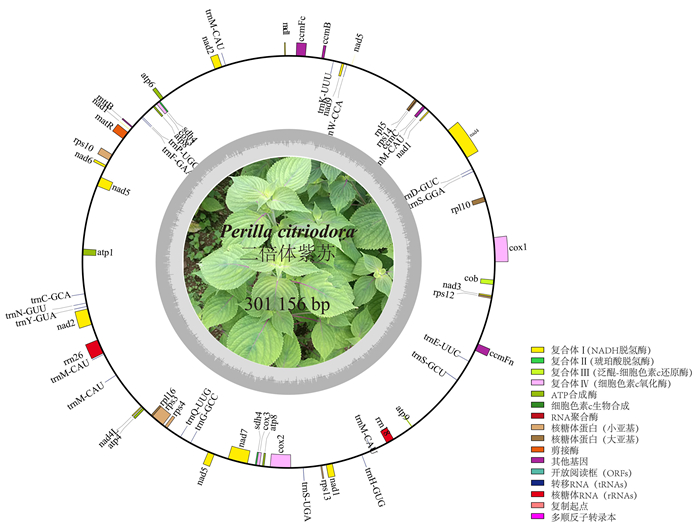
|
图 1 二倍体紫苏的线粒体基因组结构 Figure 1 Perilla citriodora mitochondrial genome structure |
| 基因组 | 基因 |
| ATP合成酶 | atp1, atp4, atp6, atp8(2), atp9 |
| 细胞色素c的生物合成 | ccmB, ccmC, ccmFc*, ccmFn |
| 乌比西诺细胞色素c还原酶 | cob |
| 细胞色素c氧化酶 | cox1*, cox2*, cox3(2) |
| 成熟酶 | matR |
| 运输膜蛋白 | mttB |
| NADH脱氢酶 | nad1****, nad2****, nad3, nad4***, nad4L, nad5****, nad6, nad7***, nad9 |
| 核糖体大亚基(LSR) | #rpl16, rpl10, rpl5 |
| 核糖体小亚基(SSR) | rps10*, rps12, rps13, rps14, rps3*, rps4 |
| 琥珀酸脱氢酶 | #sdh4(2) |
| rRNA | rrn18, rrn26 |
| tRNA | trnC-GCA, trnD-GUC, trnE-UUC, trnF-GAA, trnG-GCC, trnH-GUG, trnK-UUU, trnM-CAU (5), trnN-GUU, trnP-UGG, trnQ-UUG, trnS-GCU, trnS-GGA, trnS-UGA, trnW-CCA, trnY-GUA |
| 1) *表示内含子数; #表示伪基因; 基因后括号内的数字表示多拷贝基因的拷贝数。 | |
在二倍体紫苏线粒体基因组中, 鉴定了正向和回文2种类型的分散重复, 总共获得180个长度≥30个碱基对的分散重复。未检测到正向和互补的重复序列。这些分散重复序列广泛分布在整个基因组的基因间区域,通过Circos软件包加以可视化显示(图 2A)。180个重复序列总长度为21 528 bp,占总线粒体基因组长度的7.14%。在这些重复序列中,有87个反向重复和93个回文重复(图 2B),30~39 bp的重复最常见。在线粒体基因组中共鉴定出78个SSRs,四核苷酸重复占主导地位,占总数的35%(27个),其次是二核苷酸重复(20个)、单核苷酸重复(11个)、三核苷酸重复(9个)、五核苷酸重复(6)和六核苷酸重复(5个)。在单核苷酸SSRs中,A重复占比最高(90.91%);在二核苷酸重复中,AG碱基的重复比例最高(65.00%)(图 2C、2D)。串联重复(tandem repeats)也称为卫星DNA,其特点是重复长度为1~200碱基对,重复次数各不相同[32]。此外,本研究还鉴定到6个长度在34~46个碱基对之间的串联重复序列,这些串联重复序列都位于基因间区域。
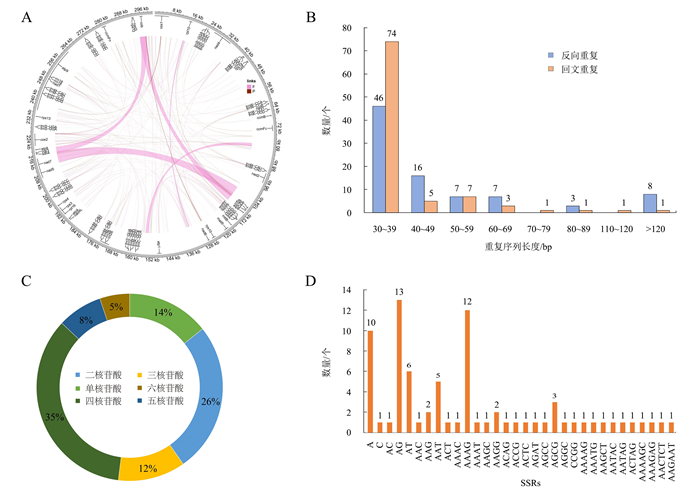
|
A.重复序列在基因组上的分布;B.分散重复的长度分布;C.SSR分析;D.不同长度重复序列的统计。 图 2 二倍体紫苏线粒体基因组的重复序列 Figure 2 Mitochondrial genomic repeat sequences of Perilla citriodora |
二倍体紫苏的线粒体基因组大小约为叶绿体基因组(152 593 bp)的1.96倍。与叶绿体相比,线粒体基因的分布相对稀疏(图 3)。基于叶绿体和线粒体基因组之间的序列相似性,本研究鉴定到70个叶绿体基因片段转移到线粒体基因组中。这些转移的片段总长度为12 680 bp,占整个线粒体基因组的4.23%。其中,线粒体基因组包括6个基因片段、26个基因间隔区域、20个rRNA基因和18个tRNA。从图 3还可见,最长序列为psbA基因片段(877 bp),从叶绿体基因组转移到了线粒体nad2和atp6的基因间区;而第二长的序列rrn基因(875 bp),则转移到了线粒体基因rrn18的基因片段和非编码区。总体上,二倍体紫苏叶绿体基因组将22个rrn16S和rrn23S序列插入到线粒体基因组中,除了4个片段完全转移到IGS区域,其中大部分转移到rrn18或rrn26基因上。
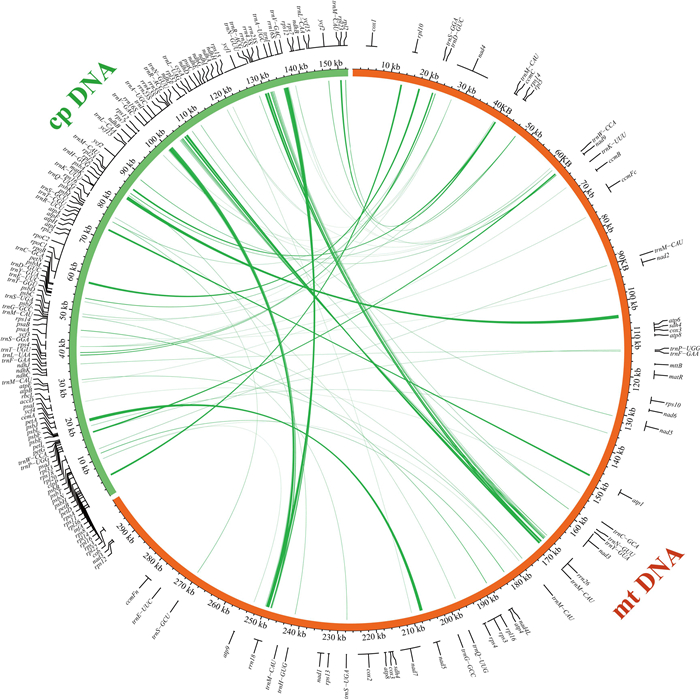
|
图 3 二倍体紫苏从叶绿体转移到线粒体的片段 Figure 3 Fragments transferred from chloroplasts to mitochondria in the Perilla citriodora genome |
二倍体紫苏基因组34个PCGs总长度为30 465 bp,包含了10 155个密码子。所有PCGs均使用AUG作为起始密码子。终止密码子UAA、UGA和UAG的利用率分别为46.88%、28.12%和25.00%,而终止密码子TAG未被使用。相对同义密码子使用(relative synonymous codon usage, RSCU)可以消除氨基酸组成对密码子使用的影响,直接反映密码子使用模式的差异。RSCU值=1,表示密码子使用率无偏;RSCU值>1, 使用频率较高; RSCU值< 1, 使用频率则较低。通过RSCU方法分析表明, 编码丙氨酸(Ala)的GCU密码子出现频率最高,平均RSCU值为1.61;RSCU>1的密码子有29个且大多以A或U结尾(图 4)。
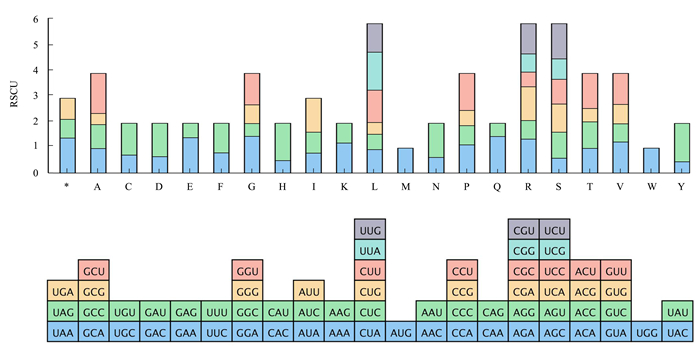
|
图 4 二倍体紫苏基因组中相对同义密码子的使用 Figure 4 Analysis of relative synonymous codon usage (RSCU) in the Perilla citriodora mitogenome |
对9个唇形科植物的选择压力进行分析发现,32个同源蛋白的平均Ka/Ks值为0.33,大多数基因表现出负选择效应,表明二倍体紫苏线粒体基因组中的大多数蛋白编码基因在进化中高度保守(图 5)。值得注意的是,ccmFn、mttB、rps10和matR基因的Ka/Ks>1,表明这些基因在进化中可能受到正选择的影响。
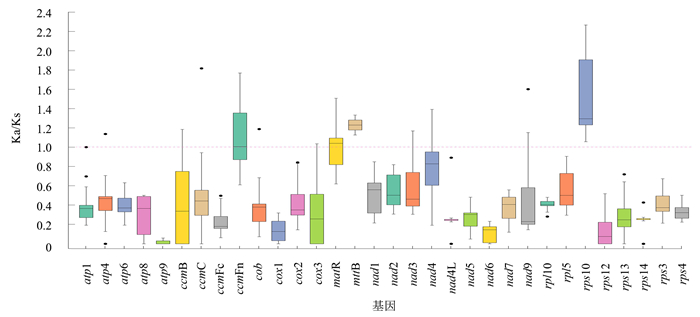
|
图 5 二倍体紫苏与其他9种植物的Ka/Ks比值箱形图 Figure 5 Boxplots of the Ka/Ks ratio of Perilla citriodora and 9 other plant species |
利用保守的线粒体蛋白编码基因重建系统发育树有助于确定绿色植物的分子进化关系[33]。基于50种典型植物线粒体基因组中的23个保守基因对二倍体紫苏进行了系统发育分析(图 6)。使用最大似然法(ML)和贝叶斯推理(BI)的系统发育重建结果表明,两种方法产生的树拓扑几乎相同,每个分支的贝叶斯后验概率(PP)和最大似然bootstrap支持值(BS)都很高且差异较小。系统发育树有力地支持了双子叶植物与单子叶植物的划分以及被子植物与裸子植物的分离,这些物种的系统发育关系与传统分类相一致;同时表明,二倍体紫苏在遗传距离上与丹参(Salvia miltiorrhiza)和Platostoma chinense最为密切相关。
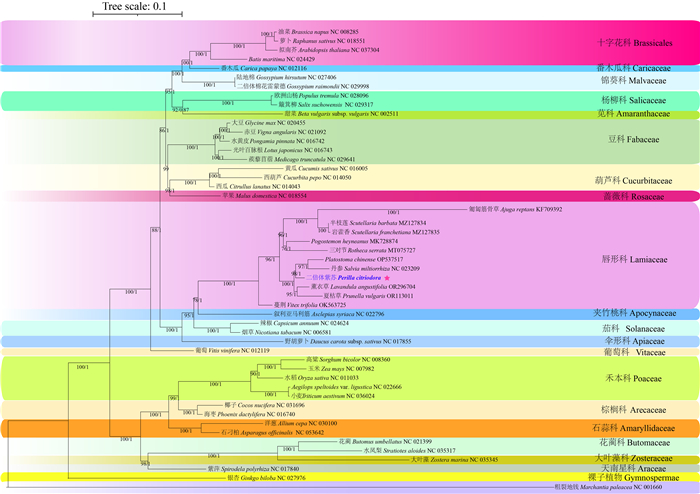
|
图 6 二倍体紫苏的系统发育树 Figure 6 The phylogenetic tree constructed for Perilla citriodora |
植物线粒体基因组具有复杂的组合、多样的结构、波动的非编码序列以及高重组性的重复序列,使得研究植物线粒体存在困难[5]。基于植物细胞器基因组的拷贝数远高于相应核基因组的事实[34],本研究利用Illumina和PacBio测序数据组装了总长度为301 156 bp的单一环状线粒体基因组。与其他唇形科植物线粒体基因组相比,二倍体紫苏的线粒体基因组大小处于中等范围,与已发表的唇形科植物夏枯草(274 779 bp)的线粒体基因组大小相似,但夏枯草的线粒体基因组是一个双环结构[35]。这表明,线粒体基因组中存在显著的结构变异和物种特异性。GC含量是评估物种的重要因素。二倍体紫苏线粒体的GC含量为45.23%,在rRNA基因中最高。这个结果类似于其他唇形科物种,如缙云黄芩(Scutellaria tsinyunensis)(45.09%)[36]、匍匐筋骨草(45.26%)[37]、三对节(45.53%)[38]以及夏枯草(43.92%)[35],表明在植物的进化过程中,线粒体基因组的GC含量相对保守。此外,本研究鉴定到59个基因,包括37个蛋白编码基因、20个tRNA基因和2个rRNA基因,基因的类型和数量与大多数陆地植物环状线粒体基因组一致。二倍体紫苏线粒体基因组超过80%是非编码区,编码区仅占其总长度的19.23%,这可能是线粒体基因组中重复序列逐渐增加所造成的[39]。
Ka和Ks的计算对于密切相关物种的系统发育重建和理解蛋白编码序列的进化动态具有重要意义[40]。本研究对Ka/Ks进行比较发现,大多数编码基因受到负选择的影响, 与Liu et al[41]报道一致。但ccmFn、mttB、rps10和matR基因的Ka/Ks>1,暗示这些编码基因在唇形科线粒体基因组的进化历史中发挥重要作用。
当外源基因插入线粒体基因组时,它们往往优先插入到基因间区域。不同物种叶绿体基因整合到线粒体基因组的DNA长度有所不同,通常在被子植物叶绿体基因组序列的1%~12%范围内,例如在山竹(Garcinia mangostana L.)中为1.7%[42],而在南瓜(Cucurbita pepo)中为11.6%[43],该现象是导致不同植物线粒体基因组编码基因数量差异的主要原因之一。因此,追踪基因转移对于探索植物线粒体基因组的进化至关重要[44]。tRNA基因序列从叶绿体到线粒体基因组的转移在植物中很常见[45]。本研究中,紫苏叶绿体的rps11和15个tRNA基因完全转移至线粒体基因组,叶绿体来源的tRNA可能具有潜在的功能补体。此外,还鉴定到大量的叶绿体基因片段迁移到线粒体基因组中, 这些叶绿体基因组衍生的片段含有在叶绿体中具有重要功能的基因,但它们是否在线粒体基因组中起作用尚不清楚。
重复序列是研究种群发育和进化分子标记的重要信息来源[46]。本研究鉴定到180个重复序列,包括87对反向重复和93对回文重复,这可能说明线粒体基因组中具有频繁的分子重组。在进化过程中,这种频繁的重组可能在动态改变线粒体基因组的结构和构象方面发挥了关键作用。此外, 本研究中还鉴定到78个SSRs分布在二倍体紫苏线粒体基因组的不同基因组区域且普遍存在A/T偏向性, SSR类型最多的是四核苷酸重复(35%), 其次是双核苷酸重复(20%), 这与大多数线粒体基因组的研究结果一致。
4 结论二倍体紫苏是一种具有重要经济和药用价值的植物。本研究对二倍体紫苏的线粒体基因组进行组装,从而实现了对细胞器基因组的全面比较,并为研究线粒体与质体之间的基因转移提供了更广阔的视角。此外,通过对该物种及其他50个植物的线粒体基因组进行系统发育分析,确认了该物种的进化地位。本研究为二倍体紫苏的遗传变异、系统进化和育种研究提供了理论参考,也为其培育、开发和利用提供指导。为更准确地确定唇形科系统发育关系,下一步有必要获得较小分类群的线粒体基因组信息。
| [1] | WANG Y, CHEN S J, CHEN J J, et al. Characterization and phylogenetic analysis of the complete mitochondrial genome sequence of Photinia serratifolia[J]. Scientific Reports, 2023, 13(1): 770. DOI: 10.1038/s41598-022-24327-x |
| [2] | 谭国飞, 李梦瑶, 罗庆, 等. 芹菜雄性不育的创制及线粒体不育候选基因鉴定[J]. 植物遗传资源学报, 2022, 23(6): 1807–1815. |
| [3] | LU G L, WANG W H, MAO J, et al. Complete mitogenome assembly of Selenicereus monacanthus revealed its molecular features, genome evolution, and phylogenetic implications[J]. BMC Plant Biology, 2023, 23(1): 541. DOI: 10.1186/s12870-023-04529-9 |
| [4] | MA Q Y, WANG Y X, LI S S, et al. Assembly and comparative analysis of the first complete mitochondrial genome of Acer truncatum bunge: a woody oil-tree species producing nervonic acid[J]. BMC Plant Biology, 2022, 22(1): 29. DOI: 10.1186/s12870-021-03416-5 |
| [5] | WANG J, KAN S L, LIAO X Z, et al. Plant organellar genomes: much done, much more to do[J]. Trends in Plant Science, 2024. DOI: 10.1016/j.tplants.2023.12.014 |
| [6] | GUALBERTO J M, MILESHINA D, WALLET C, et al. The plant mitochondrial genome: dynamics and maintenance[J]. Biochimie, 2014, 100: 107–120. DOI: 10.1016/j.biochi.2013.09.016 |
| [7] | DAY D A. Mitochondrial structure and function in plants[M]//DAY D A, MILLAR A H, WHELAN J. Plant Mitochondria: From Genome to Function. Dordrecht: Springer, 2004: 1-11. |
| [8] | CHEN J H, MA H P, FAN H L, et al. De novo assembly and comparative analysis of the mitochondrial genome of Reynoutria japonica[J]. Frontiers in Genetics, 2023, 14: 1289811. DOI: 10.3389/fgene.2023.1289811 |
| [9] | JIN J J, YU W B, YANG J B, et al. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes[J]. Genome Biology, 2020, 21(1): 241. DOI: 10.1186/s13059-020-02154-5 |
| [10] | ALLIO R, SCHOMAKER-BASTOS A, ROMIGUIER J, et al. MitoFinder: efficient automated large-scale extraction of mitogenomic data in target enrichment phylogenomics[J]. Molecular Ecology Resources, 2020, 20(4): 892–905. DOI: 10.1111/1755-0998.13160 |
| [11] | HE W C, XIANG K L, CHEN C J, et al. Master graph: an essential integrated assembly model for the plant mitogenome based on a graph-based framework[J]. Briefings in Bioinformatics, 2023, 24(1): bbac522. DOI: 10.1093/bib/bbac522 |
| [12] | BI C W, SHEN F, HAN F C, et al. PMAT: an efficient plant mitogenome assembly toolkit using low-coverage HiFi sequencing data[J]. Horticulture Research, 2024, 11(3): uhae023. DOI: 10.1093/hr/uhae023 |
| [13] | 匡卫民, 于黎. 基因组时代线粒体基因组拼装策略及软件应用现状[J]. 遗传, 2019, 41(11): 979–993. |
| [14] | 陈楠, 黄明田, 郑新宇, 等. 种植密度、施肥及种植方式对紫苏铜富集能力的影响[J]. 亚热带农业研究, 2020, 16(2): 118–125. |
| [15] | 张婉萍, 陈婕, 王恒, 等. 紫苏叶的活性成分及生物活性研究进展[J]. 食品与发酵工业, 2023, 49(20): 361–368. |
| [16] | 王馨平, 聂黎行, 康帅, 等. 紫苏叶的化学成分、药理活性和质量控制研究进展[J]. 中国药事, 2023, 37(10): 1193–1212. |
| [17] | HUANG J H, CHEN C Y, SONG Z H, et al. Effect of microwave pretreatment of perilla seeds on minor bioactive components content and oxidative stability of oil[J]. Food Chemistry, 2022, 388: 133010. DOI: 10.1016/j.foodchem.2022.133010 |
| [18] | 郭旭, 田荣荣, 张东. 紫苏油的提取工艺和药理功能研究进展[J]. 粮油食品科技, 2021, 29(5): 120–130. |
| [19] | LEE D S, NOH B S, BAE S Y, et al. Characterization of fatty acids composition in vegetable oils by gas chromatography and chemometrics[J]. Analytica Chimica Acta, 1998, 358(2): 163–175. DOI: 10.1016/S0003-2670(97)00574-6 |
| [20] | LI S S, WANG L S, SHU Q Y, et al. Fatty acid composition of developing tree peony (Paeonia section Moutan DC.) seeds and transcriptome analysis during seed development[J]. BMC Genomics, 2015, 16(1): 208. DOI: 10.1186/s12864-015-1429-0 |
| [21] | ZARU R, ORCHARD S, The Uniprot Consortium. UniProt Tools: BLAST, align, peptide search, and ID mapping[J]. Current Protocols, 2023, 3(3): e697. DOI: 10.1002/cpz1.697 |
| [22] | KOREN S, WALENZ B P, BERLIN K, et al. Canu: scalable and accurate long-read assembly via adaptive k-mer weighting and repeat separation[J]. Genome Research, 2017, 27(5): 722–736. DOI: 10.1101/gr.215087.116 |
| [23] | ZOU Q, GUO J S, JU Y, et al. Improving tRNAscan-SE annotation results via ensemble classifiers[J]. Molecular Informatics, 2015, 34(11/12): 761–770. |
| [24] | GREINER S, LEHWARK P, BOCK R. OrganellarGenomeDRAW (OGDRAW) version 1.3.1: expanded toolkit for the graphical visualization of organellar genomes[J]. Nucleic Acids Research, 2019, 47(W1): W59–W64. DOI: 10.1093/nar/gkz238 |
| [25] | BEIER S, THIEL T, MVNCH T, et al. MISA-web: a web server for microsatellite prediction[J]. Bioinformatics, 2017, 33(16): 2583–2585. |
| [26] | BENSON G. Tandem repeats finder: a program to analyze DNA sequences[J]. Nucleic Acids Research, 1999, 27(2): 573–580. |
| [27] | KRZYWINSKI M, SCHEIN J, BIROL İ, et al. Circos: an information aesthetic for comparative genomics[J]. Genome Research, 2009, 19(9): 1639–1645. |
| [28] | ZHANG D, GAO F L, JAKOVLIĆ I, et al. PhyloSuite: an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Molecular Ecology Resources, 2020, 20(1): 348–355. |
| [29] | KATOH K, ROZEWICKI J, YAMADA K D. MAFFT online service: multiple sequence alignment, interactive sequence choice and visualization[J]. Briefings in Bioinformatics, 2019, 20(4): 1160–1166. |
| [30] | HÖHLER D, PFEIFFER W, IOANNIDIS V, et al. RAxML Grove: an empirical phylogenetic tree database[J]. Bioinformatics, 2022, 38(6): 1741–1742. |
| [31] | RONQUIST F, HUELSENBECK J P. MrBayes 3: Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 2003, 19(12): 1572–1574. |
| [32] | FANG L, LIU Q, MONTEYS A M, et al. DeepRepeat: direct quantification of short tandem repeats on signal data from nanopore sequencing[J]. Genome Biology, 2022, 23(1): 108. DOI: 10.1186/s13059-022-02670-6 |
| [33] | WANG R, LUO Y J, LAN Z, et al. Insights into structure, codon usage, repeats, and RNA editing of the complete mitochondrial genome of Perilla frutescens(Lamiaceae)[J]. Scientific Reports, 2024, 14(1): 13940. DOI: 10.1038/s41598-024-64509-3 |
| [34] | ZHANG Y J, SHEN Q, LENG L, et al. Incipient diploidization of the medicinal plant Perilla within 10 000 years[J]. Nature Communications, 2021, 12(1): 5508. DOI: 10.1038/s41467-021-25681-6 |
| [35] | SUN Z H, WU Y, FAN P Y, et al. Assembly and analysis of the mitochondrial genome of Prunella vulgaris[J]. Frontiers in Plant Science, 2023, 14: 1237822. DOI: 10.3389/fpls.2023.1237822 |
| [36] | LI J L, XU Y C, SHAN Y Y, et al. Assembly of the complete mitochondrial genome of an endemic plant, Scutellaria tsinyunensis, revealed the existence of two conformations generated by a repeat-mediated recombination[J]. Planta, 2021, 254(2): 36. DOI: 10.1007/s00425-021-03684-3 |
| [37] | LIU F, FAN W S, YANG J B, et al. Episodic and guanine-cytosine-biased bursts of intragenomic and interspecific synonymous divergence in Ajugoideae (Lamiaceae) mitogenomes[J]. New Phytologist, 2020, 228(3): 1107–1114. |
| [38] | FANG X A, ZHANG S Y. The complete chloroplast genome sequence of Rotheca myricoides (Hochst.) Steane & Mabb., a traditional medicinal plant[J]. Mitochondrial DNA Part B, 2021, 6(9): 2699–2700. |
| [39] | NARDI F, CARAPELLI A, FRATI F. Repeated regions in mitochondrial genomes: distribution, origin and evolutionary significance[J]. Mitochondrion, 2012, 12(5): 483–491. |
| [40] | ZHANG Z, LI J, ZHAO X Q, et al. KaKs_Calculator: calculating Ka and Ks through model selection and model averaging[J]. Genomics, Proteomics & Bioinformatics, 2006, 4(4): 259–263. |
| [41] | LIU H R, LIU W H, XIAO Q M, et al. Comparative analysis of the complete mitochondrial genome sequence of an alpine plant Triosteum pinnatifidum[J]. Cytology and Genetics, 2023, 57(4): 335–346. |
| [42] | WEE C C, NOR MUHAMMAD N A, SUBBIAH V K, et al. Mitochondrial genome of Garcinia mangostana L. variety mesta[J]. Scientific Reports, 2022, 12(1): 9480. DOI: 10.1038/s41598-022-13706-z |
| [43] | ALVERSON A J, WEI X X, RICE D W, et al. Insights into the evolution of mitochondrial genome size from complete sequences of Citrullus lanatus and Cucurbita pepo (Cucurbitaceae)[J]. Molecular Biology and Evolution, 2010, 27(6): 1436–1448. |
| [44] | MOWER J P, SLOAN D B, ALVERSON A J. Plant mitochondrial genome diversity: the genomics revolution[M]//WENDEL J F, GREILHUBER J, DOLEZEL J, et al. Plant Genome Diversity Volume 1: Plant Genomes, their Residents, and their Evolutionary Dynamics. Vienna: Springer, 2012: 123-144. |
| [45] | BERGTHORSSON U, ADAMS K L, THOMASON B, et al. Widespread horizontal transfer of mitochondrial genes in flowering plants[J]. Nature, 2003, 424(6945): 197–201. |
| [46] | CHEVIGNY N, SCHATZ-DAAS D, LOTFI F, et al. DNA repair and the stability of the plant mitochondrial genome[J]. International Journal of Molecular Sciences, 2020, 21(1): 328. DOI: 10.3390/ijms21010328 |
 2024,Vol. 20
2024,Vol. 20


