文章信息
- 王巧, 周艳平, 郑惠成, 彭金彬, 方福钟, 邹双全
- WANG Qiao, ZHOU Yanping, ZHENG Huicheng, PENG Jinbin, FANG Fuzhong, ZOU Shuangquan
- 利用SRAP与ISSR分子标记鉴别牛樟种质
- Identification of Cinnamomum kanehirae Hayata germplasm using SRAP and ISSR molecular markers
- 亚热带农业研究, 2019, 15(3): 145-151
- Subtropical Agriculture Research, 2019, 15(3): 145-151.
- DOI: 10.13321/j.cnki.subtrop.agric.res.2019.03.001
-
文章历史
- 收稿日期: 2019-06-04
2. 莆田市荔城区西天尾镇林业站, 福建 莆田 351131;
3. 福建省林业调查规划院, 福建 福州 350003;
4. 泉州市洛江区林业局, 福建 泉州 362011;
5. 漳州市林业科学研究所, 福建 漳州 363000
2. Xitianwei Forestry Depot of Licheng District, Putian County, Putian, Fujian 351131, China;
3. Forest Inventory and Planning Institute of Fujian Province, Fuzhou, Fujian 350003, China;
4. Luojiang District Forestry Bureau, Quanzhou, Fujian 362011, China;
5. Zhangzhou Institute of Forestry, Zhangzhou, Fujian 363000, China
牛樟(Cinnamomum kanehirae Hayata)属樟科(Lauraceae)樟属(Cinnamomum)植物,又名黑樟,原产地为我国台湾[1]。牛樟主要分布于海拔200~2 000 m的山区,尤其以台湾的新竹、花东及中南部较多[2]。老龄腐朽的牛樟树干内壁寄生了极具药用价值的牛樟芝,因而深受医疗保健市场的欢迎[3]。由于过度砍伐,目前牛樟天然林仅存在于交通不便的中、高海拔地区,且多为单株分散的高龄老树,结实量少,树高体大不容易攀爬,母树间授粉不易,采种也极其困难[4]。现有的大量民间引种未经系统鉴别,导致种源混乱,阻碍了牛樟的良种保存与繁育。为加强牛樟种质资源的保护与满足市场需求,需建立有效的鉴别技术。
分子标记技术已在作物遗传多样性分析、品种鉴定及遗传图谱构建等方面得到广泛应用。相关序列扩增多态性(sequence-related amplified polymorphism, SRAP)具有遗传信息丰富、可检测的多态性强、DNA纯度要求不高、条带清晰等特点[5],已广泛应用于水稻[6]、桃[7]及芹菜[5]等研究中。简单重复序列区间(inter-simple sequence repeat, ISSR)具有遗传信息丰富、可检测的多态性强、试验操作简单并且成本较低等特点[8]。朱丽萍[8]采用ISSR分子标记技术检测牛樟、冇樟和樟树三者的族群遗传结构表明,牛樟为台湾的特有种,并认为牛樟是由樟树演变而来的。孟红岩等[9]探索了台湾牛樟总RNA的提取方法;Hung et al[10]在樟属植物中分离出了15个微卫星序列,可用于牛樟等樟属植物的鉴定;曾焕中等[11]根据樟脑素、松油醇和黄樟素含量判别是否为牛樟。目前尚未见利用SRAP与ISSR分子标记技术研究牛樟种质。福建省为适宜牛樟生长的主要地区,为此本研究在福建选取10个牛樟种质,通过SRAP与ISSR分子标记技术分析种质间的遗传多样性,以期为牛樟优质种源的保存与推广提供依据。
1 材料与方法 1.1 试验材料10个牛樟种质分别取自:①福建农林大学植物园(农大牛樟);②南安市蓬华镇苏厝村将军山(南安牛樟);③永春县东平镇冷水村(永春牛樟1);④永春县东平镇冷水村(永春牛樟2);⑤德化县龙浔镇高阳村(德化牛樟);⑥福建省林业科技试验中心(南靖牛樟);⑦漳浦县扬基生物科技有限公司(漳浦牛樟);⑧漳州市龙海林下国有林场(林场牛樟);⑨永安市贡坪采育场(永安牛樟1);⑩永安市大西坑永林种苗中心(永安牛樟2)。其中,农大牛樟与漳浦牛樟由种植人从台湾引进,并已通过台湾专家鉴定。
1.2 DNA提取与检测2016年7月初采集发育良好且无病虫害的牛樟嫩叶,采用改进的十六烷基三甲基溴化铵法(cetyltrimethyl ammonium bromide, CTAB)提取DNA[12]。用Nano Drop ND-1000核酸蛋白检测仪(Nano Drop Technologies Inc,美国)检测DNA浓度,通过1.5%琼脂糖凝胶电泳检测DNA质量[13]。
1.3 SRAP-PCR扩增与引物筛选从16个正向和16个反向引物组合的256对引物中筛选出10对具有丰富多态性的SRAP引物组合(表 1), 进行SRAP-PCR扩增试验。程序基本采用Zhou et al[14]的方法,并稍作改良。
| 编号 | 引物 | 序列 |
| 1 | Em7-Me8 | F:TGAGTCCAAACCGGACG |
| R:GACTGCGTACGAATTCAC | ||
| 2 | Em8-Me9 | F:TGAGTCCAAACCGGACT |
| R:GACTGCGTACGAATTCAG | ||
| 3 | Em10-Me2 | F:TGAGTCCAAACCGGAAA |
| R:GACTGCGTACGAATTGAC | ||
| 4 | Em10-Me16 | F:TGAGTCCAAACCGGAAA |
| R:GACTGCGTACGAATTGTC | ||
| 5 | Em11-Me8 | F:TGAGTCCAAACCGGAAC |
| R:GACTGCGTACGAATTCAC | ||
| 6 | Em12-Me12 | F:TGAGTCCAAACCGGAGA |
| R:GACTGCGTACGAATTCTC | ||
| 7 | Em13-Me5 | F:TGAGTCCAAACCGGATG |
| R:GACTGCGTACGAATTAAC | ||
| 8 | Em13-Me6 | F:TGAGTCCAAACCGGATG |
| R:GACTGCGTACGAATTGCA | ||
| 9 | Em14-Me1 | F:TGAGTCCAAACCGGATC |
| R:GACTGCGTACGAATTAAT | ||
| 10 | Em14-Me2 | F:TGAGTCCAAACCGGATC |
| R:GACTGCGTACGAATTTGC |
从哥伦比亚大学公布的100个ISSR引物中筛选出10个具有丰富多态性的ISSR引物(表 2),进行ISSR-PCR扩增试验。试验基本参照刘玉香[15]的ISSR-PCR反应体系。
| 编号 | 引物 | 序列 |
| 1 | UBC809 | AGA GAG AGA GAG AGA GG |
| 2 | UBC810 | GAG AGA GAG AGA GAG AT |
| 3 | UBC845 | CTC TCT CTC TCT CTC TRG |
| 4 | UBC856 | ACA CAC ACA CAC ACA CYA |
| 5 | UBC873 | GAC AGA CAG ACA GAC A |
| 6 | UBC888 | BDB CAC ACA CAC ACA CA |
| 7 | UBC890 | VHV GTG TGT GTG TGT GT |
| 8 | UBC895 | AGA GTT GGT AGC TCT TGA TC |
| 9 | UBC899 | CAT GGT GTT GGT CAT TGT TCC A |
| 10 | UBC900 | ACT TCC CCA CAG GTT AAC ACA |
| 1)R=(A, G), Y=(C, T), B=(C, G, T), D=(A, G, T), H=(A, C, T), V=(A, C, G)。 | ||
筛选出SRAP与ISSR引物,观察其扩增结果的电泳图。在电泳图谱中,迁移性一致的DNA条带即具有同源性。采用人工读图的方式,将电泳图上有清晰条带的记为“1”,没有条带或者条带不清晰的记为“0”。根据每对引物组合扩增出来的条带计算多态性位点数目(number of polymorphic bands, NPB)、总扩增位点数目(total number of bands, TNB)和多态性位点百分比(percentage of polymorphic bands, PPB)。公式:PPB=NPB/TNB。使用NTSYS 2.1统计分析软件中Similarity程序计算10个牛樟种质间的遗传相似系数(genetic similarity, GS),再用非加权组平均法(unweighted pair-group method using arithmetic average algorithm, UPGMA)进行聚类分析[16]。
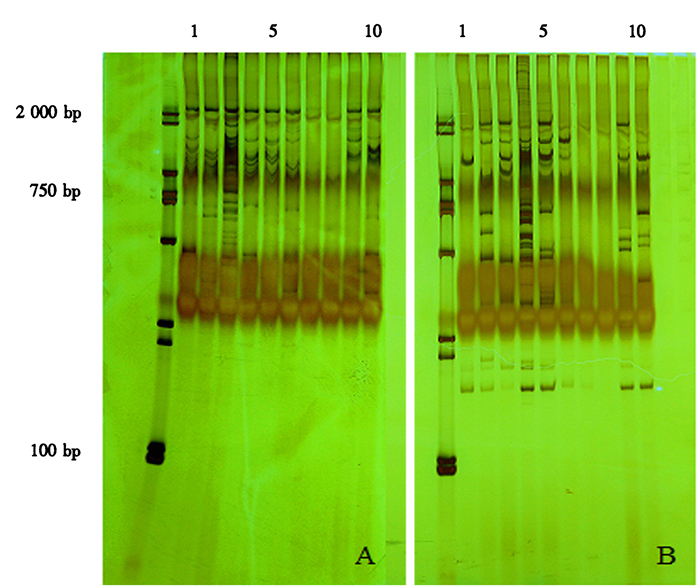
2 结果与分析 2.1 牛樟种质资源的多态性分析(1) 分别采用10对SRAP引物组合对10个牛樟种质进行DNA扩增。结果表明,片段大小主要集中在50~2 000 bp,10对引物总共扩增出251条条带,其中220条为多态性条带,多态性条带百分比为87.6%(表 3)。Em8-Me9和Em10-Me2这两对引物组合产生的多态性条带百分比最多,均达到100%(图 1)。(2)分别采用10个ISSR引物对10个牛樟种质进行DNA扩增。结果表明,扩增出来的片段大小主要集中在100~2 000 bp。10个引物总共扩增出237条条带,其中210条为多态性条带,多态性条带百分比为88.6%(表 4)。UBC895和UBC900这两个引物产生的多态性条带百分比最多,均达到100%(图 2)。
| 引物 | 总扩增带数 | 多态性带数 | 多态性条带百分比/% |
| Em7-Me8 | 18 | 15 | 83.3 |
| Em8-Me9 | 26 | 26 | 100.0 |
| Em10-Me2 | 20 | 20 | 100.0 |
| Em10-Me16 | 23 | 21 | 91.3 |
| Em11-Me8 | 28 | 26 | 92.9 |
| Em12-Me12 | 27 | 23 | 85.2 |
| Em13-Me5 | 24 | 18 | 75.0 |
| Em13-Me6 | 28 | 25 | 89.2 |
| Em14-Me1 | 33 | 29 | 87.9 |
| Em14-Me2 | 24 | 17 | 70.8 |
| 平均 | 25 | 22 | 87.6 |
| 总计 | 251 | 220 |

|
A.Em8-Me9;B.Em10-Me2。1~10分别为:农大牛樟、南安牛樟、永春牛樟1、永春牛樟2、德化牛樟、南靖牛樟、漳浦牛樟、林场牛樟、永安牛樟1、永安牛樟2。 图 1 SRAP引物Em8-Me9和Em10-Me2的扩增结果 Figure 1 The electrophoregrams amplified DNA fragments by SRAP primer Em8-Me9 and Em10-Me2 |
| 引物 | 总扩增带数 | 多态性带数 | 多态性条带百分比/% |
| UBC809 | 18 | 17 | 94.4 |
| UBC810 | 28 | 20 | 71.4 |
| UBC845 | 18 | 17 | 94.4 |
| UBC856 | 14 | 13 | 92.9 |
| UBC873 | 35 | 32 | 91.4 |
| UBC888 | 20 | 14 | 70.0 |
| UBC890 | 24 | 18 | 75.0 |
| UBC895 | 26 | 26 | 100.0 |
| UBC899 | 33 | 32 | 97.0 |
| UBC900 | 21 | 21 | 100.0 |
| 平均 | 24 | 21 | 88.6 |
| 总计 | 237 | 210 |
|
A.UBC895;B.UBC900。1~10分别为:农大牛樟、南安牛樟、永春牛樟1、永春牛樟2、德化牛樟、南靖牛樟、漳浦牛樟、林场牛樟、永安牛樟1、永安牛樟2。 图 2 ISSR引物UBC895和UBC900的扩增结果 Figure 2 The electrophoregrams of amplified DNA fragments by ISSR primer UBC895 and UBC900 |
分别根据SRAP、ISSR结果,用NTSYS 2.1软件计算10个牛樟种质的遗传相似系数(表 5、6)。从表 5可见,牛樟种质间的遗传相似系数变化范围在0.355~0.882之间,平均相似系数为0.606。从表 6可见,牛樟种质间的遗传相似系数变化范围在0.371~0.814之间,平均相似系数为0.606。
| 牛樟 | 农大 | 南安 | 永春1 | 永春2 | 德化 | 南靖 | 漳浦 | 林场 | 永安1 | 永安2 |
| 农大 | 1.000 | |||||||||
| 南安 | 0.382 | 1.000 | ||||||||
| 永春1 | 0.355 | 0.873 | 1.000 | |||||||
| 永春2 | 0.495 | 0.705 | 0.650 | 1.000 | ||||||
| 德化 | 0.364 | 0.845 | 0.882 | 0.677 | 1.000 | |||||
| 南靖 | 0.414 | 0.686 | 0.677 | 0.782 | 0.705 | 1.000 | ||||
| 漳浦 | 0.755 | 0.445 | 0.436 | 0.468 | 0.409 | 0.405 | 1.000 | |||
| 林场 | 0.436 | 0.718 | 0.673 | 0.786 | 0.673 | 0.832 | 0.482 | 1.000 | ||
| 永安1 | 0.368 | 0.623 | 0.659 | 0.591 | 0.632 | 0.627 | 0.486 | 0.623 | 1.00 | |
| 永安2 | 0.373 | 0.700 | 0.655 | 0.777 | 0.691 | 0.823 | 0.427 | 0.836 | 0.659 | 1.00 |
| 牛樟 | 农大 | 南安 | 永春1 | 永春2 | 德化 | 南靖 | 漳浦 | 林场 | 永安1 | 永安2 |
| 农大 | 1.000 | |||||||||
| 南安 | 0.467 | 1.000 | ||||||||
| 永春1 | 0.471 | 0.795 | 1.000 | |||||||
| 永春2 | 0.457 | 0.638 | 0.690 | 1.000 | ||||||
| 德化 | 0.481 | 0.814 | 0.810 | 0.729 | 1.000 | |||||
| 南靖 | 0.510 | 0.643 | 0.657 | 0.757 | 0.676 | 1.000 | ||||
| 漳浦 | 0.743 | 0.505 | 0.433 | 0.371 | 0.452 | 0.490 | 1.000 | |||
| 林场 | 0.500 | 0.557 | 0.543 | 0.576 | 0.571 | 0.6856 | 0.510 | 1.000 | ||
| 永安1 | 0.519 | 0.652 | 0.619 | 0.605 | 0.676 | 0.657 | 0.481 | 0.552 | 1.000 | |
| 永安2 | 0.524 | 0.648 | 0.633 | 0.800 | 0.681 | 0.795 | 0.467 | 0.671 | 0.719 | 1.000 |
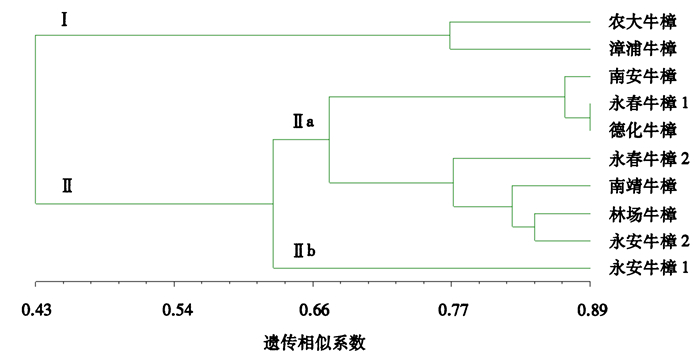
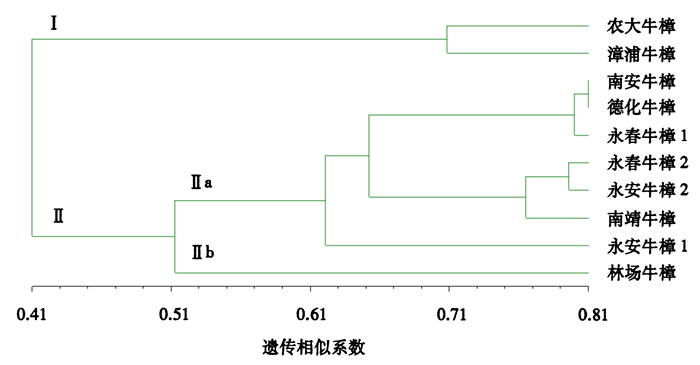
分别根据SRAP、ISSR扩增结果和10个牛樟种质间的遗传相似性,利用UPGMA法建立系统聚类分支树状图(图 3、4)。从图 3可见,在遗传相似系数为0.43时可将10个牛樟种质分成两大类,即Ⅰ类包括农大和漳浦牛樟,Ⅱ类包括南安、永春1、德化、永春2、南靖、林场、永安2和永安1牛樟。从图 4可见,在遗传相似系数为0.41时可将10个牛樟种质分成两大类,即Ⅰ类包括农大和漳浦牛樟,Ⅱ类包括南安、德化、永春1、永春2、永安2、南靖、永安1和林场牛樟。
|
图 3 10个牛樟种质的SRAP聚类图 Figure 3 A dendrogram of 10 C.kanehirae based on SRAP amplification profiles |
|
图 4 10个牛樟种质的ISSR聚类图 Figure 4 A dendrogram of 10 C.kanehirae based on ISSR amplification profiles |
(1) SRAP标记得到的10个牛樟种质间的遗传相似系数变化范围在0.355~0.882之间,平均相似系数为0.606,农大牛樟和永春牛樟1的遗传相似系数(0.355)最小,永春牛樟1和德化牛樟(0.882)最大。将农大牛樟与其余9个牛樟种质相比,农大牛樟与漳浦牛樟的遗传相似系数(0.755)最大。
(2) ISSR标记得到的10个牛樟种质间的遗传相似系数变化范围在0.371~0.814之间,平均相似系数为0.606。永春牛樟2和漳浦牛樟的遗传相似系数(0.371)最小,南安牛樟和德化牛樟的遗传相似系数(0.814)最大。将农大牛樟与其余9个牛樟种质相比,农大牛樟与漳浦牛樟的遗传相似系数(0.743)最大。将SRAP与ISSR数据整合得到的遗传相似系数变化范围在0.412~0.847之间,平均相似系数为0.603,所得结果均与SRAP一致。
2.4.2 聚类分析SRAP和ISSR分子标记的聚类分析图略有不同,具体表现为SRAP将永安牛樟1单独分在Ⅱb类,而ISSR将林场牛樟单独分在Ⅱb类。将SRAP与ISSR数据整合得到的聚类分析结果与SRAP的结果更具有一致性,除了林场牛樟和永安牛樟2顺序稍有不同,其他分类几乎相同。
利用Mantel检验SRAP、ISSR和SRAP+ISSR三者相似系数的相关性,SRAP与ISSR之间的相关系数为0.864;SRAP与SRAP+ISSR之间的相关系数为0.977;ISSR与SRAP+ISSR之间的相关系数为0.952。以上表明,SRAP和ISSR遗传相似系数都与SRAP+ISSR数据整合得到的遗传相似系数的相关性较高,SRAP标记更接近于两标记的综合遗传多样性。
3 讨论遗传相似系数对鉴别牛樟种质有着重要意义。本研究表明,SRAP分析中农大牛樟与漳浦牛樟的遗传相似系数为0.755,与其余8个种质遗传相似系数均低于0.500。ISSR分析中,农大牛樟与漳浦牛樟的遗传相似系数为0.743,与其他牛樟均低于0.530。农大牛樟与漳浦牛樟已鉴定为台湾牛樟[8, 17],本研究结果也证明了两者为同一种源。
本研究通过SRAP与ISSR分子标记技术将10个牛樟种质完全鉴别出来, 说明这两种分子标记技术对植物的鉴别均具有一定的可靠性。两者也存在一定的差异性,由于SRAP分子标记是对开放阅读框进行特异性的扩增[18],而ISSR分子标记检测的是位于反向排列的简单重复序列间的基因组DNA片段[19]。SRAP、ISSR均与SRAP+ISSR相关性较强,说明利用2种或2种以上的分子标记相结合,具有较高的可信性和一致性[20]。SRAP+ISSR的聚类分析结果与SRAP更具一致性,除林场牛樟和永安牛樟2顺序稍有不同,其他分类几乎相同。因此,在经济条件有限的情况下,仅使用SRAP分子标记便可达到良好的鉴别效果。
目前关于牛樟分子标记技术体系的研究甚少,本文首次采用SRAP与ISSR分子标记鉴定牛樟种质,为有效鉴别牛樟与杂樟奠定基础。
| [1] | 黄松根, 何坤益, 吴国伍, 等. 牛樟天然林组成与结构之调查[J]. 台湾林业科学, 1996, 11(4): 349–360. |
| [2] | 林赞标. 牛樟与冇樟[J]. 林业试验所研究报告季刊, 1993, 8(1): 11–20. |
| [3] | 黄大斌, 杨箐, 黄进华, 等. 樟芝生物学特性研究[J]. 食用菌学报, 2001, 8(2): 24–28. DOI: 10.3969/j.issn.1005-9873.2001.02.005 |
| [4] | 彭真汾, 王威, 谢倩, 等. 牛樟资源保护现状及繁育研究进展[J]. 亚热带农业研究, 2016, 12(1): 68–72. |
| [5] | LI G, QUIROS C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 2001, 103(2/3): 455–461. |
| [6] | 胡铁柱, 王玲, 冯熙路, 等. 稻瘟病菌群体的分子遗传学研究——由5个亚群体组成的广东省稻瘟病菌群体遗传结构的分析[J]. 中国农业科学, 2003, 36(12): 1476–1483. DOI: 10.3321/j.issn:0578-1752.2003.12.009 |
| [7] | AHMAD R, POTTER D, SOUTHWICK S M. Genotyping of peach and nectarine cultivars with SSR and SRAP molecular markers[J]. Journal of the American Society for Horticultural Science, 2004, 129(2): 204–210. DOI: 10.21273/JASHS.129.2.0204 |
| [8] | 朱丽萍.牛樟、冇樟及樟树之族群遗传变异及相关种属之亲缘关系研究[D].台北: 台湾大学, 2006: 143. |
| [9] | 孟红岩, 郭莺, 林文珍, 等. 台湾牛樟总RNA提取方法的建立[J]. 亚热带植物科学, 2016, 45(1): 53–56. DOI: 10.3969/j.issn.1009-7791.2016.01.011 |
| [10] | HUNG K H, LIN C H, SHIH H C, et al. Development, characterization and cross-species amplification of new microsatellite primers from an endemic species Cinnamomum kanehirae (Lauraceae) in Taiwan[J]. Conservation Genetics Resources, 2014, 6(4): 911–913. DOI: 10.1007/s12686-014-0239-z |
| [11] | 曾焕中, 贾铜安, 黄何兴.鉴定牛樟的方法: 104280482A[P].2015-01-14. |
| [12] | 曾斌, 罗淑萍, 李疆, 等. 硅胶干燥野扁桃叶片制备DNA样品及其SSR反应体系的建立[J]. 经济林研究, 2009, 27(1): 1–6. DOI: 10.3969/j.issn.1003-8981.2009.01.001 |
| [13] | 李艳香.19个绣球属种质资源的SRAP分析[D].长沙: 湖南农业大学, 2009. |
| [14] | ZHOU Y J, WANG X G, ZHANG X Q. Development and application of a SRAP marker for the identification of sex in Buchloe dactyloides[J]. Euphytica, 2011, 181(2): 261–266. DOI: 10.1007/s10681-011-0419-9 |
| [15] | 刘玉香.樟科主要属种核型研究及其亲缘关系的ISSR分析[D].南昌: 江西农业大学, 2013. |
| [16] | ROHLF F J. NTSYS-pc:numerical taxonomy and multivariate analysis system, version 2.10[J]. New York:Exeter Software, 2000. |
| [17] | 周张德堂.台湾牛樟无性繁殖技术研究[D].福州: 福建农林大学, 2013. |
| [18] | 徐宗大.玫瑰遗传多样性的SRAP分析与品种指纹图谱构建[D].泰安: 山东农业大学, 2011. |
| [19] | 沈洁.DNA序列、ISSR标记在药用植物种质鉴别中的应用[D].南京: 南京师范大学, 2005. |
| [20] | 胡迪科.雷公藤遗传多样性的ISSR和SRAP分析[D].福州: 福建农林大学, 2011. |
 2019,Vol. 15
2019,Vol. 15


