文章信息
- 王锦锋, 张健, 林占熺
- WANG Jinfeng, ZHANG Jian, LIN Zhanxi
- 应用拮抗、ITS和RAPD技术进行香菇属菌株分类
- Classification of Lentinus edodes strains by antagonistic effect, ITS and RAPD techniques
- 亚热带农业研究, 2017, 13(2): 132-138
- Subtropical Agriculture Research, 2017, 13(2): 132-138.
- DOI: 10.13321/j.cnki.subtrop.agric.res.2017.02.012
-
文章历史
- 收稿日期: 2016-11-26
2. 福建农林大学生命科学学院, 福建 福州 350002
2. College of Life Sciences, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
香菇(Lentinus edodes)属于真菌门,担子菌纲,伞菌目,口蘑科,香菇属[1], 是我国著名的食用菌。市场上香菇品种众多,但由于菌种监管不严以及私自改名等原因,当前香菇菌种命名相当混乱,同种异名、同名异种现象普遍存在[2-3]。随着香菇产品的开发,建立一套香菇菌种的保障体系尤为重要。拮抗性试验因具有直观、快速和易实现的优点,在真菌亲缘关系的初步研究中得到广泛应用[4-7],但由于很难区分出遗传上有相似背景或者亲缘关系很近的菌株,目前只限于菌株鉴定的初步研究[8-9]。为了更准确地对真菌进行分类,就需借助现代分子生物学方法。ITS序列分析技术能实质性地反映出属间、种间以及菌株间的碱基对差异,且ITS序列片段较小、易于分析,因此被广泛应用于真菌种内或种间的系统学研究,是真菌鉴别的重要手段[10-11]。RAPD-PCR具有时间短、效率高和灵敏度高等优点,可以在整个基因组DNA水平上分析不同菌株的亲缘关系,现已广泛应用于遗传作图、外缘染色体检测、种属特异性检测及物种演化等方面[12]。基于此,本研究应用拮抗试验、ITS序列分析和RAPD分子标记技术对所收集的14株香菇菌株进行亲缘关系分析,以期更好地开发利用香菇资源,为香菇的菌种检测与鉴定提供依据。
1 材料与方法 1.1 供试菌株及相关信息试验所用菌株均由国家菌草工程技术研究中心提供,具体信息见表 1。
| 菌号 | 菌株 | 温型 | t适宜出菇/℃ |
| 1 | LC236 | 中温 | 8~21 |
| 2 | LC2141 | 中高温 | 11~25 |
| 3 | LC206 | 中温 | 8~21 |
| 4 | LC202 | 中温 | 8~21 |
| 5 | LC2 | 中温 | 10~20 |
| 6 | LC109 | 中低温 | 7~20 |
| 7 | L95 | 中温 | 15~25 |
| 8 | L856 | 中温 | 10~20 |
| 9 | L66 | 中温 | 8~22 |
| 10 | L3031 | 中低温 | 7~20 |
| 11 | L087 | 中温 | 10~20 |
| 12 | L0302 | 中低温 | 7~21 |
| 13 | Cr022 | 中温 | 8~20 |
| 14 | Cr-02 | 中温 | 10~20 |
DNA提取试剂盒和胶回收盒购于OMEGA公司;2×Taq PCR Master Mix、6×Loading Buffer、大肠杆菌感受态细胞和18-T质粒载体购于TIANGEN公司;Marker Plus (D2000 Plus)购于Gentar公司;核酸染料和RNA酶购于索莱宝公司;NaCl、无水乙醇、葡萄糖、胰蛋白胨、琼脂粉、酵母提取物、异戊醇、氯仿、β-巯基乙醇等购于国药集团。
S1000TM Thermal Cycler型PCR仪和Power PacTM Basic型电泳槽购于BIO-RAD公司;Gen Box F3型高级凝胶成像系统购于SYNGENE公司。
1.3 DNA提取将香菇菌种接种到灭菌后的常规PDA液体培养基中,于28 ℃、150 r·min-1恒温摇床中培养15 d,收集新鲜菌丝体,液氮研磨后使用OMEGA DNA提取试剂盒提取样品DNA。
1.4 拮抗试验将不同菌株活化后,在同一平板上按“品”字形接种3个菌株。25 ℃恒温下培养,观察菌丝间的拮抗反应情况并记录。
1.5 ITS试验与RAPD试验 1.5.1 ITS试验选用2条特异引物ITS4和ITS5对14个菌株的基因组DNA进行特异性扩增(表 2)。25 μL反应体系为:12.5 μL 2×Taq PCR Master Mix、1 μL引物ITS4、1 μL引物ITS5、1 μL DNA样品、9.5 μL ddH2O。ITS扩增反应程序为:95 ℃预变性3 min,35个循环,包括94 ℃变性30 s、55 ℃退火30 s和72 ℃延伸1 min,最后72 ℃延伸7 min。反应结束后,取10 μL PCR产物于1.5%琼脂凝胶上电泳后拍照、记录、送检。
| 引物 | 序列(5′-3′) |
| ITS4 | TCCTCCGCTTATTGATATGC |
| ITS5 | GGAAGTAAAAGTCGTAACAAGG |
选用9个随机引物对14个菌株进行扩增,所用引物序列参考文献[13](表 3)。25 μL反应体系为:12.5 μL 2×Taq PCR Master Mix、1 μL引物、1 μL DNA样品、9.5 μL ddH2O。RAPD扩增反应程序为:95 ℃预变性5 min,40个循环,包括94 ℃变性45 s、35 ℃退火45 s和72 ℃延伸2 min,最后72 ℃延伸7 min。反应结束后,取10 μL PCR产物于1.5%琼脂凝胶上电泳后拍照并记录。
| 编号 | 引物 | 序列(5′-3′) |
| 1 | A3 | AGTCAGCCAC |
| 2 | A4 | AATCGGGCTG |
| 3 | D18 | GAGAGCCAAC |
| 4 | G19 | GTCAGGGCAA |
| 5 | H07 | CTGCATCGTG |
| 6 | H13 | GACGCCACAC |
| 7 | B11 | GTAGACCCGT |
| 8 | RA01 | AGCGCCATTG |
| 9 | RA02 | CAGCACCCAC |
用Gel-Pro Analyzer和UPGMA(NTSYS2.1) 软件进行聚类分析,构建树状图[10]。
2 结果与分析 2.1 拮抗试验分析拮抗试验结果见表 4。由表 4可知,LC206(3号)与LC2(5号);LC202(4号)与5号菌株、L087(11号);5号菌株与L856(8号);LC109(6号)与L95(7号);11号菌株与Cr-02(14号)间没有拮抗反应或拮抗性反应极不明显,说明其亲缘关系很近。LC236(1号)与L66(9号);LC2141(2号)与3、5、7、8、9号菌株以及L3031(10号)、Cr022(13号);3号与7、8、9、13号菌株;4号与8、13、14号菌株;5号与7、9、13、14号菌株;7号与8、9、13号菌株;8号与9、13号菌株;9号与13号菌株;13号和14号菌株之间虽存在拮抗反应,但表现很弱,较难分辨。而其他各菌株之间则存在着不同程度的拮抗反应,这表明菌株之间的亲缘关系较为疏远。不同种之间存在着拮抗反应,同种不同菌株之间也可能存在拮抗反应。菌株之间没有拮抗反应或拮抗反应极不明显并不能确定是同一菌株,但菌株之间存在拮抗反应则可以确定一定不是同一菌株。因此,本研究利用ITS分析和RAPD技术进一步鉴定。
| 菌号 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 1 | - | |||||||||||||
| 2 | +++ | - | ||||||||||||
| 3 | +++ | + | - | |||||||||||
| 4 | +++ | +++ | ++ | - | ||||||||||
| 5 | +++ | + | - | - | - | |||||||||
| 6 | +++ | ++ | +++ | +++ | +++ | - | ||||||||
| 7 | +++ | + | + | +++ | + | - | - | |||||||
| 8 | +++ | + | + | + | - | +++ | + | - | ||||||
| 9 | + | + | + | +++ | + | +++ | + | + | - | |||||
| 10 | +++ | + | +++ | +++ | +++ | +++ | ++ | +++ | +++ | - | ||||
| 11 | ++ | +++ | +++ | - | ++ | +++ | +++ | ++ | ++ | +++ | - | |||
| 12 | +++ | +++ | ++ | +++ | +++ | +++ | +++ | +++ | +++ | +++ | +++ | - | ||
| 13 | +++ | + | + | + | + | +++ | + | + | + | +++ | +++ | +++ | - | |
| 14 | +++ | +++ | ++ | + | + | +++ | +++ | ++ | +++ | +++ | - | +++ | + | - |
| 1)“-”表示未观察到拮抗现象;“+”表示拮抗线很弱,较难分辨;“++”表示平板难以观察到拮抗线,但背面能观察到拮抗线;“+++”表示拮抗线明显。1~14号菌株分别为:LC236、LC2141、LC206、LC202、LC2、LC109、L95、L856、L66、L3031、L087、L0302、Cr022和Cr-02。 | ||||||||||||||
采用特异性引物ITS4和ITS5对供试菌株DNA进行PCR扩增,均获得约750 bp(包括18S、28S、ITS1、ITS2和5.8S RNA片段)的单一条带。经切胶回收后进行序列分析。将测序结果在NCBI上进行比对,将比对后的结果除去18S和28S序列后,每个菌株的ITS序列长度为667 bp(表 5),其中ITS1和5.8S RNA完全一样,而差别集中在ITS2部分。
| 菌号 | ITS1 | 5.8S RNA | ITS2 | |||||
| 长度/bp | G+C/% | 长度/bp | G+C/% | 长度/bp | G+C/% | |||
| 1 | 237 | 36.71 | 157 | 47.13 | 273 | 33.70 | ||
| 2 | 237 | 36.71 | 157 | 47.13 | 274 | 32.85 | ||
| 3 | 237 | 36.71 | 157 | 47.13 | 274 | 33.21 | ||
| 4 | 237 | 36.71 | 157 | 47.13 | 273 | 33.33 | ||
| 5 | 237 | 36.71 | 157 | 47.13 | 274 | 32.85 | ||
| 6 | 237 | 36.71 | 157 | 47.13 | 273 | 33.33 | ||
| 7 | 237 | 36.71 | 157 | 47.13 | 274 | 33.21 | ||
| 8 | 237 | 36.71 | 157 | 47.13 | 274 | 33.21 | ||
| 9 | 237 | 36.71 | 157 | 47.13 | 274 | 33.21 | ||
| 10 | 237 | 36.71 | 157 | 47.13 | 274 | 33.21 | ||
| 11 | 237 | 36.71 | 157 | 47.13 | 273 | 33.33 | ||
| 12 | 237 | 36.71 | 157 | 47.13 | 273 | 33.70 | ||
| 13 | 237 | 36.71 | 157 | 47.13 | 274 | 33.21 | ||
| 14 | 237 | 36.71 | 157 | 47.13 | 273 | 33.33 | ||
| 1)1~14号菌株分别为:LC236、LC2141、LC206、LC202、LC2、LC109、L95、L856、L66、L3031、L087、L0302、Cr022和Cr-02。 | ||||||||
将14株供试菌株的ITS序列依次与GenBank中已登录的序列进行相似性比对,结果见表 6。14株菌株均为香菇属菌株,无外源种菌株,其中4、11和14号菌株登录号相同,为AB286067.1;2、3、5、7、8、9号和13号菌株登录号相同, 为DQ497071.1。菌株在Genbank中的登录号相同,表明菌株同源性高,即它们之间具有高度亲缘性。Genbank比对结果与拮抗试验结果存在一定的差异,但基本一致。造成差异的原因可能在于通过人为判断拮抗反应的强弱,较为主观,难免会造成误差。
| 菌号 | 登录号 |
| 1 | AB286065.1 |
| 2 | DQ497071.1 |
| 3 | DQ497071.1 |
| 4 | AB286067.1 |
| 5 | DQ497071.1 |
| 6 | AB286064.1 |
| 7 | DQ497071.1 |
| 8 | DQ497071.1 |
| 9 | DQ497071.1 |
| 10 | FJ582641.1 |
| 11 | AB286067.1 |
| 12 | AB286063.1 |
| 13 | DQ497071.1 |
| 14 | AB286067.1 |
| 1)1~14号菌株分别为:LC236、LC2141、LC206、LC202、LC2、LC109、L95、L856、L66、L3031、L087、L0302、Cr022和Cr-02。 | |
通过选取的9个引物对14个香菇菌株进行RAPD扩增,均出现多态性的DNA扩增片段,条带较为清晰,扩增效果好。9个引物一共扩增出120条条带,其中89条条带具有多态性,多态性条带比例达74.17%。单个引物平均扩增出13条条带,片段大小介于200~3 000 bp之间。不同引物RAPD扩增结果见图 1。
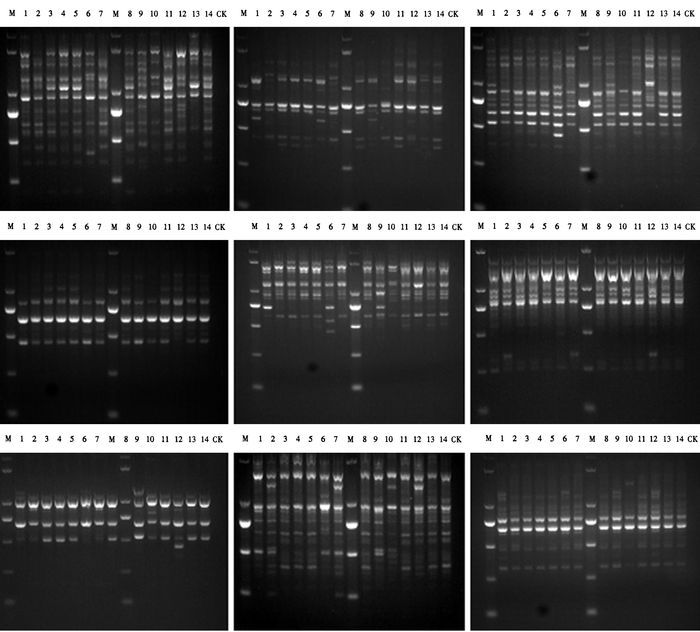
|
图 1 RAPD引物扩增结果 Figure 1 Electrophoregrams of amplified RAPD products 由左到右、由上到下,分别为1~9号引物。 |
由图 1可以看出,在不同引物扩增下,多数菌株分子量大小和条带数量都有较为明显的区别,但也有少数菌株(如3号和5号菌株)具有相同的带型。对9种引物的扩增图谱进行综合比对后,14种菌株均可区分开来。根据条带的有无,对RAPD-PCR产物电泳图谱中的条带进行分析,采用UPGMA软件进行聚类分析(图 2)并计算遗传相似距离矩阵(表 7)。
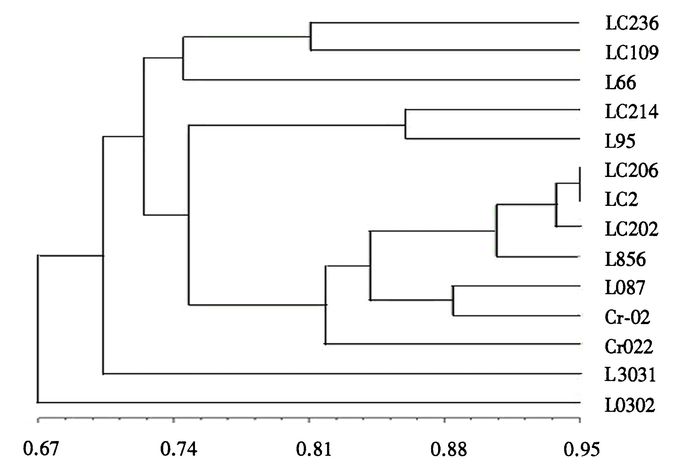
|
图 2 RAPD扩增结果构建的系统发育树 Figure 2 A phylogenetic tree constructed based on amplified RAPD products 相似系数 |
| 菌号 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 1 | 1.000 0 | |||||||||||||
| 2 | 0.775 0 | 1.000 0 | ||||||||||||
| 3 | 0.816 7 | 0.775 0 | 1.000 0 | |||||||||||
| 4 | 0.825 0 | 0.766 6 | 0.941 6 | 1.000 0 | ||||||||||
| 5 | 0.808 3 | 0.783 3 | 0.958 3 | 0.950 0 | 1.000 0 | |||||||||
| 6 | 0.816 7 | 0.691 6 | 0.733 3 | 0.741 6 | 0.725 0 | 1.000 0 | ||||||||
| 7 | 0.808 3 | 0.866 6 | 0.791 6 | 0.783 3 | 0.800 0 | 0.708 3 | 1.000 0 | |||||||
| 8 | 0.791 7 | 0.750 0 | 0.908 3 | 0.916 6 | 0.916 6 | 0.725 0 | 0.783 3 | 1.000 0 | ||||||
| 9 | 0.783 3 | 0.691 6 | 0.733 3 | 0.708 3 | 0.758 3 | 0.716 6 | 0.675 0 | 0.725 0 | 1.000 0 | |||||
| 10 | 0.725 0 | 0.716 6 | 0.708 3 | 0.733 3 | 0.733 3 | 0.675 0 | 0.750 0 | 0.733 3 | 0.691 6 | 1.000 0 | ||||
| 11 | 0.716 7 | 0.708 3 | 0.866 6 | 0.858 3 | 0.858 3 | 0.700 0 | 0.708 3 | 0.841 6 | 0.733 3 | 0.686 6 | 1.000 0 | |||
| 12 | 0.671 6 | 0.683 3 | 0.708 3 | 0.700 0 | 0.683 3 | 0.671 6 | 0.700 0 | 0.676 6 | 0.671 6 | 0.676 6 | 0.708 3 | 1.000 0 | ||
| 13 | 0.725 0 | 0.716 6 | 0.825 0 | 0.800 0 | 0.816 6 | 0.658 3 | 0.700 0 | 0.816 6 | 0.725 0 | 0.683 3 | 0.825 0 | 0.683 3 | 1.000 0 | |
| 14 | 0.691 7 | 0.733 3 | 0.841 6 | 0.850 0 | 0.850 0 | 0.675 0 | 0.733 3 | 0.816 6 | 0.675 0 | 0.700 0 | 0.891 6 | 0.733 3 | 0.866 6 | 1.000 0 |
| 1)1~14号菌株分别为:LC236、LC2141、LC206、LC202、LC2、LC109、L95、L856、L66、L3031、L087、L0302、Cr022和Cr-02。 | ||||||||||||||
图 2表明,当相似系数达到0.81时,1号和6号菌株划分为第1分支,2号和7号菌株为第2分支,3、4、5、8、11、13、14号菌株为第3分支,9、10、12号菌株被独立分离出来。而当相似系数在0.74以上时,9号菌株并入第1分支,第2和第3分支合并,10号、12号菌株仍相对独立。综合来看,10号、12号菌株与其他菌株相似系数小,说明其亲缘关系较远,而3号和5号菌株相似系数超过了95%,说明两者亲缘性非常近,很可能来源于同一菌株。
由表 7可见,14株香菇菌株相互间的遗传相似距离在0.658 3~0.958 3之间,其中3号和5号菌株相似距离最大(0.958 3),6号与13号菌株相似距离最小(0.658 3),1、6、9号与12号菌株相似距离仅0.671 6,即亲缘性最近的是3号与5号菌株。该结果与拮抗性试验和ITS序列分析结果基本一致。
3 结论香菇是最重要的食用菌之一,在中国已有上千年的栽培历史。由于地域、分类标准及来源等原因,造成香菇分类及命名混乱。在香菇属建立起来之后,分类学家们对香菇进行分类,但由于子实体的形态会随外界环境因素及地域的影响而变化,仅根据形态学进行分类并不能建立稳定的分类系统[14]。因此,为了保证菌株的可持续栽培,建立一个可靠的遗传多样性评价方法尤为重要[15]。
本研究通过拮抗性试验对14个香菇菌株进行了初步鉴定,结果表明,3号与5号菌株;4号与5、11号菌株;5号与8号菌株;6号与7号菌株;11号与14号菌株间没有拮抗反应或拮抗性反应极不明显,说明其亲缘关系很近,其余菌株间均存在着不同程度的拮抗反应,表明它们之间亲缘性关系存在差异。ITS序列分析结果表明,2、3、5、7、8、9号和13号菌株在GenBank中的序列号相同,均为DQ497071.1;4、11和14号序列号相同,为AB286067.1。在GenBank中的序列号相同,说明这些菌株可能来源于同一菌株,但不能确定是同一菌株。RAPD分析结果表明,14个菌株之间的亲缘性均较近,但12号菌株与其他菌株之间均较远,6号与13号菌株亲缘性最远;而3号和5号菌株亲缘性最近,超过0.95。综合来看,3号和5号菌株在拮抗试验中未表现拮抗反应,ITS序列分析中两者同源性达到0.95,RAPD聚类分析中相似系数超过0.95,3种方法研究结果相一致,说明两者源于同一菌株,即同种异名。
本研究对14株香菇菌株进行了初步分类与鉴定,更具体的分类与鉴定还有待进一步研究。
| [1] | 张寿橙, 赖敏男. 中国香菇栽培历史与文化[M]. 上海: 上海科学技术出版社, 1994: 3-5. |
| [2] | 黄年来, 林志彬, 陈国良, 等. 中国食药用菌学(下篇)[M]. 上海: 上海科学技术文献出版社, 2010. |
| [3] | 佩格勒DN, 杨TWK, 姚一建. 香菇的学名及其在当代真菌分类中的位置[J]. 真菌学报, 1993, 12(3): 226–231. |
| [4] | 唐传红, 苏春丽, 张劲松, 等. 灵芝属分类学研究进展[J]. 食用菌学报, 2007, 14(3): 86–90. |
| [5] | ALKA M, ABHAY K P, POOJA S, et al. Mycoparasites of Ganoderma lucidum (Leyss:Fr) karst and their botanical management[J]. Proceedings of the National Academy of Sciences, India Section B:Biological Sciences, 2013, 83(1): 119–123. DOI: 10.1007/s40011-012-0087-0 |
| [6] | WANG X C, XI R J, LI Y, et al. The species identity of the widely cultivated Ganoderma, 'G.lucidum' (Ling-zhi), in China[J]. PLoS One, 2012, 7(7): e40857. DOI: 10.1371/journal.pone.0040857 |
| [7] | CHEN S L, XU J, LIU C, et al. Genome sequence of the model medicinal mushroom Ganoderma lucidum[J]. Nature Communications, 2012, 3: 913. DOI: 10.1038/ncomms1923 |
| [8] | 张宇, 郭良栋. 真菌DNA条形码研究进展[J]. 菌物学报, 2012, 31(6): 809–820. |
| [9] | PARK Y J, KWON O J, SON E, et al. Genetic diversity analysis of Ganoderma species and development of a specific marker for identification of medicinal mushroom Ganoderma lucidum[J]. African Journal of Microbiology Research, 2012, 6(25): 5417–5425. |
| [10] | 白树猛, 田黎. ITS序列分析在真菌分类鉴定和分子检测中的应用[J]. 畜牧与饲料科学, 2009, 30(1): 52–53, 189. |
| [11] | WHITE T J, BRUNS T D, LEE S, et al. Analysis of phylogenetic relationships by amplification and direct sequencing of ribosomal RNA genes[M]//INNIS M A, GELFAND D H, SNINSKY J J, et al. PCR protocols:a guide to methods and applications. New York, NY, USA:Academic Press, 1990:15-22. |
| [12] | 李翠翠, 郭立忠, 卢伟东, 等. RAPD和SRAP分子标记在真姬菇菌种鉴定中的应用[J]. 食用菌学报, 2009, 16(1): 21–25. |
| [13] | 龚利娟. 冷季型香菇(Lentinula edodes)菌种的分子鉴定[D]. 长春: 吉林农业大学, 2005. |
| [14] | RICHTER C, WITTSTEIN K, KIRK P M, et al. An assessment of the taxonomy and chemotaxonomy of Ganoderma[J]. Fungal Diversity, 2015, 71(1): 1–15. DOI: 10.1007/s13225-014-0313-6 |
| [15] | 唐传红, 张劲松, 陈明杰, 等. 利用拮抗试验和RAPD对灵芝属菌株进行分类研究[J]. 微生物学通报, 2005, 32(5): 72–76. |
 2017,Vol. 13
2017,Vol. 13


