文章信息
- 郭亚辉, 姚卫蓉, 裴仁军, 周晓东, 胡继明, 钱和
- GUO Yahui, YAO Weirong, PEI Renjun, ZHOU Xiaodong, HU Jiming, QIAN He
- G四链体在生物传感器中的应用
- Application of G- Quadruplexes for the Construction of Biosensors
- 武汉大学学报(理学版), 2017, 63(1): 9-21
- Journal of Wuhan University(Natural Science Edition), 2017, 63(1): 9-21
- http://dx.doi.org/10.14188/j.1671-8836.2017.01.002
-
文章历史
- 收稿日期:2015-12-24

2. 中国科学院 苏州纳米技术与纳米仿生研究所,江苏 苏州 215123;
3. 武汉大学 化学与分子科学学院/生物医学分析化学教育部重点实验室,湖北 武汉 430072
2. Suzhou Institute of Nano- Tech and Nano- Bionics, Chinese Academy of Sciences, Suzhou 215123, Jiangsu, China;
3. College of Chemistry and Molecular Sciences / Key Laboratory of Analytical Chemistry for Biology and Medicine of Ministry of Education, Wuhan University, Wuhan 430072, Hubei, China
生物传感器的概念起源于1962年Clark和Lyons设计的葡萄糖检测电极,即基于葡萄糖氧化酶与葡萄糖的相互作用而产生电信号用以检测样品中葡萄糖的浓度[1].生物传感器主要包括两个部分:信号识别单元和信号转化单元.识别单元一般对待测物具有高特异性强亲和力的结合作用,早期传感器一般使用特异性的酶作为识别单元,后来发展到使用有机小分子、抗体、多肽、适配体甚至细胞等.信号转化单元则将识别结合等相互作用转化为各种物理化学信号,比如荧光、颜色、电化学信号等[2].生物传感器广泛应用于许多重要的分析科学领域,如疾病诊断与医学成像、环境污染监测、刑事侦查、军事反恐、食品安全分析等.据文献预计,2015年生物传感器的全球市场产值可能达到120亿美元[3].得益于DNA特殊的物理化学性质,其在生物传感器中的应用已引起了广大研究者的关注,并且发展十分迅速.
DNA即脱氧核糖核酸,是由4种脱氧核糖核苷酸(A/T/C/G)组成的长链聚合物,作为一种生物大分子聚合物,负载着大量的遗传信息,是生命的遗传物质[4, 5].不仅如此,由于DNA一些独特的物理化学特性,其在生物传感器方面的应用研究也吸引了越来越多的研究者.首先,随着DNA合成技术的发展,可通过人工合成的方式得到任意人为设定序列的DNA,DNA链间和链端的碱基可以通过化学键合的方式修饰上氨基、羧基、巯基等活性基团,以及荧光染料分子、电化学活性物质分子、蛋白质、抗体、不同的功能性纳米材料等;其次,DNA具有强大的信息储存能力[6],其每一个碱基位点上都有4个变量(A/T/C/G),一段具有N个碱基的序列具有4N种编码信息;此外,DNA是理想的纳米级自组装材料,其调控分辨率达亚纳米级(0.34 nm,即一个碱基的长度),如利用固定在纳米材料表面的DNA对纳米材料进行组装[7, 8],或者基于DNA折纸技术[9]建造纳米级精度的单分子检测样品固定平台[10, 11] 等;DNA还具有多元化的结合活性,如Watson-Crick碱基配对(A=T,C≡G)、嘧啶的N3(T-Hg2+-T,C-Ag+-C)[12, 13]、嘌呤的N7(G四链体,DNA三链)[14, 15]、C6O(可与Ag/Co/Cu等过渡金属作用)[16]以及带负电的磷酸骨架[17];基于DNA组装技术(霍利迪结、四通结等)的DNA折纸术也使得DNA的应用从二维尺度扩展到三维尺度[18, 19].
作为一种特殊的DNA结构,G四链体可与多种离子、小分子、蛋白、酶甚至细胞等相互作用,并且具有结构多样性,作为信号输出单元还具有多种免标记信号输出方式(荧光、化学发光、电化学、比色等),基于G四链体结构可设计不同的信号产生策略,其在生物传感器的构建中具有巨大的应用潜力.
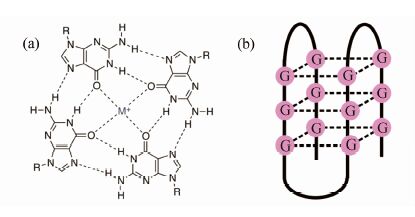
1 G四链体的特点由富G序列形成的G四链体结构(G4)广泛存在于人体染色体的端粒序列与原癌基因的启动子序列,G四链体结构可以抑制端粒酶的活性,从而抑制癌细胞的增殖能力[20, 21],还可抑制原癌基因的转录活性[22],因此在生物医学以及癌症领域中的研究非常重要.G四链体结构是由富含G碱基的DNA所形成,如图1所示,4个G碱基通过Hoogsteen氢键(N1-O6,N2-N7;N1/N2为氢键供体,O6/N7为氢键受体)形成一个G平面,两个以上G平面堆积形成G四链体结构,单价阳离子(如Na+/K+)可以通过络合鸟嘌呤的O6未共用电子对而稳定G四链体的形成[23, 24].
|
| 图 1 G四链体结构 (a) 4个鸟嘌呤形成的一个G平面;(b) 3个G平面构成的G四链体结构 Figure 1 The structure of G4 (a) A G-quartet formed by four guanines;(b) A typical G4 structure with three G-quartets |
除一价离子外,二价金属离子、有机分子配体和染料、酶和蛋白以及细胞等都可以诱导富G序列形成G四链体结构或结合G四链体结构.现已报道的可以诱导或结合G四链体结构的一价离子有Li+[25]、Na+[23]、K+[24]、Rb+[26]、Cs+[27]、NH4+[28];二价金属离子包括:Pb2+[29]、Ca2+[30]、Zn2+[31]、Ba2+[32]、Sr2+[33];三价离子有:Tb3+[34]、Eu3+[35];有机分子配体有四甲基吡啶卟啉(TMPyP4),N-甲基卟啉二丙酸IX(NMM),原卟啉IX(PPIX),二乙烯吡啶咔唑(BMVC)等[36];酶和蛋白有:G4游离酶1(G4R1)[37]、尖毛虫端粒结合蛋白β亚基(βTBP)[38]、酿酒酵母阻抑激活物蛋白(RAP1)[39]、凝血酶(Thrombin)[40]、血管内皮生长因子(VEGF)[41]、核仁蛋白[42, 43] 等.其中,G4R1与G四链体的Kd值达到pmol\5L-1级.富G序列AS1411形成的G四链体结构可以结合几乎所有的癌细胞系,并显示抗增殖诱导凋亡作用[44],具有抗癌活性[45].
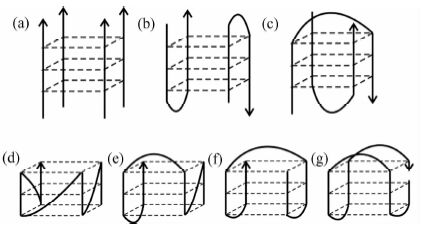
1.2 结构和构象多样性此外,G四链体具有结构多样性.重复G碱基的数目及组合、G平面的数量、G碱基之间连接碱基的序列及长度、组成四链体结构的链数目、G四链体的空间构型等这些可变因素都使得G四链体具有结构多样性.如图2所示,由一条完整的序列分子内折叠形成的G四链体结构称为分子内G四链体;基于链间Hoogsteen氢键作用由多条链形成的G四链体称为分子间G四链体.当组成G四链体结构的4“股”G序列取向相同时(即同为5′→3′或3′→5′)为平行构型;当至少有1“根”G序列取向不同时,即为反平行构型.图2分别图示了分子间、分子内、平行、反平行几种四链体构型.图2(a)为由4条富G碱基的DNA链组成的分子间平行构型G四链体,(b),(c)为由2条富G序列DNA组成的双发卡分子间G四链体构型,(d)所展示的为分子内平行构型的G四链体,(e)为分子内混合型G四链体,(f),(g)为分子内反平行G四链体构型.
|
| 图 2 G四链体结构和构象的多样性 a)~(c) 分子间G四链体构型;(d)~(g) 分子内G四链体 Figure 2 Structure diversity of G4 (a)~(c) Intermolecular G-quadruplex structures;(d)~(g) Intramolecular G4 structures with |
许多研究显示,对于同一富G序列DNA不同离子形成的G四链体构象不同,且不同的有机配体对G四链体的结构和构象具有选择性的结合[46, 47].如Li等[48]报道阴离子卟啉染料(PPIX)对平行结构的G四链体构象具有特异性结合作用,基于PPIX对G四链体平行构象的选择性,构建了检测K+的生物传感器.由于对Na+诱导形成的反平行结构不具有结合活性,该生物传感器即使在100 mmol\5L-1 Na+浓度条件下,对K+的检出限也能达到0.5 mmol\5L-1.
1.3 多种信号表达方式G四链体作为信号输出模块具有多种信号输出方式,本文将其总结为3类.
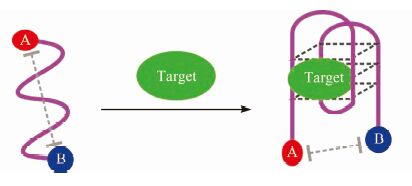
1) 第一类为标记模式.如图3所示,未加入待测物时,富G序列通常处于松散单链状态,一个碱基的长度约为0.34 nm,一段24 bp的富G序列长度约为8.2 nm,5′端与3′端活性作用位点距离较远;当加入待测物,待测物诱导富G序列形成紧密的G四链体结构,而G四链体结构长度约为1.5 nm [49],使两端作用位点靠近,产生相应的作用信号.A/B可以分别是有机荧光染料、无机纳米颗粒、电化学活性物质、电化学界面等,可标记两端也可只标记一端.Ueyama等[50] 将一条富G碱基的DNA两端分别共价标记荧光分子6-FAM和猝灭分子6-TAMRA,当加入K+,富G序列从松散单链状态转换成结构紧密的G四链体结构,使5′/3′两端靠近,两个修饰的荧光分子发生荧光共振能量转移(FRET)效率增强,得到相应的荧光信号用以检测溶液相中的K+.Yue等[51] 基于此原理逆向思维构建了检测DNA的纳米探针,此探针一端修饰有纳米金颗粒,另一端标记有荧光分子6-FAM.当未加入目标序列时,G四链体结构可以形成,富G序列末端标记的荧光分子靠近纳米金表面,6-FAM作为荧光供体纳米金作为受体发生FRET导致荧光猝灭;当目标序列加入时,目标序列与富G序列形成互补双链破坏G四链体结构,使紧密的G四链体结构变成长的刚性DNA双链,使6-FAM远离纳米金表面,纳米金猝灭6-FAM的效率变低,从而得到恢复的6-FAM的荧光信号.另一种机理为碳纳米管、石墨烯、纳米金等[17, 52, 53]与单链之间存在π-π堆积或静电吸附等相互作用吸附单链,当待测物的加入诱导富G序列形成G四链体结构后,由于碱基被G四链体立体结构保护起来,使碱基无法暴露在外与材料相互作用从而解吸附.如Wei等[54] 使用一条标记有6-FAM荧光分子的G四链体构建了酶辅放大检测赭曲霉毒素A(OTA)的荧光生物传感器.OTA的加入会诱导富G序列形成四链体结构将DNA从纳米石墨表面解吸附,同时在DNA酶的作用下将DNA剪切释放标记在DNA上的6-FAM荧光分子,在OTA不断的解吸附作用下,得到放大增强的荧光信号.
|
| 图 3 基于信号分子标记的信号输出方式 Figure 3 Signal output by employing G4 terminally-labeled with signal moieties |
2) 第二类为使用G四链体结合染料的免标记信号输出方式.根据结合后荧光变化特点可将这些荧光染料分为两类:① 结合G四链体结构后荧光增强的化合物;② 结合G四链体结构后荧光强度大大降低的化合物.可以与G四链体结合的染料按照化合物类型分可以分为:花菁类染料(噻唑橙及其衍生物[55, 56]、SYBR Green I[57]、DODC[58]、ETC[59] 等),苯并咪唑衍生物(Hoechst 33258[60]、EtBzEt衍生物[61]等),乙啡啶衍生物(溴化乙锭[62]等),三苯甲烷类染料(孔雀绿[63]、结晶紫[64]等),咔唑类染料(BMVC[65]、CBz-2Py[66] 等),卟啉类染料(NMM[67]、TMPyP4[68]、TrPyP4[69]、PPIX[48]等),具有聚集诱导发光性质的染料(TTAPE[70]、Silole 6[71]等),天然化合物(小檗碱及其衍生物[72]、槲皮黄酮[73]、喹哗酮类生物碱[74] 等),金属配合物(铂[75]/钌[76] 等过渡金属配合物、酞菁类染料的金属螯合物[77]、镧系金属化合物[35] 等),以及一些其他杂环类化合物(MOQ2[78]、BOQ1[79] 等)和氨茴环霉素类药物(阿霉素[80]、萨巴比星[81] 等).如Zhang等[82] 基于ZnPPIX可以与G四链体结构结合并且荧光增强的性质,使用G四链体结构作为免标记的荧光信号输出模块,构建了免标记荧光生物传感器用于检测三磷酸腺苷(ATP).
3) 第三类为基于G4/Hemin拟酶的多种信号输出方式(包括标记、免标记以及混合方式).Hemin(Fe(Ⅲ)-protoporphyrin IX,氯高铁血红素)是过氧化氢酶的活性辅助因子,Hemin自身也具有催化过氧化氢氧化的活性,但是活性远远低于使用Hemin作为辅助因子的酶,比如辣根过氧化氢酶[83, 84].Travascio等研究者发现Hemin可以与G四链体结构结合形成G四链体/Hemin复合物,此G四链体/Hemin复合物的催化效率是单独Hemin催化效率的约250倍[85],具有类似辣根过氧化氢酶的活性,被称为基于G四链体的DNA拟酶(DNAzyme).G四链体/Hemin拟酶的出现,使得许多基于辣根过氧化酶的氧化比色反应都可以使用G四链体/Hemin拟酶代替.如图4(a)所示,常见的氧化比色检测中的氧化底物ABTS(2,2′-Azinobis-(3-ethylbenzthiazoline-6-sulphonate))在H2O2的存在下氧化生成有色的氧化产物,H2O2氧化无色的ABTS2-为绿色的ABTS-,ABTS-最大吸收波长为420 nm.G四链体/Hemin拟酶的形成可以催化加速H2O2的氧化效率,得到明显增强的吸收变化信号.G四链体/Hemin拟酶除了可以催化H2O2氧化ABTS2-、TMB等变色,还可以催化H2O2氧化鲁米诺产生化学发光.如图4(b)所示,在H2O2和鲁米诺的存在条件下,只有Hemin存在时,其催化效率非常低,产生很弱的化学发光,G四链体形成后,与Hemin结合形成的DNA拟酶复合物具有辣根过氧化酶活性,大大增强Hemin 的催化活性,得到放大的鲁米诺化学发光信号.Pelossof等[86]首次基于G四链体/Hemin拟酶构建了一个生物电催化的电化学放大检测方法.固定在电极表面的G四链体/Hemin拟酶会催化H2O2氧化产生阴极电流,随着H2O2的浓度增加,阴极电流越来越大,相比单独存在的Hemin,电流得到了大大的增强.
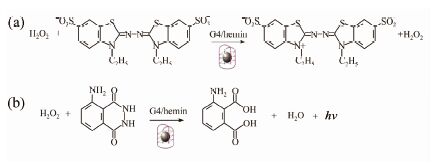
|
| 图 4 G四链体/Hemin拟酶催化双氧水氧化 (a) ABTS2-与(b) 鲁米诺反应方程式 Figure 4 Chemical reaction equations of ABTS2- (a) and Luminol (b) oxidization catalyzed by G4/Hemin DNAzyme |
G四链体作为信号输出模块在生物传感器中的应用非常广泛,用于荧光、化学发光、电化学、比色、拉曼等检测方法的构建[87, 88].得益于G四链体多元的作用活性、结构和构象多样性以及多种信号输出方式,其在生物传感器中的应用十分广泛.作者基于大量的文献总结,将其在生物传感器中的应用主要分为五种策略:直接作用、封闭/解封闭、分段G四链体探针、聚合酶辅助放大策略和表面富集策略.
2.1 与G四链体结构直接作用的策略前文已经提到许多物质可以与富G序列作用,诱导并稳定G四链体结构的形成.这一类型分析方法的第一种方案主要是基于离子、小分子、蛋白等可以诱导稳定G四链体的形成,从而与相应的信号输出配体作用,产
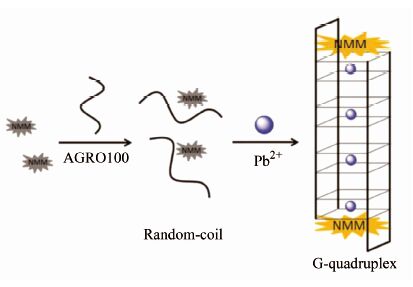
生信号输出.如图5,Xu等[89]使用NMM作为免标记荧光信号配体,构建了基于G四链体的Pb2+荧光生物传感器.当检测体系中加入Pb2+时,诱导富G序列AGRO100形成分子间G四链体结构,NMM从而与之结合,产生增强的荧光信号,其检测线性区间为0~1 000 nmol\5L-1,检出限为3 nmol\5L-1.
基于此策略,本课题组Guo等[90]使用氧化石墨烯及染料噻唑橙(TO)设计了免标记检测Zn2+的荧光生物传感器,Zn2+的加入会诱导富G序列H22形成G四链体结构并从氧化石墨烯表面脱离,进而与TO结合并增强其荧光发射强度.该方法的线性范围为0~30 μmol\5L-1,检出限达0.71 μmol\5L-1,可用于血清中Zn2+浓度的测定.Yan等[91] 使用N掺杂的TiO2作为可见光区域光敏吸收材料,基于G4/Hemin复合物对光致电流的猝灭作用,构建了检测K+的光电化学生物传感器.检测体系中K+的加入会诱导松散状态的富G序列PS2.M形成G四链体结构,并结合游离Hemin形成G4/Hemin复合物,由于Hemin作为电子受体与N掺杂的TiO2发生了电子转移,从而减弱了光生电流强度.光电生物传感器由于激发信号(光)与检测信号(电)的完全分离,具有检测背景低灵敏度高的优点,有很好的应用前景.
2.2 G四链体序列被双链封闭/解封闭策略这是基于G四链体结构的生物传感器中比较常用的策略,即将G四链体结构作为传感器的免标记信号输出模块,一般是经过方法的前面几步反应后,最后的信号表达步骤.Hu等[92] 基于此策略构建了一个免标记荧光检测核糖核酸酶H活性的分析方法,及其抑制剂浓度的免标记荧光分析方法.一段互补RNA序列可以与一条形成G四链体结构的富G序列互补形成双链,封闭G碱基抑制其形成G四链体结构,当加入RNase H后会将RNA序列剪切,解除对G碱基的封闭,在K+存在下此富G序列形成G四链体结构,以NMM(N-甲基卟啉二丙酸Ⅸ,与G四链体作用后荧光有大于20倍增强)作为荧光信号输出配体,得到增强的荧光信号.此方法,简单不需要繁琐的操作、免标记成本低且只需30 min即可完成检测.如图6所示,本课题组Guo等[87]设计了一个含有富G序列的DNA茎环结构,并构建了检测病毒核酸的免标记荧光生物传感器.茎环探针由于分子内碱基互补作用富G序列被封闭无法形成G四连体,当加入目标序列的核酸分子时,核酸分子与环部序列互补配对形成刚性双链结构,从而打开茎环结构,解除对富G序列的封闭作用,在溶液中K+的作用下形成G四连体结构,结合对G四连体特异性的染料NMM并增强荧光其荧光发射强度.此传感器为均相检测方法,即混即测,操作简单,30 min即可完成,线性范围为0~160 nmol\5L-1.
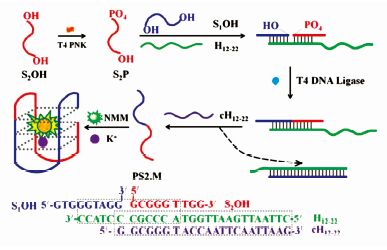
Zhou等[93] 基于足点置换反应设计了免标记检测T4 PNK磷酸化酶活性的荧光生物传感器.如图7所示,在磷酸化酶的作用下S2P的5′-OH被磷酸化,随后首尾相接分别与模版链部分互补配对,在连接酶的作用下,两段富含G碱基的序列被连接为完整的一条可以形成G四链体的DNA,但此时由于模板链的封闭作用G四链体并不能形成,当加入与模版链完全互补的辅助链cH12-22后,由于足点置换反应,富G序列被完全置换下来,解除封闭,在K+的作用下,形成G四链体并增强染料NMM的荧光强度.
分段G四链体一般由两段富G序列(一般是由完整的G四链体序列切割而成)组成,富G序列分为两部分,一部分是可以形成G四链体结构的富G序列片段;另一部分是侧翼链,一般是目标序列DNA结合部分或者待测物的适配体序列.侧翼链与目标序列DNA结合,或者由于待测物与含适配体序列的侧翼链的结合,起到“桥梁”的作用使两段富G序列靠近,形成DNA分子间的Hoogsteen氢键,组成分段G四链体结构.Kolpashchikov[94]以分段G四链体为探针,Hemin为G四链体配体,以G四链体/Hemin复合物催化H2O2氧化DAB(3,3′-二氨基联苯胺)变色为信号输出,检测了单核苷酸的突变.此探针的信噪比(S/N)约为10,可以可视化比色鉴别目标序列的单个碱基突变.Ren等[95] 使用一种逆向设计思路构建了一个免标记生物传感器,用以检测生物分子(ATP/NAD+)、DNA连接酶、单核苷酸突变.该研究首先将“桥梁”链用两小段DNA封闭(即形成互补双链),当连接酶辅助因子(ATP/NAD+)未加入到检测体系中、连接酶失活、或者模板分子存在碱基突变时,两小段DNA不会被连接酶连接成完整的DNA,而会被两富G序列的与“桥梁”链完全互补的侧翼替换,两段富G序列以“桥梁”链为模板形成分子间分段G四链体结构,增强信号配体NMM的荧光强度;反之,两小段DNA会被连接,形成完整的序列,富G序列无法将其替换下来,分子间G四链体无法形成,信号配体NMM没有荧光增强.
本课题组Guo等[88] 通过对一系列序列进行荧光强度进行对比后,进行不同比例的切割,以卟啉染料NMM作为G四链体的结构表征荧光染料,得到一组信噪比大于20的分段G四链体探针,并使用此探针作为免标记信号输出,构建了一组检测T4 DNA连接酶与T4 DNA磷酸化活性的方案.如图8所示,当T4 DNA 5′磷酸化酶以及T4 DNA 连接酶存在时,引物B2的5′端羟基将被磷酸化酶活化为磷酸基团,继而在连接酶的催化作用下,引物B1的3′端羟基与引物B2的5′端磷酸基形成磷酸二酯键,B1/B2被连接为完整的B0桥链.由于足点置换反应,两条富G序列的结合部分会与完整的B0链完互补,从而将H15替换下来,而连接之后的B0会使两条富G序列相互靠近,形成分子间G四链体结构,从而增强NMM的荧光,得到荧光增强信号.该检测方案不需要复杂的合成提纯步骤,不需要材料及电极等的修饰活化,步骤较少、操作简单,对T4 DNA连接酶和T4 DNA PNK酶的检出限分别达到0.012 U\5mL-1和0.17 U\5mL-1.
外加待测物的加入会激活基于聚合酶(使DNA复制链按模板顺序延长)的活性,或者释放聚合引物序列促进引物与模板的互补,抑或形成一段完整聚合模板(即富C序列),在聚合酶的作用下以富C序列为模板合成扩增并释放出大量富G序列,在缓冲溶液体系中(一般含有K+/Na+等),与相应的信号输出配体结合后得到放大的免标记信号输出.
1) 第一种聚合酶辅助信号放大方法为滚环扩增方法(rolling circle amplification,RCA),即以圆形DNA为模板,在聚合酶的作用下,合成与模板互补的连续重复序列,达到序列放大的目的.如图9所示,Xue等[96]基于滚环扩增的放大策略构建了定量ATP/NAD+的荧光传感器,以一个哑铃状DNA结构为探针,以NMM为G四链体结合配体作为免标记荧光输出信号分子.ATP/NAD+作为连接酶的辅助因子使连接酶将哑铃探针链接为一个完整的环状DNA,在Phi29聚合酶的作用下以环状DNA为模板(含富G序列的互补序列)合成连续的大量的富G序列.随后形成的大量G四链体结构与特异性染料NMM作用,得到放大增强的荧光输出信号.该方法可以检测到0.5 pmol\5L-1的ATP以及100 fmol\5L-1的NAD+,并且可以用于细胞培养液和细胞内的实际样品浓度检测.
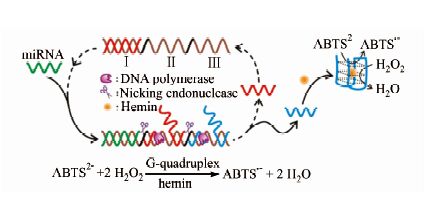
Jiang等[97] 采用类似的放大策略,以Hemin为G四链体配体,构建了放大检测连接酶以及磷酸化酶的比色传感器,该方法检出限分别达到了0.001 9和0.001 8 U\5mL-1.Zhang等[98] 研究者基于DNA脱氧核酶和滚环扩增建立了一个二次放大检测miRNA的电化学生物传感器,miRNA的加入会启动一个基于Pb2+-DNA脱氧核酶的放大步骤,释放大量滚环扩增步骤的引物,然后引物启动滚环扩增步骤,合成大量的富G序列形成G四链体,继而以Hemin/G四链体拟酶催化H2O2氧化鲁米诺发光增强产生的电化学发光信号.该方法的检出限达到了0.3 fmol\5L-1,重现性好(RSD<5%),选择性高,可以应用于细胞裂解液中miRNA的检测.
1) 第二种聚合酶辅助的信号放大方法为聚合酶与限制性内切酶双酶辅助的链扩增方法.He等[99]设计了一种双酶辅助的放大检测策略,并基于此放大策略构建了一个免标记连接酶活性的比色检测方法.该方法以G四链体为免标记信号输出模块,以Hemin/G四链体催化H2O2氧化ABTS2-得到吸收变化信号.Nt.BbvC Ⅰ是一种限制性内切酶,只剪切特定序列双链中的单链,内切酶在剪切位点将链切断后,聚合酶随后从切点的3′末端以未切序列(富G序列的互补序列)为模板延伸合成DNA,随着聚合的延长继而将下游片段替换下来,合成的DNA随后又被切断替换,此过程循环发生,从而产生大量的富G序列,达到序列放大的目的,进而得到放大的配体作用信号.如图10所示,Wang等(2013)[100]研究者基于此双酶(剪切酶-聚合酶)放大策略构建了一个miRNA的免标记比色传感器.此检测方法巧妙地设计了两个剪切位点,同时放大目标miRNA的序列以及形成G四链体结构的富G序列.模板DNA分为3部分,分别是miRNA互补序列、miRNA序列和富C序列.当目标miRNA加入到检测体系中,miRNA结合模板作为聚合酶的引物在模板上合成互补序列;随后限制性内切酶分别在剪切位点切断合成DNA,释放具有miRNA序列的DNA进入下一个循环,同时释放合成的富G序列,形成G四链体/Hemin拟酶复合物,催化H2O2氧化ABTS2-形成绿色的ABTS-.Xue等[101] 以DNA甲基化后被酶剪切释放的DNA为聚合酶的合成模板引物,使用相同的放大策略(双酶双放大)以荧光染料ZnPPIX为G四链体作用荧光信号输出配体,检测了DNA甲基化酶的活性,该方法对甲基化酶的检出限达到8.6×10-5 U\5mL-1.
富G序列组装富集策略一般用于构建电化学、光电化学、表面等离子体共振等生物传感器,基本程序是使用纳米金等纳米材料负载或者基于一些DNA组装技术,将富G序列富集到检测器表面,加入电化学活性的配体、催化活性配体(Hemin)、或者光学活性配体与G四链体结构作用后,达到免标记信号输出放大的目的.
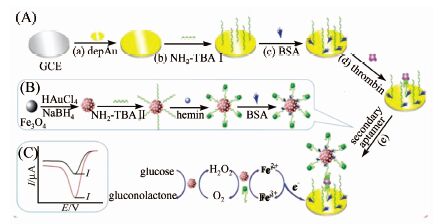
Jing等[102] 基于Fe3O4-Au纳米簇组装微球,使用凝血酶的两个适配体序列构建了凝血酶的电化学多级放大生物传感器.Fe3O4-Au纳米簇组装微球具有很大的表面积,因此可以负载大量富G序列到电极表面,以G四链体/Hemin拟酶催化H2O2产生电荷电子转移得到增强的电化学信号.如图11所示,当体系中加入待测蛋白凝血酶时,凝血酶与电极表面的适配体及微球表面修饰的适配体形成三明治夹心结构,将纳米微球聚集到电极表面.由于Fe3O4表面的Au纳米簇具有拟葡萄糖氧化酶活性,可以催化水中溶解氧氧化葡萄糖产生H2O2.继而大量负载在纳米微球表面的富G序列形成的G四链体/Hemin 拟酶,与Fe3O4核共同催化H2O2还原发生电子转移,产生电化学信号.此传感器的检测线性范围为0.1 pmol\5L-1~20 nmol\5L-1,检出限低至0.013 pmol\5L-1,对类似物具有很好的选择性,并且可用于实际血清样品的分析.
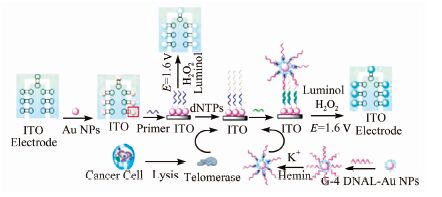
如图12所示,Zhang等[103]基于纳米金负载富G序列信号放大策略,在微阵列芯片表面构建了一个可视化电化学发光传感器检测端粒酶的活性.该研究在ITO玻片表面集成了7个独立检测单元,可以同时得到7组可视化检测信号.将纳米金固定在检测单元里,同时纳米金表面修饰了端粒酶引物序列,加入从HL-60癌细胞(人早幼粒白血病细胞)溶解产物中提取的端粒酶后,端粒酶将引物延长,随后延长的序列通过与DNAzyme-L-纳米金表面的序列形成互补双链,将其富集到电极表面.由于DNAzyme-L-纳米金表面负载了大量的富G序列,同时修饰了鲁米诺,在G四链体/Hemin拟酶的催化下,H2O2氧化鲁米诺产生电化学发光,得到可视化信号.得益于此方法极高的灵敏度,其最低可以可视化检测到313个癌细胞的端粒酶活性.
此基于大量富G序列组装、富集的检测策略一般用于界面上的反应及检测,因为游离分散的G四链体结构与富集到反应界面的G四链体结构所引起的信号变化不同,而富集到界面的G四链体结构在与其特异性作用配体结合后,可以达到催化放大、电荷转移等放大信号输出的作用.
3 总结及展望以上总结了大部分基于G四链体结构的生物传感器的构建策略,但是不能概括所有.G四链体具有多元结合活性、结构和构象多样性以及多种信号表达方法(包括修饰信号分子的标记方法、使用荧光染料的免标记方法和使用DNAzyme 的方法)的特点,在生物传感器中已显示出巨大的应用能力及潜力.但目前该研究领域仍存在一些问题和挑战:第一,由于G四链体多元的结合活性,在一些复杂样品体系的检测中,传感器的选择性较差,信号容易受到干扰;第二,缺少高信噪比、高特异性的荧光染料,目前发现的一些荧光染料既可与四链体结构作用,又可与双链或三链等核酸结构作用;第三,虽然已有很多研究成果,但商品化的检测试剂盒和检测标准体系尚待建立.
笔者认为,基于G四链体的生物传感器未来研究的方向有:1) 基于G四链体多元的结合活性和结构多样性,利用逻辑门的概念进行多目标物的检测和鉴别;2) 发展多信号输出的比率传感器,降低干扰物的影响,增加输出信号的可靠性;3) 多种检测策略的组合使用,借助核酸酶和具有良好性能的纳米材料等发展具有高灵敏且快速的检测方法.未来结合DNA纳米技术、适配体技术、生物酶工具、新功能纳米材料等将大大促进生物传感器的发展及其在分析化学和生物医学分析中的应用.
| [1] | CLARK L C, LYONS C. Electrode systems for continuous monitoring in cardiovascular surgery[J]. Annals of the New York Academy of Sciences, 1962, 102 : 29–45. DOI:10.1111/j.1749-6632.1962.tb13623.x. |
| [2] | LIU J W, CAO Z H, LU Y. Functional nucleic acid sensors[J]. Chemical Reviews, 2009, 109(5) : 1948–1998. DOI:10.1021/cr030183i. |
| [3] | KIRSCH J, SILTANEN C, ZHOU Q, et al. Biosensor technology: Recent advances in threat agent detection and medicine[J]. Chemical Society Reviews, 2013, 42(22) : 8733–8768. DOI:10.1039/c3cs60141b. |
| [4] | WATSON J D, CRICK F H. Genetical implications of the structure of deoxyribonucleic acid[J]. Nature, 1953, 171(4361) : 964–967. |
| [5] | GRIFFITH F. The significance of pneumococcal types[J]. Journal of Hygiene, 1928, 27(2) : 113–159. DOI:10.1017/S0022172400031879 |
| [6] | GOLDMAN N, BERTONE P, CHEN S, et al. Towards practical, high-capacity, low-maintenance information storage in synthesized DNA[J]. Nature, 2013, 494(7435) : 77–80. DOI:10.1038/nature11875. |
| [7] | MIRKIN C A, LETSINGER R L, MUCIC R C, et al. A DNA-based method for rationally assembling nanoparticles into macroscopic materials[J]. Nature, 1996, 382(6592) : 607–609. DOI:10.1038/382607a0. |
| [8] | QI H, GHODOUSI M, DU Y N, et al. DNA-directed self-assembly of shape-controlled hydrogels[J]. Nature Communications, 2013, 4 : 2275. DOI:10.1038/Ncomms3275. |
| [9] | SEEMAN N C. Nanomaterials based on DNA[J]. Annual Review of Biochemistry, 2010, 79 : 65–87. DOI:10.1146/annurev-biochem-060308-102244. |
| [10] | TINNEFELD P. Single-molecule detection: Breaking the concentration barrier[J]. Nature Nanotechnology, 2013, 8(7) : 480–482. DOI:10.1038/nnano.2013.122. |
| [11] | ACUNA G P, MOLLER F M, HOLZMEISTER P, et al. Fluorescence enhancement at docking sites of DNA-directed self-assembled nanoantennas[J]. Science, 2012, 338(6106) : 506–510. DOI:10.1126/science.1228638. |
| [12] | LI T, DONG S J, WANG E K. Label-free colorimetric detection of aqueous mercury ion (Hg2+) using Hg2+-modulated G-quadruplex-based DNAzymes[J]. Analytical Chemistry, 2009, 81(6) : 2144–2149. DOI:10.1021/ac900188y. |
| [13] | ONO A, CAO S Q, TOGASHI H, et al. Specific interactions between silver(Ⅰ) ions and cytosine-cytosine pairs in DNA duplexes[J]. Chemical Communications, 2008(39) : 4825–4827. DOI:10.1039/b808686a. |
| [14] | SEN D, GILBERT W. Formation of parallel four-stranded complexes by guanine-rich motifs in DNA and its implications for meiosis[J]. Nature, 1988, 334(6180) : 364–366. DOI:10.1038/334364a0. |
| [15] | CHRISTOPHE D. H-form DNA and the hairpin-triplex model[J]. Nature, 1988, 333(6170) : 214. DOI:10.1038/333214a0. |
| [16] | ZHOU X H, KONG D M, SHEN H X. Ag+ and cysteine quantitation based on G-quadruplex-hemin DNAzymes disruption by Ag+[J]. Analytical Chemistry, 2010, 82(3) : 789–793. DOI:10.1021/ac902421u. |
| [17] | XIA F, ZUO X L, YANG R Q, et al. Colorimetric detection of DNA, small molecules, proteins, and ions using unmodified gold nanoparticles and conjugated polyelectrolytes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(24) : 10837–10841. DOI:10.1073/pnas.1005632107. |
| [18] | HAN D R, PAL S, YANG Y, et al. DNA gridiron nanostructures based on four-arm junctions[J]. Science, 2013, 339(6126) : 1412–1415. DOI:10.1126/science.1232252. |
| [19] | PEI H, LIANG L, YAO G B, et al. Reconfigurable three-dimensional DNA nanostructures for the construction of intracellular logic sensors[J]. Angewandte Chemie-International Edition, 2012, 51(36) : 9020–9024. DOI:10.1002/anie.201202356. |
| [20] | JING N J, LI Y D, XIONG W J, et al. G-quartet oligonucleotides: A new class of signal transducer and activator of transcription 3 inhibitors that suppresses growth of prostate and breast tumors through induction of apoptosis[J]. Cancer Research, 2004, 64(18) : 6603–6609. DOI:10.1158/0008-5472.Can-03-4041. |
| [21] | VERDUN R E, KARLSEDER J. Replication and protection of telomeres[J]. Nature, 2007, 447(7147) : 924–931. DOI:10.1038/Nature05976. |
| [22] | SIDDIQUI-JAIN A, GRAND C L, BEARSS D J, et al. Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99 : 11593–11598. DOI:10.1073/pnas.182256799. |
| [23] | KETTANI A, BOUAZIZ S, GORIN A, et al. Solution structure of a Na cation stabilized DNA quadruplex containing G.G.G.G and G.C.G.C tetrads formed by G-G-G-C repeats observed in adeno-associated viral DNA[J]. Journal of Molecular Biology, 1998, 282(3) : 619–636. DOI:10.1006/jmbi.1998.2030. |
| [24] | HAIDER S, PARKINSON G N, NEIDLE S. Crystal structure of the potassium form of an Oxytricha nova G-quadruplex[J]. Journal of Molecular Biology, 2002, 320 : 189–200. DOI:10.1016/S0022-2836(02)00428-X. |
| [25] | CREZE C, RINALDI B, HASER R, et al. Structure of a d(TGGGGT) quadruplex crystallized in the presence of Li+ ions[J]. Acta Crystallographica Section D, Biological Crystallography, 2007, 63(6) : 682–688. DOI:10.1107/S0907444907013315. |
| [26] | IDA R, WU G. Solid-state 87Rb NMR signatures for rubidium cations bound to a G-quadruplex[J]. Chemical Communications, 2005, 14(34) : 4294–4296. DOI:10.1039/b505674h. |
| [27] | MARINCOLA F C, VIRNO A, RANDAZZO A, et al. Effect of rubidium and cesium ions on the dimeric quaduplex formed by the Oxytricha nova telomeric repeat oligonucleotide d(GGGGTTTTGGGG)[J]. Nucleosides Nucleotides Nucleic Acids, 2007, 26(8-9) : 1129–1132. DOI:10.1080/15257770701521821. |
| [28] | NAGESH N, CHATTERJI D. Ammonium ion at low concentration stabilizes the G-quadruplex formation by telomeric sequence[J]. Journal of Biochemical and Biophysical Methods, 1995, 30(1) : 1–8. DOI:10.1016/0165-022x(94)00057-K. |
| [29] | SMIRNOV I, SHAFER R H. Lead is unusually effective in sequence-specific folding of DNA[J]. Journal of Molecular Biology, 2000, 296(1) : 1–5. DOI:10.1006/jmbi.1999.3441. |
| [30] | MIYOSHI D, NAKAO A, SUGIMOTO N. Structural transition from antiparallel to parallel G-quadruplex of d(G4T4G4) induced by Ca2+[J]. Nucleic Acids Research, 2003, 31(4) : 1156–1163. DOI:10.1093/nar/gkg211 |
| [31] | WEI C, TANG Q, LI C. Structural transition from the random coil to quadruplex of AG(3)(T(2)AG(3))(3) induced by Zn2+[J]. Biophysical Chemistry, 2008, 132(2-3) : 110–113. DOI:10.1016/j.bpc.2007.10.013. |
| [32] | ZHANG D N, HUANG T, LUKEMAN P S, et al. Crystal structure of a DNA/Ba2+ G-quadruplex containing a water-mediated C-tetrad[J]. Nucleic Acids Research, 2014, 42(21) : 13422–13429. DOI:10.1093/Nar/Gku1122. |
| [33] | CHEN F M. Sr2+ facilitates intermolecular G-quadruplex formation of telomeric sequences[J]. Biochemistry, 1992, 31(15) : 3769–3776. DOI:10.1021/bi00130a006 |
| [34] | NAGESH N, BHARGAVA P, CHATTERJI D. Terbium(Ⅲ)-induced fluorescence of four-stranded G4-DNA[J]. Biopolymers, 1992, 32(10) : 1421–1424. DOI:10.1002/bip.360321015. |
| [35] | GALEZOWSKA E, GLUSZYNSKA A, JUSKOWIAK B. Luminescence study of G-quadruplex formation in the presence of Tb3+ ion[J]. Journal of Inorganic Biochemistry, 2007, 101(4) : 678–685. DOI:10.1016/j.jinorgbio.2006.12.013. |
| [36] | VUMMIDI B R, ALZEER J, LUEDTKE N W. Fluorescent probes for G-Quadruplex Structures[J]. Chembiochem: A European Journal of Chemical Biology, 2013, 14(5) : 540–558. DOI:10.1002/cbic.201200612. |
| [37] | CREACY S D, ROUTH E D, IWAMOTO F, et al. G4 resolvase 1 binds both DNA and RNA tetramolecular quadruplex with high affinity and is the major source of tetramolecular quadruplex G4-DNA and G4-RNA resolving activity in HeLa cell lysates[J]. Journal of Biological Chemistry, 2008, 283(50) : 34626–34634. DOI:10.1074/jbc.M806277200. |
| [38] | FANG G W, CECH T R. The beta subunit of Oxytricha telomere-binding protein promotes G-quartet formation by telomeric DNA[J]. Cell, 1993, 74(5) : 875–885. DOI:10.1016/0092-8674(93)90467-5 |
| [39] | GIRALDO R, RHODES D. The yeast telomere-binding protein RAP1 binds to and promotes the formation of DNA quadruplexes in telomeric DNA[J]. The EMBO Journal, 1994, 13(10) : 2411–2420. |
| [40] | BOCK L C, GRIFFIN L C, LATHAM J A, et al. Selection of single-stranded-DNA molecules that bind and inhibit human thrombin[J]. Nature, 1992, 355(6360) : 564–566. DOI:10.1038/355564a0. |
| [41] | NONAKA Y, SODE K, IKEBUKURO K. Screening and improvement of an anti-VEGF DNA aptamer[J]. Molecules, 2010, 15(1) : 215–225. DOI:10.3390/molecules15010215. |
| [42] | ISHIKAWA F, MATUNIS M J, DREYFUSS G, et al. Nuclear proteins that bind the premessenger RNA 3′ splice-site sequence R(Uuag/G) and the human telomeric DNA-sequence D(Ttaggg)N[J]. Molecular and Cellular Biology, 1993, 13(7) : 4301–4310. DOI:10.1128/MCB.13.7.4301 |
| [43] | DICKINSON L A, KOHWI-SHIGEMATSU T. Nucleolin is a matrix attachment region DNA-binding protein that specifically recognizes a region with high base-unpairing potential[J]. Molecular and Cellular Biology, 1995, 15(1) : 456–465. DOI:10.1128/MCB.15.1.456 |
| [44] | BATES P J, LABER D A, MILLER D M, et al. Discovery and development of the G-rich oligonucleotide AS1411 as a novel treatment for cancer[J]. Experimental and Molecular Pathology, 2009, 86(3) : 151–164. DOI:10.1016/j.yexmp.2009.01.004. |
| [45] | KOCHETKOVA M, IVERSEN P O, LOPEZ A F, et al. Deoxyribonucleic acid triplex formation inhibits granulocyte macrophage colony-stimulating factor gene expression and suppresses growth in juvenile myelomonocytic leukemic cells[J]. Journal of Clinical Investigation, 1997, 99(12) : 3000–3008. DOI:10.1172/JCI119495. |
| [46] | HE H Z, WANG M, CHAN D S, et al. A parallel G-quadruplex-selective luminescent probe for the detection of nanomolar calcium(Ⅱ) ion[J]. Methods, 2013, 64(3) : 212–217. DOI:10.1016/j.ymeth.2013.07.021. |
| [47] | GUO Y H, SUN Y, SHEN X Q, et al. Label-free Detection of Zn2+ Based on G-quadruplex[J]. Analytical Sciences: The International Journal of the Japan Society for Analytical Chemistry, 2015, 31 : 1–5. DOI:10.2116/analsci.31.1 |
| [48] | LI T, WANG E K, DONG S J. Parallel G-quadruplex-specific fluorescent probe for monitoring DNA structural changes and label-free detection of potassium ion[J]. Analytical Chemistry, 2010, 82(18) : 7576–7580. DOI:10.1021/ac1019446. |
| [49] | PERSSON B N J, LANG N D. Electron-hole-pair quenching of excited-states near a metal[J]. Physical Review B, 1982, 26(10) : 5409–5415. DOI:10.1103/PhysRevB.26.5409. |
| [50] | UEYAMA H, TAKAGI M, TAKENAKA S. A novel potassium sensing in aqueous media with a synthetic oligonucleotide derivative. Fluorescence resonance energy transfer associated with Guanine quartet-potassium ion complex formation[J]. Journal of the American Chemical Society,, 2002, 124(48) : 14286–14287. DOI:10.1021/ja026892f |
| [51] | YUE Q L, SHEN T F, WANG C N, et al. Construction of a controllable Forster resonance energy transfer system based on G-quadruplex for DNA sensing[J]. Biosensors and Bioelectronics, 2013, 40(1) : 75–81. DOI:10.1016/j.bios.2012.06.026. |
| [52] | WANG J C, WANG Y S, XUE J H, et al. An ultrasensitive label-free assay of 8-hydroxy-2′-deoxyguanosine based on the conformational switching of aptamer[J]. Biosensors and Bioelectronics, 2014, 58 : 22–26. DOI:10.1016/j.bios.2014.02.047. |
| [53] | LI M, ZHOU X J, GUO S W, et al. Detection of lead (Ⅱ) with a “turn-on” fluorescent biosensor based on energy transfer from CdSe/ZnS quantum dots to graphene oxide[J]. Biosensors and Bioelectronics, 2013, 43 : 69–74. DOI:10.1016/j.bios.2012.11.039. |
| [54] | WEI Y, ZHANG J, WANG X, et al. Amplified fluorescent aptasensor through catalytic recycling for highly sensitive detection of ochratoxin A[J]. Biosensors and Bioelectronics, 2015, 65 : 16–22. DOI:10.1016/j.bios.2014.09.100. |
| [55] | MONCHAUD D, ALLAIN C, TEULADE-FICHOU M P. Development of a fluorescent intercalator displacement assay (G4-FID) for establishing quadruplex-DNA affinity and selectivity of putative ligands[J]. Bioorganic & Medicinal Chemistry Letters, 2006, 16(18) : 4842–4845. DOI:10.1016/j.bmcl.2006.06.067. |
| [56] | ALLAIN C, MONCHAUD D, TEULADE-FICHOU M P. FRET templated by G-quadruplex DNA: A specific ternary interaction using an original pair of donor/acceptor partners[J]. Journal of the American Chemical Society, 2006, 128(36) : 11890–11893. DOI:10.1021/Ja062193h. |
| [57] | XU H, GAO S L, YANG Q, et al. Amplified fluorescent recognition of G-quadruplex folding with a cationic conjugated polymer and DNA intercalator[J]. ACS Applied Materials & Interfaces, 2010, 2(11) : 3211–3216. DOI:10.1021/Am1006854. |
| [58] | PARAMASIVAN S, BOLTON P H. Mix and measure fluorescence screening for selective quadruplex binders[J]. Nucleic Acids Research, 2008, 36(17) : e106. DOI:10.1093/nar/gkn487. |
| [59] | YANG Q F, XIANG J F, YANG S, et al. Verification of specific G-quadruplex structure by using a novel cyanine dye supramolecular assembly: Ⅰ. Recognizing mixed G-quadruplex in human telomeres[J]. Chemical Communications, 2009, 9 : 1103–1105. DOI:10.1039/B820101c. |
| [60] | MAITI S, CHAUDHURY N K, CHOWDHURY S. Hoechst 33258 binds to G-quadruplex in the promoter region of human c-myc[J]. Biochemical and Biophysical Research Communications, 2003, 310(2) : 505–512. DOI:10.1016/j.bbrc.2003.09.052. |
| [61] | JAIN A K, BHATTACHARYA S. Interaction of G-quadruplexes with nonintercalating duplex-DNA minor groove binding ligands[J]. Bioconjugate Chemistry, 2011, 22(12) : 2355–2368. DOI:10.1021/Bc200268a. |
| [62] | GUO Q, LU M, MARKY L A, et al. Interaction of the dye ethidium bromide with DNA containing guanine repeats[J]. Biochemistry, 1992, 31(9) : 2451–2455. DOI:10.1021/bi00124a002 |
| [63] | BHASIKUTTAN A C, MOHANTY J, PAL H. Interaction of malachite green with guanine-rich single-stranded DNA: Preferential binding to a G-quadruplex[J]. Angewandte Chemie International Edition, 2007, 46(48) : 9305–9307. DOI:10.1002/anie.200703251. |
| [64] | KONG D M, MA Y E, WU J, et al. Discrimination of G-quadruplexes from duplex and single-stranded DNAs with fluorescence and energy-transfer fluorescence spectra of crystal violet[J]. Chemistry, 2009, 15(4) : 901–909. DOI:10.1002/chem.200801441. |
| [65] | CHANG C C, WU J Y, CHIEN C W, et al. A fluorescent carbazole derivative: High sensitivity for quadruplex DNA[J]. Analytical Chemistry, 2003, 75(22) : 6177–6183. DOI:10.1021/ac034789i. |
| [66] | DUMAT B, BORDEAU G, FAUREL-PAUL E, et al. N-phenyl-carbazole-based two-photon fluorescent probes: Strong sequence dependence of the duplex vs quadruplex selectivity[J]. Biochimie, 2011, 93(8) : 1209–1218. DOI:10.1016/j.biochi.2011.05.035. |
| [67] | ARTHANARI H, BASU S, KAWANO T L, et al. Fluorescent dyes specific for quadruplex DNA[J]. Nucleic Acids Research, 1998, 26(16) : 3724–3728. DOI:10.1093/nar/26.16.3724 |
| [68] | HAN F X G, WHEELHOUSE R T, HURLEY L H. Interactions of TMPyP4 and TMPyP2 with quadruplex DNA. Structural basis for the differential effects on telomerase inhibition[J]. Journal of the American Chemical Society, 1999, 121(15) : 3571–3570. DOI:10.1021/Ja984153m. |
| [69] | WEI C Y, HAN G Y, JIA G Q, et al. Study on the interaction of porphyrin with G-quadruplex DNAs[J]. Biophysical Chemistry, 2008, 137(1) : 19–23. DOI:10.1016/j.bpc.2008.06.006. |
| [70] | HONG Y, HAUSSLER M, LAM J W, et al. Label-free fluorescent probing of G-quadruplex formation and real-time monitoring of DNA folding by a quaternized tetraphenylethene salt with aggregation-induced emission characteristics[J]. Chemistry, 2008, 14(21) : 6428–6437. DOI:10.1002/chem.200701723. |
| [71] | LUO J, XIE Z, LAM J W, et al. Aggregation-induced emission of 1-methyl-1,2,3,4,5-pentaphenylsilole[J]. Chemical Communications, 2001(18) : 1740–1741. DOI:10.1039/b105159h |
| [72] | ARORA A, BALASUBRAMANIAN C, KUMAR N, et al. Binding of berberine to human telomeric quadruplex-spectroscopic, calorimetric and molecular modeling studies[J]. The FEBS Journal, 2008, 275(15) : 3971–3983. DOI:10.1111/j.1742-4658.2008.06541.x. |
| [73] | SUN H X, TANG Y L, XIANG J F, et al. Spectroscopic studies of the interaction between quercetin and G-quadruplex DNA[J]. Bioorganic & Medicinal Chemistry Letters, 2006, 16(13) : 3586–3589. DOI:10.1016/j.bmcl.2006.03.087. |
| [74] | TAN J H, OU T M, HOU J Q, et al. Isaindigotone derivatives: A new class of highly selective ligands for telomeric G-quadruplex DNA[J]. Journal of Medicinal Chemistry, 2009, 52(9) : 2825–2835. DOI:10.1021/Jm801600m. |
| [75] | MA D L, CHE C M, YAN S C. Platinum(Ⅱ) complexes with dipyridophenazine ligands as human telomerase inhibitors and luminescent probes for G-quadruplex DNA[J]. Journal of the American Chemical Society, 2009, 131(5) : 1835–1846. DOI:10.1021/Ja806045x. |
| [76] | SHI S, GENG X T, ZHAO J, et al. Interaction of [Ru(bpy)2 (dppz)]2+ with human telomeric DNA: Preferential binding to G-quadruplexes over i-motif[J]. Biochimie, 2010, 92(4) : 370–377. DOI:10.1016/j.biochi.2010.01.003. |
| [77] | ALZEER J, VUMMIDI B R, ROTH P J C, et al. Guanidinium-modified phthalocyanines as high-affinity G-quadruplex fluorescent probes and transcriptional regulators[J]. Angewandte Chemie-International Edition, 2009, 48(49) : 9362–9365. DOI:10.1002/anie.200903685. |
| [78] | MERGNY J L, LACROIX L, TEULADE-FICHOU M P, et al. Telomerase inhibitors based on quadruplex ligands selected by a fluorescence assay[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(6) : 3062–3067. DOI:10.1073/pnas.051620698. |
| [79] | TEULADE-FICHOU M P, CARRASCO C, GUITTAT L, et al. Selective recognition of G-quadruplex telomeric DNA by a bis(quinacridine) macrocycle[J]. Journal of the American Chemical Society, 2003, 125(16) : 4732–4740. DOI:10.1021/ja021299j. |
| [80] | MANET I, MANOLI F, ZAMBELLI B, et al. Affinity of the anthracycline antitumor drugs Doxorubicin and Sabarubicin for human telomeric G-quadruplex structures[J]. Physical Chemistry Chemical Physics, 2011, 13(2) : 540–551. DOI:10.1039/c0cp00898b. |
| [81] | MANET I, MANOLI F, ZAMBELLI B, et al. Complexes of the antitumoral drugs Doxorubicin and Sabarubicin with telomeric G-quadruplex in basket conformation: ground and excited state properties[J]. Photochemical & Photobiological Sciences, 2011, 10(8) : 1326–1337. DOI:10.1039/c1pp05065f. |
| [82] | ZHANG Z X, SHARON E, FREEMAN R, et al. Fluorescence detection of DNA, adenosine-5′-triphosphate (ATP), and telomerase activity by zinc(Ⅱ)-protoporphyrin IX/G-quadruplex labels[J]. Analytical Chemistry, 2012, 84(11) : 4789–4797. DOI:10.1021/ac300348v. |
| [83] | JONES P, MANTLE D, DAVIES D M, et al. Hydroperoxidase activities of ferrihemes: heme analogues of peroxidase enzyme intermediates[J]. Biochemistry, 1977, 16(18) : 3974–3978. DOI:10.1021/bi00637a006 |
| [84] | PORTSMOUTH D, BEAL E A. The peroxidase activity of deuterohemin[J]. European Journal of Biochemistry, 1971, 19(4) : 479–487. DOI:10.1111/ejb.1971.19.issue-4 |
| [85] | TRAVASCIO P, LI Y, SEN D. DNA-enhanced peroxidase activity of a DNA-aptamer-hemin complex[J]. Chemistry and Biology, 1998, 5(9) : 505–517. DOI:10.1016/S1074-5521(98)90006-0 |
| [86] | PELOSSOF G, TEL-VERED R, ELBAZ J, et al. Amplified biosensing using the horseradish peroxidase-mimicking DNAzyme as an electrocatalyst[J]. Analytical Chemistry, 2010, 82(11) : 4396–4402. DOI:10.1021/Ac100095u. |
| [87] | GUO Y H, XU P, HU H, et al. A label-free biosensor for DNA detection based on ligand-responsive G-quadruplex formation[J]. Talanta, 2013, 114(30) : 138–142. DOI:10.1016/j.talanta.2013.04.003. |
| [88] | GUO Y H, WANG Q L, WANG Z L, et al. Label-free detection of T4 DNA ligase and polynucleotide kinase activity based on toehold-mediated strand displacement and split G-quadruplex probes[J]. Sensors and Actuators B Chemical, 2015, 214 : 50–55. DOI:10.1016/j.snb.2015.03.013. |
| [89] | XU L J, SHEN X Q, HONG S N, et al. Turn-on and label-free fluorescence detection of lead ions based on target-induced G-quadruplex formation[J]. Chemical Communications, 2015, 51(38) : 8165–8168. DOI:10.1039/c5cc01590a. |
| [90] | GUO Y H, SUN Y, SHEN X Q, et al. Quantification of Zn(II) using a label-free sensor based on graphene oxide and G-quadruplex[J]. Analytical Methods, 2015, 7 : 9615–9618. DOI:10.1039/c5ay01840d. |
| [91] | YAN X, WANG K W, XIE D P, et al. A photoelectrochemical sensor through quenching of photoinduced electrons based on a G-quadruplex/hemin complex[J]. Analytical Methods, 2015, 7(9) : 3697–3700. DOI:10.1039/c5ay00245a. |
| [92] | HU D, PU F, HUANG Z Z, et al. A quadruplex-based, label-free, and real-time fluorescence assay for RNase H activity and inhibition[J]. Chemistry, 2010, 16(8) : 2605–2610. DOI:10.1002/chem.200902166. |
| [93] | ZHOU L, SHEN X Q, SUN N, et al. Label-free fluorescence light-up detection of T4 polynucleotide kinase activity using the split-to-intact G-quadruplex strategy by ligation-triggered and toehold-mediated strand displacement release[J]. The Analyst, 2015, 140(16) : 5450–5453. DOI:10.1039/c5an01032b. |
| [94] | KOLPASHCHIKOV D M. Split DNA enzyme for visual single nucleotide polymorphism typing[J]. Journal of the American Chemical Society, 2008, 130(10) : 2934–2935. DOI:10.1021/ja711192e. |
| [95] | REN J T, WANG J H, WANG J, et al. Inhibition of G-quadruplex assembling by DNA ligation: A versatile and non-covalent labeling strategy for bioanalysis[J]. Biosensors and Bioelectronics, 2014, 51 : 336–342. DOI:10.1016/j.bios.2013.07.059. |
| [96] | XUE Q W, WANG L, JIANG W. A novel label-free cascade amplification strategy based on dumbbell probe-mediated rolling circle amplification-responsive G-quadruplex formation for highly sensitive and selective detection of NAD+ or ATP[J]. Chemical Communications, 2013, 49(26) : 2640–2642. DOI:10.1039/C3cc39064k. |
| [97] | JIANG H X, KONG D M, SHEN H X. Amplified detection of DNA ligase and polynucleotide kinase/phosphatase on the basis of enrichment of catalytic G-quadruplex DNAzyme by rolling circle amplification[J]. Biosensors and Bioelectronics, 2014, 55 : 133–138. DOI:10.1016/j.bios.2013.12.001. |
| [98] | ZHANG P, WU X Y, YUAN R, et al. An “Off-On” electrochemiluminescent biosensor based on DNAzyme-assisted target recycling and rolling circle amplifications for ultrasensitive detection of microRNA[J]. Analytical Chemistry, 2015, 87(6) : 3202–3207. DOI:10.1021/ac504455z. |
| [99] | HE K Y, LI W, NIE Z, et al. Enzyme-regulated activation of DNAzyme: A novel strategy for a label-free colorimetric DNA ligase assay and ligase-based biosensing[J]. Chemistry, 2012, 18(13) : 3992–3999. DOI:10.1002/chem.201102290. |
| [100] | WANG X P, YIN B C, WANG P, et al. Highly sensitive detection of microRNAs based on isothermal exponential amplification-assisted generation of catalytic G-quadruplex DNAzyme[J]. Biosensors and Bioelectronics, 2013, 42 : 131–135. DOI:10.1016/j.bios.2012.10.097. |
| [101] | XUE Q W, LV Y Q, XU S L, et al. Highly sensitive fluorescence assay of DNA methyltransferase activity by methylation-sensitive cleavage-based primer generation exponential isothermal amplification-induced G-quadruplex formation[J]. Biosensors and Bioelectronics, 2015, 66 : 547–553. DOI:10.1016/j.bios.2014.12.017. |
| [102] | JING P, XU W J, YI H Y, et al. An amplified electrochemical aptasensor for thrombin detection based on pseudobienzymic Fe3O4-Au nanocomposites and electroactive hemin/G-quadruplex as signal enhancers[J]. The Analyst, 2014, 139(7) : 1756–1761. DOI:10.1039/c3an02237d. |
| [103] | ZHANG H R, WANG Y Z, WU M S, et al. Visual electrochemiluminescence detection of telomerase activity based on multifunctional Au nanoparticles modified with G-quadruplex deoxyribozyme and luminol[J]. Chemical Communications, 2014, 50(83) : 12575–12577. DOI:10.1039/c4cc06302c |
 2017, Vol. 63
2017, Vol. 63