文章信息
- 段慧芳, 刘娟
- DUAN Huifang, LIU Juan
- 密集卷积神经网络和辅助特征相结合的乳腺组织病理图像有丝分裂检测方法
- Mitosis Detection in Breast Tissue Pathological Images by Combining Additional Features into the DenseNet
- 武汉大学学报(理学版), 2019, 65(5): 434-440
- Journal of Wuhan University(Natural Science Edition), 2019, 65(5): 434-440
- http://dx.doi.org/10.14188/j.1671-8836.2019.05.003
-
文章历史
- 收稿日期:2018-11-23

《2018全球癌症统计数据》[1]显示, 在所有癌症中, 乳腺癌发病率(11.6%)位居第二。乳腺癌的及早发现和及时治疗是提高存活率的关键。因此, 乳腺癌的组织学分级对选择合适的治疗方案和评估治疗效果至关重要。诺丁汉评级系统(Nottingham grading system)[2]是目前最常见的对乳腺癌严重程度进行评级的系统。其中有丝分裂核的个数是乳腺癌分级的重要指标。因此, 准确识别并统计有丝分裂核的个数有助于提高乳腺癌评级结果的准确度。
经苏木精-伊红(hematoxylin-eosin, HE)染色的切片上的有丝分裂核具有特定的形态学特征:核深染、核增大、核膜欠清晰、边缘呈毛刺状。在临床实验中, 从HE切片中统计有丝分裂核的个数是极具挑战性的工作, 往往需要经验丰富的病理学家在40×显微镜下的可见区域(被称为高能量区域, high power field, HPF)内通过肉眼观察, 该过程费时费力, 且主观性强, 结果受经验差异、疲劳程度等因素影响。因此, 开发一个能够自动检测有丝分裂, 统计结果客观的计算机辅助系统是非常必要的。
近年来, 有丝分裂的自动检测算法研究越来越受到学者们的重视。各种国际比赛[3~6]中相继出现有关有丝分裂的课题, 如ICPR2012[3], MICCAI2013[4], ICPR2014[5]等举办的有丝分裂检测竞赛, 其中ICPR2012竞赛的数据在比赛结束后仍然可以公开获得, 因此很多学者继续基于该数据集尝试不同的有丝分裂识别算法。目前, 有丝分裂自动检测方法主要分为两类。1)基于传统机器学习的方法[7~10]。该类方法利用传统的机器学习方法识别有丝分裂核, 首先需要结合有丝分裂核在图像中的特点, 人工设计特征, 再利用监督学习算法构建预测模型。如Humayun[7]利用形态学特征、纹理特征和决策树分类算法训练分类器, 在测试集上的F分数为0.72;Tashk等[9]首先提取候选目标的CLBP (complete local binary pattern)特征, 然后利用支持向量机(SVM)算法构建分类器以识别有丝分裂核, 分类器性能的F分数为0.71;Nateghi等[10]选用Haralick纹理特征等330维特征和SVM对候选区域进行分类, F分数达到0.784 7。传统机器学习在小数据集上就能取得较好的结果, 但是其对特征的选择非常敏感。2)基于深度学习的方法[11~17]。近年来, 卷积神经网络(convolutional neural network, CNN)[18~21]在图像识别领域取得了较好的效果, 在很多相关领域得到广泛关注。因此, 有很多学者提出利用深度学习算法检测有丝分裂。Ciresan等[11]使用最大池卷积神经网络作为检测算法, F分数达到0.78。Wang等[12]提出组合卷积神经网络模型和辅助特征(形态、颜色、纹理特征)的级联方法, 识别候选集中的有丝分裂核, 所得F分数为0.734 5。由于有丝分裂核的个数远远小于非有丝分裂核的个数, 针对训练集中数据不均衡问题, Wahab等[14]在CNN结构基础上, 提出一个两阶段的模型, 该模型在测试数据集上的F分数达到0.79。综上所述, 深度学习方法可以自主学习特征, 但是存在样本不足时模型泛化能力不佳的问题, 其中传统的CNN还存在梯度消失、参数量大、参数冗余等问题。针对以上问题, Huang等[22]提出一种密集连接的卷积网络(DenseNet), 该网络结构可以缓解梯度消失, 加强特征传播, 鼓励特征复用, 极大地减少了参数量。而且在训练数据集不大的情况下, DenseNet仍取得了不错的效果, 在医学图像处理方面具有广阔的应用前景。
因此, 针对传统人工检测方法耗时费力, 受主观因素影响较大以及一般性卷积神经网络和传统机器学习方法对特征选择较敏感等问题, 本文提出利用DenseNet和人工辅助特征(传统手工设计特征)相结合的方法识别有丝分裂核。
1 本文方法本文提出的有丝分裂检测算法首先分割候选目标, 获取候选集; 然后对候选集进行数据增强, 以扩增样本; 最后构建DenseNet和辅助特征相结合的有丝分裂预测模型, 包括密集卷积神经网络的设计和对候选目标辅助特征(强度特征、形态学特征和纹理特征)的提取。
1.1 获取候选目标由于有丝分裂现象只出现在细胞核中, 因此可通过分割细胞核提取候选目标。在被HE染色过的切片中, 核区域的颜色往往比非核区域颜色深, 细胞核的分割主要依赖这一特性。首先, 为了不遗漏包含有丝分裂的核区, 本文将训练集中的35张RGB图像转换成Blue-Ratio(BR)图像[23]以突出细胞核部分, 计算方法如下:
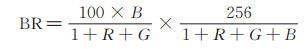
|
(1) |
其中, R、G、B分别表示红色、绿色和蓝色通道。
然后, 为平滑噪声, 采用高斯低通滤波对BR图像进行模糊处理。接着, 用全局阈值对模糊后的图像进行分割, 得到所有的候选区域。由于有丝分裂核比较少, 候选区域中非有丝分裂核远远多于有丝分裂核。最后, 为尽可能多地剔除非有丝分裂核, 本文根据有丝分裂核的尺寸一般不会比非有丝分裂核小的特点, 使用二值阈值和形态学操作去除面积比较小的候选区域。对每个保留下来的连通区域, 以其质心为中心, 剪切适当大小的区域作为候选目标, 构成后续分类使用的样本。图 1(a)为通过RGB图转换得到的BR图像。图 1(b)为分割结果二值图与原RGB图的叠加图, 即候选目标在RGB图上的显示, 其中的黄色部分表示经上述步骤提取的候选目标, 绿色圆圈是标注的有丝分裂核。

|
| 图 1 (a) BR图;(b)候选目标在RGB图上的显示 Fig. 1 (a) BR image; (b) Display of candidate targets on the RGB image |
在深度学习中, 为了避免出现过拟合, 通常需要输入充足的数据, 而在医学图像研究领域, 标注好的数据较少, 所以需要通过数据增强操作得到更多的数据, 达到扩增数据集的目的, 使得深度学习网络具有更好的泛化性能。常用的数据增强方法有旋转、翻转、平移等, 本文采用旋转、上下翻转和左右翻转操作以扩增数据集。
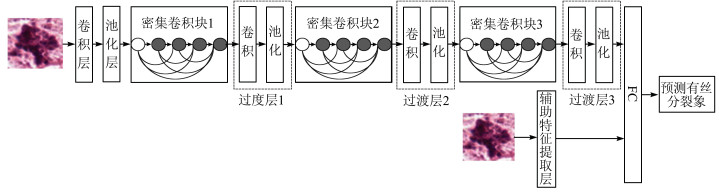
1.3 DenseNet和辅助特征相结合的有丝分裂预测模型本文提出融合辅助特征的DenseNet网络预测模型, 以补充浅层学习特征。其中, DenseNet包括3个密集卷积块。具体网络结构如图 2所示。从图 2中可以看出, 原始图像首先进入由卷积、池化、密集卷积块、过渡层、全连接层(fully align="center"onnected layers, FC)等组成的DenseNet中, 由DenseNet自主提取深度学习特征, 同时原始图像进入辅助特征提取层, 提取图像的形态学特征、强度特征、纹理特征(共43维辅助特征), 然后将深度学习特征与辅助特征共同送入全连接层, 实现有丝分裂的检测。下文将详细介绍DenseNet网络结构以及辅助特征的选择。
|
| 图 2 融合辅助特征的DenseNet结构 Fig. 2 DenseNet structure with the additional features |
传统的卷积神经网络由输入层、卷积层、池化层、全连接层和输出层等组成, 卷积层和池化层交替连接。原始图像进入卷积层, 由卷积层进行特征提取, 获得的特征图再进入池化层, 实现对特征图的降采样处理, 最后由全连接层整合卷积层提取的特征并进行分类。卷积神经网络的提出使得图像分类方法取得了一系列突破[18~21], 但是随着网络深度的增加, 网络训练中会出现梯度消失的问题。
与传统卷积神经网络相比, DenseNet[22]将每一层与之前的所有层相连接, 综合利用了浅层复杂度低的特征和高层复杂度高的特征, 实现特征的重复利用, 使网络更容易得到具有更好泛化性能的决策函数, 具有一定的抗过拟合特性, 有效地缓解了梯度消失的问题。因此本文首先基于DenseNet网络构建有丝分裂分类模型, 提取深度学习特征。具体的DenseNet结构如表 1所示, 主要包含的模块有卷积层、池化层、密集卷积块、过渡层和全连接层。其中, M表示过滤器个数; Conv表示卷积核; Max表示最大池化; 增长率表示每层卷积核的个数; 过渡层由于包含卷积和池化两个操作, 在表中以两行表示。各模块功能介绍如下。
| 层 | 输出特征图大小 | 过滤器尺寸 | 步长 | 增长率 |
| 卷积层 | 32M×70×70 | 2×2Conv | 1 | 32 |
| 池化层 | 24M×35×35 | 2×2Max | 2 | |
| 密集卷积块1 | 104M×35×35 | (3×3Conv)×6 | 1 | 12 |
| 过渡层1 | 104M×32×32 | 4×4Conv | 1 | 104 |
| 104M×16×16 | 2×2Max | 2 | ||
| 密集卷积块2 | 200M×16×16 | (3×3Conv)×8 | 1 | 12 |
| 过渡层2 | 200M×14×14 | 3×3Conv | 1 | 200 |
| 200M×7×7 | 2×2Max | 1 | ||
| 密集卷积块3 | 260M×7×7 | (3×3Conv)×5 | 1 | 12 |
| 过渡层3 | 260M×6×6 | 2×2Conv | 1 | 260 |
| 260M×3×3 | 2×2Max | 2 | ||
| 全连接层 | 2 |
卷积层:一个卷积层包含若干个卷积核, 用以提取图像中不同位置的信息, 并将提取的特征送到下一层卷积中。假设原始输入图像为X, 本文用Yi表示卷积神经网络第i层的特征图, 其中Y0=X。假设Yi-1是卷积层, Yi的产生公式可以描述为:
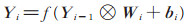
|
(2) |
其中, Wi表示第i层卷积核的权值向量; ⊗表示卷积核和第i-1层图像或者特征图进行卷积操作。卷积的输出与第i层的偏移向量bi相加, 最终通过非线性的激励函数f (x)得到第i层的特征图Yi。
池化层:也称下采样层, 通常跟在卷积层之后, 依据一定的下采样规则对特征图进行池化操作。池化的功能主要有两种:1)对特征图进行降维; 2)在一定程度上保持特征的尺度不变特性, 减小过拟合, 提高算法的容错性和鲁棒性。常见的下采样规则有两种:取平均值(mean-pooling)和取最大值(max-pooling)。本文采用max-pooling方式。
密集卷积块:在密集卷积块中, 以每层之前所有层的输出作为输入。对于L层的传统卷积神经网络, 连接数为L, 对于DenseNet, 连接数则为L(L+1)/2, 这样可以充分利用之前层的所有信息, 同时缩短了前层和后层之间的连接, 有效地解决了随着网络的加深而产生的梯度消失问题[22]。本文设置密集卷积块的增长率为12。第l层特征图生成公式如下:
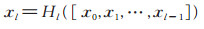
|
(3) |
其中, [x0, x1, …, xl-1]表示将0, 1, …, l-1层中产生的特征图以通道的维度进行合并(concatenation); Hl是复合函数, 例如批归一化(BN)、修正线性单元(ReLU)、池化(Pooling)、卷积(Conv)。
过渡层:两个相邻密集块之间的层, 可以通过卷积和池化改变特征图的大小, 同时, 为了进一步提高模型的紧凑型, 可以通过过渡层减少特征图的数量。
FC:全连接是指每个神经元和前面的所有神经元都相连。本文模型只有一层全连接层, 该层将深度学习特征与辅助特征整合并进行分类。
1.3.2 辅助特征目前用于HE染色的组织病理图像中有丝分裂识别的特征主要包括形态学特征、强度特征、基于灰度共生矩阵(gray level co-occurrence matrix, GLCM)的纹理特征[7, 12, 24, 25]等。灰度共生矩阵反映的是图像灰度关于方向、相邻间隔、变化幅度的综合信息。通过灰度共生矩阵可以分析图像的局部模式和排列规则等特征, 并且基于灰度共生矩阵获取二次统计量, 可以得到图像的纹理特征。
本文拟引入的特征应是对深度学习模型学习到的特征的有益补充, 通过实验分析, 选择基于灰度共生矩阵的22维纹理特征, 可以和深度学习模型得到的特征形成互补。另外, 考虑到深度学习模型最开始学习到的浅层特征对最后的分类器的影响小, 本文另外引入了强度特征(2维)和形态学特征[26, 27](19维)。综上, 本文一共引入了43维辅助特征, 如表 2所示。
| 类别 | 维度 | 特征 |
| 形态学特征 | 19 | Area, Perimeter, Compactness, Eccentricity, E-xtent, Perimeter_equal_di-ameter, Form_factor, ConvexArea, Area_equal_diameter, Solidity, Roundness, AspectRatio, Rectangularity, EulerNumber, MajorAxisLength, Mi-norAxisLength, Concavity, Hausdorff dim-ension, Smoothness |
| 强度特征 | 2 | Mean,Skewness |
| 纹理特征 | 22 | All Features from GLCM |
传统的交叉熵损失函数中不同类的权重相同, 在不同类训练样本严重不均衡时, 学习到的分类器更倾向于多数类。本文使用的训练集中正负样本数量严重不均衡, 为解决该问题, 提出代价敏感损失函数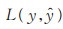
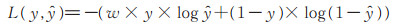
|
(4) |
其中, 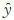
本文所用数据来自ICPR2012有丝分裂检测竞赛提供的公开数据集[3]。该数据集来自5个病人乳腺组织的HE染色后的病理切片。在40×高倍显微镜下, 病理学家从每张切片(面积为512 μm ×512 μm)中选取10个HPFs拍照成图, 一共得到50张2 084×2 084的图片, 图片分辨率为0.245 6 μm/pixel。经有经验的病理学家标注, 这些图片中总共包含了330个有丝分裂核, 其中35张用来训练, 共含有226个有丝分裂核, 15张用来测试, 共含有104个有丝分裂核。
从35张用于训练的图片中共提取5 736个候选目标, 其中237个正样本(对应226个有丝分裂核), 5 499个负样本。以像素点为单位剪切的每个样本大小为71×71。由于训练集较小, 尤其是其中正样本个数太少, 需要对正样本和负样本进行数据增强以扩增训练集。因此, 本文通过旋转45°/90°/135°/ 180°/225°/270°、上下翻转、左右翻转操作对正样本进行增强, 得到2 133个正样本。而对负样本, 则随机旋转一定角度进行增强, 最终得到10 998个负样本。
2.2 性能评价指标为评估本文检测方法的性能, 采用查全率(recall)、查准率(precision)、F分数(F-measure)3个指标, 计算方法如下:
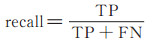
|
(5) |
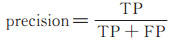
|
(6) |
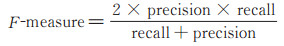
|
(7) |
其中, TP是检测为正样本实际也是正样本的个数, FP是检测为负样本实际是正样本的个数, FN是检测为正样本实际是负样本的个数。一般来说, 查准率和查全率是相互制约的, 而F-measure值平衡这两个度量值, 综合评价算法性能。
2.3 模型训练本文采用随机梯度下降算法最小化代价函数(如(8)式), 以获得最优的模型参数。(8)式中, J (θ)表示代价函数, 该值越小, 表示预测函数越准确。
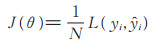
|
(8) |
其中, N表示样本数; yi为第i个样本的样本类标, 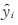
训练时, 将每次输入的样本数设为101, 进行300轮训练迭代, 学习率设为0.01, 对正样本错误分类的惩罚系数w设为2。
2.4 性能测试对于训练好的模型, 采用独立的测试集评估其性能。首先从15张用于测试的图片中共提取2 598个候选目标作为测试样本, 其中正样本104个。对每个样本分别进行90°/180°/270°旋转操作, 将原样本和3张变体样本依次送入训练好的模型进行预测, 计算4次预测概率的平均值作为该样本的预测概率。如果4次预测概率的平均值大于0.5, 就认为该样本是有丝分裂核, 否则不是。
在独立测试集上, 本文方法查准率、查全率、F分数分别为0.840, 0.770, 0.801 9, 其中查全率和F分数均获得了较好的结果, 证明本文提出的有丝分裂自动检测方法获得很好的检测效果。部分检测效果如图 3所示, 图中绿色圆圈为正确检测的结果(TP, 8个), 红色圆圈为误检测成有丝分裂核的结果(FN, 1个), 黄色圆圈表示漏检测的有丝分裂核(FP, 1个)。
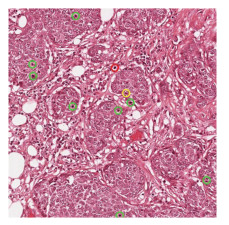
|
| 图 3 本文方法的检测结果 Fig. 3 Test results of the method in this paper |
为验证增加辅助特征和变体预测对实验结果的影响以及本文方法的有效性, 在实验数据集(见2.1节)上, 将方法1、2以及文献[8, 10~12, 14]算法的检测结果与本文方法检测结果进行对比。其中, 方法1:基于无融合辅助特征的DenseNet检测方法; 方法2:融合辅助特征, 但是在测试时没有将对每张图片进行变体预测求平均作为最终预测结果的检测方法。对比检测结果如表 3所示。
由表 3可以看出, 方法1的查准率、F分数都远远低于融合了辅助特征的方法2和本文方法, 证明了融合辅助特征可提高检测准确性。本文方法与方法2的区别在于, 本文方法在将训练好的模型应用于测试图片时, 将测试集提取的每个候选集进行3次旋转操作, 将原候选集和每次旋转之后所得图像分别进行预测, 对4次预测概率求平均获得最终的预测结果, 该方法提高了模型的鲁棒性, 增加了模型的旋转不变性, 其查准率、F分数都高于方法2。
文献[8]和[10]采用手工设计特征与传统机器学习相结合的方法, 而文献[11, 12, 14]基于深度学习实现对有丝分裂的检测。由表 3可以看出, 在查准率方面, 文献[11]的查准率最高, 本文次之, 但文献[11]的查全率有待提高, 而且, 文献[11]采用了基于像素点级的分类策略, 模型的训练和最后的测试都需要很长时间, 而与文献[11]相比, 本文方法检测耗时较少。在查全率方面, 本文方法的查全率较高, 说明在本文方法的检测结果中, 检测到的真有丝分裂核个数占所有手动标注有丝分裂核个数的比例较高。在综合评价查准率和查全率的F分数方面, 本文方法的F分数最高, 充分证明了本文方法的有效性。
在缓解样本不平衡问题上, 文献[14]使用两个传统CNN构建分类模型, 前一个CNN剔除部分明显不是有丝分裂核的样本, 以此减少负样本数量, 但缓解不平衡问题需要的训练模型开销较大, 比较复杂; 而本文直接采用代价敏感损失函数, 实现简单。
综上, 本文直接采用代价敏感损失函数缓解样本不均衡问题, 且采用DenseNet和辅助特征相融合的方式构建训练模型, 有效利用浅层特征, 因此取得了较好的预测结果。
3 结语本文方法将DenseNet与辅助特征相结合, 同时针对训练样本中正负样本不均衡的问题, 使用代价敏感损失函数, 在一定程度上缓解了该问题, 相比其他方法得到了较高的F分数、查准率和查全率。在有丝分裂候选集提取部分, 本文方法对部分候选集分割不够准确, 导致特征提取有误差, 在未来的工作中, 有待提出更好的候选集提取算法。
| [1] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer Journal for Clinicians, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [2] |
BLOOM H J, RICHARDSON W W. Histological grading and prognosis in breast cancer:A study of 1409 cases of which 359 have been followed for 15 years[J]. British Journal of Cancer, 1957, 11(3): 359-377. DOI:10.1038/bjc.1957.43 |
| [3] |
ROUX L.Mitosis Detection in Breast Cancer Histological Images[EB/OL].[2019-02-01].http://ludo17.free.fr/mitos_2012.
|
| [4] |
VETA M.Medical Image Computing and Computer-Assisted Intervention(MICCAI2013)[EB/OL].[2019-02-01].http://www.miccai2013.org.
|
| [5] |
ROUX L.22nd International Conference on Pattern Recognition(ICPR2014)[EB/OL].[2019-02-01].https://mitos-atypia-14.grand-challenge.org.
|
| [6] |
VETA M.Tumor Proliferation Assessment Challenge 2016[EB/OL].[2019-02-01].http://tupac.tue-image.nl.
|
| [7] |
HUMAYUN I. Automated mitosis detection in histopathology using morphological and multi-channel statistics features[J]. Journal of Pathology Informatics, 2013, 4(1): 247-252. DOI:10.4103/2153-3539.112695 |
| [8] |
HUMAYUN I, SEPEHR J, LUDOVIC R, et al. Automated mitosis detection using texture, SIFT features and HMAX biologically inspired approach[J]. Journal of Pathology Informatics, 2013, 4(1): 291-295. DOI:10.4103/2153-3539.109870 |
| [9] |
TASHK A, HELFROUSH M S, DANYALI H, et al.An automatic mitosis detection method for breast cancer histopathology slide images based on objective and pixelwise textural features classification[C]//The 5th Conference on Information and Knowledge Technology.New York: IEEE Press, 2013: 406-410.DOI: 10.1109/IKT.2013.6620101.
|
| [10] |
NATEGHI R, DANYALI H, SADEGHHELFROUSH M, et al.Automatic detection of mitosis cell in breast cancer histopathology images using genetic algorithm[C]//2014 21st Iranian Conference on Biomedical Engineering.New York: IEEE Press, 2015: 1-6.DOI: 10.1109/ICBME.2014.7043883.
|
| [11] |
CIREŞAN D C, GIUSTI A, GAMBARDELLA L M, et al.Mitosis detection in breast cancer histology images with deep neural networks[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention(MICCAI 2013).Heidelberg: Springer, 2013: 411-418.
|
| [12] |
WANG H B, CRUZ-ROA A, BASAVANHALLY A, et al.Cascaded ensemble of convolutional neural networks and handcrafted features for mitosis detection[C]//Proceedings of SPIE-The International Society for Optical Engineering, 2014, 9041(2): 90410B-1-90410B-10.DOI: 10.1117/12.2043902.
|
| [13] |
CHEN H, DOU Q, WANG X, et al.Mitosis detection in breast cancer histology images via deep cascaded networks[C]//Proceedings of the 30th AAAI Conference on Artificial Intelligence.Arizona: AAAI Press, 2016: 1160-1166.
|
| [14] |
WAHAB N, KHAN A, LEE Y S. Two-phase deep convolutional neural network for reducing class skewness in histopathological images based breast cancer detection[J]. Computers in Biology & Medicine, 2017, 85: 86-97. DOI:10.1016/j.compbiomed.2017.04.012 |
| [15] |
LI C, WANG X G, LIU W Y, et al. Weakly supervised mitosis detection in breast histopathology images using concentric loss[J]. Medical Image Analysis, 2019, 53: 165-178. DOI:10.1016/j.media.2019.01.013 |
| [16] |
PAENG K, HWANG S, PARK S, et al.A Unified Framework for Tumor Proliferation Score Prediction in Breast Histopathology[EB/OL].[2019-02-15].https://arxiv.org/abs/1612.07180.
|
| [17] |
ZERHOUNI E, LÁNYI D, VIANA M, et al.Wide residual networks for mitosis detection[C]//IEEE 14th International Symposium on Biomedical Imaging.New York: IEEE Press, 2017: 924-928.
|
| [18] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E.ImageNet classification with deep convolutional neural networks[C]//Proceedings of the IEEE International Conference on Computer Vision.New York: IEEE Press, 2015: 1520-1528.
|
| [19] |
SIMONYAN K, ZISSERMAN A.Very Deep Convolutional Networks for Large-Scale Image Recognition[EB/OL].[2018-11-12].https://arxiv.org/pdf/1409.1556.pdf.
|
| [20] |
SZEGEDY C, LIU W, JIA Y, et al.Going deeper with convolutions[C]//2015 IEEE Conference on Computer Vision and Pattern Recognition (CVPR).New York: IEEE Press, 2015: 1-9.DOI: 10.1109/CVPR.2015.7298594.
|
| [21] |
HE K M, ZHANG X Y, REN S Q, et al.Deep residual learning for image recognition[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition(CVPR).New York: IEEE Press, 2016: 770-778.DOI: 10.1109/CVPR.2016.90.
|
| [22] |
HUANG G, LIU Z, MAATEN L V D, et al.Densely connected convolutional networks[C]//2017 IEEE Conference on Computer Vision and Pattern Recognition.New York: IEEE Press, 2017: 2261-2269.DOI: 10.1109/CVPR.2017.243.
|
| [23] |
CHANG H, LOSS L A, PARVIN B.Nuclear Segmentation in H & E Sections via Multi-Reference Graph Cut(MRGC) [EB/OL].[2019-02-02].https://pdfs.semanticscholar.org/d538/cc7019bb00a0e39b978dc5e0e40861cc75d2.pdf.
|
| [24] |
KHAN A M, ELDALY H, RAJPOOT N M. A gammagaussian mixture model for detection of mitotic cells in breast cancer histopathology images[J]. Journal of Pathology Informatics, 2013, 4(1): 11-16. DOI:10.4103/2153-3539.112696 |
| [25] |
唐娇, 梁毅雄, 邹北骥, 等. 基于级联分类器的乳腺癌病理学图像中有丝分裂检测[J]. 计算机应用研究, 2016, 33(12): 3876-3879. TANG J, LIANG Y X, ZOU B J, et al. Mitosis detection in breast cancer histopathology with cascaded classifier algorithm[J]. Application Research of Computers, 2016, 33(12): 3876-3879. DOI:10.3969/j.issn.1001-3695.2016.12.079(Ch) (Ch). |
| [26] |
MUTHU R K M, PAL M, PAUL R R, et al. Computer vision approach to morphometric feature analysis of basal cell nuclei for evaluating malignant potentiality of oral submucous fibrosis[J]. Journal of Medical Systems, 2012, 36(3): 1745-1756. DOI:10.1007/s10916-010-9634-5 |
| [27] |
KRISHNAN M M R, CHOUDHARY A, CHAKRABORTY C, et al. Texture based segmentation of epithelial layer from oral histological images[J]. Micron, 2011, 42(6): 632-641. DOI:10.1016/j.micron.2011.03.00 |
 2019, Vol. 65
2019, Vol. 65





