文章信息
- 刘军立, 红凌
- LIU Junli, HONG Ling
- 基于转录组数据分析杜氏盐藻能量代谢途径
- Energy Metabolic Pathways in Dunaleilla salina Using the Transcriptome Data
- 武汉大学学报(理学版), 2017, 64(1): 85-94
- Journal of Wuhan University(Natural Science Edition), 2017, 64(1): 85-94
- http://dx.doi.org/10.14188/j.1671-8836.2018.01.010
-
文章历史
- 收稿日期:2016-12-12

利用微藻生产生物柴油具有培养简单、周期短、可大规模培养等优势.微藻通过光合作用捕获的太阳能主要以脂质(脂肪酸、三酰基甘油)、淀粉的形式存在.脂质能够与低级醇发生酯交换反应合成脂肪酸甲酯和甘油[1];淀粉经过一系列的发酵可用于生产可再生清洁能源醇类(例如乙醇、正丁醇等[2]).杜氏盐藻(Dunaliella salina)是一种单细胞绿藻,无细胞壁,在营养元素(例如氮等)缺乏或高盐、强光胁迫下,可大量积累脂质,尤其是三酰基甘油(TAG).三酰基甘油是一种能量分子,主要储存于脂滴(lipid droplets)中,由大脂滴蛋白(MLDP)包裹,能够缓解环境胁迫对微藻的影响[3].
已有研究主要侧重于通过改变盐藻的生长条件(如:盐、紫外诱导、营养元素等)选择培养脂质含量高的突变藻株[4].目前,采用基因工程手段、基因操作和代谢调控构建工程藻,可有目的地调控基因的表达,获取优质藻株.通过同源或异源表达脂肪酸生物合成的关键酶,可诱使微藻积累大量的脂肪酸[5, 6],如:在小球藻中异源表达酵母的NAD+依赖型甘油3-磷酸脱氢酶(GPD1),或TAG生物合成相关的酶均会诱使微藻大量累积TAG[7].在谷物(例如玉米、水稻、小麦)中上调表达葡萄糖-1-磷酸腺苷转移酶(AGPase)能提高农作物中淀粉的含量[8].然而,并不是过量表达所有关键酶,就会取得预期效果,如在烟草中高表达3-酮脂酰-酰基载体蛋白合成酶Ⅲ(KASⅢ),虽然提高了烟草中棕榈酸的含量,却降低了其合成脂肪酸的速率[9].因此,挖掘杜氏盐藻遗传信息,搭建其核心代谢网络并解析其调控机制,才可能通过基因工程手段对杜氏盐藻进行有效的遗传操作.
本文通过杜氏盐藻转录组的功能注释及聚类分析,预测了能量分子(脂质、淀粉)的代谢途径,分析了脂肪酸、三酰基甘油以及淀粉代谢路径中的关键酶,将脂质与淀粉代谢通路与其亚种D. tertiolecta的相关代谢通路[10]进行比较,阐述了淀粉与脂类代谢之间的关系,可望为采用基因工程技术构建优质工程藻提供候选基因和理论依据,为大量可持续性生产生物柴油打下基础.
1 材料与方法 1.1 实验材料杜氏盐藻由中国科学研究院淡水水生生物研究所馈赠,在DM培养基中培养.DM培养基组分:NaCl 87.69 g·L-1(1.5 mol·L-1), NaNO3 0.42 g·L-1,NaH2PO4·2H2O 0.156 g·L-1,NaHCO3 0.084 g·L-1,KCl 0.074 g·L-1,MgSO4·7H2O 1.23 g·L-1,CaCl2·2H2O 0.044 g·L-1,1%柠檬酸铁1 mL和A5溶液0.5 mL.其中,A5溶液组分:H3BO3 2.86 g·L-1,MnCl2·4H2O 1.81 g·L-1,ZnSO4·7H2O 0.22 g·L-1,CuSO4·5H2O 0.079 g·L-1以及(NH4)6Mo7O24·4H2O 0.039 g·L-1,培养条件为:12 h/12 h(光照/黑暗),26 ℃,光照强度为2 600 lx.
1.2 mRNA的分离和链特异性cDNA文库的构建收集不同生长时期(适应期、对数期、平台期)的杜氏盐藻,利用TriZol试剂(Invitrogen公司)抽提总RNA,采用Dynabeads®聚(dT)25磁珠纯化法(NEB公司)从总RNA中纯化分离mRNA.用琼脂糖凝胶电泳和安捷伦2100生物分析仪(Agilent公司)来评估mRNA的完整性,NanoDrop分光光度仪(Thermo Scientific公司)检测mRNA的浓度.达到RNA测序实验要求的样品即可用来构建链特异性cDNA文库.首先通过盐离子(Mg2+)随机打断mRNA使之片段化,并以片段化的mRNA为模板,反转录合成cDNA文库第一链;在合成第二条链时加入磷酸尿苷(dUTP),合成第二条链.合成cDNA双链后,按照Illumina HiSeqTM 2000(Illumina公司)说明书上机进行测序,由Novogene公司完成.
1.3 原始序列的获取及序列从头重新拼接高通量测序获取的原始序列通过perl scripts软件(Novogene公司)的纯化处理,去除含有接头、未识别碱基含量N≥10%和低质量的序列.其中质量值Q≤5的碱基数占原始转录本碱基数50%以上的序列为低质量序列.经过纯化处理的序列为高质量序列,可用于序列拼接.首先采用Trinity软件[11]将序列初步拼接成基因簇.参数设定:min_kmer_cov设置为2,其他参数为系统默认值.选取基因簇中最长的转录本序列,代表该基因簇,定义为重叠群(contigs).采用CodonCode(5.0.2)比对软件将contigs进一步组装成两端不能再延长的序列,即单一基因(unigenes).参数设置为系统默认值.具体的转录组拼接过程见图 1.

|
| 图 1 杜氏盐藻转录组拼接分析过程 Figure 1 Assembly and analysis of the Dunaliella transcriptome |
根据序列相似性,采用本地比对搜索软件(BLAST)将杜氏盐藻unigenes与美国国家生物技术信息(NCBI)数据库中的蛋白质数据库进行比对,获取其蛋白质注释信息(图 1).如果E≤1×10-5则认定相比较的序列之间具有较高的相似度.蛋白质数据库包括非冗余蛋白质数据库和Swiss-prot蛋白质数据库.Unigenes的聚类分析包括基因本体论(GO)注释,真核同源分类注释(KOG)以及京都基因和基因组百科全书(KEGG)分析(图 1).利用Blast2GO(V2.5)分析软件,根据基因产物的相关分子功能、生物学途径以及细胞学组分对unigenes进行GO注释[12].KOG注释利用基因组特异最佳匹配的方法快捷富集直系同源蛋白[13].KEGG是一个系统分析基因与基因组信息的数据库[14].应用KEGG自动注释服务器(KAAS)[15],杜氏盐藻unigenes被进一步富集分析,并获取序列所参与的代谢过程.
2 结果与讨论 2.1 杜氏盐藻转录组注释及富集分析通过双末端深度测序,共获取30 445 912条原始序列.表 1为杜氏盐藻测序数据质量基本统计信息,显示杜氏盐藻超过Q20质量标准的碱基占96.83%,远高于要求的80%,说明测序质量良好.经序列纯化,过滤掉1 119 196条低质量序列后,剩余的29 326 716条序列为高质量序列.这些序列被进一步拼接为39 820条unigenes.其中,有13 143(33.73%)条unigenes与蛋白质数据库序列高度同源;18 963个unigenes被GO注释;8 908个unigenes被分配到26个KOG亚组中;5 260个unigenes被匹配到245个KEGG参考标准途径中.
| 序列数目 | 碱基数目/GB | 序列长度/bp | 错误率/% | Q20a/% | Q30b/% | GC含量/% |
| 30 445 912 | 5.86 | 100 | 0.04 | 96.83 | 90.89 | 52.77 |
| 注:a指测序后在对碱基识别的错误率是1%,定义为Q20;b指对碱基识别的错误率为0.1%时,定义为Q30 | ||||||
利用微藻生产生物柴油主要由其生物质积累量和单位生物质的脂质含量[16]所决定, 因此,本研究主要关注的是杜氏盐藻脂质、淀粉的代谢途径以及二者之间的关系.
2.2 杜氏盐藻脂质代谢途径 2.2.1 脂肪酸代谢1) 脂肪酸生物合成途径
通过杜氏盐藻转录组功能注释以及聚类分析发现,杜氏盐藻脂肪酸的生物合成是一系列的催化合成饱和及不饱和脂肪酸的过程,参与脂肪酸生物合成的酶列于表 2,其过程示于图 2.
| 酶 | 简写 | KOa编号 | ECb编号 |
| 脂肪酸的生物合成 | |||
| 乙酰-CoA羧化酶生物素羧基载体蛋白 | BCCP | K02160 | — |
| 乙酰-CoA羧化酶/生物素羧化酶亚基 | accC | K01961 | 6.4.1.2,6.3.4.14 |
| 乙酰-CoA羧化酶羧基转移酶α亚基 | α-CT | K01962 | 6.4.1.2 |
| 乙酰-CoA羧化酶羧基羧基转移酶β亚基 | β-CT | K01963 | 6.4.1.2 |
| 乙酰-CoA羧化酶/生物素羧化酶 | BC | K11262 | 6.4.1.2,6.3.4.14 |
| 丙二酰-CoA ACP转移酶 | MAT | K00645 | 2.3.1.39 |
| 3-酮脂酰-ACP合成酶Ⅱ | KAS Ⅱ | K09458 | 2.3.1.179 |
| 3-酮脂酰-ACP合成酶Ⅲ | KAS Ⅲ | K00648 | 2.3.1.180 |
| 3-酮脂酰-ACP还原酶 | KAR | K00059 | 1.1.1.100 |
| 3-羟脂酰-ACP脱水酶 | HAD | K02372 | 4.2.1.59 |
| 脂酰-ACP硫酯酶A | FatA | K10782 | 3.1.2.14 |
| 不饱和脂肪酸的生物合成 | |||
| δ-9酰基-ACP去饱和酶 | DESA1 | K03921 | 1.14.19.2 |
| 硬脂酰-CoA δ-9去饱和酶 | SCD | K00507 | 1.14.19.1 |
| ω-6脂肪酸δ-12去饱和酶2/6 | FAD2/6 | K10256 | 1.14.19.- |
| ω-3脂肪酸δ-15去饱和酶 | FAD8 | K10257 | 1.14.19.- |
| 酰基-CoA硫酯酶YciA | YciA | K10806 | 3.1.2.- |
| 脂肪酸的延伸 | |||
| 长链-3-羟脂酰-ACP脱水酶 | PHS1 | K10703 | 4.2.1.134 |
| 十六酰基蛋白硫酯酶 | PPT | K01074 | 3.1.2.22 |
| 2-烯脂酰-CoA还原酶 | MECR | K07512 | 1.3.1.38 |
| 长链脂肪酸延伸蛋白6 | ELOVL6 | K10203 | 2.3.1.199 |
| 长链烯酰-CoA还原酶 | TER | K10258 | 1.3.1.93 |
| 注:a代表KEGG数据库中这种酶的同源序列号;b代表国际酶学委员会编号 | |||

|
| 图 2 杜氏盐藻脂肪酸合成途径 A:丙二酰-ACP的生物合成;B:丁酰-ACP的生物合成;C:饱和脂肪酸的生物合成;D:不饱和脂肪酸的生物合成.实线框代表在本转录组中发现的酶;虚线框中的酶在本转录组中未发现 Figure 2 Construction of fatty acid biosynthesis pathway in Dunaliella salina A: Biosynthesis of the malonyl-ACP; B: Biosynthesis of the butyryl-ACP; C: Biosynthesis of the saturated fatty acids; D: Biosynthesis of the unsaturated fatty acids. Solid box represents the enzymes we have identified in our transcriptome; Dash box represents the enzymes we have not identified in our transcriptome |
图 2A展示丙二酰-酰基载体蛋白(ACP)的合成.脂肪酸合成的起始原料是乙酰-CoA,主要来自糖酵解产物丙酮酸.由于脂肪酸的合成发生在细胞液中,因此乙酰-CoA需要从线粒体转运到胞液中.乙酰辅酶A(乙酰-CoA)羧化酶(ACCase)催化乙酰-CoA和碳酸盐合成丙二酰-CoA.之后丙二酰-CoA酰基载体蛋白转移酶(MAT EC:2.3.1.39)催化丙二酰基的转移反应,生成丙二酰-ACP.ACCase是催化合成脂肪酸的关键酶,催化脂肪酸合成的起始反应.它由4个亚基组成,包括生物素羧化酶亚基(BC EC:6.4.1.2 6.3.4.14),生物素羧基载体蛋白(BCCP)亚基以及两个BCCP羧基转移酶的亚基α-CT(EC:6.4.1.2)和β-CT(EC:6.4.1.2).
图 2B展示脂肪酸延伸时,丙二酰-ACP的碳架上增加两个碳,合成丁酰-ACP的过程.催化该过程的酶包括3-酮脂酰-ACP合成酶Ⅲ(KASⅢ)、3-酮脂酰-ACP还原酶(KAR EC:1.1.1.100)、3-羟脂酰-ACP脱水酶(HAD EC:4.2.1.59)和烯脂酰-ACP还原酶(EAR),分别催化脂肪酸的缩合、还原、脱水以及再还原4步反应,最终合成丁酰-ACP.3-酮脂酰-ACP合成酶Ⅲ(KAS Ⅲ EC:2.3.1.180)催化起始缩合反应,是脂肪酸延伸反应的关键酶.
图 2C展示脂肪酸延伸时,在4碳的基础上延伸合成不同长度的脂肪酸-ACP的过程.6到16碳的缩合反应该由3-酮脂酰-ACP合成酶Ⅰ(KASⅠ)催化.然而,在杜氏盐藻转录组中并没有检测到编码KASⅠ蛋白的转录本.3-酮脂酰-ACP合成酶Ⅱ(KAS Ⅱ EC:2.3.1.179)催化由16碳延伸至18碳的脂肪酸缩合反应.由于在杜氏盐藻中不存在碳链大于18个碳的脂肪酸[3, 16],在本研究中也没有发现相应酶的转录本.
根据脂肪酸的饱和度,脂肪酸可分为饱和脂肪酸以及不饱和脂肪酸.杜氏盐藻不饱和脂肪酸的合成(图 2D)主要发生在叶绿体内,一般由酰基-ACP饱和酶在脂肪酸碳架中脱氢形成双键.在本研究中发现了3种饱和酶(表 2),分别是δ-9酰基-ACP饱和酶(DESA1 EC:1.14.19.2)、ω-6脂肪酸δ-12饱和酶(FAD2/6 EC:1.14.19.-)和ω-3脂肪酸δ-15饱和酶(FAD8 EC:1.14.19.-).DESA1作用于棕榈酸(C18:0)的9号碳位,合成油酸(C18:1);FAD2/6作用于油酸的12号碳位,合成亚油酸(C18:2);FAD8作用于亚油酸的15号碳位,合成亚麻酸(C18:3).研究显示[17],在吉兰泰杜氏盐藻中,饱和脂肪酸所占比例为38.8%;不饱和脂肪酸比例高达51%,以C18:2(亚油酸)和C18:3(亚麻酸)为主,占总脂肪酸含量的27.1%.这两种不饱和脂肪酸为必需脂肪酸,因此杜氏盐藻广泛用于功能食品新材料的开发.
2) 脂肪酸分解代谢
杜氏盐藻脂肪酸降解相关的酶在表 3中展示.脂酰-CoA合成酶(ACSL EC: 6.2.1.3)催化脂肪酸生成脂酰-CoA,接着由脂酰-CoA脱氢酶(ACD EC:1.3.8.7)催化生成β-烯酯酰-CoA(图 3A).目前发现参与β-氧化的酶在杜氏盐藻中皆存在,包括乙酰-CoA氧化酶(ACOX EC:1.3.3.6)、烯酰-CoA水合酶(ECH EC:4.2.1.17),3-羟烷基-CoA脱氢酶(CHAD EC:1.1.1.35)和乙酰-CoA酰基转移酶1(ACAT1 EC:2.3.1.16).β-烯酯酰-CoA通过脱氢、水合、还原和硫解四个步骤从酰基上依次脱掉2个碳(图 3B).脂肪酸再进一步β-氧化降解生成的乙酰-CoA参与柠檬酸(TCA)循环,最终被降解生成CO2和水(图 3C).
| 酶 | 简写 | KO编号 | EC编号 |
| 脂酰辅酶A合成酶 | ACSL | K01897 | 6.2.1.3 |
| 脂酰辅酶A氧化酶 | ACOX | K00232 | 1.3.3.6 |
| 脂酰辅酶A脱氢酶 | ACD | K00249,K06445 | 1.3.8.7,1.3.99.- |
| 烯脂酰辅酶A合成酶 | MFP2 | K10527 | 4.2.1.17,1.1.1.35,1.1.1.211 |
| 3-羟丁酸脂酰辅酶A | FadB | K01825 | 1.1.1.35,4.2.1.17,5.1.2.3,5.3.3.8 |
| β-酮硫解酶1 | ACAT1 | K07513 | 2.3.1.16 |
| 脂酰辅酶A C-酰基转移酶2 | ACAT2 | K00626 | 2.3.1.9 |
| 线粒体3, 2-顺式-烯脂酰辅酶A异构酶 | ECI | K13238 | 5.3.3.8 |
| 乙醇脱氢酶 | ADH | K00121 | 1.1.1.1 |

|
| 图 3 杜氏盐藻脂肪酸分解代谢途径 A: β-烯脂酰-CoA的生物合成;B: β-烯脂酰-CoA(n-2)的生物合成;C:脂酰-CoA(n-2)的β-氧化.实线框代表在本转录组中发现的酶.“n”代表脂肪酸中碳的数目 Figure 3 Construction of fatty acid catabolic pathway in Dunaliella salina A: Biosynthesis of the enoy-CoA; B: Biosynthesis of the acyl-CoA(n-2); C: β-oxidation pathway of the acyl-CoA(n-2). Solid box represents the enzymes we have identified in our transcriptome. The letter "n" represents the number of the carbon number of the fatty acid |
1) 三酰基甘油生物合成途径
在杜氏盐藻转录组KEGG聚类分析的基础上,搭建杜氏盐藻三酰基甘油的生物合成途径.三酰基甘油的合成是在甘油3-磷酸的碳架上依次添加酰基的一系列酶促反应(图 4A),参与该通路的酶列于表 4中.

|
| 图 4 杜氏盐藻三酰基甘油代谢通路的构建 A: TAG的生物合成,①酰基-CoA非依赖型TAG合成通路;②酰基-CoA非依赖型TAG合成通路.B: TAG的降解过程.实线框代表在本转录组中发现的酶 Figure 4 Construction of TAG metabolic pathway in Dunaliella salina A: biosynthesis of the TAG, ① Acyl-CoA-dependent synthesis of TAG; ② Acyl-CoA-independent synthesis of the TAG. B: Catabolism of the TAG. Solid box represents the enzymes we have identified in our transcriptome |
| 酶 | 简写 | KO编号 | EC编号 |
| 三酰基甘油的生物合成 | |||
| D-甘油酸3-激酶 | GLYCTK | K15918 | 2.7.1.31 |
| 二羟基丙酮激酶 | DAK | K00863 | 2.7.1.29 |
| 甘油激酶 | GK | K00864 | 2.7.1.30 |
| 甘油-3-磷酸O-酰基转移酶 | GPAT/ PlsX | K13506 | 2.3.1.15 |
| 1-酰基-sn-甘油-3-磷酸酰基转移酶 | AGPAT/ LPT1 | K00655 | 2.3.1.51 |
| ATP依赖型二酰基甘油激酶 | DGK | K00901 | 2.7.1.107 |
| 磷脂酸磷酸酯酶 | PP | K15728 | 3.1.3.4 |
| 二酰基甘油O-酰基转移酶1 | DGAT1 | K11155 | 2.3.1.20,2.3.1.75,2.3.1.76 |
| 二酰基甘油O-酰基转移酶2 | DGAT2 | K11160 | 2.3.1.20,2.3.1.75 |
| 二酰基甘油O-酰基转移酶3 | DGAT3 | K14456 | 2.3.1.20,2.3.1.22 |
| 磷脂:二酰基甘油酰基转移酶 | PDAT | K00679 | 2.3.1.158 |
| 三酰基甘油分解代谢 | |||
| 胆盐刺激脂酶 | CEL | K12298 | 3.1.1.3,3.1.1.13 |
| 胰酯酶相关蛋白2 | PLRP2 | K14075 | 3.1.1.3 |
| 胃三酰基甘油酯酶 | LIPF | K14452 | 3.1.1.3 |
| 三酰基甘油酯酶 | TAGL | K14674 | 3.1.1.3,3.1.1.13,3.1.1.4,2.3.1.51 |
其中,甘油-3-磷酸O-酰基转移酶(GPAT/ PlsX EC:2.3.1.15)和1-酰基-sn-甘油-3-磷酸酰基转移酶(AGPAT/LPT1 EC:2.3.1.51)分别催化1号碳和2号碳的酯化反应,生成单酰基-甘油3-磷酸和1, 2-二酰基-甘油3-磷酸.之后,1, 2-二酰基-甘油3-磷酸在磷脂酸磷酸酯酶(PP EC:3.1.3.4)催化下去磷酸化生成二酰基甘油(DAG).接着,二酰基甘油O-酰基转移酶(DGAT EC:2.3.1.20)参与DAG的3号碳的酯化反应,生成三酰基甘油(TAG).由DGAT催化的酯化反应是合成储存型油脂的限速反应[17],是合成三酰基甘油的关键酶.DGAT是一种膜蛋白,定位于内质网(ER)上,分为三次跨膜蛋白(DGAT1)和两次跨膜蛋白(DGAT2)[17].与DGAT1相比,DGAT2优先利用底物酰基-CoA合成TAG[18].在转基因烟草[19]、花生[20]中上调DGAT,导致其大量合成三酰基甘油.本研究发现了3种DGAT酶,分别是DGAT1、DGAT2和DGAT3(DGAT2的亚型).以上合成TAG的碳骨架皆来自酰基-CoA(图 4A①),为传统的酰基-CoA依赖型TAG合成通路.另外,在杜氏盐藻中还发现了一条非酰基-CoA依赖型TAG合成通路(图 4A②).该通路通过磷脂二酰基甘油酰基转移酶(PDAT EC:2.3.1.158)催化磷脂酸,合成DAG,最终合成TAG.该通路的磷脂酸大部分来自于类囊体膜[21].
2) 三酰基甘油的分解代谢
三酰基甘油的降解是一个去酰基化生成甘油、脂肪酸以及脂肪酸再降解的过程(图 4B).其分解代谢相关的酶列于表 4中.三酰基甘油和二酰基甘油在三酰基甘油酯酶(TAGL EC:3.1.1.3 3.1.1.4)的催化下分别生成DAG和溶血磷脂,并伴随游离脂肪酸的产生.而催化溶血磷脂进一步去脂酰基水解的酶——单酰基甘油酯酶(MGLL EC:3.1.1.23)并没有在本转录组中发现.
2.3 杜氏盐藻淀粉的代谢 2.3.1 淀粉的生物合成途径在杜氏盐藻转录组功能注释以及聚类分析的基础上,搭建杜氏盐藻淀粉生物合成途径(图 5A).淀粉生物合成相关的酶列于表 5中.杜氏盐藻淀粉合成的主要原料为二磷酸腺苷-葡萄糖(ADP-Glc),由葡萄糖-1-磷酸腺苷转移酶(AGPase EC:2.7.7.27)催化葡萄糖1-磷酸(G1P)焦磷酸化生成.AGPase由两个大亚基和两个小亚基组成,具有胞质型和叶绿体型两个异构体[22].研究发现,AGPase能调节淀粉代谢关键产物的产生[23],为关键酶.淀粉合成酶(SS EC:2.4.1.21)以ADP-Glc为底物合成直链淀粉,各个葡萄糖(G1c)之间以α-1, 4糖苷键连接.支链淀粉则是在1, 4-α-葡聚糖分支酶(BE EC: 2.4.1.18)的催化下,在直链淀粉延伸的同时,以α-1, 6糖苷键延伸合成.SS有可溶性淀粉合成酶(SSS)和颗粒性淀粉合成酶(GBSS)两种.一般认为淀粉的生物合成,并不受SS活性调节,而是通过调节AGPase活性来实现的.AGPase大亚基突变,淀粉合成受到抑制[24].因此,抑制该酶活性,可调节杜氏盐藻淀粉的含量.

|
| 图 5 杜氏盐藻淀粉代谢通路的构建 A:淀粉的生物合成;B:淀粉的降解过程.实线框代表在本转录组中发现的酶;虚线框中的酶在本转录组中未发现 Figure 5 Construction of starch metabolic pathway in Dunaliella salina A: biosynthesis of the starch; B: catabolism of the starch. Solid box represents the enzymes we have identified in our transcriptome. Dash box represents the enzymes we have not identified in our transcriptome |
| 酶 | 简写 | KO编号 | EC编号 |
| 淀粉的生物合成 | |||
| 磷酸丙糖异构酶 | TPI | K01803 | 5.3.1.1 |
| 果糖二磷酸醛缩酶 | ALDO | K01623 | 4.1.2.13 |
| 果糖1,6-二磷酸酶 | FBP | K03841 | 3.1.3.11 |
| 葡萄糖-6-磷酸异构酶 | GPI | K01810 | 5.3.1.9 |
| 葡萄糖-6-磷酸酶 | G6PC | K01084 | 3.1.3.9 |
| 葡萄糖磷酸变位酶 | PGM | K01835 | 5.4.2.2 |
| 葡萄糖-1-磷酸腺苷转移酶 | AGPase | K00975 | 2.7.7.27 |
| 淀粉合成酶 | SS | K00703 | 2.4.1.21 |
| 1, 4-α-葡聚糖分支酶 | BE | K00700 | 2.4.1.18 |
| β-呋喃果糖苷酶 | ScaA | K01193 | 3.2.1.26 |
| 己糖激酶 | HK | K00844 | 2.7.1.1 |
| 淀粉的降解 | |||
| 淀粉磷酸化酶 | Spase | K00688 | 2.4.1.1 |
| α-淀粉酶 | α-AMY | K01176 | 3.2.1.1 |
| β-淀粉酶 | β-AMY | K01177 | 3.2.1.2 |
| 4-α-葡聚糖转移酶 | MalQ | K00705 | 2.4.1.25 |
淀粉通过水解和磷酸化两种方式来降解,生成葡萄糖(图 5B).葡萄糖以糖酵解形式为有机体提供能量.淀粉分解代谢相关的酶列于表 5中.通过杜氏盐藻转录组分析,发现α-淀粉酶(α-AMY EC:3.2.1.1)和β-淀粉酶(β-AMY EC:3.2.1.2),这两个酶可降解淀粉生成糊精和麦芽糖(表 4).
在水稻中干扰α-amy基因,可缓解种子中淀粉的分解代谢,进而改善种子的质量[25].糊精和麦芽糖在糊精-6-葡糖苷水解酶(D6G EC:3.2.1.10)和麦芽糖苷酶(MG EC:3.2.1.20)的作用下降解生成葡萄糖.然而,在本转录组中并未发现这两种酶的转录本.另外,淀粉磷酸化酶(Spase EC:2.4.1.1)可水解淀粉中的α-1, 4糖苷键生成G1P.根据Spase在细胞中的不同定位,可分为Spase1和Spase2两种酶,分别定位于叶绿体和细胞质中.Spase1通过调节其临近的淀粉合成系统来操控玉米淀粉的合成[26];而在拟南芥中的研究显示Spase2影响着淀粉的分解代谢[27].由此可推断Spase是调节淀粉代谢的关键酶.因此,可通过基因敲除技术敲除spase2或α-amy基因,或许可缓解淀粉降解,提高杜氏盐藻淀粉含量.
2.4 淀粉代谢与脂质代谢的关系斜生栅藻Scenedesmus obliquus在氮元素缺乏的情况下,前期TAG和淀粉含量均有所上升,后期淀粉含量开始下降;在光合效率不变的情况下,该藻突变株在同种处理条件下,淀粉含量很少,TAG含量明显增加[28].这说明在淀粉合成受到抑制时,光合作用固定的碳用于合成脂质.淀粉降解生成的葡萄糖可进一步酵解产生丙酮酸、磷酸烯醇式丙酮酸(PEP)以及乙酰-CoA.而乙酰-CoA是合成脂肪酸和三酰基甘油的重要原料.因此认为,淀粉的降解产物可用以合成脂质(脂肪酸和TAG),此二者的关系见图 6.
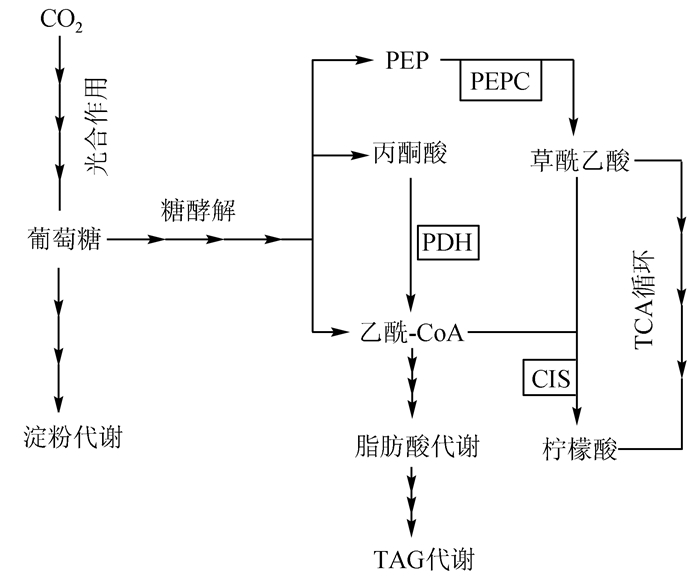
|
| 图 6 杜氏盐藻淀粉与脂质代谢的关系 实线框代表在本转录组中发现的酶 Figure 6 Relationship of the metabolism between starch and lipid in Dunaliella salina. Solid box represents the enzymes we have identified in our transcriptome |
通过杜氏盐藻转录组注释,发现一个单一基因编码细菌型磷酸烯醇式丙酮酸羧化酶(PEPC EC:4.1.1.31),见表 6.该酶能不可逆的羧化PEP,生成草酰乙酸,主要用于柠檬酸(TCA)循环[29].草酰乙酸和乙酰CoA在柠檬酸合成酶(CIS EC:2.3.3.1)的催化下生成柠檬酸,是柠檬酸循环的重要组成部分.抑制PEPC或柠檬酸合成酶,会导致柠檬酸循环下降,节省的乙酰-CoA可用于合成脂质[30~32].因此可以推测,促进杜氏盐藻淀粉降解或抑制淀粉的生物合成,可能会提高其脂质含量,用于生产生物柴油.
| 酶 | 简写 | KO编号 | EC编号 |
| 磷酸烯醇式丙酮酸羧化酶 | PEPC | K01595 | 4.1.1.31 |
| 柠檬酸合成酶 | CIS | K01647 | 2.3.3.1 |
| 丙酮酸脱氢酶 | PDH | K00156 | 1.2.5.1 |
本研究采用高通量测序技术对杜氏盐藻进行转录组测序,获取大量的杜氏盐藻转录本.在杜氏盐藻转录组功能注释和聚类分析的基础上,预测了能量分子(脂质、淀粉)的代谢途径,分析了脂肪酸和三酰基甘油的合成、淀粉的代谢路径中的关键酶(例如Accase,DGAT,AGPase和Spase),为基因工程技术提供了候选基因和理论依据,增强了遗传操作的可行性及成功率.
另外,在三酰基甘油合成途径的分析中发现,杜氏盐藻除了存在传统的酰基-CoA依赖型合成通路外,还存在一条酰基-CoA非依赖型合成通路.通过进一步分析发现,淀粉分解代谢产物可用于合成脂质.抑制淀粉的分解代谢(例如干扰α-amy或spase2基因),或高表达合成通路的关键基因(例如AGPase),可导致淀粉含量的积累.而抑制淀粉的合成和促进淀粉的分解代谢,会导致杜氏盐脂质的累积.然而,本文并未解析这些能量分子代谢网络调控的机制,这些都有待进一步研究.
| [1] |
黄雄超. 微藻油脂的提取及制备生物柴油的研究[D]. 泉州: 华侨大学, 2012. HUANG X C. Investigation on Extraction and Preparation of Biodiesel from Microalgae Lipid[D]. Xiamen: Huaqiao University, 2012(Ch). http://cdmd.cnki.com.cn/Article/CDMD-10385-1013000975.htm |
| [2] |
王海栋. 生产生物燃料的技术难题[J]. 发酵科技通讯, 2013, 42(3): 5-9. WANG H D. The research progress of the activity of marine microbes[J]. Ferm Commun Technology, 2013, 42(3): 5-9. |
| [3] |
DAVIDI L, KATZ A, PICK U. Characterization of major lipid droplet proteins from Dunaliella[J]. Planta, 2012, 236(1): 19-33. DOI:10.1007/s00425-011-1585-7 |
| [4] |
陈昱, 林锦明, 刘广发. 高脂杜氏藻的诱变筛选与分析[J]. 厦门大学学报(自然科学版), 2008, 47(3): 397-401. CHEN Y, LIN J M, LIU G F. Screening and analysis of high lipid Dunaliella salina[J]. J Xiamen University(Nat Sci), 2008, 47(3): 397-401. |
| [5] |
RUENWAI R, CHEEVADHANARAK S, LAOTENG K. Overexpression of acetyl-CoA carboxylase gene of Mucor rouxii enhanced fatty acid content in Hansenula polymorpha[J]. Molecular Biotechnology, 2009, 42(3): 327-332. DOI:10.1007/s12033-009-9155-y |
| [6] |
DAVIS M S, SOLBIATI J, CRONAN J E. Overproduction of acetyl-CoA carboxylase activity increases the rate of fatty acid biosynthesis in Escherichia coli[J]. J Biol Chem, 2000, 275(37): 28593-28598. DOI:10.1074/jbc.M004756200M004756200 |
| [7] |
HSIEH H J, SU C H, CHIEN L J. Accumulation of lipid production in Chlorella minutissima by triacylglycerol biosynthesis-related genes cloned from Saccharomyces cerevisiae and Yarrowia lipolytica[J]. Journal of Microbiology, 2012, 50(3): 526-534. DOI:10.1007/s12275-012-2041-5 |
| [8] |
TUNCEL A, OKITA T W. Improving starch yield in cereals by over-expression of ADPglucose pyrophosphorylase: Expectations and unanticipated outcomes[J]. Plant Science, 2013, 211: 52-60. DOI:10.1016/j.plantsci.2013.06.009S0168-9452(13)00146-5 |
| [9] |
DEHESH K, TAI H, EDWARDS P, et al. Overexpression of 3-ketoacyl-acyl-carrier protein synthase Ⅲs in plants reduces the rate of lipid synthesis[J]. Plant Physiology and Biochemistry, 2001, 125: 1103-1114. DOI:10.1104/pp.125.2.1103 |
| [10] |
RISMANI-YAZDI H, HAZNEDAROGLU B Z, BIBBY K, et al. Transcriptome sequencing and annotation of the microalgae Dunaliella tertiolecta: Pathway description and gene discovery for production of next-generation biofuels[J]. BMC Genomics, 2011, 12: 148-164. DOI:10.1186/1471-2164-12-148 |
| [11] |
CONESA A, GOTZ S, GARCIA-GOMEZ J M, et al. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics, 2005, 21(18): 3674-3676. DOI:10.1093/bioinformatics/bti610 |
| [12] |
ASHBURNER M, BALL C A, BLAKE J A, et al. Gene ontology: Tool for the unification of biology[J]. Nature Genetics, 2000, 25: 25-29. DOI:10.1038/75556 |
| [13] |
TATUSOV R L, FEDOROVA N D, JACKSON J D, et al. The COG database: An updated version includes eukaryotes[J]. BMC Bioinformatics, 2003, 4: 28-41. DOI:10.1186/1471-2105-4-411471-2105-4-41 |
| [14] |
Kanehisa M, Goto S. KEGG: Kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Research, 2000, 27(1): 29-34. |
| [15] |
MORIYA Y, ITOH M, OKUDA S, et al. KAAS: An automatic genome annotation and pathway reconstruction server[J]. Nucleic Acids Research, 2007, 35: 182-185. DOI:10.1093/nar/gkm321 |
| [16] |
CHEN M, TANG H, MA H, et al. Effect of nutrients on growth and lipid accumulation in the green algae Dunaliella tertiolecta[J]. Bioresoure Technology, 2011, 102(2): 1649-1655. DOI:10.1016/j.biortech.2010.09.062S0960-8524(10)01575-0 |
| [17] |
TURCHETTO-ZOLET A C, MARASCHIN F S, MORAIS G L D, et al. Evolutionary view of acyl-CoA diacylglycerol acyltransferase (DGAT), a key enzyme in neutral lipid biosynthesis[J]. BMC Evolutionary Biology, 2011, 11: 263-276. DOI:10.1186/1471-2148-11-263 |
| [18] |
ZHOU X R, SHRESTHA P, YIN F, et al. AtDGAT2 is a functional acyl-CoA: Diacylglycerol acyltransferase and displays different acyl-CoA substrate preferences than AtDGAT1[J]. FEBS Letters, 2013, 587(15): 2371-2376. DOI:10.1016/j.febslet.2013.06.003 |
| [19] |
NOOKARAJU A, PANDEY S K, FUJINO T, et al. Enhanced accumulation of fatty acids and triacylglycerols in transgenic tobacco stems for enhanced bioenergy production[J]. Plant Cell Reports, 2014, 33(7): 1041-1052. DOI:10.1007/s00299-014-1582-y |
| [20] |
PENG Z, LI L, YANG L, et al. Overexpression of peanut diacylglycerol acyltransferase 2 in Escherichia coli[J]. PLoS One, 2013, 8(4): 61363-61373. DOI:10.1371/journal.pone.0061363PONE-D-12-36179 |
| [21] |
FAN J, YAN C, ZHANG X, et al. Dual role for phospholipid:diacylglycerol acyltransferase: Enhancing fatty acid synthesis and diverting fatty acids from membrane lipids to triacylglycerol in Arabidopsis leaves[J]. Plant Cell, 2013, 25(9): 3506-3518. DOI:10.1105/tpc.113.117358tpc.113.117358 |
| [22] |
DAWAR C, JAIN S, KUMAR S. Insight into the 3D structure of ADP-glucose pyrophosphorylase from rice (Oryza sativa L.)[J]. Journal of Molecular Modeling, 2013, 19: 3351-3367. DOI:10.1007/s00894-013-1851-7 |
| [23] |
ASENCION DIEZ M D, DEMONTE A M, GUERRERO S A, et al. The ADP-glucose pyrophosphorylase from Streptococcus mutans provides evidence for the regulation of polysaccharide biosynthesis in Firmicutes[J]. Molecular Microbiology, 2013, 90(5): 1011-1027. DOI:10.1111/mmi.12413 |
| [24] |
TANG X J, PENG C, ZHANG J, et al. ADP-glucose pyrophosphorylase large subunit 2 is essential for storage substance accumulation and subunit interactions in rice endosperm[J]. Plant Science, 2016, 249: 70-83. DOI:10.1016/j.plantsci.2016.05.010 |
| [25] |
HAKATA M, KURODA M, MIYASHITA T, et al. Suppression of α-amylase genes improves quality of rice grain ripened under high temperature[J]. Plant Biotechnology Journal, 2012, 10: 1110-1117. DOI:10.1111/j.1467-7652.2012.00741.x |
| [26] |
SUBASINGHE R M, LIU F, POLACK U C, et al. Multimeric states of starch phosphorylase determine protein-protein interactions with starch biosynthetic enzymes in amyloplasts[J]. Plant Physiology and Biochemistry, 2014, 83C: 168-179. DOI:10.1016/j.plaphy.2014.07.016 |
| [27] |
MALINOVA I, MAHLOW S, ALSEEKH S, et al. Double knockout mutants of arabidopsis grown under normal conditions reveal that the plastidial phosphorylase isozyme participates in transitory starch metabolism[J]. Plant Physiology, 2014, 164(2): 907-921. DOI:10.1104/pp.113.227843pp.113.227843 |
| [28] |
BREUER G, JAEGER L D, ARTUS V P, et al. Superior triacylglycerol (TAG) accumulation in starchless mutants of Scenedesmus obliquus: (Ⅱ) Evaluation of TAG yield and productivity in controlled photobioreactors[J]. Biotechnology for Biofuels, 2014, 7: 70-80. DOI:10.1186/1754-6834-7-701754-6834-7-70 |
| [29] |
SANCHEZ R, CEJUDO F J. Identification and expression analysis of a gene encoding a bacterial-type phosphoenolpyruvate carboxylase from Arabidopsis and rice[J]. Plant Physiology, 2003, 132(2): 949-57. DOI:10.1104/pp.102.019653 |
| [30] |
SEBEI K, OUERGHI Z, KALLEL H, et al. Evolution of phosphoenolpyruvate carboxylase activity and lipid content during seed maturation of two spring rapeseed cultivars (Brassica napus L.)[J]. Comptes Rendus Biologies, 2006, 329(9): 719-725. DOI:10.1016/j.crvi.2006.06.002 |
| [31] |
TIAN Q L, SHI D J, JIA X H, et al. Recombinant expression and functional analysis of a Chlamydomonas reinhardtii bacterial-type phosphoenolpyruvate carboxylase gene fragment[J]. Biotechnology Letters, 2014, 36(4): 821-827. DOI:10.1007/s10529-013-1418-9 |
| [32] |
DENG X, CAI J, FEI X. Effect of the expression and knockdown of citrate synthase gene on carbon flux during triacylglycerol biosynthesis by green algae Chlamydomonas reinhardtii[J]. BMC Biochemistry, 2013, 14: 38-48. DOI:10.1186/1471-2091-14-381471-2091-14-38 |
 2017, Vol. 64
2017, Vol. 64





