文章信息
- 陈凯, 王佩, 吕应堂, 高嵩
- CHEN Kai, WANG Pei, LV Yingtang, GAO Song
- 利用GpC DNA甲基化酶M.CviPI高效表达限制性内切酶PstⅠ
- High-Efficiency Expression of Restriction Endonuclease PstⅠ Utilizing GpC DNA Methylase M.CviPI
- 武汉大学学报(理学版), 2017, 63(4): 361-367
- Journal of Wuhan University(Natural Science Edition), 2017, 63(4): 361-367
- http://dx.doi.org/10.14188/j.1671-8836.2017.04.013
-
文章历史
- 收稿日期:2017-01-16

2. 淮海工学院 江苏省海洋生物产业技术协同创新中心,江苏 连云港 222005;
3. 武汉大学 生命科学学院,湖北 武汉 430072;
4. 江苏省海洋资源开发研究院,江苏 连云港 222005
2. Co-Innovation Center of Jiangsu Marine Bio-industry Technology, Huaihai Institute of Technology, Lianyungang 222005, Jiangsu, China;
3. College of Life Sciences, Wuhan University, Wuhan 430072, Hubei, China;
4. Jiangsu Marine Resources Development Research Institute, Lianyungang 222005, Jiangsu, China
限制性核酸内切酶是一类能够识别DNA的特异序列并在识别位点之内或之外剪切双链DNA的核酸内切酶,简称限制酶[1].它们产生于细菌细胞内,能将外来DNA切割,限制异源DNA的侵入,但不对自身DNA产生损害,以此保护自身不被噬菌体侵染[2].1970年,Smith分离出第一种限制性内切酶Ⅱ型,开辟了对基因分离、基因克隆及序列分析的新研究[3~6].
由于细菌体内存在“限制-修饰”现象,即自身的DNA被甲基化酶修饰,从而避免所产生的限制酶对自身DNA片段的切割,达到抵御外来噬菌体侵染的目的.所以在原核表达体系中单一的重组表达限制酶,会使宿主因DNA被切割而死亡[3, 7].目前,研究者主要通过寻找每种限制酶的专一性甲基化酶对重组表达宿主的DNA进行保护,从而获得重组限制酶[8, 9].但这种方法需要使用特异性甲基化酶,应用范围狭窄,技术手段也较为繁琐.
DNA甲基化酶Methylase CviPI(M.CviPI)是一种来源于小球藻Chlorella病毒NYs-1的甲基转移酶[10],能将双链二核苷酸识别序列GpC中的胞嘧啶残基(C5) 甲基化.由于许多限制酶的识别位点包含GpC序列,推测对GpC甲基化样式敏感,不能切割带有此甲基化样式的DNA.利用这种通用型甲基化酶M.CviPI替代专一性甲基化酶对宿主DNA进行甲基化修饰,具有应用于一系列识别位点中带有GpC序列的限制性内切酶表达的潜在可能性.
本研究选用大肠杆菌表达宿主ER2566,向其中导入DNA甲基化酶M.CviPI,并让该酶持续性表达,使宿主DNA中形成GpC的甲基化样式.再利用这种菌株实现常用限制酶PstⅠ的重组表达,然后通过Ni-亲和层析及阴离子交换层析二步色谱方式纯化,获得具有快速酶切性质、高活力和高质量的限制性内切酶PstⅠ,可望为进一步优化大批含有GpC序列的限制性内切酶的制备工艺提供有益参考.
1 材料与方法 1.1 实验材料 1.1.1 菌种和质粒大肠杆菌DH5α和ER2566购自美国菌种保藏中心(ATCC);质粒pACYC184购自武汉淼灵生物科技有限公司;活性测定底物λ DNA购自美国Thermo Fisher Scientific公司;质粒pET-28b、pUC19由本实验室提供.
1.1.2 仪器和试剂引物合成、基因合成及测序由苏州金唯智生物技有限公司完成;细胞高压破碎仪购自广州聚能生物科技有限公司; GenoSens 1850型凝胶成像仪(Clinx)购自上海勤翔科学仪器有限公司.
Ni-亲和层析基质和阴离子交换层析基质(Toyopearl)购自东曹(上海)生物科技有限公司.限制性内切酶、CutOne缓冲液、T4 DNA连接酶、抗生素、异丙基-β-D-硫代吡喃半乳糖苷(IPTG)、BSA和三磷酸腺苷(ATP)等生化试剂购自江苏愚公生命科技有限公司;B-PER细菌蛋白提取试剂购自Thermo Fisher Scientific公司;蛋白分子量标准购自金斯瑞生物科技有限公司;胶回收试剂盒购自广州飞扬生物工程有限公司(Omega Bio-tek);质粒提取试剂盒(Axygen)购自Corning公司.
裂解缓冲液:10 mmol·L-1 Tris-HCl, pH 7.4, 250 mmol·L-1 NaCl, 5%甘油, 5 mmol·L-1咪唑, 1 mmol·L-1苯甲基磺酰氟.Ni亲和层析所使用的平衡缓冲液:10 mmol·L-1 Tris-HCl, pH 7.4, 250 mmol·L-1 NaCl, 5%甘油, 5 mmol·L-1咪唑;漂洗缓冲液:10 mmol·L-1 Tris-HCl, pH 7.4, 250 mmol·L-1 NaCl, 5%甘油, 20 mmol·L-1咪唑;洗脱缓冲液:10 mmol·L-1 Tris-HCl, pH 7.4, 250 mmol·L-1 NaCl, 5%甘油, 400 mmol·L-1咪唑.阴离子交换层析使用的平衡缓冲液:10 mmol·L-1 Tris-HCl, pH 7.4, 250 mmol·L-1 NaCl, 5%甘油, 1 mmol·L-1 DTT,0.1 mmol·L-1 EDTA;步级梯度洗脱液分别为250,300,500 mmol·L-1 NaCl.贮存缓冲液:10 mmol·L-1 Tris-HCl, pH 7.4, 250 mmol·L-1 NaCl, 50%甘油, 1 mmol·L-1 DTT, 0.1 mmol·L-1 EDTA.配制上述缓冲液的试剂均购自上海国药集团化学试剂有限公司,其纯度均为分析纯.
1.2 实验方法 1.2.1 甲基化酶M.CviPI组成型表达菌株的构建甲基化酶M.CviPI的DNA序列(GenBank:AAC64006) 通过基因合成获得,连接至克隆载体pUC57上,构建pUC57-M.CviPI质粒.合成序列全长1 279 bp,5′-端的增添序列包括:Hind Ⅲ酶切位点和E. coli pltL启动子序列(GenBank:M88256.1),3′-端的增添序列包括:终止密码子(TAA)和E. coli rrnB T1终止子序列(GenBank:CP010229.1),以及Hind Ⅲ酶切位点.pUC57-M.CviPI经Hind Ⅲ单酶切后,含M.CviPI基因的片段以T4 DNA连接酶连接至相同限制性内切酶酶切后的pACYC184载体上,并转化大肠杆菌DH5α感受态细胞进行质粒扩增,然后提取重组质粒pACYC184-M.CviPI.此质粒经测序正确后,转入感受态细胞ER2566,并于含35 mg·L-1氯霉素的LB平板上生长,获得甲基化酶M.CviPI组成型表达菌株ER2566 [pACYC184-M.CviPI],利用大肠杆菌pltL启动子持续表达甲基化酶M.CviPI.
1.2.2 限制性内切酶PstⅠ重组表达菌株的构建限制性内切酶PstⅠ的DNA序列(GenBank:P00640) 通过基因合成获得,连接至克隆载体pUC57上,构建pUC57-PstⅠ质粒.合成序列全长994 bp,包括5′-端添加的BamH Ⅰ酶切位点和3′-端添加的终止密码子(TAA)以及XhoⅠ酶切位点.pUC57-PstI经BamHⅠ和XhoⅠ双酶切后,带有PstⅠ基因的片段以T4 DNA连接酶连接至相同限制性内切酶酶切后的pET-28b载体上,使PstⅠ基因的表达框与载体上的多聚组氨酸(6×His)纯化标签的表达框一致.连接产物转化进入甲基化酶M.CviPI组成型表达菌株ER2566 [pACYC184-M.CviPI]感受态细胞,并于含75 mg·L-1卡那霉素和35 mg·L-1氯霉素的LB平板上生长,获得限制性内切酶PstⅠ重组表达菌株ER2566 [pACYC184-M.CviPI, pET-PstI].限制性内切酶PstⅠ的基因序列经测序确认正确,诱导后表达为N-端融合多聚组氨酸纯化标签的重组限制酶PstⅠ.
1.2.3 重组限制酶PstⅠ的诱导及粗提液活性检测将限制性内切酶PstⅠ重组表达菌株ER2566 [pACYC184-M.CviPI, pET-PstI]接种于含75 mg·L-1卡那霉素和35 mg·L-1氯霉素的液体LB培养基中,于37 ℃以200~250 r/min振荡培养至OD600 nm值为0.6~0.8,然后加入终浓度为0.5 mmol·L-1的IPTG,于16 ℃以200~250 r/min振荡培养16 h.以8 000g离心10 min收集菌体后,使用B-PER细菌蛋白提取试剂对菌体总蛋白进行提取.取1~3 μL细菌蛋白粗提液,在10 μL CutOne缓冲液(51 mmol·L-1乙酸钾, 22 mmol·L-1 Tris-AC, 10 mmol·L-1乙酸镁, 1 mg·mL-1 BSA, pH 8.0) 反应体系中,与PstⅠ的酶切底物λ DNA于37 ℃温育15 min,然后使用1%琼脂糖凝胶电泳检测底物被细菌蛋白粗提液消化的情况.
1.2.4 重组限制酶PstⅠ的表达及纯化重组限制酶PstⅠ经IPTG诱导后,培养液以8 000g离心10 min收集菌体.菌体以裂解缓冲液充分重悬后,以细胞高压破碎仪破碎.不可溶组分以43 000g低温离心30 min去除,含有限制酶PstⅠ的上清可溶组分经过Ni亲和层析初步纯化和阴离子交换层析精细纯化.Ni亲和层析初步纯化PstⅠ:首先上清液流经Ni柱时重组限制酶PstⅠ被Ni-基质吸附保留下来,然后使用含咪唑的洗脱液进行特异性洗脱,得到初步纯化限制酶PstⅠ洗脱液.阴离子交换层析精细纯化:将初步得到的限制酶PstⅠ洗脱液与阴离子交换基质结合,然后使用步级梯度缓冲液洗脱.对上述层析各步骤的样品取样,使用12%的变性聚丙烯酰胺凝胶电泳(SDS-PAGE)进行检测,最后将含有重组限制酶PstⅠ的洗脱组分合并,透析至贮存缓冲液中,并添加BSA至终浓度0.2 mg·mL-1,于-20 ℃保存.整个纯化过程于4 ℃或冰水浴中进行.
1.2.5 重组限制酶PstⅠ的活力测定按照快速酶切的酶活力标准定义:37 ℃下,在20 μL反应体系中,1 μL(1 U)酶能够在15 min内完全酶切1 μg λ DNA,将重组限制酶PstⅠ进行梯度稀释后,在20 μL CutOne缓冲液反应体系中,与PstⅠ的酶切底物λ DNA于37 ℃温育15 min,将能够把底物完全酶切所需的酶量定义为1 U.使用1%琼脂糖凝胶电泳检测底物的酶切情况,根据酶切完全时对应的稀释度计算重组限制酶PstⅠ的活力.
1.2.6 星号活性及非特异性DNA酶污染检测将重组限制酶PstⅠ进行梯度稀释后,在20 μL CutOne缓冲液反应体系中,与底物λ DNA于37 ℃分别温育60 min和16 h,使用1%琼脂糖凝胶电泳检测底物的酶切情况.
1.2.7 酶切-再连接检测使用20 U重组限制酶PstⅠ在20 μL CutOne缓冲液反应体系中,与0.2 μg底物DNA分子(894 bp,带有1个PstⅠ酶切位点,位于420 bp处)于37 ℃温育15 min.酶切完成后,于80 ℃温育20 min使限制酶PstⅠ失活,然后在反应液中补充ATP至终浓度0.5 mmol·L-1,并使用T4 DNA连接酶于22 ℃连接1 h.连接产物于65 ℃温育10 min失活后,加入20 U重组限制酶PstⅠ进行再次酶切(37 ℃温育15 min).酶切、连接及再次酶切的效率以1%琼脂糖凝胶电泳检测,并用凝胶成像分析系统进行量化分析.
1.2.8 蓝白斑实验检测重组限制酶PstⅠ活性使用8 U或16 U重组限制酶PstⅠ在20 μL CutOne缓冲液反应体系中,与1 μg pUC19质粒DNA于37 ℃温育15 min.酶切完成后,于80 ℃温育20 min使限制酶PstⅠ失活,然后在反应液中补充ATP至终浓度0.5 mmol·L-1,并使用T4 DNA连接酶于22 ℃连接1 h.连接产物转化进入DH5α大肠杆菌感受态细胞中,并涂布于含氨苄抗性的IPTG/X-gal平板,37 ℃培养过夜.统计所得白斑(白色菌落)与蓝斑(蓝色菌落)的数目并计算白斑比例.将重组限制酶PstⅠ酶切线性化后的pUC19质粒(未进行连接反应)作为对照,同样转化DH5α大肠杆菌感受态细胞中,并于含氨苄抗性的IPTG/X-gal平板上培养过夜,用于蓝斑背景的统计.
2 结果分析 2.1 构建限制性内切酶PstⅠ重组表达菌株针对限制性内切酶PstⅠ的重组表达,首先构建了用于保护宿主DNA的甲基化酶组成型表达质粒pACYC184-M.CviPI(图 1A).该质粒含有甲基化酶M.CviPI的基因,并且该基因被大肠杆菌组成型表达元件pltL启动子和rrnB T1终止子所调控.带有此质粒的大肠杆菌宿主细胞能够持续表达甲基化酶M.CviPI,在宿主DNA上的GpC位点形成甲基化.由于PstⅠ的识别位点中包含GpC序列,这种GpC甲基化样式预计能够保护宿主DNA免受限制酶PstⅠ的切割.大肠杆菌ER2566细胞株经转化质粒pACYC184-M.CviPI,形成了适于表达限制性内切酶PstⅠ的细胞株ER2566 [pACYC184-M.CviPI].我们推测,此细胞株能够持续表达甲基化酶M.CviPI,其宿主的DNA受到GpC甲基化样式保护,不会由于限制性内切酶PstⅠ的表达而死亡.
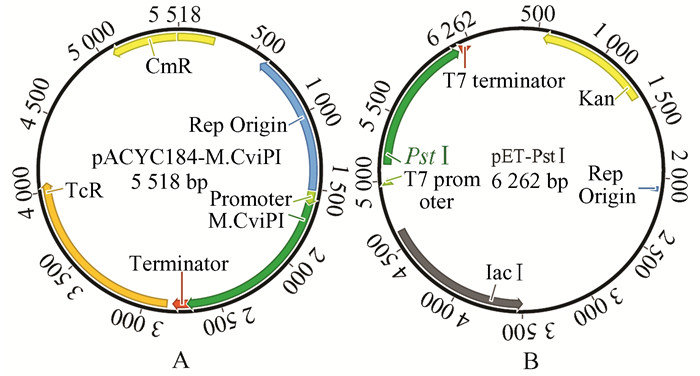
|
| 图 1 pACYC184-M.CviPI(A)和pET-PstⅠ(B)表达载体构建图谱圆环表示环型表达载体,数字表示载体的分子大小 Figure 1 Schematic of the pACYC184-M.CviPI(A) and pET-PstⅠ(B) constructs The circular plasmid DNA molecules are represented by circles, with numbers indicating the lengths of the molecules (in bp) |
然后,构建限制性内切酶PstⅠ的重组表达质粒pET-PstⅠ(图 1B).限制酶PstⅠ的基因被插入到pET-28b载体的表达区域,置于强诱导型启动子T7启动子的调控之下,并与载体上的多聚组氨酸纯化标签融合,形成了带有N-端融合多聚组氨酸纯化标签的重组PstⅠ基因.将插入PstⅠ基因的环状DNA分子pET-PstⅠ转化进入ER2566 [pACYC184-M.CviPI]感受态细胞中,获得了限制性内切酶PstⅠ重组表达菌株ER2566 [pACYC184-M.CviPI, pET-PstⅠ].由于pACYC184和pET质粒的复制子来源不同,可相容共存于同一个细胞株系中.环状DNA分子pET-PstⅠ转入DNA受到GpC甲基化保护的宿主ER2566 [pACYC184-M.CviPI]菌株,可获得转化子;相反,转入DNA未受GpC甲基化样式保护的宿主菌株(如DH5α等)则不能获得转化子.此现象表明,T7启动子调控下的PstⅠ基因在非诱导条件下的微量本底表达即足以杀死DNA未受甲基化保护的宿主细胞,而GpC甲基化却能保护宿主DNA免受限制酶PstⅠ的切割.
2.2 限制酶PstⅠ重组表达蛋白粗提液的酶活力实验室检测限制酶PstⅠ酶切活性的常用底物为λ DNA,在λ DNA上存在28个限制性内切酶PstⅠ的酶切位点.在对PstⅠ重组表达菌株ER2566 [pACYC184-M.CviPI, pET-PstⅠ]以IPTG进行小量诱导后,通过差速离心收集菌体,破碎细胞并粗提总蛋白.对蛋白粗提液进行酶切活性检测,结果显示,底物λ DNA被切割出多条特异性条带,表明重组表达的限制酶PstⅠ具有功能活性(图 2).
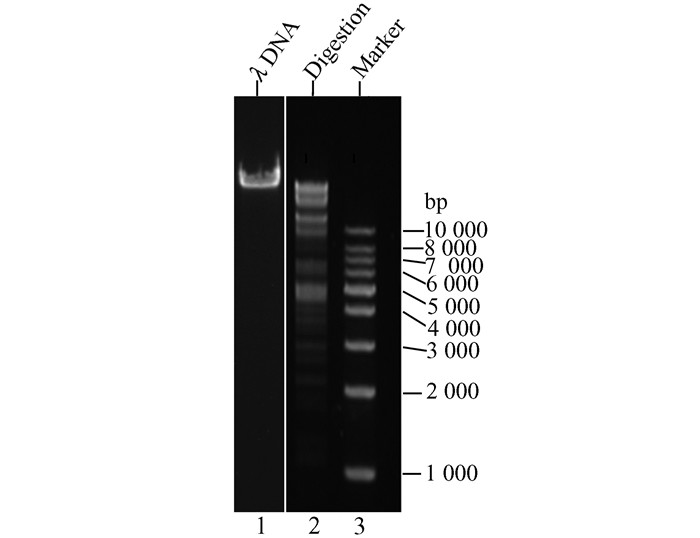
|
| 图 2 细胞粗提液酶切λ DNA的凝胶电泳图 1:底物λ DNA;2:1 μL细胞粗提液酶切λ DNA后的片段;3:DNA分子量标准(1 kb DNA Ladder) Figure 2 Agarose gel electrophoresis of λ DNA digested by the cell extract 1:Substrate λ DNA; 2: the specific DNA fragments digested from the substrate λ DNA by 1 μL of the cell extract; 3: DNA molecular weight standard (1 kb DNA Ladder) |
在验证了重组表达的限制酶PstⅠ具有功能活性后,对ER2566 [pACYC184-M.CviPI, pET-PstⅠ]菌株进行大规模诱导,大量表达重组限制酶PstⅠ结果显示,诱导后菌液在39×103处有明显条带,表明限制酶PstⅠ有明显表达(图 3A).经Ni-亲和层析纯化,限制酶PstⅠ获得纯度~85%的纯化效果(图 3B).将收集的洗脱液经阴离子交换层析进行精细纯化后,重组限制酶PstⅠ的蛋白纯度提升至90%以上(图 3C),阴离子交换层析同时也有去除宿主DNA残留的效果.
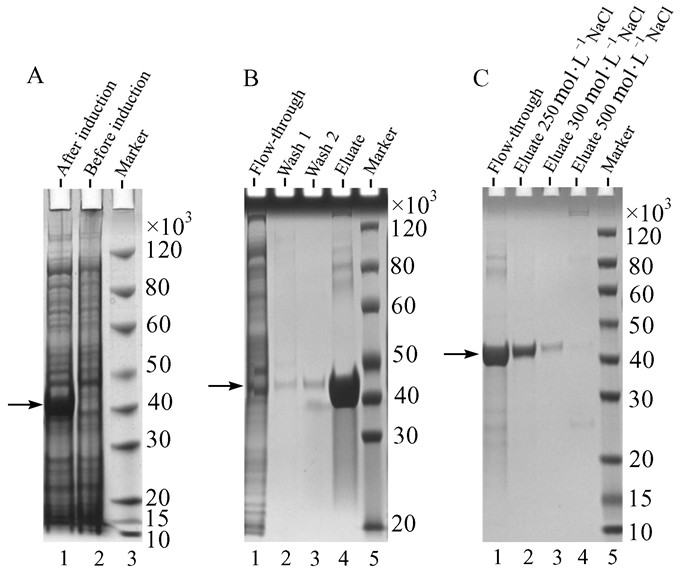
|
| 图 3 重组限制酶PstⅠ表达及纯化的凝胶电泳图 A:重组限制酶PstⅠ的表达检测;B:重组限制酶PstⅠ的Ni亲和层析纯化结果;C:重组限制酶PstⅠ的阴离子交换层析纯化结果 Figure 3 Expression and purification of the recombinant PstⅠ A: SDS-PAGE analysis of the induction of recombinant PstⅠ; B: SDS-PAGE analysis of the purification of recombinant PstⅠ by Ni-affinity chromatography; C: SDS-PAGE analysis of the purification of recombinant PstⅠ by ion-exchange chromatography |
对纯化后的重组限制酶PstⅠ进行活力检测,通过系列梯度稀释样品,对1 μg λ DNA进行酶切.结果表明,PstⅠ样本在稀释1 600倍后的酶切活力为1 U·μL-1(图 4).一次典型的对2 L诱导菌液进行的纯化可获得4.25 mg重组限制酶PstⅠ,总活力2 720 000 U,比活力640 000 U·mg-1,产率1 360 000 U·L-1.上述数据依据15 min内完全酶切1 μg λ DNA为1 U的活性定义计算,比以往报道中[6, 11]制备的PstⅠ的酶切速度提升了4倍(已报道的限制酶PstⅠ的制备均将1 h内完全酶切1 μg λ DNA的酶量定义为1 U).可见本研究制备的重组限制酶PstⅠ具有快速酶切性能.
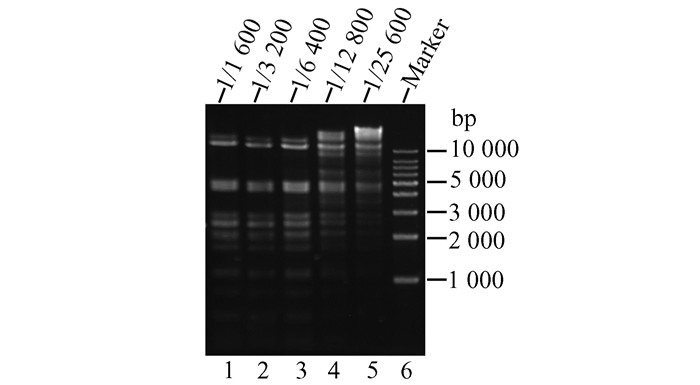
|
| 图 4 系列梯度稀释的重组限制酶PstⅠ活力检测凝胶电泳图 Figure 4 Determination of enzymatic activity of the serial diluted recombinant PstⅠ sample |
依据限制酶生产的行业标准,限制酶制备的质量控制除了对限制酶的活性有要求外,还要求无其它干扰酶切的活性.长时间孵育及星号活性检测、酶切-再连接检测和蓝白斑检测是常用的限制酶质量控制手段[6].
图 5A为重组限制酶PstⅠ长时间孵育及星号活性检测的凝胶电脉图,可见在温育60 min后,过量(32 U,对应稀释度1/50) 的酶切并未造成底物DNA产生非特异性降解条带.在过长时间(16 h)温育后,8 U(对应稀释度1/200) 酶量的酶切未产生非特异性降解条带,而高达16 U酶量的酶切仅造成了轻微的非特异性降解.此结果表明重组限制酶PstⅠ在过量酶切和过长时间酶切条件下,均不会产生星号活性及非特异性DNA降解.
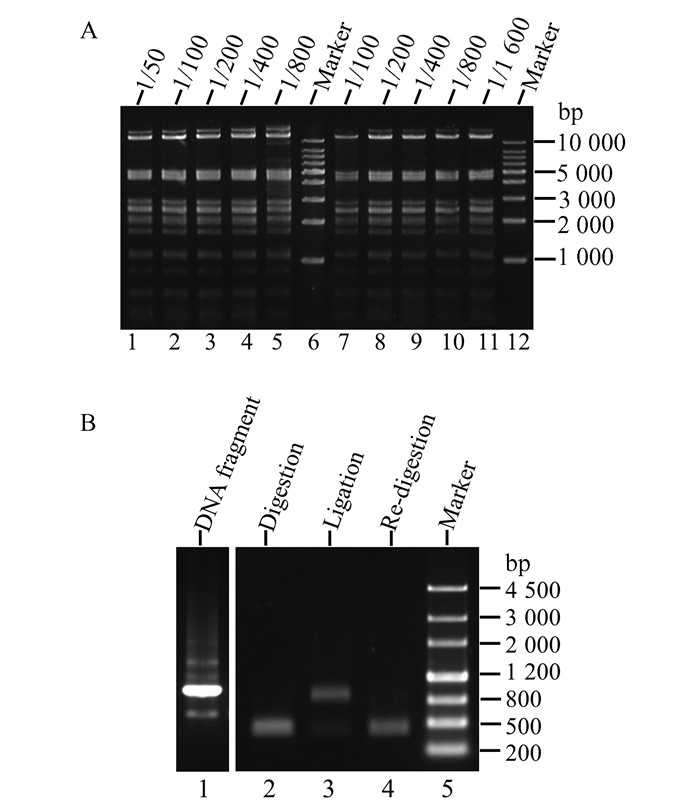
|
| 图 5 重组限制酶PstⅠ质量控制的凝胶电泳图 A:长时间孵育及星号活性检测,系列梯度稀释1 μL重组限制酶PstⅠ样品分别在37 ℃孵育1 h(泳道1~5)、16 h(泳道7~11) 时酶切1 μg λ DNA的琼脂糖凝胶电泳图;B:酶切-再连接检测 Figure 5 Quality control of the recombinant PstⅠ A: Prolonged incubation and star activity assay. Agarose gel shows the digestion of 1 μg λ DNA by 1 μL of the serial diluted recombinant PstⅠ sample that incubated at 37 ℃ for 60 min (lanes 1-5) or 16 h (lanes 7-11); B: Ligation and recut assaya |
在酶切-再连接检测中,使用20 U重组限制酶PstⅠ与底物DNA分子在37 ℃温育15 min后,所得的酶切片段使用T4 DNA连接酶进行连接,获得了大于95%的重新连接.所得的连接产物中大于95%可以被重组限制酶PstⅠ再次切开(图 5B).此结果表明,重组限制酶PstⅠ切割DNA形成的粘性末端准确并且完整.
蓝白斑检测结果显示,所得白斑(白色菌落)比例分别为0.035%(8 U)和0.072%(16 U),表明重组限制酶PstⅠ切割DNA形成的粘性末端十分完整(表 1).
| 酶量 | 白斑数 | 总斑数 | 蓝斑背景 | 白斑比例/% |
| 8U | 2 | 8 954 | 3 288 | 0.035 |
| 16U | 6 | 9 538 | 1 212 | 0.072 |
上述检测结果与NEB公司公布的质量控制标准比较,可知本研究获得的重组限制酶PstⅠ达到了的NEB公司(http://www.neb.com)的商业化限制酶标准.
3 讨论及结论本研究利用甲基化酶M.CviPI的甲基化修饰位点GpC包含在一大批限制酶识别序列之中的特征,构建了宿主DNA具有GpC甲基化样式的表达菌株,并应用于常用限制酶PstⅠ的表达.
DNA的甲基化样式包括只在原核细胞中发现的N4mC和N6mA型甲基化[12],以及在原核和真核细胞中都有发现的5mC型甲基化[13].对于某一特定的限制酶,其甲基化敏感性可能只针对其识别位点中特定碱基、特定类型的甲基化,也可能会对识别位点的其他甲基化样式敏感.本研究中获得限制酶PstⅠ,其自身“限制-修饰”系统的甲基化为N6mA型,但甲基化酶M.CviPI形成的5mC仍然能够保护其表达宿主的DNA,达到成功表达限制酶PstⅠ的目的.因此,本研究构建的M.CviPI甲基化菌株对于其他识别序列中含有GpC的限制性内切酶的重组表达也具有潜在的应用价值.
E.coil pltL启动子和rrnB T1终止子这两种表达调控元件能有效调控基因在大肠杆菌细胞中的持续稳定表达,在多种大肠杆菌工程菌的改造中被应用于外源蛋白的组成型表达[14~16].因此,本文将这两个表达调控元件应用于甲基化酶M.CviPI的表达载体上,实现了宿主细胞DNA在限制酶诱导表达前完成甲基化,避免被重组表达的限制酶切割,顺利完成表达过程.
菌株ER2566是一种mcrC基因和mrr基因缺失的工程菌.mcrC基因编码的mcrC蛋白能特异性结合外源DNA上被甲基化的胞嘧啶特异序列G5mC上,然后由mcrB蛋白切割外源DNA[17].而mrr基因能严格限制被甲基化的外源DNA的介入,同时对N6-腺嘌呤甲基化酶和C5-胞嘧啶甲基化酶活性有明显的抑制作用[18].这两种基因缺失的菌株ER2566能够承受外源甲基化酶基因的转入.在引入甲基化酶M.CviPI基因后,菌株ER2566被用于转化限制酶PstⅠ的表达质粒并成功获得了转化子.而其他菌株(如DH5α等)由于DNA未受GpC甲基化样式的保护,转化限制酶PstⅠ的表达质粒未能产生转化子,这与之前的研究[11, 19]中观察到的现象一致.以上结果表明,通过转入甲基化酶M.CviPI的基因有效实现了对宿主DNA的GpC甲基化样式修饰,避免了限制酶PstⅠ对其DNA的切割造成的毒性影响.而在DNA未受甲基化保护的菌株中,T7启动子调控下的PstⅠ基因在非诱导条件下的微量本底表达即可将宿主细胞杀死.
本研究仅利用Ni-亲合层析及阴离子交换层析两步纯化,使重组限制酶PstⅠ的纯度达到90%以上.同时,3项经典限制酶质量控制检测的结果表明,获得的重组限制酶PstⅠ品质优良,达到了NEB公司商业化限制酶的品质标准.本研究获得的限制酶PstⅠ实现了在15 min内完全消化1 μg底物λ DNA,达到了快速酶切的标准.而传统Ⅱ型限制酶的纯化通常先利用硫酸链霉素或聚乙烯亚胺除去核酸,然后经过P-磷酸纤维素阳离子交换柱、DEAE-纤维素弱阴离子交换柱、羟基磷灰石层析柱和依据分子大小分离的凝胶层析柱等多步柱层析,才能获得性能优良的限制酶[6, 20].与传统限制酶纯化技术相比,本研究在工艺上实现了很大的简化.
4 展望研究者在1998年便完成了M.CviPI甲基化酶的克隆及表达[10],识别并作用于GpC位点,具有保护DNA免受多种限制酶切割的可能性.依据美国NEB公司的官网显示,其Time-Saver系列193种(2017年1月份数据,仍更新中)商业化限制酶中,包括但不限于AluⅠ、BglⅠ、HindⅢ、NheⅠ、NotⅠ、NsiⅠ、PstⅠ、SacⅠ、SphⅠ等在内的54种常用限制酶的识别位点中包含GpC序列(http://www.neb.com).后期,使用本研究构建的M.CviPI甲基化菌株可对这一系列GpC甲基化敏感的限制酶进行表达尝试,使用这一菌株能对其进行高效表达.因此,基于M.CviPI甲基化酶的限制酶表达菌株对于识别位点中包含GpC序列的一系列限制酶的高效表达具有潜在的应用价值.
| [1] | 萨姆布鲁克J, 拉塞尔D W. 分子克隆实验指南[M]. 北京: 科学出版社, 2002: 1623-1624. SAMBROOK J, RUSSELL D W. Molecular Cloning: A Laboratory Manual[M]. Beijing: Science Press, 2002: 1623-1624(Ch). |
| [2] | NAITO T, KUSANO K, KOBAYASHI I. Selfish behavior of restriction-modification systems[J]. Science, 1995, 267(5199) : 897–899. DOI:10.1126/science.7846533 |
| [3] | LOENEN W A, DRYDEN D T, RALEIGH E A, et al. Highlights of the DNA cutters: A short history of the restriction enzymes[J]. Nucleic Acids Res, 2014, 42(1) : 3–19. DOI:10.1093/nar/gkt990 |
| [4] | WILLIAMS R J. Restriction endonucleases: Classification, properties, and applications[J]. Molecular Biotechnology, 2003, 23(3) : 225–243. DOI:10.1385/MB:23:3:225 |
| [5] | PINGOUD A, FUXREITER M, PINGOUD V, et al. Type Ⅱ restriction endonucleases: Structure and mechanism[J]. Cell Mol Life Sci, 2005, 62(6) : 685–707. DOI:10.1007/s00018-004-4513-1 |
| [6] | PINGOUD A, WILSON G G, WENDE W. Type Ⅱ restriction endonucleases—A historical perspective and more[J]. Nucleic Acids Research, 2014, 42(12) : 7489–7527. DOI:10.1093/nar/gku447 |
| [7] | LUNNEN K D, BARSOMIAN J M, CAMP R R, et al. Cloning type-Ⅱ restriction and modification genes[J]. Gene, 1988, 74(1) : 25–32. DOI:10.1016/0378-1119(88)90242-9 |
| [8] | ZHU Z Y, QUIMBY A, GUAN S X, et al. High Fidelity Restriction Endonucleases: US, US2011015 1450A1[P]. 2011-06-23. |
| [9] | ZHU Z Y, BLANCHARD A, XU S X, et al. High Fidelity Restriction Endonucleases: US, US200900 29376A1[P]. 2009-01-29. |
| [10] | XU M, KLADDE M P, VAN ETTEN J L, et al. Cloning, characterization and expression of the gene coding for a cytosine-5-DNA methyltransferase recognizing GpC[J]. Nucleic Acids Research, 1998, 26(17) : 3961–3966. DOI:10.1093/nar/26.17.3961 |
| [11] | WALDER R Y, WALDER J A, DONELSON J E. The organization and complete nucleotide sequence of the PstⅠ restriction-modification system[J]. Journal of Biological Chemistry, 1984, 259(12) : 8015–8026. |
| [12] | CHENG X. DNA modification by methyltransferases[J]. Curr Opin Struct Biol, 1995, 5(1) : 4–10. DOI:10.1016/0959-440X(95)80003-J |
| [13] | JELTSCH A. Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases[J]. Chembiochem, 2002, 3(4) : 274–293. DOI:10.1002/chin.200222292 |
| [14] | KONSTANTINOVIC M, MAKSIMOVIC V, NIKCEVIC G, et al. Hybrid PLtl promoter with dual regulation control[J]. DNA and Cell Biology, 1991, 10(5) : 389–395. DOI:10.1089/dna.1991.10.389 |
| [15] | CHEONG D E, CHOI J H, SONG J J, et al. Construction of non-invasively constitutive expression vectors using a metagenome-derived promoter for soluble expression of proteins[J]. Bioprocess and Biosystems Engineering, 2013, 36(6) : 667–676. DOI:10.1007/s00449-013-0890-x |
| [16] | SABIDO A, MARTÍNEZ L M, DE ANDA R, et al. A novel plasmid vector designed for chromosomal gene integration and expression: Use for developing a genetically stable Escherichia coli melanin production strain[J]. Plasmid, 2013, 69(1) : 16–23. DOI:10.1016/j.plasmid.2012.04.005 |
| [17] | ZHENG L, BRAYMER H D. Overproduction and purification of McrC protein from Escherichia coli K-12[J]. Journal of Bacteriology, 1991, 173(12) : 3918–3920. DOI:10.1128/jb.173.12.3918-3920.1991 |
| [18] | WAITE-REES P A, KEATING C J, MORAN L S, et al. Characterization and expression of the Escherichia coli Mrr restriction system[J]. Journal of Bacteriology, 1991, 173(16) : 5207–5219. DOI:10.1128/jb.173.16.5207-5219.1991 |
| [19] | 舒伟良. Ⅱ型限制性内切酶PstⅠ在合成生物学中的应用[D]. 汕头: 汕头大学, 2011. SHU W L. The Application of Type Ⅱ Restriction Endonuclease Pst Ⅰ to Synthetic Biology[D]. Shantou: Shantou University, 2011(Ch). |
| [20] | ROBERTS R J. Restriction endonucleases[J]. CRC Critical Reviews in Biochemistry, 1976, 4(2) : 123–164. DOI:10.3109/10409237609105456 |
 2017, Vol. 63
2017, Vol. 63





