文章信息
- 王加丽, 杨怡, 瞿旭东
- WANG Jiali, YANG Yi, QU Xudong
- 理性设计提高乙醇脱氢酶LbADH的催化活性
- Improvement of Catalytic Activity of Alcohol Dehydrogenase LbADH by Rational Design
- 武汉大学学报(理学版), 2017, 63(1): 45-50
- Journal of Wuhan University(Natural Science Edition), 2017, 63(1): 45-50
- http://dx.doi.org/10.14188/j.1671-8836.2017.01.005
-
文章历史
- 收稿日期:2016-03-07

生物催化剂的应用非常广泛,包括制药、医疗、食品、农业、造纸、基础化学品、能源生产和矿业等领域.酶作为生物催化剂具有经济、环保等突出优势,但也普遍存在稳定性较低等缺点[1].常用于酶工程改造的方法包括理性设计和非理性设计.非理性设计不需要完全了解酶的结构与功能之间的关系[2, 3],但需要筛选大量的突变体和发展高通量的筛选方法[4].而理性设计则要求对酶的催化机制和空间结构有充分的了解,通过少量的突变就可以对酶的结构进行精确调控[5].因此,在对酶的结构信息了解的基础上,理性设计具有突变率高、简单易行的优势[6, 7].
革兰氏阳性短乳杆菌Lactobacillus brevis (L. brevis)来源的乙醇脱氢酶LbADH是一种烟酰胺腺嘌呤二核苷磷酸(nicotinamide-adenine dinucleotide phosphate,NADP)依赖型的短链还原酶,催化活性依赖于Mg2+[8, 9].LbADH是手性醇生物催化合成的模式酶,具有广泛的用途[10, 11].人们尝试用多种方法提高LbADH在工业应用中的催化效率,如:在对LbADH吸附固定化过程中采取逐步干燥的方法[12];LbADH在非水相体系中的反应提高酶活力[13];通过构建双相连续反应体系解决LbADH的底物不溶于水相的问题[14].但野生型LbADH稳定性较低、不耐储存的缺点,使其在工业应用中的反应速度、溶剂浓度等方面受到限制.据报道,LbADH的热稳定性对催化反应起决定性作用[15],因此,提高LbADH的催化活性、热稳定性使其适用于工业化生产具有重要意义.前人多通过改变LbADH对辅酶的依赖性[16]或对LbADH催化体系进行改造[17, 18]来提高酶活力,但通过理性设计对LbADH进行改造来提高其活性和热稳定性的研究尚未见报道.
本研究通过分子模拟软件Discovery Studio 4.0对LbADH(GenBank登录号CAD66638)的蛋白空间结构进行分析,得出一系列与四聚体酶单体间相互作用相关的关键位点,并通过实验对这些位点进行突变,改变酶单体间的疏水作用力,影响盐桥键的形成,以期获得高活性高热稳定性的突变株.
1 实验部分 1.1 材料与仪器材料:表达载体pET28a、表达菌株大肠杆菌DH5α、大肠杆菌BL21(DE3)、L. brevis ATCC367由本实验室保存;Bradford法蛋白浓度测定试剂盒、琼脂糖凝胶DNA回收试剂盒、SanPrep柱式质粒DNA小量抽提试剂盒、还原型烟酰胺腺嘌呤二核苷磷酸(NADPH)、卡那霉素购自上海生工生物工程技术公司,蛋白质Marker,DNA Marker,T4 DNA连接酶、高保真DNA聚合酶、各种限制性内切酶购自大连宝生物工程公司;Ni-NTA亲和层析填料购自Qiagen公司;其他试剂均为国产或进口分析纯产品.本实验中引物合成和测序工作由金唯智生物科技公司完成.
仪器:Lambda25型紫外-可见分光光度计(PerkinElmer公司)、C1000型PCR仪(BioRad公司)、GelDOC型蛋白凝胶成像仪(BioRad公司)、G:BOX F3型DNA凝胶成像仪(Syngene公司).
1.2 方法 1.2.1 氨基酸定点突变及表达载体的构建提取L. brevis ATCC 367基因组DNA,以基因组DNA为模板,设计引物LbADH-For、LbADH-Rev扩增LbADH全长基因,两端引物上分别设计有限制性酶切位点NdeⅠ和HindⅢ(表S1).PCR获得的DNA片段经NdeⅠ和HindⅢ酶切后克隆至pET28a的相应位点,构建成N端带有多聚组氨酸标签(6×His-tag)的LbADH的表达质粒.基因的定点突变采用重叠延伸法[19]获得.首先利用For引物(表1)和pET28a上的T7终止子引物T7-Rev以上述pET8a-LbADH质粒为模板,扩增出包含突变位点的后部片段;其次利用Rev引物(表1)和pET28a上的T7启动子引物T7-For扩增出包含有突变位点的上部分段.而后再将两个DNA片段互为模板,利用T7-For和T7-Rev引物扩增出包含有相应突变位点的全长LbADH基因.最后通过NdeⅠ和HindⅢ酶切将突变基因克隆至pET28a,构建成表达载体pET28a-LbADH-K210R,pET28a-LbADH-S63V,pET28a-LbDAH-F146L,并测序验证.
| 引物 | 序列(5′→3′) |
| K210R-For | AACGGACCCGCACGCCAATGGGCCATATCG |
| K210R-Rev | TTGGCGTGCGGGTCCGTTGTGACATCGC |
| S63V-For | CCAACATGATGTTTCCGATGAAGACGGCTGG |
| S63V-Rev | CATCGGAAACATCATGTTGGAAAAATTGAATCTG |
| F146L-For | GCAAGGCTTGGTGGGTGATCCTAGCTTAG |
| F146L-Rev | TCACCCACCAAGCCTTCGATCGAAGACATG |
| T7-For | ACGACTCACTATAGGGGAATTGTG |
| T7-Rev | AAGGGGTTATGCTAGTTATTGCTC |
| LbADH-For | GCATATGTCAAACCGGTTAGATGGAAAAG |
| LbADH-Rev | GAAGCTTATTGAGCGGTATAACCACCATCA |
将表达载体分别转化到大肠杆菌BL21(DE3)中,挑取其单菌落接种到含有50 μg·mL-1卡那霉素的3 mL LB培养基中,于37 ℃过夜培养.取2 mL已培养好的菌转接至含有相应抗生素的500 mL LB中,37 ℃培养至光密度值OD600 nm值达到0.5时,冰上放置10 min使其冷却,加入诱导剂IPTG(异丙基硫代半乳糖苷) 至0.1 mmol·L-1,16 ℃诱导18 h.离心收集菌体,用pH 7.5的破菌缓冲液(300 mmol·L-1氯化钠、5 mmol·L-1 咪唑、20 mmol·L-1 HEPES、10%甘油)清洗并重悬菌体.将均匀的菌体置于冰上,超声破碎后,离心取上清液即粗酶液与Ni-NTA柱结合,并用浓度分别为25,50,100,300 mmol·L-1的pH 7.5咪唑缓冲液洗脱,收集洗脱液.将收集的各管洗脱液进行SDS-PAGE(十二烷基硫酸钠-聚丙烯酰胺凝胶电泳)鉴定,低温超滤离心浓缩蛋白.
1.2.3 酶活力的测定使用Bradford法[20]蛋白浓度测定试剂盒测定酶浓度,取牛血清白蛋白(BSA)标准品稀释10,20,40,50倍以其在595 nm下的吸光度值(A595 nm)做标准曲线.同样,将酶分别稀释5,10,20,40倍测A595 nm,根据标准曲线计算出酶浓度. 酶活力测定反应条件为:200 mmol·L-1丙酰乙酸乙酯10 μL,10 mmol·L-1 NADPH 10 μL,pH 7.5磷酸盐缓冲液970 μL,适量的酶,总体积共计1 mL.酶活力单位(U)定义为:在上述反应条件下,每min转化1 μmol 丙酰乙酸乙酯所需乙醇脱氢酶量为1个酶活力单位.
1.2.4 酶动力学的测定酶的动力学参数测定时,NADPH的浓度保持在0.1 mmol·L-1,底物丙酰乙酸乙酯浓度在0.2~3 mmol·L-1的范围内变化.反应通过加入一定量的酶启动,检测340 nm处的特征峰吸收峰的变化.采用双倒数作图法(Lineweaver-Burk)计算动力学参数米氏常数和转换数.
2 结果与讨论 2.1 突变位点的选择通过序列对比和晶体结构分析,选择LbADH位点63位丝氨酸(S63)、210位赖氨酸(K210)和146位苯丙氨酸(F146)进行突变,各突变位点的空间位置如图1所示.其中,K210突变为精氨酸(R)形成盐桥键突变体LbADH K210R,S63,F146分别突变为缬氨酸(V),亮氨酸(L)形成疏水突变体LbADH S63V,LbADH F146L.从晶体结构上看,K210与149位的天冬氨酸(N149)距离为1 nm,是一个较为合适的构成盐键的距离[21].赖氨酸的NH3+的正电性不及精氨酸中的胍基,将赖氨酸突变成精氨酸后可增强正电性,从而增强四聚体酶的单体表面的盐桥键.通过序列对比,发现许多同源蛋白的63位为缬氨酸,但LbADH中该位点为丝氨酸.从晶体结构上看,S63位于蛋白内部,将S63突变为缬氨酸有利于提高蛋白的疏水作用从而提高蛋白的稳定性.位于四聚体酶的中心的F146突变为亮氨酸可增加单体间的疏水作用.虽然异亮氨酸的疏水性大于亮氨酸,但由于亮氨酸和苯丙氨酸在结构上更为相似,故将F146突变设计为亮氨酸更符合理性设计的原则.

|
| 图 1 LbADH中突变位点的空间位置 Figure 1 Spatial location of mutation sites in LbADH |
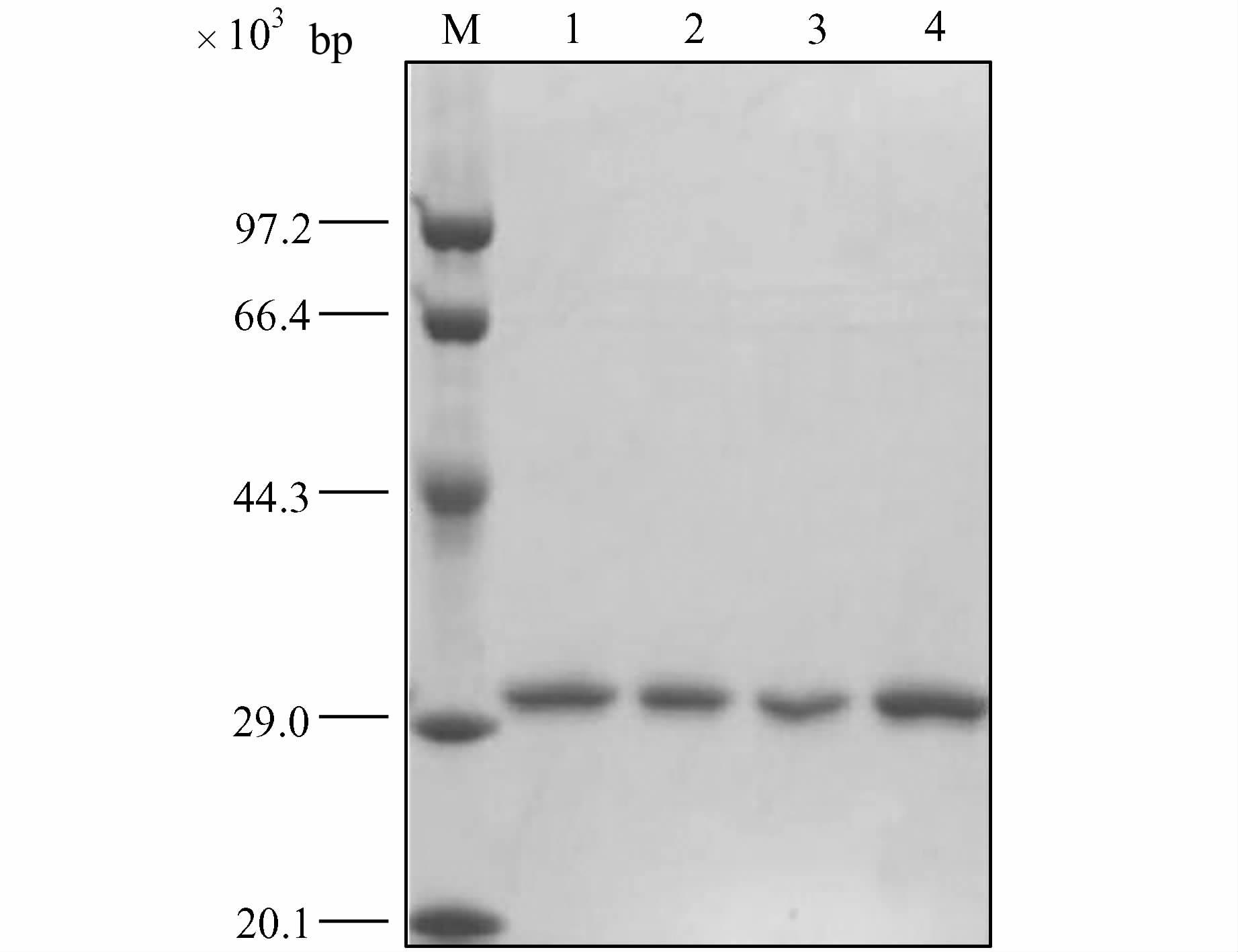
以L. brevis ATCC 367基因组为模板,通过PCR扩增获得LbADH基因片段.片段经NdeⅠ,HindⅢ酶切后克隆至pET28a相应位点中,得到表达质粒pET28a-LbADH.随后以该质粒为模板,采用重叠延伸法克隆包含有LbADH K210R,LbADH S63V,LbADH F146L的全长基因片段,片段再经NdeⅠ,HindⅢ酶切连入pET28a载体,转化大肠杆菌DH5α,经PCR和测序验证获得LbADH相应位点突变的表达质粒.将验证正确的表达载体分别转化到大肠杆菌BL21(DE3)中诱导表达并纯化.将浓缩蛋白通过SDS-PAGE电泳分析,如图2所示,在29×103 bp附近出现LbADH及突变体蛋白,与预计理论大小相符.
|
| 图 2 LbADH及突变体蛋白表达的SDS-PAGE图 Figure 2 SDS-PAGE analysis of LbADH and its mutants M:Protein marker;1:LbADH;2:LbADH S63V;3:LbADH F146L;4:LbADH K210R |
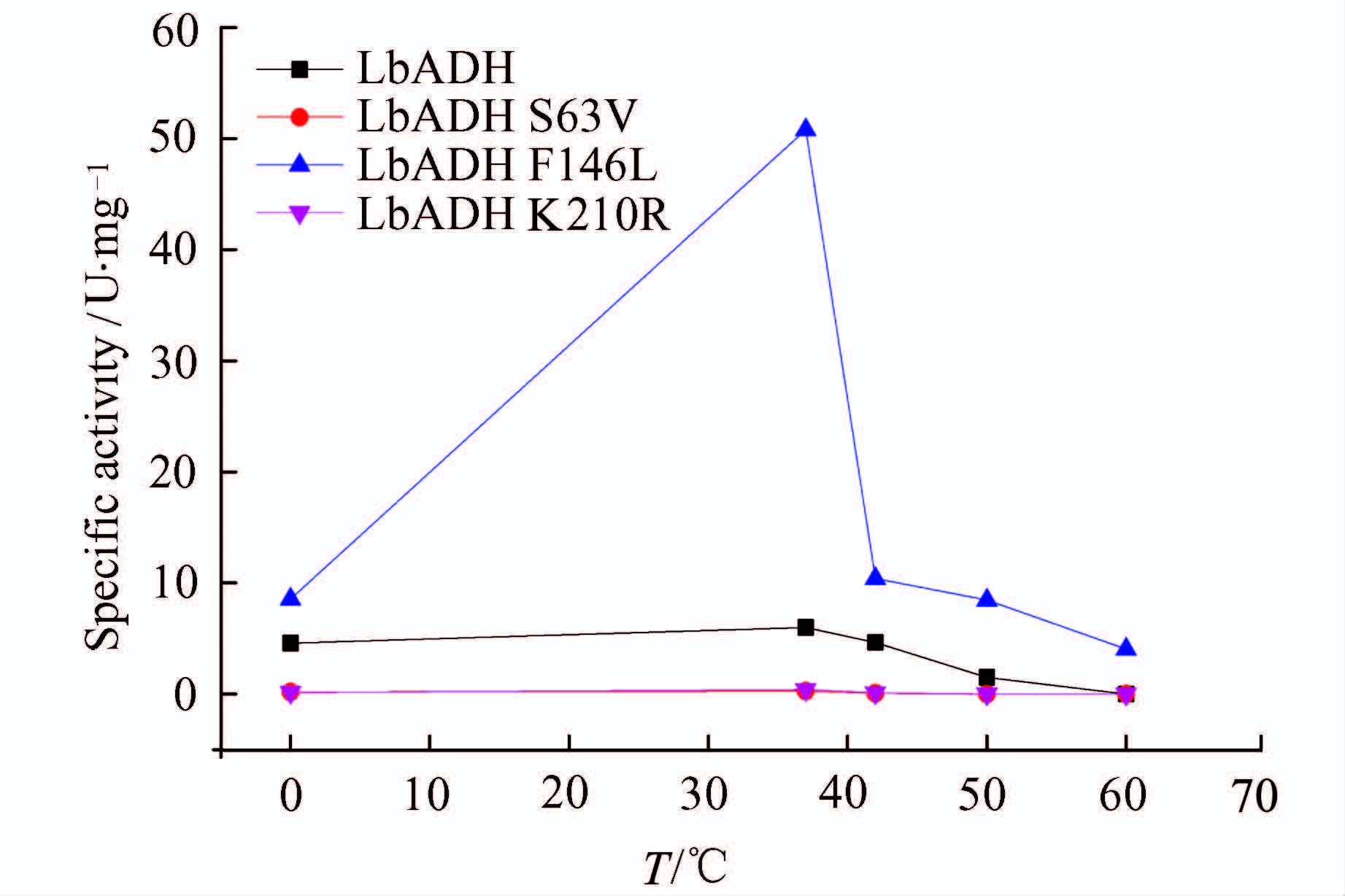
以具有代表性的潜手性酮丙酰乙酸乙酯作为反应底物,验证LbADH及其突变体的催化性能,然后测其粗酶液的酶活力.与LbADH比较,盐桥键突变体LbADH K210R、疏水突变体LbADH S63V的酶活力降低.而疏水突变体LbADH F146L的比活力在37 ℃的反应条件下为LbADH的8.5倍,在50 ℃下为LbADH的5.6倍(图3),说明LbADH F146L在酶活力显著提高的同时具有一定热稳定性,在较高温度下仍能发挥生物催化功能.通过理性设计,Kim等也获得了催化活性和热稳定性同时提高的植酸酶[22].在LbADH的空间结构中,F146位于四聚体酶的内部,且在4个单体中距离较近.将F146突变为亮氨酸可增大疏水作用,有利于四聚体的形成和稳定.酶的热稳定性需要活性中心的周边区域具有一定的刚性,从而抵御高温环境的破坏[23].因此,选择理性设计的突变位点时,应在不影响酶活性中心的基础上,尽量提高其周边区域的刚性.
|
| 图 3 LbADH野生酶及突变体的比活力分析 Figure 3 Analysis of the specific activity of LbADH and its mutants |
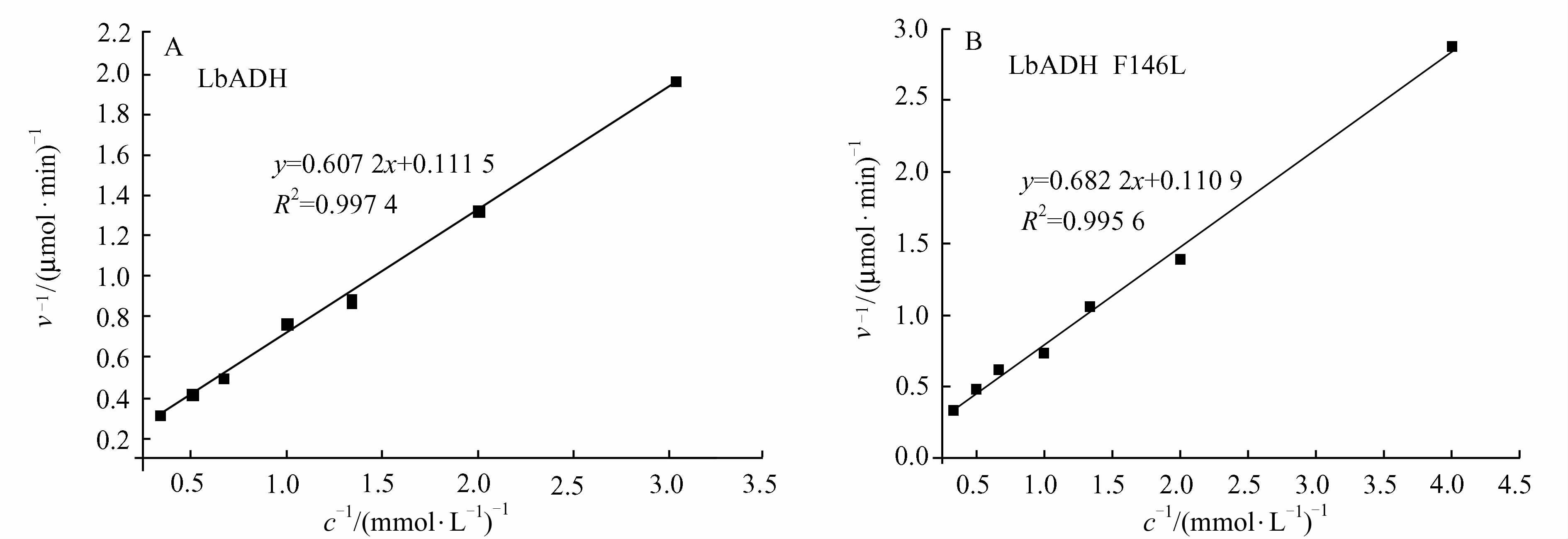
对LbADH及突变体LbADH F146L粗酶液的酶活力测定结果表明,LbADH F146L的催化活性及热稳定性均显著提高.进一步研究了其动力学性能,绘制LbADH和LbADH F146L的动力学双倒数曲线(图4),拟合得到的两个线性回归方程的相关系数R2均大于0.99,表明线性关系良好,计算得到较为精确的米氏常数Km (mmol·L-1)和转换数Kcat (s-1)的值(表2).
|
| 图 4 LbADH(A)和LbADH F146L(B)的双倒数曲线 Figure 4 Double-reciprocal courses of LbADH (A)and LbADH F146L(B) |
| 酶 | Km /L·mmol-1 | Kcat/s-1 | Kcat/Km /L\5mol-1·s-1 |
| LbADH | 5.45 | 76.1 | 1.40×104 |
| LbADH F146L | 6.15 | 1.23×104 | 1.99×106 |
突变体LbADH F146L的Km值与野生酶相比略有提高,即突变体对底物的亲和力有所降低.文献[24]研究表明,酶活力的提高,往往伴随着酶对底物亲和性的降低,与本文结论一致.与野生酶相比,突变体LbADH F146L的Kcat/Km值为野生酶的142倍.
上述结果表明,与Schlieben等[16]的研究结果相比,本研究在不改变辅酶的基础上,大幅度提高了乙醇脱氢酶的催化效率.
3 结论本研究使用LbADH作为催化手性醇的模式酶,并采用理性设计对其进行改造.研究中得到催化活性及热稳定性同时提高的疏水突变体LbADH F146L,其在37 ℃和50 ℃下的催化活性分别是野生酶的8.5倍和5.6倍,LbADH F146L的催化效率是野生酶142倍.
LbADH F146L催化活性提高的同时,热稳定性和催化效率也随之提高,进一步适应了工业化生产的要求.该研究结果证实了计算机模拟在理性设计中的可行性,可望为研究LbADH的结构和功能之间的关系提供理论参考.
| [1] | BRANNIGAN J A, WILKINSON A J. Protein engineering 20 years on[J]. Nature Reviews Molecular Cell Biology, 2002, 3(12) : 964–970. DOI:10.1038/nrm975 |
| [2] | BORNSCHEUER U T, POHL M. Improved biocatalysts by directed evolution and rational protein design[J]. Current Opinion in Chemical Biology, 2001, 5(2) : 137–143. DOI:10.1016/S1367-5931(00)00182-4. |
| [3] | 孙志浩, 柳志强. 酶的定向进化及其应用[J]. 生物加工过程, 2005, 3(3) : 7–13. SUN Z H, LIU Z Q. Methods and application of directed enzyme evolution[J]. Chinese Journal of Bioprocess Engineering, 2005, 3(3) : 7–13(Ch). |
| [4] | 王凡业, 薛文漪. 酶活性设计的新方法--半理性设计[J]. 应用化工, 2006, 35(8) : 634–636. WANG F Y, XUE W Y. Using a new approach to engineer enzyme activity-Semi-rational design[J]. Applied Chemical Industry, 2006, 35(8) : 634–636(Ch). |
| [5] | SIEGEL J B, ZANGHELLINI A, LOVICK H M, et al. Computational design of an enzyme catalyst for a stereoselective bimolecular Diels-Alder reaction[J]. Science, 2010, 329(5989) : 309–313. DOI:10.1126/science.1190239 |
| [6] | RÖTHLISBERGER D, KHERSONSKY O, WOLLACOTT A M, et al. Kemp elimination catalysts by computational enzyme design[J]. Nature, 2008, 453(7192) : 190–195. DOI:10.1038/nature06879 |
| [7] | JIANG L, ALTHOFF E A, CLEMENTE F R, et al. De novo computational design of retro-aldol enzymes[J]. Science, 2008, 319(5868) : 1387–1391. DOI:10.1126/science.1152692. |
| [8] | HUMMEL W. Large-scale applications of NAD (P)-dependent oxidoreductases: Recent developments[J]. Trends in Biotechnology, 1999, 17(12) : 487–492. DOI:10.1016/S0167-7799(98)01207-4 |
| [9] | NIEFIND K, MVLLER J, RIEBEL B, et al. The crystal structure of R-specific alcohol dehydrogenase from Lactobacillus brevis suggests the structural basis of its metal dependency[J]. Journal of Molecular Biology, 2003, 327(2) : 317–328. DOI:10.1016/S0022-2836(03)00081-0 |
| [10] | DI GENNARO P, BERNASCONI S, ORSINI F, et al. Multienzymatic preparation of 3-[(1R)-1-hyd-roxyethyl] benzoic acid and (2S)-hydroxy (phenyl) ethanoic acid[J]. Tetrahedron: Asymmetry, 2010, 21(15) : 1885–1889. DOI:10.1016/j.tetasy.2010.07.007 |
| [11] | LI B J, LI Y X, BAI D G, et al. Whole-cell biotransformation systems for reduction of prochiral carbonyl compounds to chiral alcohol in Escherichia coli[J]. Scientific Reports, 2014, 4 : 6750–6754. DOI:10.1038/srep06750 |
| [12] | TRIVEDI A, HEINEMANN M, SPIESS A C, et al. Optimization of adsorptive immobilization of alcohol dehydrogenases[J]. Journal of Bioscience and Bioengineering, 2005, 99(4) : 340–347. DOI:10.1263/jbb.99.340 |
| [13] | THOREY P, KN EZ, HABULIN M. Alcohol dehydrogenase in non-aqueous media using high-pressure technologies: Reaction set-up and deactivation determination[J]. Journal of Chemical Technology and Biotechnology, 2010, 85(7) : 1011–1016. DOI:10.1002/jctb.2411 |
| [14] | LEUCHS S, NONNEN T, DECHAMBRE D, et al. Continuous biphasic enzymatic reduction of aliphatic ketones[J]. Journal of Molecular Catalysis B: Enzymatic, , 2013, 88 : 52–59. DOI:10.1016/j.molcatb.2012.09.017 |
| [15] | KULISHOVA L, DIMOULA K, JORDAN M, et al. Factors influencing the operational stability of NADpH-dependent alcohol dehydrogenase and an NADH-dependent variant thereof in gas/solid reactors[J]. Journal of Molecular Catalysis B: Enzymatic, 2010, 67(3) : 271–283. DOI:10.1016/j.molcatb.2010.09.005 |
| [16] | SCHLIEBEN N H, NIEFIND K, MVLLER J, et al. Atomic resolution structures of R-specific alcohol dehydrogenase from Lactobacillus brevis provide the structural bases of its substrate and cosubstrate specificity[J]. Journal of Molecular Biology, 2005, 349(4) : 801–813. DOI:10.1016/j.jmb.2005.04.029 |
| [17] | MACHIELSEN R, LOOGER L L, RAEDTS J, et al. Cofactor engineering of Lactobacillus brevis alcohol dehydrogenase by computational design[J]. Engineering in Life Sciences, 2009, 9(1) : 38–44. DOI:10.1002/elsc.200800046 |
| [18] | NAIK H G, YENIAD B, KONING C E, et al. Investigation of asymmetric alcohol dehydrogenase (ADH) reduction of acetophenone derivatives: Effect of charge density[J]. Organic & Biomolecular Chemistry, 2012, 10(25) : 4961–4967. DOI:10.1039/C2OB06870B |
| [19] | 赵炜, 崔红晶, 方炳雄, 等. PCR重叠延伸法结合分段克隆技术构建高GC含量基因突变载体[J]. 海南医学院学报, 2012, 18(10) : 1349–1356. ZHAO W, CUI H J, FANG B X, et al. Mutate gene of high GC content with gene splicing by overlap extension and fragmentation cloning method[J]. Journal of Hainan Medical University, 2012, 18(10) : 1349–1356(Ch). |
| [20] | BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1-2) : 248–254. DOI:10.1016/0003-2697(76)90527-3 |
| [21] | MÜLLER M A, BRUNIE L, BÄCHER A S, et al. Cytoplasmic salt bridge formation in integrin αvβ3 stabilizes its inactive state affecting integrin-mediated cell biological effects[J]. Cellular Signalling, 2014, 26(11) : 2493–2503. DOI:10.1016/j.cellsig.2014.07.013 |
| [22] | KIM M S, LEI X G. Enhancing thermostability of Escherichia coli phytase AppA2 by error-prone PCR[J]. Applied Microbiology and Biotechnology, 2008, 79(1) : 69–75. DOI:10.1007/s00253-008-1412-7 |
| [23] | NAKAZAWA H, OKADA K, ONODERA T, et al. Directed evolution of endoglucanase Ⅲ (Cel12A) from Trichoderma reesei[J]. Applied Microbiology and Biotechnology, 2009, 83(4) : 649–657. DOI:10.1007/s00253-009-1901-3 |
| [24] | KUNO A, SHIMIZU D, KANEKO S, et al. Significant enhancement in the binding of p-nitrophenyl-β-D-xylobioside by the E128H mutant F/10 xylanase from Streptomyces olivaceoviridis E-86[J]. FEBS Letters, 1999, 450(3) : 299–305. DOI:10.1016/S0014-5793(99)00498-6 |
 2017, Vol. 63
2017, Vol. 63





