文章信息
- 余幼林, 余军林, 沈雄山, 胡超华, 卢婷, 张文杰, 杨青青, 刘程浩, 杨峰
- Yu Youlin, Yu Junlin, Shen Xiongshan, Hu Chaohua, Lu Ting, Zhang Wenjie, Yang Qingqing, Liu Chenhao, Yang Feng
- 生物信息学分析甲状腺癌免疫基因构建预后评估模型及对免疫细胞浸润的影响
- Construction of a prognostic evaluation model based on bioinformatics analysis of immune genes in thyroid cancer and its influence on immune cell infiltration
- 实用肿瘤杂志, 2022, 37(1): 50-59
- Journal of Practical Oncology, 2022, 37(1): 50-59
基金项目
- 湖北省卫生计生委联合基金项目(WJ2018H0098);武汉科技大学附属孝感医院(孝感市中心医院)院级科研项目(201818)
-
通信作者
- 余幼林,E-mail:yuyoulin2800@163.com
-
文章历史
- 收稿日期:2021-02-09
2. 武汉科技大学附属孝感医院肿瘤科, 湖北 孝感 432000;
3. 锦州医科大学研究生院临床系, 辽宁 锦州 121000
2. Department of Oncology, Xiaogan Hospital Affiliated Wuhan University of Science and Technology, Xiaogan 432000, China;
3. Department of Clinical Medicine, Graduate School, Jinzhou Medical University, Jinzhou 121000, China
目前,甲状腺癌是内分泌肿瘤发病率最高的恶性肿瘤,死亡率低,预后相对较好[1]。但对于局部晚期或复发转移性甲状腺癌现有治疗方法仍不能有效改善患者预后。因此,针对甲状腺癌发生和发展分子机制的新治疗方法如免疫疗法仍在探索[2]。已有研究表明,免疫相关的基因可以用于预测甲状腺癌患者的预后并作为治疗靶标[3]。因此,了解甲状腺癌微环境中的免疫细胞及免疫基因的作用尤为重要。免疫细胞广泛分布在甲状腺癌的微环境中,在肿瘤发展的不同阶段形成不同的肿瘤微环境促进或抑制肿瘤发生和发展。有研究表明,甲状腺常见的自身免疫性疾病慢性淋巴细胞性甲状腺炎可以促进甲状腺乳头状癌的进展[4]。甲状腺乳头状癌存在BRAF V600E突变与肿瘤组织中的程序性死亡配体1/程序性死亡受体1(programmed cell death ligand 1/programmed cell death protein 1,PD-L1/PD-1)表达量正相关,应用免疫检查点抑制剂可以有效杀伤甲状腺肿瘤细胞[5]。
免疫基因作为甲状腺癌预后分子标志物及甲状腺癌免疫治疗潜在靶标备受关注[6-8]。当前,分化型甲状腺癌术后AJCC TNM分期和复发危险度分层用于预测患者预后,指导个体化治疗,制定基于甲状腺癌患者临床病理资料,缺乏分子检测内容。本研究通过生物信息学方法筛选出预后相关的免疫基因并构建预后模型和危险评分系统。从基因分子层面评估甲状腺癌预后,并进一步分析甲状腺癌免疫基因调控网络,为甲状腺癌的免疫相关机制研究及免疫靶向药物的研发提供新思路。
1 资料与方法 1.1 数据库提取数据2021年1月18日从癌症基因组图谱(The Cancer Genome Atlas,TCGA;https://portal.gdc.cancer.gov/)下载甲状腺癌患者的临床资料和转录组数据,从ImmoPort Resources(https://www.immport.org/resources/)数据库中下载免疫相关的基因,从Cistrome Project(http://www.cistrome.org/)数据库中下载转录因子数据,从TIMER2.0下载免疫细胞浸润数据。通过R语言(4.0.3版本)“wilcox.test”筛选在正常组织和甲状腺癌组织中表达差异的免疫基因及转录因子,进一步分析免疫基因与转录因子相互作用的关系,运用cytoscape软件构建调控网络。采用单因素和多因素Cox分析并构建免疫基因预后模型,评估模型与临床病理特征和甲状腺癌预后的相关性。探讨免疫基因对肿瘤免疫细胞(B细胞、CD4+ T细胞、CD8+ T细胞、中性粒细胞、巨噬细胞及树突状细胞)浸润的影响。
1.2 统计学分析甲状腺癌组织与正常组织间的免疫基因和转录因子表达差异采用R语言的wilcox检验。免疫基因与转录因子、预后模型与临床病理特征及浸润性免疫细胞的相关性采用Spearman相关性检验。预后模型与肿瘤预后的相关性采用单因素及多因素Cox分析。R语言绘制全部的图像。以P<0.05为差异具有统计学意义。
2 结果 2.1 甲状腺癌组织与正常组织之间免疫相关基因表达的差异从TCGA数据中获取甲状腺癌(n=510)及正常甲状腺(n=58)的mRNA数据。510例甲状腺癌中,男性139例,女性371例;年龄15~89岁,中位年龄46岁。差异表达分析筛选出2 695个基因,其中下调基因1 228个,上调基因1 467个[错误发现率(false discovery rate,FDR)<0.05,log2差异倍数(fold change,FC)>1或<-1]。从ImmoPort Resources数据库获取免疫相关基因2 483个,筛选出甲状腺癌组织和正常组织差异表达的272个免疫相关基因,其中下调基因116个,上调基因156个(FDR<0.05,log2FC>1或<-1;图 1A~1B)。
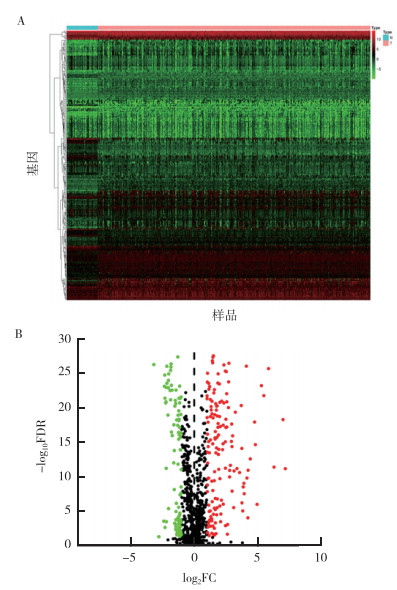
|
| 注 A:热图;B:火山图;热图中绿色代表低表达,红色代表高表达,顶端的浅红色代表肿瘤组织(tumor,T),蓝色代表正常组织(normal,N);火山图中绿色代表低表达,红色代表高表达,黑色代表差异无统计学意义(P>0.05);FDR:错误发现率(false discovery rate);FC:差异倍数(fold change) 图 1 甲状腺癌免疫相关基因差异表达分析 Fig.1 Analysis of differentially expressed immune-related genes of thyroid cancer |
单因素Cox分析差异表达的免疫相关基因结果表明,共13个免疫相关基因与预后相关(均P<0.01,图 2),分别为CXCL5、COLEC10、A100A9、MMP12、FABP4、APOD、MARCO、AQP9、NFATC2、FGF7、RETN、SPP1和GHR。
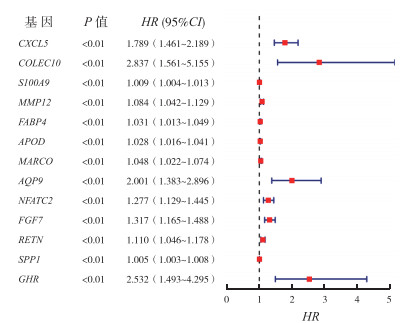
|
| 图 2 基于单因素Cox回归分析的13个肿瘤免疫相关基因的森林图 Fig.2 Forest map of 13 tumor-related immune genes based on univariate Cox analysis |
从TCGA数据中获取mRNA数据中筛选出肿瘤组织和正常组织差异表达的36个转录因子,其中下调基因22个,上调基因14个(FDR<0.05,log2FC>1或<-1,图 3A~3B)。
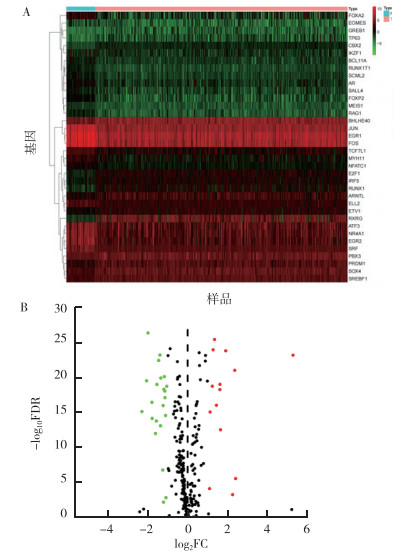
|
| 注 A:热图;B:火山图;热图中绿色代表低表达,红色代表高表达,顶端的浅红色代表肿瘤组织(tumor,T),蓝色代表正常组织(normal,N);火山图中绿色代表低表达,红色代表高表达,黑色代表差异无统计学意义(P>0.05);FDR:错误发现率(false discovery rate);FC:差异倍数(fold change) 图 3 甲状腺癌转录因子差异表达分析 Fig.3 Analysis of differentially expressed transcription factors of thyroid cancer |
预后相关的差异表达的免疫相关基因与转录因子相关性分析结果表明,共13个转录因子与11个预后相关的免疫基因相关(均r>0.3或<-0.3,均P<0.01,表 1)。其中,免疫相关基因AQP9与BHLHE40、IKZF1、IRF5和RUNX1正相关;免疫相关基因COLEC10与EGR1、ELL2、FOXA2和TCF7L1正相关,且与IRF5、RUNX1、RXRG和SOX4负相关;免疫相关基因CXCL5与RUNX1负相关;免疫相关基因FABP4与IRF5和SOX4负相关,且与TCF7L1正相关;免疫相关基因FGF7与TCF7L1正相关;免疫相关基因GHR与EGR1、FOS、JUN和TCF7L1正相关,且与IRF5、RUNX1和RXRG负相关;免疫相关基因MARCO与IRF5和RUNX1正相关;免疫相关基因NFATC2与ELL2、IKZF1和MYH11正相关;免疫相关基因RETN与RUNX1正相关;免疫相关基因S100A9与RUNX1正相关。两者之间的调控网络见图 4。
| TF | IRG | r值 | P值 |
| BHLHE40 | AQP9 | 0.316 | <0.01 |
| IKZF1 | AQP9 | 0.338 | <0.01 |
| IRF5 | AQP9 | 0.443 | <0.01 |
| RUNX1 | AQP9 | 0.476 | <0.01 |
| EGR1 | COLEC10 | 0.317 | <0.01 |
| ELL2 | COLEC10 | 0.306 | <0.01 |
| FOXA2 | COLEC10 | 0.315 | <0.01 |
| IRF5 | COLEC10 | -0.368 | <0.01 |
| RUNX1 | COLEC10 | -0.410 | <0.01 |
| RXRG | COLEC10 | -0.314 | <0.01 |
| SOX4 | COLEC10 | -0.354 | <0.01 |
| TCF7L1 | COLEC10 | 0.399 | <0.01 |
| RUNX1 | CXCL5 | 0.405 | <0.01 |
| IRF5 | FABP4 | -0.302 | <0.01 |
| SOX4 | FABP4 | -0.302 | <0.01 |
| TCF7L1 | FABP4 | 0.505 | <0.01 |
| TCF7L1 | FGF7 | 0.354 | <0.01 |
| EGR1 | GHR | 0.407 | <0.01 |
| FOS | GHR | 0.349 | <0.01 |
| IRF5 | GHR | -0.430 | <0.01 |
| JUN | GHR | 0.371 | <0.01 |
| RUNX1 | GHR | -0.326 | <0.01 |
| RXRG | GHR | -0.420 | <0.01 |
| TCF7L1 | GHR | 0.533 | <0.01 |
| IRF5 | MARCO | 0.448 | <0.01 |
| RUNX1 | MARCO | 0.426 | <0.01 |
| ELL2 | MMP12 | 0.317 | <0.01 |
| ELL2 | NFATC2 | 0.444 | <0.01 |
| IKZF1 | NFATC2 | 0.482 | <0.01 |
| MYH11 | NFATC2 | 0.315 | <0.01 |
| RUNX1 | RETN | 0.339 | <0.01 |
| RUNX1 | S100A9 | 0.358 | <0.01 |
| 注 TF:转录因子(transcription factor);IRG:免疫相关基因(immune related gene) | |||
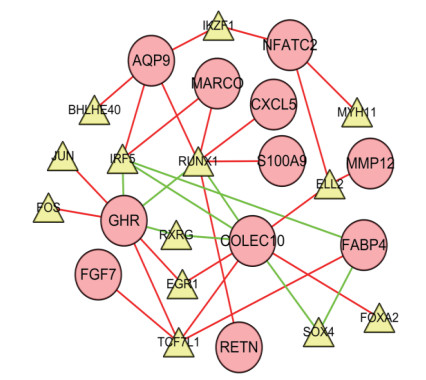
|
| 注 圆圈代表免疫相关基因;三角形代表转录因子;红色线条代表正相关;绿色代表负相关 图 4 预后相关的差异表达的免疫相关基因与转录因子的调控网络 Fig.4 Regulatory network of differentially expressed immune-related genes and transcription factors related to prognosis |
多因素Cox分析具有统计学意义的13个免疫相关基因结果显示,与预后独立相关的基因有6个,分别是CXCL5、COLEC10、S100A9、MMP12、APOD和FGF7(均P<0.01,表 2)。将6个基因联合构建预后评估模型进行风险评分。风险得分(risk score,RS)=β1X1+β2X2+……+βnXn,β表示基因相关系数,X表示基因表达量。
| IRG | r值 | HR | 95%CI | P值 |
| CXCL5 | 0.832 | 2.299 | 1.716~3.080 | <0.01 |
| MMP12 | 1.623 | 5.067 | 2.289~11.216 | <0.01 |
| S100A9 | 0.008 | 1.008 | 1.003~1.012 | 0.001 |
| COLEC10 | -0.172 | 0.842 | 0.741~0.956 | 0.008 |
| APOD | 0.030 | 1.030 | 1.011~1.049 | 0.002 |
| FGF7 | 0.345 | 1.413 | 1.182~1.688 | <0.01 |
| 注 IRG:免疫相关基因(immune related gene) | ||||
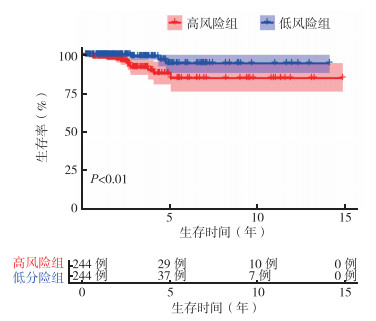
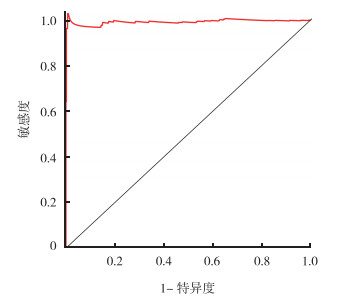
通过风险评分的中位数将患者分为高风险组组和低风险组,高风险组和低风险组患者的10年总生存(overall survival,OS)率分别为85.4%和95.6%(P<0.05,图 5)。接收者操作特征(receiver operating characteristic,ROC)曲线显示,该预后评估模型具有良好的敏感度和特异度[曲线下面积(area under curve,AUC)=0.992,图 6]。
|
| 图 5 Kaplan-Meier法分析基于免疫相关基因预后风险分组的甲状腺癌患者总生存曲线 Fig.5 Kaplan-Meier survival curve of patients with thyroid cancer: grouping based on the prognostic risk score of immune-related genes |
|
| 图 6 甲状腺癌免疫基因预后评估模型ROC曲线 Fig.6 ROC curve of prognostic evaluation model of immune-related genes for thyroid cancer |
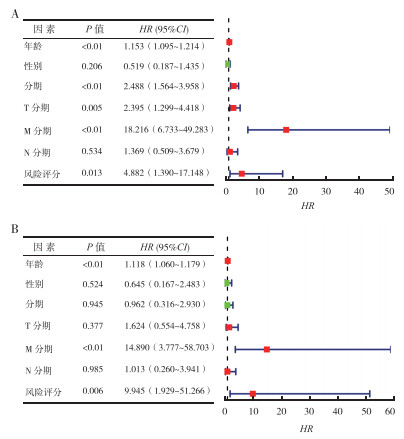
单因素及多因素Cox分析TCGA数据库获取患者的临床病理特征资料及免疫基因预后模型风险评分。单因素Cox分析表明,年龄、临床分期、远处转移及风险评分是OS的相关影响因素(均P<0.05,图 7A)。年龄增大、临床Ⅲ/Ⅳ期、远处转移及高风险组是OS的危险因素。多因素分析表明,年龄、远处转移及风险评分是OS的独立预后因素(均P<0.05,图 7B)。
|
| 注 A:单因素Cox分析森林图;B:多因素Cox分析森林图 图 7 甲状腺癌预后相关影响因素的单因素及多因素Cox分析 Fig.7 Univariate and multivariate Cox analysis of prognostic factors of thyroid cancer |
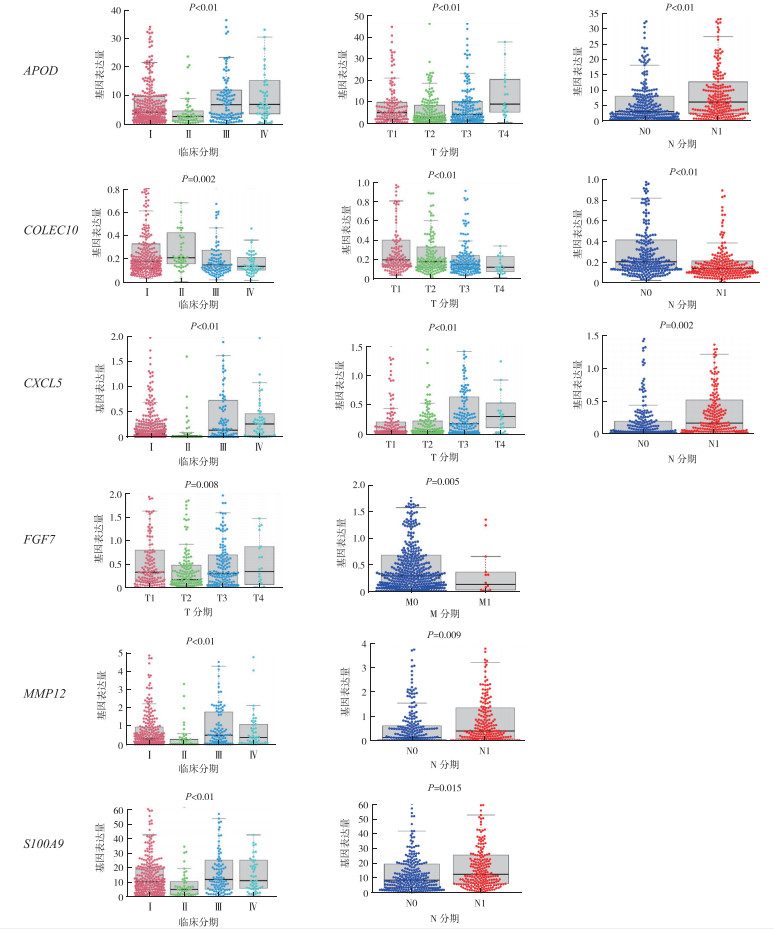
独立预后相关的6个免疫基因CXCL5、COLEC10、S100A9、MMP12、APOD和FGF7及风险评分与临床病理特征进行相关性分析结果表明,COLEC10、CXCL5和APOD与T、N和临床分期相关(均P<0.05,图 8);FGF7与T和M分期相关(均P<0.05);MMP12和S100A9与N和临床分期相关(均P<0.05)。CXCL5和APOD在N1、Ⅲ/Ⅳ和T4期患者中高表达,而COLEC10与之相反。FGF7在T3和T4期患者中高表达,在M1期患者中低表达。MMP12和S100A9在N1和Ⅲ/Ⅳ期患者中高表达。
|
| 图 8 免疫相关基因与临床病理特征的相关性分析 Fig.8 Analysis of correlation between immune-related genes and clinicopathological characteristics |
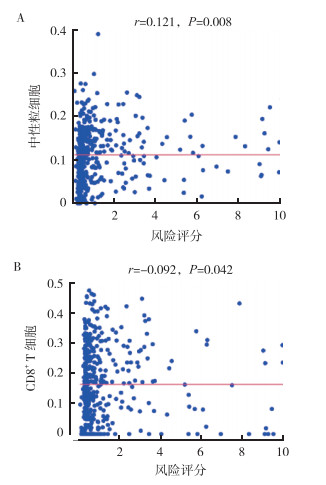
从TIMER 2.0下载免疫细胞浸润数据,分析风险评分与6种浸润性免疫细胞(B细胞、CD4+ T细胞、CD8+ T细胞、中性粒细胞、巨噬细胞及树突状细胞)浸润丰度的相关性。结果显示,风险评分与中性粒细胞和CD8+ T细胞的浸润数量存在相关性(均P<0.05,图 9)。风险值越高,中性粒细胞浸润数量增多,而CD8+ T细胞浸润数量则减少。
|
| 注 A:风险评分与中性粒细胞浸润丰度的相关性;B:风险评分与CD8+ T细胞浸润丰度的相关性 图 9 风险评分与浸润性免疫细胞含量的相关性分析 Fig.9 Correlation analysis of risk score and infiltrating immune cells |
肿瘤发生与原癌基因激活,与肿瘤免疫逃逸有关,因此了解肿瘤相关免疫细胞与免疫基因相互关系对肿瘤免疫研究具有重要的意义。近年来,甲状腺癌免疫研究主要集中在免疫检查点和肿瘤微环境[9-10]。对免疫相关基因与甲状腺癌预后的研究涉及较少。本研究主要分析免疫相关基因与甲状腺预后的关系,并构建预后评估模型和危险评分系统及免疫调控网络,为进一步理解甲状腺癌免疫基因调控机制提供理论依据和评估甲状腺癌预后提供参考。
在本研究中,通过TCGA数据库、免疫信息学数据库(ImmoPort Resources)和Cistrome Project数据库联合分析免疫基因与预后和转录因子相关性,最终筛选出11个甲状腺癌免疫相关基因,构建调控网络,为甲状腺癌免疫基因作用机制的研究提供思路。通过单因素和多因素Cox分析确定具有统计学意义(均P<0.05)的6个基因(CXCL5、COLEC10、S100A9、MMP12、APOD和FGF7)构建预后评估模型,预测甲状腺癌OS准确度较高(AUC=0.992)。结合临床病理特征进一步验证了该模型是独立有效预后预测因子,模型风险值越高,则预后不佳。模型中结果显示,风险值越高,浸润性中性粒细胞越多,浸润性CD8+ T细胞则减少。
模型中6个免疫基因(CXCL5、COLEC10、S100A9、MMP12、APOD和FGF7)具有各自生物学特征,与肿瘤患者临床预后密切相关。CXCL5属于CXC趋化因子的一种,是CXCR2受体激动剂。在免疫细胞、肿瘤细胞和上皮细胞等多种细胞类型中均有表达。在肿瘤微环境中,CXCL5趋化作用募集免疫细胞,发挥抗肿瘤免疫作用。同时也促进肿瘤血管生成,参与肿瘤生长和转移。肿瘤过表达CXCL5预示患者预后不良[11]。在甲状腺乳头状癌细胞中过表达CXCL5通过激活CXCL5-CXCR2轴促进肿瘤增殖[12]。除此之外,CXCL5-CXCR2轴还激活多种细胞旁路如Raf/MEK/ERK和ERK/Egr-1/Snail等影响肿瘤细胞的增殖、侵袭和转移。CXCL5、CXCR2及趋化因子信号旁路可作为肿瘤治疗靶点。本研究证实,CXCL5过表达提示甲状腺癌预后不良,可作为预后生物分子标志物。COLEC10基因是编码C-凝集素家族成员,该基因产物是一种胞质蛋白,属于特殊的集合蛋白,作为先天免疫和触发补体级联的传感器,与细胞上的特定配体结合发挥趋化因子,参与调控细胞迁移。在肿瘤组织中,低表达COLEC10预示预后不良[13]。本研究发现,低表达的COLEC10与甲状腺癌分期较晚和淋巴结转移相关,潜在机制可能与肿瘤免疫调节相关。S100A9基因编码的蛋白是低相对分子质量的钙结合蛋白,属于S100蛋白家族的成员之一。S100A9可结合高级糖基化终末产物受体,激活自然杀伤细胞,发挥免疫应答[14]。有研究表明,肿瘤浸润性单核细胞/巨噬细胞通过上调癌细胞中的S100A8和S100A9表达促进肿瘤侵袭和迁移[15]。S100A8和S100A9是肿瘤细胞侵袭性的重要介质,可作为肿瘤治疗潜在靶点。另外,S100A9与基质金属蛋白酶存在互作网络作用,可上调基质金属蛋白酶,诱导上皮间质转化,激活Wnt/β-catenin信号通路等促进肿瘤细胞增殖、迁移和侵袭[16]。在本研究中发现S100A9高表达提示预后不良。MMP12基因编码MMP12蛋白,是一类含有锌钙离子的蛋白水解酶类,可降解细胞外基质成分和血管成分,属于MMP家族成员之一。MMP在肿瘤细胞迁移、增殖、细胞凋亡和诱导肿瘤血管生成中发挥重要作用,并参与肿瘤免疫逃逸[17]。MMP12蛋白主要是由炎性免疫细胞如巨噬细胞和单核细胞等主动分泌的,在正常组织中少量或不表达,而在肿瘤组织中高表达[18]。在肿瘤进展过程中,MMP-12可以降解结缔组织和基底膜,诱导肿瘤血管生成,促使肿瘤生长和转移扩散[19]。MMP-12参与肿瘤免疫调节潜在机制是MMP-12可能通过IL-6信号传导途径上调PD-L1的表达[20]。PD-L1与PD-1结合,抑制T细胞活化,从而导致肿瘤的免疫逃逸[21]。本研究结果显示:MMP12高表达预示预后不良。与淋巴结转移和临床分期较晚密切相关。APOD基因编码的载脂蛋白D(APOD)是一种多配基多功能高密度脂蛋白,在人体多种组织中都有表达,包括肾上腺、胰腺、肾脏、胎盘、脾、肺、卵巢、睾丸、脑、外周神经和脑脊液。乳腺癌高表达APOD提示预后不良[22]。在甲状腺癌中,APOD高表达与肿瘤体积大、临床分期较晚和淋巴结转移密切相关。成纤维细胞生长因子存在多个亚群,包括FGF1、FGF2、FGF4和FGF7等,在不同类别肿瘤中表达存在差异。FGF7与硫酸乙酰肝素蛋白多糖和FGFRs结合旁分泌FGFs,影响肿瘤微环境[23]。在胃癌中,FGF7促进胃癌的侵袭和迁移,高表达FGF7预示患者预后不良[24]。在本研究中也发现类似结果,潜在的机制是通过调节肿瘤微环境中的免疫细胞功能发挥作用。
本研究中,CXCL5与APOD在N1、Ⅲ/Ⅳ及T4期患者中高表达,而COLEC10与之相反。FGF7在T3和T4期患者中高表达,在M1期患者中低表达。MMP12和S100A9在N1及Ⅲ/Ⅳ期患者中高表达。在调控网络中,CXCL5和S100A9基因存在共同正向调控转录因子RUNX1。RUNX1作为一种转录因子,发挥促癌和抑制癌双重作用。在不同癌症与癌症亚型和肿瘤发展的不同阶段表现的作用不同。在多数乳腺癌中,RUNX1抑制肿瘤形成[25]。在血液肿瘤中,RUNX1突变可以促成肿瘤形成[26]。RUNX1还通过激活Wnt/β-catenin信号通路促进结直肠癌转移[27]。本研究中,RUNX1作为转录因子,作用下游靶基因CXCL5和S100A9,CXCL5发挥趋化作用,S100A9募集MDSCs,形成肿瘤微环境免疫抑制。协同作用促进肿瘤血管生成,转移前生态位的形成。与临床特征肿瘤临床分期较晚,发生肿瘤转移相符。
MMP12和COLEC10基因存在共同正向调控转录因子ELL2。ELL2是RNA聚合酶Ⅱ的延伸因子,驱动B细胞向浆细胞的分化成熟,促进与浆细胞分泌相关的重链免疫球蛋白的产生发挥抑制肿瘤的作用。在前列腺癌中,其作为一种抑癌基因与RB结合,从而抑制肿瘤进展[28]。在前列腺腺癌组织中,ELL2常表达下调,在良性前列腺增生中表达上调。ELL2及其通路基因可能在前列腺癌的发生和发展中起重要作用[29]。本研究中,ELL2作用下游靶基因MMP12和COLEC10。MMP12基因可促进肿瘤生长、侵袭和转移,并参与肿瘤免疫逃逸。COLEC10基因是编码C-凝集素家族成员,作为先天免疫和触发补体级联的传感器,可以激活免疫反应。甲状腺癌低表达COLEC10减低抗肿瘤免疫反应。协同肿瘤免疫逃逸,甲状腺癌高表达MMP12,低表达COLEC10,两者协同参与肿瘤免疫逃逸,促进肿瘤转移。本研究临床特征表现为在分期较晚和转移甲状腺癌中,MMP12高表达,COLEC10低表达。
COLEC10和FGF7基因存在共同正向调控转录因子TCF7L1。TCF7L1是一种胚胎干细胞标志基因,在多种侵袭性癌症类型中表达上调,通过调节Keap1/NRF2信号通路促进肿瘤增殖[30]。同时作为Wnt信号通路的关键因子,形成β-catenin-TCF7L1复合物可激活转录并调节下游靶基因促进肿瘤形成和生长[31]。本研究中,TCF7L1作用下游靶基因COLEC10和FGF7。COLEC10基因是编码C-凝集素家族成员和免疫反应效应因子,可以激活免疫反应。甲状腺癌低表达COLEC10协同肿瘤免疫逃逸。FGF7高表达甲状腺癌促进肿瘤增殖、侵袭和转移。这可能是潜在协同调节机制。本次研究临床特征表现为甲状腺癌中低表达COLEC10,肿瘤体积较大,FGF7高表达。
然而,单个免疫相关基因的预后预测价值有限,因此,本研究联合6个独立预后的免疫相关基因构建预后评估模型,并予以风险评分,大幅提高甲状腺癌预后预测价值。在高风险组中,患者的10年OS率为85.4%,在低风险组中,10年OS率为95.6%,该模型有较高准确度(AUC=0.992),可作为甲状腺癌患者预后评估的预测因子。在模型中,风险评分与肿瘤微环境中的中性粒细胞呈正相关(r=0.121,P=0.008),与CD8+ T细胞呈负相关(r=-0.092,P=0.042)。高风险组患者预后不佳与浸润性CD8+ T细胞减少相关。中性粒细胞是促肿瘤因子,通过合成和分泌相关蛋白酶(如中性粒细胞弹性蛋白酶和基质金属蛋白酶)促进肿瘤增殖、侵袭及转移[32],并产生大量的促炎因子,如MCP-1、IL-8和IL-6及其他细胞因子如IL-1β、TNF-α、IL-12、G-CSF和VEGF等促肿瘤增殖[33]。CD8+ T细胞是靶向抑制杀伤肿瘤最重要的免疫细胞,在癌症进展期间,肿瘤微环境中的免疫抑制因子导致CD8+ T细胞功能减弱和缺失,肿瘤获得性免疫抵抗,促进肿瘤的生长和转移[34]。免疫细胞在肿瘤免疫微环境形成和演进过程发挥重要作用,免疫基因调控免疫细胞生物学特性,对肿瘤发生和发展具有促进或抑制双重作用。目前针对甲状腺癌的免疫治疗仍处在探索阶段[35]。
综上所述,本研究筛选出6个独立的预后免疫相关基因(CXCL5、COLEC10、S100A9、MMP12、APOD和FGF7),可作为预后预测的生物分子标志物,并构建了预后评估模型及危险评分系统,予以风险评分分高低危险组预测甲状腺癌10年OS率,具有较高准确度。并且,模型阐释了肿瘤预后与浸润性免疫细胞之间关系,为甲状腺癌的免疫相关机制研究和免疫靶向药物的研发提供了新思路。然而,本研究仍存在一定的局限性:(1)本研究的数据来源于TCGA数据库的RNA测序结果,缺少体内和体外实验;(2)该预后评估模型仍需大量的多中心样本进行验证。后续本研究组将继续开展相关工作。
| [1] |
Wang J, Yu F, Shang Y, et al. Thyroid cancer: incidence and mortality trends in China, 2005-2015[J]. Endocrine, 2020, 68(1): 163-173. DOI:10.1007/s12020-020-02207-6 |
| [2] |
Farkona S, Diamandis EP, Blasutig IM. Cancer immunotherapy: the beginning of the end of cancer?[J]. BMC Med, 2016, 14: 73. DOI:10.1186/s12916-016-0623-5 |
| [3] |
Gunda V, Gigliotti B, Ndishabandi D, et al. Combinations of BRAF inhibitor and anti-PD-1/PD-L1 antibody improve survival and tumour immunity in an immunocompetent model of orthotopic murine anaplastic thyroid cancer[J]. Br J Cancer, 2018, 119(10): 1223-1232. DOI:10.1038/s41416-018-0296-2 |
| [4] |
Babli S, Payne RJ, Mitmaker E, et al. Effects of chronic lymphocytic thyroiditis on the clinicopathological features of papillary thyroid cancer[J]. Eur Thyroid J, 2018, 7(2): 95-101. DOI:10.1159/000486367 |
| [5] |
Bai Y, Guo T, Huang X, et al. In papillary thyroid carcinoma, expression by immunohistochemistry of BRAF V600E, PD-L1, and PD-1 is closely related[J]. Virchows Arch, 2018, 472(5): 779-787. DOI:10.1007/s00428-018-2357-6 |
| [6] |
Qin XJ, Lin X, Xue G, et al. CXCL10 is a potential biomarker and associated with immune infiltration in human papillary thyroid cancer[J]. Biosci Rep, 2021, 29, 41(1): BSR20203459. |
| [7] |
Zhi J, Yi J, Tian M, et al. Immune gene signature delineates a subclass of thyroid cancer with unfavorable clinical outcomes[J]. Aging, 2020, 12(7): 5733-5750. DOI:10.18632/aging.102963 |
| [8] |
Ma M, Lin B, Wang M, et al. Immunotherapy in anaplastic thyroid cancer[J]. Am J Transl Res, 2020, 12(3): 974-988. |
| [9] |
Kurimoto C, Inaba H, Ariyasu H, et al. Predictive and sensitive biomarkers for thyroid dysfunctions during treatment with immune-checkpoint inhibitors[J]. Cancer Sci, 2020, 111(5): 1468-1477. DOI:10.1111/cas.14363 |
| [10] |
Jung CK. Crosstalk between the tumor microenvironment and immune response in thyroid cancer[J]. Gland Surg, 2019, 8(3): 294-297. DOI:10.21037/gs.2019.05.08 |
| [11] |
Zhang W, Wang H, Sun M, et al. CXCL5/CXCR2 axis in tumor microenvironment as potential diagnostic biomarker and therapeutic target[J]. Cancer Commun, 2020, 40(2/3): 69-80. |
| [12] |
Cui D, Zhao Y, Xu J. Activation of CXCL5-CXCR2 axis promotes proliferation and accelerates G1 to S phase transition of papillary thyroid carcinoma cells and activates JNK and p38 pathways[J]. Cancer Biol Ther, 2019, 20(5): 608-616. DOI:10.1080/15384047.2018.1539289 |
| [13] |
Zhang B, Wu H. Decreased expression of COLEC10 predicts poor overall survival in patients with hepatocellular carcinoma[J]. Cancer Manag Res, 2018, 10: 2369-2375. DOI:10.2147/CMAR.S161210 |
| [14] |
Narumi K, Miyakawa R, Ueda R, et al. Proinflammatory proteins S100A8/S100A9 activate NK cells via interaction with RAGE[J]. J Immunol, 2015, 194(11): 5539-5548. DOI:10.4049/jimmunol.1402301 |
| [15] |
Lim SY, Yuzhalin AE, Gordon-Weeks AN, et al. Tumor-infiltrating monocytes/macrophages promote tumor invasion and migration by upregulating S100A8 and S100A9 expression in cancer cells[J]. Oncogene, 2016, 35(44): 5735-5745. DOI:10.1038/onc.2016.107 |
| [16] |
Zhang W, Chen M, Cheng H, et al. The role of calgranulin B gene on the biological behavior of squamous cervical cancer in vitro and in vivo[J]. Cancer Manag Res, 2018, 10: 323-338. DOI:10.2147/CMAR.S153036 |
| [17] |
Gonzalez-Avila G, Sommer B, Mendoza-Posada DA, et al. Matrix metalloproteinases participation in the metastatic process and their diagnostic and therapeutic applications in cancer[J]. Crit Rev Oncol Hematol, 2019, 137: 57-83. |
| [18] |
Ella E, Harel Y, Abraham M, et al. Matrix metalloproteinase 12 promotes tumor propagation in the lung[J]. J Thorac Cardiovasc Surg, 2018, 155(5): 2164-2175. DOI:10.1016/j.jtcvs.2017.11.110 |
| [19] |
Conlon GA, Murray GI. Recent advances in understanding the roles of matrix metalloproteinases in tumour invasion and metastasis[J]. J Pathol, 2019, 247(5): 629-640. DOI:10.1002/path.5225 |
| [20] |
Gao H, Zhou X, Li H, et al. Role of matrix metallopeptidase 12 in the development of hepatocellular carcinoma[J]. J Invest Surg, 2021, 34(4): 366-372. DOI:10.1080/08941939.2019.1637975 |
| [21] |
Juneja VR, McGuire KA, Manguso RT, et al. PD-L1 on tumor cells is sufficient for immune evasion in immunogenic tumors and inhibits CD8 T cell cytotoxicity[J]. J Exp Med, 2017, 214(4): 895-904. DOI:10.1084/jem.20160801 |
| [22] |
Jankovic-Karasoulos T, Bianco-Miotto T, Butler MS, et al. Elevated levels of tumour apolipoprotein D independently predict poor outcome in breast cancer patients[J]. Histopathology, 2020, 76(7): 976-987. DOI:10.1111/his.14081 |
| [23] |
Katoh M. FGFR inhibitors: Effects on cancer cells, tumor microenvironment and whole-body homeostasis (Review)[J]. Int J Mol Med, 2016, 38(1): 3-15. DOI:10.3892/ijmm.2016.2620 |
| [24] |
Huang T, Wang L, Liu D, et al. FGF7/FGFR2 signal promotes invasion and migration in human gastric cancer through upregulation of thrombospondin-1[J]. Int J Oncol, 2017, 50(5): 1501-1512. DOI:10.3892/ijo.2017.3927 |
| [25] |
Hong D, Fritz AJ, Gordon JA, et al. RUNX1-dependent mechanisms in biological control and dysregulation in cancer[J]. J Cell Physiol, 2019, 234(6): 8597-8609. DOI:10.1002/jcp.27841 |
| [26] |
Yokota A, Huo L, Lan F, et al. The clinical, molecular, and mechanistic basis of RUNX1 mutations identified in hematological malignancies[J]. Mol Cells, 2020, 43(2): 145-152. |
| [27] |
Li Q, Lai Q, He C, et al. RUNX1 promotes tumour metastasis by activating the Wnt/β-catenin signalling pathway and EMT in colorectal cancer[J]. J Exp Clin Cancer Res, 2019, 38(1): 334. DOI:10.1186/s13046-019-1330-9 |
| [28] |
Qiu X, Pascal LE, Song Q, et al. Physical and functional interactions between ELL2 and RB in the suppression of prostate cancer cell proliferation, migration, and invasion[J]. Neoplasia, 2017, 19(3): 207-215. DOI:10.1016/j.neo.2017.01.001 |
| [29] |
Wang Z, Pascal LE, Chandran UR, et al. ELL2 is required for the growth and survival of AR-negative prostate cancer cells[J]. Cancer Manag Res, 2020, 12: 4411-4427. DOI:10.2147/CMAR.S248854 |
| [30] |
Zhang B, Wu J, Cai Y, et al. TCF7L1 indicates prognosis and promotes proliferation through activation of Keap1/NRF2 in gastric cancer[J]. Acta Biochim Biophys Sin, 2019, 51(4): 375-385. DOI:10.1093/abbs/gmz015 |
| [31] |
Yuan YH, Zhou J, Zhang Y, et al. Identification of key genes and pathways downstream of the β-catenin-TCF7L1 complex in pancreatic cancer cells using bioinformatics analysis[J]. Oncol Lett, 2019, 18(2): 1117-1132. |
| [32] |
Szczerba BM, Castro-Giner F, Vetter M, et al. Neutrophils escort circulating tumour cells to enable cell cycle progression[J]. Nature, 2019, 566(7745): 553-557. DOI:10.1038/s41586-019-0915-y |
| [33] |
Lechner MG, Liebertz DJ, Epstein AL, et al. Characterization of cytokine-induced myeloidderived suppressor cells from normal human peripheral blood mononuclear cells[J]. J Immunol, 2010, 185(4): 2273-2284. DOI:10.4049/jimmunol.1000901 |
| [34] |
Wang T, Shen Y, Luyten S, et al. Tissue-resident memory CD8+ T cells in cancer immunology and immunotherapy[J]. Pharmacol Res, 2020, 159: 104876. DOI:10.1016/j.phrs.2020.104876 |
| [35] |
马丹妮, 赵春雷. 碘难治性分化型甲状腺癌分子靶向治疗药物研究进展[J]. 实用肿瘤杂志, 2021, 5(36): 468-474. |
 2022, Vol. 37
2022, Vol. 37


